2. 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008
硝化作用长期以来被认为是一个两步的过程,首先由氨氧化细菌(Ammonia-oxidizing bacteria, AOB)和古菌(Ammonia-oxidizing archaea,AOA)将氨氧化为亚硝酸盐,再由亚硝酸盐氧化细菌(Nitrite-oxidizing bacteria, NOB)将其氧化为硝酸盐,从而完成整个硝化过程。但根据代谢过程中的动力学理论,由一种微生物来完成氨的两步氧化过程是可行的[1]。2015年底,科学家在水生生态系统中发现了全程氨氧化微生物(Complete ammonia oxidizers,Comammox),能够直接完成从氨氮到硝氮的氧化,其系统发育分类地位可归属于经典的NOB中的硝化螺菌属(Nitrospira)[2-3]。通过比对已经发表和尚未发表的海量宏基因组数据库以及最新的分子生态学研究,发现Comammox广泛分布于土壤、淡水生态系统、污水和饮用水处理系统中[2-5]。但是,Comammox如何适应复杂环境并与传统的AOA和AOB相互竞争共存,目前仍不清楚,是现在研究的热点和难点。
氨氧化过程是在O2存在的条件下,微生物将NH3氧化为亚硝酸盐和硝酸盐的过程。因此,环境中NH3和O2浓度的变化极有可能是驱动Comammox和AOA及AOB的关键环境因子。例如,土壤酸碱度是影响氨氧化微生物生态位分异的重要因素,其主要原因是H+介导的化学平衡使得酸性土壤中NH3浓度通常在nmol·L-1水平,中性和碱性土壤中在µmol·L-1水平[6],同时,AOA对NH3的亲和力远高于AOB[7]。因此,AOA通常主导酸性土壤氨氧化[8],而AOB则在中性和碱性土壤中发挥作用[9-10]。也有研究发现,AOA能够更好地适应低O2胁迫,从而在水稻土氨氧化过程中发挥重要作用[11-12]。最新的研究则表明,全程氨氧化细菌Nitrospira inopinata对底物NH3的亲和力远高于大多数可培养的AOA物种,能够更好地适应极低NH3浓度的胁迫环境[13]。然而,在复杂土壤环境中,Comammox对O2和NH4+-N的环境适应机制仍鲜有报道。
稻田淹水导致其O2浓度远低于旱地土壤,而施肥则可能提升NH3浓度刺激氨氧化微生物的生长。据此,本研究依托西南大学1989年设置的中性紫色水稻土长期定位试验站,针对植稻淹水(夏季植稻施肥、全年淹水)和休耕冬干(不植稻、不施肥,仅在夏季植稻期间淹水,冬季排水落干)两种不同处理下的水稻土,研究施肥和水分管理对水稻土硝化潜势及氨氧化微生物丰度的影响规律。
1 材料与方法 1.1 研究区概况研究区位于国家紫色土长期定位试验基地(106°26´E,30°26´N),地处重庆市北碚区,属亚热带季风气候,年均气温18.30 ℃,年均降雨量1 105 mm,年均日照时数1 277 h,年均无霜期334 d。土壤类型为中性紫色水稻土。
1.2 研究方法长期定位试验始于1989年,设植稻淹水(PF)和休耕冬干(FD)两种种植模式。植稻淹水处理夏季植稻、全年淹水。夏季按照传统方法翻耕田地后种植水稻,施肥量为尿素273.1 kg·hm-2;过磷酸钙500.3 kg·hm-2;氯化钾150.1 kg·hm-2;过磷酸钙作为底肥一次施用,尿素2/3做底肥,1/3做追肥,氯化钾底肥和追肥各1/2。休耕冬干处理全年不种植任何作物,夏季的水分管理与种植水稻相同,4月灌水,8月排水,但不施肥、不耕作、不种植,后期做进一步排水处理,全年约7个月处于落干状态。
于2017年11月4日在两种种植模式地块分别采集表层(0~5 cm)土壤。采集后立即进行氨氧化微生物丰度的测定,部分土样风干后测定其基本理化性质。
1.3 硝化潜势的测定硝化潜势的测定采用好气培养法[14],稍作修改:称取15 g鲜土置于三角瓶中,加入(NH4)2SO4 调节NH4+-N浓度至0.4 g·kg-1(干土质量计)。将三角瓶置于摇床,室温25 ℃振荡24 h。在第2、4、22、24小时分别取10 mL的振荡悬浊液,以2 mol·L-1KCl溶液浸提过滤,流动分析仪测定悬浊液中NO3--N的含量。以培养时间为横坐标,NO3--N含量为纵坐标,求出斜率,即土壤硝化潜势。
1.4 DNA的提取采用FastDNA® SPIN Kit for Soil(MP Biomedicals)试剂盒提取土壤基因组总DNA,称取0.5 g土壤样品,按试剂盒提供的操作步骤进行,将提取的DNA保存于-20 ℃用于后续分析。
1.5 实时荧光定量PCR采用实时荧光定量PCR法在QuantStudioTM 6 Flex定量PCR仪(Thermo Fisher Scientific,Singapore)上测定AOA、AOB及Comammox分支A(Clade A)和分支B(Clade B)amoA基因的拷贝数。实时荧光定量PCR所用的扩增引物如表 1所示。AOA、AOB、Comammox Clade A、Comammox Clade B的amoA基因定量PCR所用反应体系均为20 μL,主要包含:待扩增的模板DNA1 μL、上下引物及ROX染料各0.4 μL,Taq DNA聚合酶10 μL、灭菌水7.8 μL。通过液体LB培养基培养含有目的基因的克隆子,按试剂盒(MiniBEST Plasmid Purification Kit)说明书方法提取、纯化质粒后,在NanoDrop®ND-1000 UV-Vis分光光度计上测定质粒的浓度,并计算出目的基因的拷贝数。最后用TE缓冲溶液将质粒连续稀释6~8个梯度以制作定量PCR的标准曲线。AOA、AOB的实时荧光定量PCR扩增程序皆为95 ℃预变性30 s;95 ℃变性30 s,54 ℃退火30 s,72 ℃延伸45 s,40个循环。Comammox Clade A、Clade B的实时荧光定量PCR扩增程序皆为95 ℃预变性3 min;95 ℃变性30 s,52 ℃退火45 s,72 ℃延伸1 min,45个循环。
|
|
表 1 实时荧光定量PCR扩增引物 Table 1 Real-time PCR Primers used in the study |
采用Excel 2010和SPSS 13等软件对数据进行处理分析。利用Origin 8.5软件作图。
2 结果 2.1 长期不同施肥和淹水管理对水稻土理化性质及硝化潜势的影响植稻淹水及休耕冬干两种不同模式下水稻土的基本性质如表 2所示。长期植稻淹水管理下水稻土的含水量为102.59%,远高于对照处理的34.07%(休耕冬干)。植稻淹水和休耕冬干水稻土中铵态氮含量分别为17.77、4.09 mg·kg-1;硝态氮的含量分别为13.86、4.71 mg·kg-1;全氮含量分别为1.76、0.92 g·kg-1。休耕冬干水稻土有机质含量显著高于植稻淹水处理,分别为20.51、17.55 g·kg-1。长期施肥显著刺激了水稻土的硝化潜势,植稻淹水和休耕冬干水稻土分别为25.0、2.11 mg·kg-1·d-1,两者相差高达12倍。
|
|
表 2 不同管理模式下水稻土的基本理化性质 Table 2 Basic physico-chemical properties of the paddy soils relative to treatment |
植稻淹水及休耕冬干这两种水稻土中氨氧化微生物的丰度如图 1所示。长期施肥和淹水管理显著影响了氨氧化微生物类群的丰度。相比于休耕冬干管理,植稻淹水刺激了Comammox Clade A、AOA和AOB的生长,三者增长倍数分别为9倍、3倍、42倍。两种水稻土中,Comammox的丰度均高于AOA和AOB。植稻淹水中Comammox丰度分别是AOA的8.5倍、AOB的77.3倍,而休耕冬干中Comammox丰度分别是AOA的4.1倍、AOB的490.3倍。
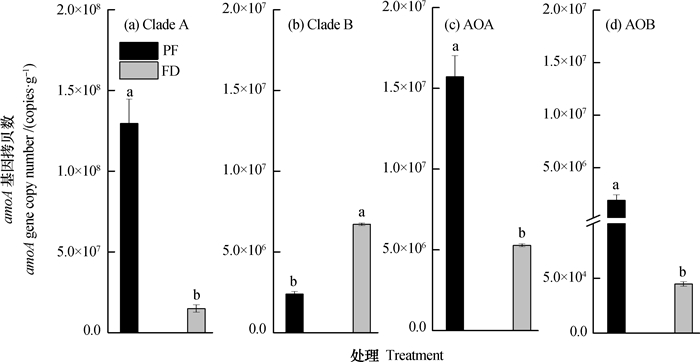
|
注:误差线表示标准误,n=3。不同字母表示P < 0.05水平上差异显著 Note: Error bars represent standard errors, n=3.Different letters mean significant differences at 0.05 level 图 1 不同管理模式下水稻土中氨氧化微生物的丰度 Fig. 1 Abundance of the ammonia-oxidizing microorganisms in paddy soils relative to treatment |
如图 1所示,植稻淹水中Comammox Clade A的丰度高于休耕冬干水稻土,而Clade B的丰度却低于休耕冬干,但两种水稻土中,Comammox Clade A的丰度均要高于Clade B。植稻淹水中Comammox Clade A和Clade B的amoA基因的拷贝数(以干土计,下同)分别为1.30×108 copies·g-1、2.39×106copies·g-1;而休耕冬干中Comammox Clade A和Clade B的amoA基因的拷贝数分别为1.50×107copies·g-1、6.71×106copies·g-1(图 1a,图 1b)。
由图 1c、图 1d可知,植稻淹水中AOA的amoA基因拷贝数为1.57×107copies·g-1,远高于休耕冬干水稻土的5.28×106copies·g-1。AOB的amoA基因拷贝数为1.88×106copies·g-1,也高于休耕冬干。这两种水稻土中AOA的丰度均高于AOB。
3 讨论水稻土是我国重要的农业土地资源,约占全国耕地面积的1/5。本研究中,与休耕冬干相比,28年长期植稻淹水管理极可能导致水稻土生态位发生显著变化,进而影响氨氧微生物的生理生态功能。长期的施肥和植稻管理,显著促进了水稻土的硝化潜势,使其增幅高达12倍之多。实时荧光定量PCR分析表明,两种水稻土均能检测到Comammox、AOA和AOB,并且其数量均为Comammox > AOA > AOB。尽管Comammox丰度高并不意味着其在水稻土硝化过程中发挥重要作用,但其功能意义却将是未来研究的重点。例如,植稻淹水条件下,大量施肥导致铵态氮浓度显著高于休耕冬干,极高的水分含量也可能导致氧气浓度显著低于后者。植稻淹水土壤中,高氨低氧则可能刺激了Comammox Clade A的生长,而非Clade B,导致两者的比值高达54.1,远远高于休耕冬干水稻土中的比值2.2(表 3)。事实上,已有研究针对旱地土壤仅检测到Comammox Clade B,而未发现Clade A[18]。这些结果也许暗示着Comammox Clade A更适应低氧的淹水环境,而Clade B可能在高氧的旱地土壤中具有生理活性。
|
|
表 3 不同管理模式下水稻土中氨氧化微生物的数量比 Table 3 Ratio of ammonia-oxidizing microorganisms in paddy soils relative to treatment |
施肥通常能够增加土壤铵态氮浓度并刺激土壤硝化活性。植稻淹水中铵态氮的浓度达到了17.77 mg·kg-1,是休耕冬干水稻土的4倍多。底物浓度对氨氧化微生物的相对丰度和分布起着重要的作用,新发现的Comammox被证实能够更好地适应于极低氨浓度的环境。本研究在休耕冬干水稻土中检测到的Comammox Clade B的amoA基因的拷贝数也确实要高于底物浓度较高的植稻淹水水稻土。相比于植稻淹水,休耕冬干水稻土每年4—8月淹水,随后处于落干状态。土壤水分也是影响硝化微生物活性的重要因素,通过控制空气中O2的扩散而间接影响好氧微生物的活性。目前已知的所有氨氧化微生物皆为好氧生长类型,也有研究指出AOA和AOB对水分条件和氧气的要求不同[19-20],但Comammox对氧气胁迫的适应机制尚不清楚。尽管无法区分施肥和淹水管理对氨氧化微生物功能的相对影响,但全年淹水的植稻水稻土中Comammox Clade A更高,表明其能更好适应氧气胁迫,并可能具有更为广泛的底物适应范围。
尽管植稻淹水和休耕冬干水稻土中AOA和AOB数量均显著低于Comammox,但数量上的优势并不一定具有功能意义,目前的研究并不能排除AOA和AOB在硝化过程中发挥了重要作用。事实上已有的研究表明不同母质水稻土中,AOA和AOB对硝化作用的相对贡献明显不同[12]。此外,本研究表明与AOB相比,AOA适应低氨胁迫的能力更强。例如,尽管植稻淹水中AOA和AOB的丰度均要高于休耕冬干水稻土,但在休耕冬干水稻土中,两者丰度的比值却高达118.3,远高于植稻淹水中的9.2,说明在铵态氮浓度较低的休耕冬干水稻土中,AOA较之AOB具有更强的竞争优势(表 3)。这些结果与已有的报道一致,在土壤生态系统中较低的铵态氮浓度下能够观察到更高的AOA:AOB的比值[21]。
农业种植模式、化肥施用、灌溉管理以及土壤类型等均会对微生物数量、活性与种群结构产生较大影响[22-25]。而硝化作用是农田氮循环的关键过程,其发生与速率主要受氨氧化微生物的丰度和活性制约。Comammox的发现表明在农田土壤硝化过程仍有大量亟待解决的科学问题。首先,由于氨被完全氧化成硝酸盐可以释放出更高的能量,那么在什么情况下Comammox对硝化作用的贡献会超过别的硝化微生物?以往对Comammox的忽视是否导致过高估计了传统硝化微生物的功能?此外,尤为重要的是要了解Comammox与传统氨氧化微生物(AOA和AOB)的生态位划分及其功能意义。例如,以往稳定性同位素示踪氨氧化微生物DNA试验中,发现Nitrospira中类群在13C-DNA中的丰度远高于AOA和AOB[12],但这些微生物是否为Comammox并在土壤氨氧化过程中发挥了重要作用,仍待进一步研究。
4 结论在植稻淹水和休耕冬干水稻土中都检测到了较高丰度的Comammox,表明Comammox广泛分布于水稻土壤中,并有可能在水稻土的硝化过程中发挥重要作用。此外,水稻土中Comammox Clade A的丰度要高于Clade B,长期的施肥和淹水管理会对水稻土中Comammox的两个分支的丰度产生一定影响。施肥种植可能刺激了Comammox Clade A、AOA和AOB的生长。
| [1] |
Costa E, Pérez J, Kreft J U. Why is metabolic labour divided in nitrification? . Trends in Microbiology, 2006, 14(5): 213-219. DOI:10.1016/j.tim.2006.03.006
(  0) 0) |
| [2] |
Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria . Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461
(  0) 0) |
| [3] |
van Kessel M A H J, Speth D R, Albertsen M, et al. Complete nitrification by a single microorganism . Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459
(  0) 0) |
| [4] |
Pinto A J, Marcus D N, Ijaz U Z, et al. Metagenomic evidence for the presence of Comammox Nitrospira-like bacteria in a drinking water system . MSphere, 2015. DOI:10.1128/mSphere.00054-15
(  0) 0) |
| [5] |
Palomo A, Jane F S, Gülay A, et al. Metagenomic analysis of rapid gravity sand filter microbial communities suggests novel physiology of Nitrospira spp . ISME Journal, 2016, 10(11): 2569-2581. DOI:10.1038/ismej.2016.63
(  0) 0) |
| [6] |
Prosser J I, Nicol G W. Archaeal and bacterial ammonia-oxidisers in soil:the quest for niche specialisation and differentiation . Trends in Microbiology, 2012, 20(11): 523-531. DOI:10.1016/j.tim.2012.08.001
(  0) 0) |
| [7] |
Martens-Habbena W, Berube P M, Urakawa H, et al. Ammonia oxidation kinetics determine niche separation of nitrifying Archaea and Bacteria . Nature, 2009, 461(7266): 976-979. DOI:10.1038/nature08465
(  0) 0) |
| [8] |
Lu L, Han W, Zhang J, et al. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea . ISME Journal, 2012, 6(10): 1978-1984. DOI:10.1038/ismej.2012.45
(  0) 0) |
| [9] |
Jia Z J, Conrad R. Bacteria rather than Archaea dominate microbial ammonia oxidation in an agricultural soil . Environmental Microbiology, 2009, 11(7): 1658-1671. DOI:10.1111/j.1462-2920.2009.01891.x
(  0) 0) |
| [10] |
Xia W, Zhang C, Zeng X, et al. Autotrophic growth of nitrifying community in an agricultural soil . ISME Journal, 2011, 5(7): 1226-1236. DOI:10.1038/ismej.2011.5
(  0) 0) |
| [11] |
Jiang X J, Hou X Y, Zhou X, et al. pH regulates key players of nitrification in paddy soils . Soil Biology & Biochemistry, 2015, 81: 9-16.
(  0) 0) |
| [12] |
Wang B, Zhao J, Guo Z, et al. Differential contributions of ammonia oxidizers and nitrite oxidizers to nitrification in four paddy soils . ISME Journal, 2015, 9(5): 1062-1075. DOI:10.1038/ismej.2014.194
(  0) 0) |
| [13] |
Kits K D, Sedlacek C J, Lebedeva E V, et al. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle . Nature, 2017, 549(7671): 269-272. DOI:10.1038/nature23679
(  0) 0) |
| [14] |
Chu H, Fujii T, Morimoto S, et al. Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil . Applied and Environmental Microbiology, 2007, 73(2): 485-491. DOI:10.1128/AEM.01536-06
(  0) 0) |
| [15] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations . Applied and Environmental Microbiology, 1997, 63(12): 4704-4712.
(  0) 0) |
| [16] |
Beman J M, Francis C A. Diversity of ammonia-oxidizing archaea and bacteria in the sediments of a hypernutrified subtropical estuary:Bahía del Tóbari, Mexico . Applied and Environmental Microbiology, 2006, 72(12): 7767-7777. DOI:10.1128/AEM.00946-06
(  0) 0) |
| [17] |
Pjevac P, Schauberger C, Poghosyan L, et al. AmoA-targeted polymerase chain reaction primers for the specific detection and quantification of comammox Nitrospira in the environment . Frontiers in Microbiology, 2017. DOI:10.3389/fmicb.2017.01508
(  0) 0) |
| [18] |
Thion C, Pjevac P, Meng A, et al. Determining the influence of soil pH on the abundance, diversity and activity of nitrifiers:Ammonia oxidisers, nitrite oxidisers and comammox . Poster, 2017. DOI:10.13140/RG.2.2.24628.55680
(  0) 0) |
| [19] |
Szukics U, Abell G C J, Hödl V, et al. Nitrifiers and denitrifiers respond rapidly to changed moisture and increasing temperature in a pristine forest soil . FEMS Microbiology Ecology, 2010, 72(3): 395-406. DOI:10.1111/j.1574-6941.2010.00853.x
(  0) 0) |
| [20] |
Szukics U, Hackl E, Zechmeister-Boltenstern S, et al. Rapid and dissimilar response of ammonia oxidizing archaea and bacteria to nitrogen and water amendment in two temperate forest soils . Microbiological Research, 2012, 167(2): 103-109. DOI:10.1016/j.micres.2011.04.002
(  0) 0) |
| [21] |
Verhamme D T, Prosser J I, Nicol G W. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms . ISME Journal, 2011, 5(6): 1067-1071. DOI:10.1038/ismej.2010.191
(  0) 0) |
| [22] |
Wertz S, Leigh A K K, Grayston S J. Effects of long-term fertilization of forest soils on potential nitrification and on the abundance and community structure of ammonia oxidizers and nitrite oxidizers . FEMS Microbiology Ecology, 2012, 79(1): 142-154. DOI:10.1111/j.1574-6941.2011.01204.x
(  0) 0) |
| [23] |
钟文辉, 蔡祖聪, 尹力初, 等. 种植水稻和长期施用无机肥对红壤氨氧化细菌多样性和硝化作用的影响. 土壤学报, 2008, 45(1): 105-111. Zhong W H, Cai Z C, Yin L C, et al. Effects of rice cultivation and long term application of inorganic fertilizers on ammonium oxidizers diversity and nitrification of red soils (In Chinese). Acta Pedologica Sinica, 2008, 45(1): 105-111. DOI:10.3321/j.issn:0564-3929.2008.01.014 (  0) 0) |
| [24] |
Hansel C M, Fendorf S, Jardine P M, et al. Changes in bacterial and archaeal community structure and functional diversity along a geochemically variable soil profile . Applied and Environmental Microbiology, 2008, 74(5): 1620-1633. DOI:10.1128/AEM.01787-07
(  0) 0) |
| [25] |
蔡祖聪, 赵维. 土地利用方式对湿润亚热带土壤硝化作用的影响. 土壤学报, 2009, 46(5): 795-801. Cai Z C, Zhao W. Effects of land use types on nitrification in humid subtropical soils of China (In Chinese). Acta Pedologica Sinica, 2009, 46(5): 795-801. DOI:10.3321/j.issn:0564-3929.2009.05.006 (  0) 0) |
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
 2019, Vol. 56
2019, Vol. 56


