2. 中国科学院大学, 北京 100049
土壤是地球上最多样化的生物栖息地之一, 不仅包含较大的生物体, 如线虫、蚂蚁或鼹鼠等, 还包含大量的细菌和真菌等微生物群体。每克土壤中的微生物数以亿计, 物种类型达10万余种, 这些海量的微生物与复杂的土壤环境总称为土壤微生物组, 其中蕴藏的巨大微生物多样性被认为是地球元素循环过程的重要驱动力[1-3]。传统的土壤微生物多样性研究手段, 如实验室培养法, 能分离鉴定的土壤微生物种类数量较少[4], 近年来随着高通量测序等新技术的快速发展, 大规模、高效、低价检测微生物多样性成为了可能, 同时极大地推动了土壤微生物学研究。
不过面对高通量测序产生的海量数据, 如何进行管理、加工和分析又成为新的课题, 迫使微生物学家不得不加入大数据俱乐部[5]。这种努力又反之促进了微生物领域专业数据库和参考数据库的发展。目前得到广泛应用的微生物数据大部分存储在公共的宏基因组在线数据库平台中, 例如美国阿贡实验室开发的MG-RAST[6-7]、美国能源部联合基因组研究所的整合微生物基因组和宏基因组(IMG/M)[8]、以及metaMicrobesOnline[9]、宏基因组病毒信息学资源(VIROME)[10]、MGnify(原EBI Metagenomics)[11]等。这些在线数据库平台大都提供内置的注释管道, 通过将用户提交的测序数据与后台的参考测序数据库进行比对, 进行物种分类及功能注释。常用的微生物参考测序数据库包括SEED subsystem, COG, KO, NOG, ggNOG, M5RNA, KEGG, TrEMBL, SEED, PATRIC, SwissProt, GenBank, RefSeq, TIGRfam, TIGR, MetaCyc, GO, NCBI Taxonomy, Database of reference genomes, RDP, Greengenes, MGOL, UniRef 100, BacMap, GOLD等[12]。虽然已建成了较多的微生物数据库和参考数据库, 但也有研究指出, 为了全面地解码生态系统, 需要新的工具、框架和假设来分析、存储、可视化和共享数据集, 单个平台不足以进行整体宏基因组学分析, 较长的阅读序列、精确的组装和注释管道是未来宏基因组学研究的发展方向[13]。
对于土壤微生物数据, 常用的参考库包括Greengenes[14], Unite[15], Silva[16], RDP[17], Ez-Taxon[18], eggNOG[19], KEGG[20]等。然而, 已有通用平台大都聚焦于提供微生物数据存储、管理、访问、注释等基础性服务, 缺乏对土壤微生物所处环境数据的系统收集和标准化整合。此外, 对于土壤微生物组研究领域关注的问题, 如环境因素对土壤微生物群落的影响、土壤微生物空间分布格局等, 仍难以提供有效的模型工具支撑。
2014年开始, 中国科学院实施了战略性先导科技专项(B类)“土壤–微生物系统功能及其调控”, 该项目的研究目标之一就是构建服务于我国土壤微生物组研究的专业数据集成和分析平台, 目前平台已完成数据集成整合、可视化分析、空间制图等功能, 本文主要针对平台的架构设计和已实现功能进行介绍。
1 平台架构设计中国土壤微生物组数据平台直接服务于“土壤–微生物系统功能及其调控”先导专项, 为便于专项研究产生的海量数据资源的整合集成, 兼顾未来的持续建设发展, 平台采用了基于B/S的可扩展架构设计, 具体包括基础设施层、数据资源层、应用支撑层、管理业务层、用户服务层五个层次, 以及配套标准规范体系及运维保障体系, 其总体架构如图 1所示。
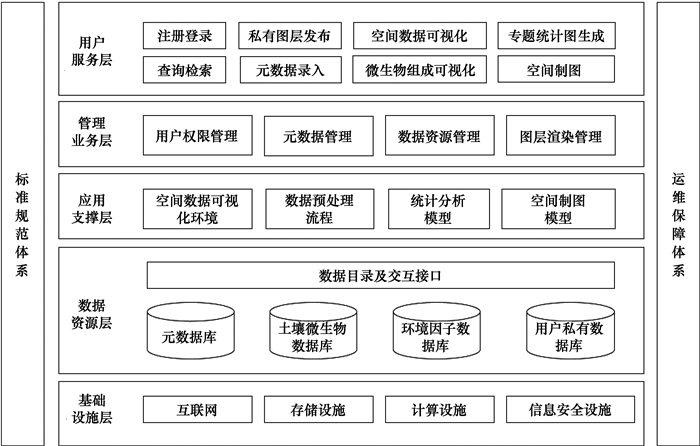
|
图 1 中国土壤微生物组数据平台总体架构 Fig. 1 Framework of the China soil microbiome data platform |
基础设施层包括网络、服务器、防火墙等硬件设施, 需要为平台提供足够的数据存储能力、计算能力、网络带宽及信息安全保障。考虑到土壤微生物组数据具备一定的大数据特征[21], 本平台通过虚拟化等技术将10余台服务器集群的硬件资源池化, 以作为土壤微生物组数据存储及计算分析的基础。同时采用独立的Web服务器用于响应用户请求、提供空间数据引擎及调用模型算法等, 从而提升服务器的安全性和可扩展性。
1.2 数据资源层数据资源层是平台数据资源管理的基础, 具体包括元数据库、土壤微生物数据库、土壤微生物环境因子数据库、用户私有数据库、统一数据目录及数据交互接口等数据管理模块。平台采用基于用户的数据资源权限控制方式, 所整合的数据资源分开放与私有两大类, 其中开放数据资源可被全部用户访问, 而私有数据资源由用户通过数据交互接口集成至平台, 只限上传者本人访问使用。数据目录由元数据库提取数据库关键描述信息生成, 数据交互接口则负责数据层与其他层次的数据交互, 包括数据资源的检索、集成、修改等。
1.3 应用支撑层应用支撑层为平台提供运行环境、工作流程和模型算法等条件保障, 具体包含空间数据可视化环境、数据预处理流程、统计分析模型和空间制图模型等模块。其中空间数据可视化环境采用了成熟的空间数据引擎ArcGIS Server开发实现, 确保所集成微生物组空间数据运行环境的稳健性和跨浏览器兼容性。数据预处理流程主要包含数据标准化、空间化等入库前预处理步骤。统计分析模型集成了微生物数据及环境数据的常用统计方法, 如相对丰度柱状图、时间序列统计图等; 制图模型模块则集成了土壤学领域常用的空间制图方法, 包括反距离权重法、克里金法等。
1.4 管理业务层管理业务层为管理员持续管理运营平台提供有效工具, 具体包括用户权限管理、元数据管理、数据资源管理、图层渲染管理等模块。管理员通过相关模块持续更新平台开放数据资源, 并确保其元数据完整、图层渲染规则合适。考虑到海量、异构土壤微生物组数据及相关元数据管理的复杂性, 在实现相关模块基础管理配置功能的同时还完善了对数据资源的排序、筛选等辅助功能。
1.5 用户服务层用户服务层直接与用户交互, 本平台采用基于B/S的技术架构, 通过Web直接向用户提供数据服务与模型计算服务, 解决了不同操作系统环境下常见的兼容性问题。本层次具体包括用户注册登录、数据查询检索、私有图层发布、元数据录入、空间可视化、微生物组成可视化、空间统计、空间制图等功能模块, 满足土壤微生物数据及环境因子数据整合集成、可视化浏览以及空间分析制图等需求。
2 数据库建设 2.1 数据资源整合数据资源的整合建库是平台为用户提供土壤微生物组研究相关数据及模型计算服务的基础。鉴于土壤微生物组是土壤中所有微生物及其栖息环境的总称[3], 平台收集整合的数据资源主要包括土壤微生物组数据及环境因子数据两部分。整合的数据资源通过元数据库及数据字典进行统一描述和规范, 包括数据来源、数据精度、投影坐标体系、土壤分类体系、数据生产时间等, 其建设依据“土壤科学数据元数据”(GB/T 32739-2016)国标, 确保平台数据具有良好的完整性与可用性。
在平台整合集成的数据资源如图 2所示, 其中土壤微生物数据主要来源于专项实施中产生的海量土壤微生物数据, 具体包括微生物组成和丰度数据、微生物多样性数据和测序序列数据, 由于采用了统一规范的采样及分析方法, 所集成的微生物数据质量较好。而环境因子数据主要依托中国土壤数据库(http://vdb3.soil.csdb.cn/)和土壤科学数据中心(http://soil.geodata.cn/), 具体涵盖从90 m、100 m到1 000 m不同分辨率多尺度的土壤类型因子、土壤理化因子、气候环境因子、地形环境因子、生物环境因子、人为因子等土壤微生物栖息环境有关的数据。其中土壤类型因子包含发生分类、系统分类、美国系统分类、WRB分类等多种土壤分类体系数据。土壤理化因子包含土壤pH、有机质、全氮、全磷等主要土壤属性含量数据。气候环境因子包含年均温度及年均降水量等。地形环境因子包含地形、坡度等。生物环境因子包含植被指数、覆盖度、生物量等。人为因子则包含土地利用、行政区划等。平台整合的数据资源为用户开展土壤微生物组相关研究, 如环境因素对土壤微生物群落的影响、土壤微生物空间分布格局等提供了有力的数据支撑。
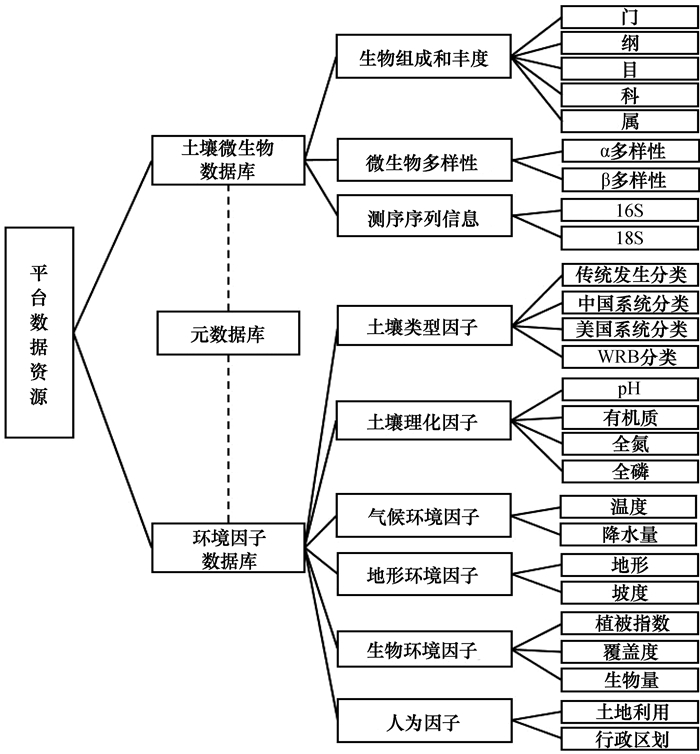
|
图 2 中国土壤微生物组数据平台数据资源体系 Fig. 2 Data resource system of the China soil microbiome data platform |
数据库采用了PostgreSQL数据库代替传统的“关系型数据库+ArcSDE”模式, 使平台可直接通过数据库管理空间数据。与传统的空间数据管理模式相比, PostgreSQL数据库不仅具有开源免费的优势, 还具有更优秀的空间数据管理性能, 更适合管理土壤微生物组数据, 具体体现在以下四方面:
可扩展性强:第三方开源软件很多, 有利于提升系统能力的可扩展性。针对本平台基础设施层分布式集群的架构, 通过第三方开源工具可以方便地解决集群数据传输中读写分离、负载均衡、数据水平拆分等问题。
功能完善:对空间数据存储和分析功能的支持完善, 而本平台整合的数据资源以空间数据为主, 涉及有关的空间数据分析功能, 如空间关系分析、拓扑分析等, 在PostgreSQL数据库中均有相应的SQL函数支持。
兼容性好:属于跨平台的数据库软件, 在各主流操作系统环境上均能部署应用, 同时主流的GIS平台软件如ArcGIS、MapInfo、PostGIS等均支持PostgreSQL数据库, 这为本平台后续集成其他GIS平台的优势功能提供了数据库层面的基础。
存取效率高:传统的空间数据管理, 如Oracle结合ArcSDE, 是原生的关系型数据库和外挂扩展的空间数据结构的结合, 而采用PostgreSQL数据库统一管理关系型和空间数据, 是原生的关系型数据库和原生的空间数据结构的天然统一, 从而提升平台对海量土壤微生物组数据的存取效率。
3 平台功能建设在数据资源整合及数据库建设完成的基础上, 平台基于.NET Web开发框架、IIS发布服务器、C#开发语言, 遵循高内聚、低耦合的功能模块实现原则, 采用ArcGIS Server作为空间数据管理及相关分析功能的开发引擎, 初步建成了基于B/S的中国土壤微生物组数据平台(http://159.226.101.185/microbe), 实现了数据管理、数据可视化、数据分析、用户管理四部分功能模块。建成的功能模块包括数据服务前台与业务管理后台, 为平台持续提供数据服务、维护与更新数据资源提供了支撑保障, 建成的平台功能结构如图 3所示。
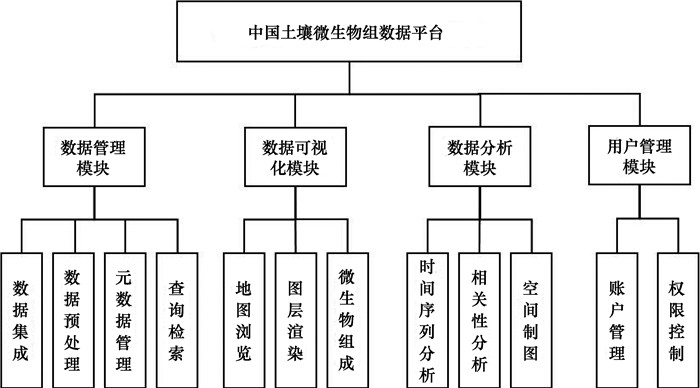
|
图 3 中国土壤微生物组数据平台功能结构图 Fig. 3 Function structure of the China soil microbiome data platform |
数据管理模块用于支撑平台数据资源的发布、管理及检索, 具体包括数据集成、数据预处理、元数据管理、查询检索等功能。支持集成到平台的数据格式除常用的空间数据格式shp和tif外, 同时支持csv、xls、xlsx等多种常用数据格式。数据集成到平台时需要填写元数据信息, 包括数据生产者、数据生产时间、投影坐标体系等。集成到平台的数据经过空间化、地理坐标转换等预处理流程后统一在前台发布。
3.2 数据可视化模块数据可视化模块主要满足平台空间数据浏览展示需求, 包括地图浏览、图层渲染、微生物组成等可视化功能。其中地图浏览功能集成了图层目录、图层选取、视图切换、点查、测距、测面等地图浏览常用工具。图层渲染功能根据属性字段的类别不同或数值大小, 渲染得到不同颜色图斑或不同大小散点进行展示, 从而直观地表达某一属性字段的分布情况, 如图 4a中不同图斑颜色代表了不同土壤类型。微生物组成可视化功能通过叠加柱状图在地图上可视化地展示目标位置土壤微生物的组成及丰度, 并可与平台其他数据如土壤类型图等进行叠加展示, 土壤微生物组成可视化效果如图 4b所示。
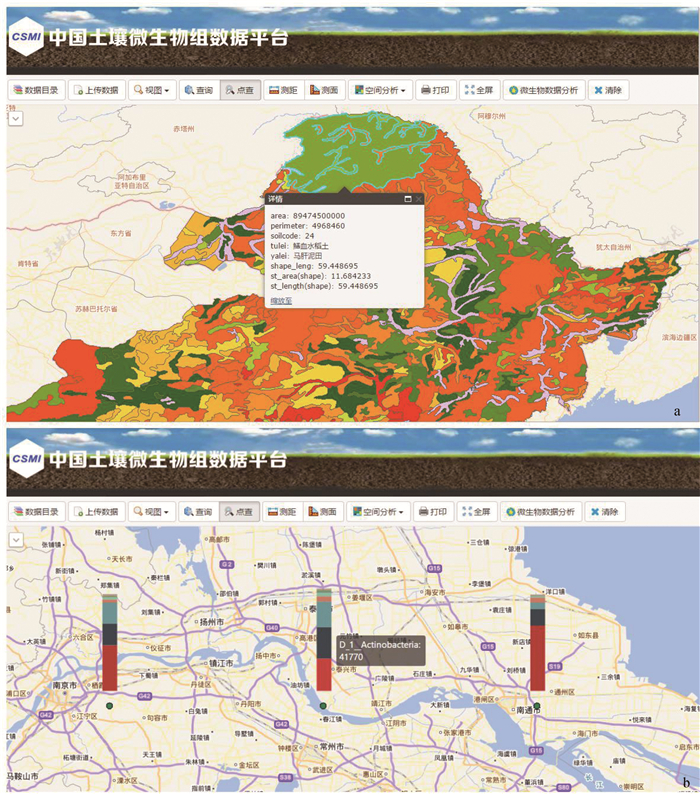
|
图 4 平台数据可视化模块 Fig. 4 Data visualization module of the platform |
平台数据分析模块通过集成土壤微生物组数据常用的统计分析模型, 满足用户开展土壤微生物组数据分析研究需求。具体包括时间序列分析、相关性分析以及空间制图等功能。其中时间序列分析通过折线图结合区域范围选择工具, 分析展示目标属性在选定区域随时间变化的特征。相关性分析通过象限散点图工具分析不同属性字段间的相关关系。分析得到相关关系后, 通过制图数据生成工具结合平台数据资源, 得到用于空间制图的属性图层, 最终通过选取合适的制图模型得到目标属性的分布图, 相关功能具体应用示例见本文第4节。
3.4 用户管理模块用户管理模块负责平台账户管理与用户权限控制, 为平台访问安全和信息共享安全提供保障。用户分管理员与普通用户两类角色, 不同角色用户拥有不同的操作权限。普通用户登录平台后, 可以使用平台私有数据集成、元数据录入等功能管理自己的数据, 并可开展私有数据与平台公开数据的相关性分析、空间分布制图等研究。管理员用户则拥有对平台开放数据资源管理的权限, 包括数据发布、图层渲染规则设置等, 确保平台稳定运行。
4 平台应用示例中国土壤微生物组数据平台建设的核心思路是在整合土壤微生物数据及环境因子数据基础上, 通过集成方法模型, 为土壤微生物组研究领域关注的问题提供数据及模型支撑。本节以表 1数据为示例, 从数据集成、相关性分析、空间制图等环节, 展示平台在土壤微生物多样性空间分布研究的应用。
|
|
表 1 土壤微生物多样性分布研究示例数据 Table 1 Demonstration data of the research on soil microbial diversity distribution |
示例区域为安徽省宣城市, 原始数据以常用的xlsx格式保存, 包含样品编号、经纬度、pH以及OTUs属性字段。用户登录平台后, 通过前台“上传数据”工具选取原始数据, 指定数据存放目录及经纬度字段(图 5a), 同时并补充元数据信息, 其中元数据主要包含数据生产者、数据生产时间、数据简要描述等(图 5b)。平台检查数据格式以及指定信息无误后, 通过空间化步骤将用户上传的关系型数据转换成统一地理坐标的空间数据入库并发布, 成功发布后用户即可开展可视化的地图浏览及分析(图 5c)。
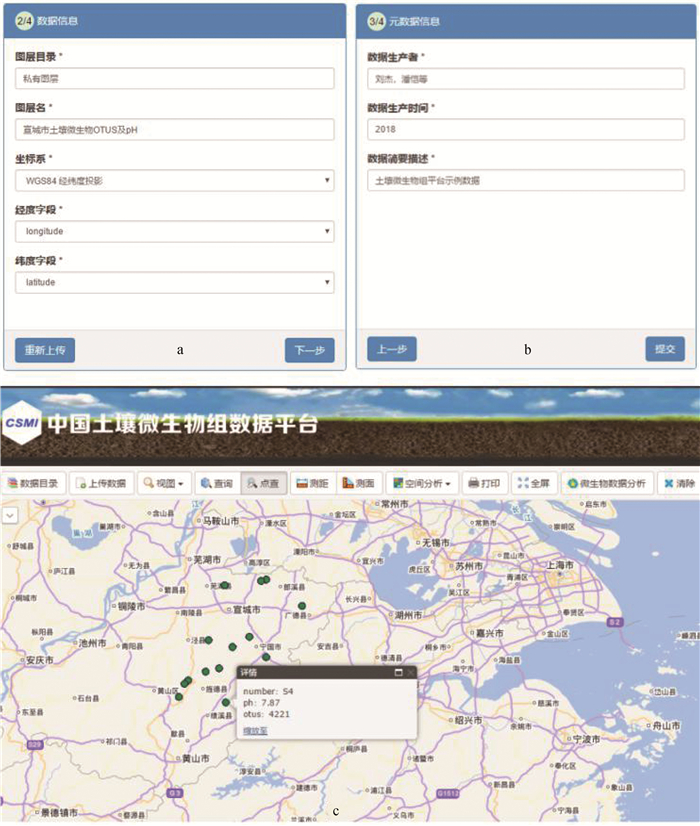
|
图 5 数据集成示例 Fig. 5 Example of data integration |
数据集成到平台后, 通过象限图工具建立土壤微生物OTUs与环境因子pH之间的关系模型, 通过指定目标字段OTUs和pH, 生成的象限图效果如图 6所示。通过象限图初步发现在研究区域内的土壤微生物OTUs与pH具备一定的线性关系, 进一步计算得到线性回归方程为:Y=831.68 X-1971.8, 其中Y代表OTUs, X代表pH, 对应决定系数R2为0.868 9。
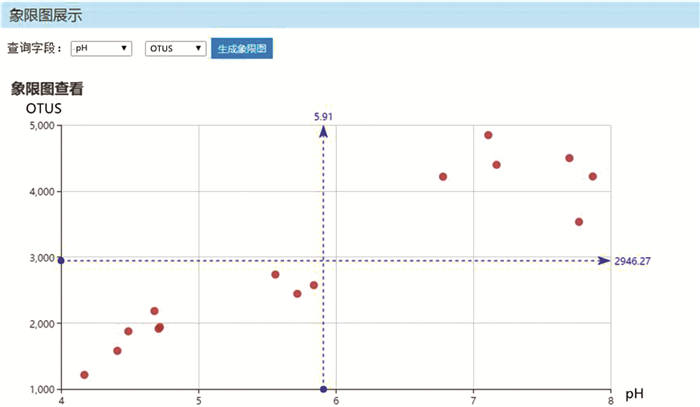
|
图 6 数据相关性分析示例 Fig. 6 Example of data correlation analysis |
基于得到的土壤微生物OTUs与pH的关系模型, 结合平台已整合的土壤理化因子数据库中的全国第二次土壤普查典型剖面pH属性数据, 可以进一步开展宣城市土壤微生物OTUs全国第二次土壤普查空间分布的研究, 获取该时期宣城市土壤OTUs空间分布图, 具体包括制图数据准备和空间制图两个步骤。
首先依据得到的线性相关关系和全国第二次土壤普查典型剖面pH属性数据, 通过“制图数据生成”工具导入计算模型(图 7a), 生成用于绘制普查期间宣城市OTUs空间分布图的数据图层。完成数据准备工作后, 利用平台“空间插值制图”功能, 选择合适的制图方法如反距离权重法或克里金法, 绘制得到普查时期的宣城市土壤微生物OTUs分布图(图 7b), 并可以与现阶段采集样品绘制得到的OTUs分布图开展更深层次的时空变化分析等研究。

|
图 7 空间分布制图示例 Fig. 7 Example of spatial distribution mapping |
平台目前已完成架构设计、数据库建设以及数据集成整合、可视化分析、空间制图等主要功能, 初步具备了服务我国土壤微生物组研究的能力基础, 但在未来数据资源持续丰富以及相关计算制图模型持续完善上仍有一些问题值得分析和讨论。
1) 数据资源。平台目前集成的土壤环境因子数据主要依托于中国土壤数据库及土壤科学数据中心中的第二次土壤普查成果, 然而该调查距今已近40年, 亟需现势性更强的土壤环境因子数据资源, 从而进一步提升对我国土壤微生物组研究支撑的能力。
2) 相关性分析。平台初步实现了基于二维象限图工具的土壤微生物数据与环境因子数据相关性分析功能, 未来需集成更丰富的数据分析模型, 支撑涵盖线性及非线性关系的土壤微生物-环境因子挖掘建模研究。
3) 空间制图。平台目前已集成了多种土壤学领域常用的空间制图模型, 包括反距离权重模型、克里金模型等, 但尚缺少适用性强的数字土壤制图模型。下一步将结合数字土壤制图领域新近研究进展, 研究实现适用于土壤微生物多样性空间分布的数字土壤制图模型。
6 结语中国土壤微生物组数据平台是战略性先导科技专项(B类)“土壤–微生物系统功能及其调控”的一项重要成果。在数据整合方面, 通过统一标准规范系统收集整合了我国土壤微生物数据及环境因子数据。在功能建设方面, 通过应用先进的空间数据库技术和WebGIS技术, 初步实现了数据集成、可视化分析、空间制图等功能。目前平台已集成约10GB的土壤微生物数据和多尺度的各环境因子数据, 并在专项团队内部开放试运行, 随着平台数据资源及功能的不断丰富和完善, 期望将其建设成为我国土壤微生物组研究领域的重要公共支撑平台。
| [1] |
Torsvik V, Øvreås L. Microbial diversity and function insoil:From genes to ecosystems . Current Opinion in Microbiology, 2002, 5(3): 240-245. DOI:10.1016/S1369-5274(02)00324-7
(  0) 0) |
| [2] |
Veresoglou S D, Halley J M, Rillig M C. Extinction risk of soil biota . Nature Communications, 2015, 6: 8862-8871. DOI:10.1038/ncomms9862
(  0) 0) |
| [3] |
朱永官, 沈仁芳, 贺纪正, 等. 中国土壤微生物组:进展与展望. 中国科学院院刊, 2017, 32(6): 554-565. Zhu Y G, Shen R F, He J Z, et al. China soil microbiome initiative:Progress and perspective (In Chinese). Bulletin of Chinese Academy of Sciences, 2017, 32(6): 554-565. (  0) 0) |
| [4] |
Achtman M, Wagner M. Microbial diversity and the genetic nature of microbial species . Nature Reviews Microbiology, 2008, 6: 431-440. DOI:10.1038/nrmicro1872
(  0) 0) |
| [5] |
Marx V. Biology:The big challenges of big data . Nature, 2013, 498: 255-260. DOI:10.1038/498255a
(  0) 0) |
| [6] |
Glass E M, Wilkening J, Wilke A, et al. Using the Metagenomics RAST server(MG-RAST)for analyzing shotgun metagenomes . Cold Spring Harbor Protocols, 2010. DOI:10.1101/pdb.prot5368
(  0) 0) |
| [7] |
Meyer F, Paarmann D, D'Souza M, et al. The metagenomics RAST server-A public resource for the automatic phylogenetic and functional analysis of metagenomes . BMC Bioinformatics, 2008, 9: 386-393. DOI:10.1186/1471-2105-9-386
(  0) 0) |
| [8] |
Chen I A, Markowitz V M, Chu K, et al. IMG/M:Integrated genome and metagenome comparative data analysis system . Nucleic Acids Research, 2017, 45(D1): 507-516. DOI:10.1093/nar/gkw929
(  0) 0) |
| [9] |
Chivian D, Dehal P S, Keller K, et al. metaMicrobesOnline:Phylogenomic analysis of microbial communities . Nucleic Acids Research, 2013, 41(D1): 648-654.
(  0) 0) |
| [10] |
Wommack K E, Bhavsar J, Polson S W, et al. VIROME:A standard operating procedure for analysis of viral metagenome sequences . Standards in Genomic Sciences, 2012, 6: 427-439. DOI:10.4056/sigs.2945050
(  0) 0) |
| [11] |
Mitchell A, Bucchini F, Cochrane G, et al. EBI metagenomics in 2016-An expanding and evolving resource for the analysis and archiving of metagenomic data . Nucleic Acids Research, 2016, 44(D1): 595-603. DOI:10.1093/nar/gkv1195
(  0) 0) |
| [12] |
Dudhagara P, Bhavsar S, Bhagat C, et al. Web resources for metagenomics studies . Genomics, Proteomics & Bioinformatics, 2015, 13(5): 296-303.
(  0) 0) |
| [13] |
Thomas T, Gilbert J, Meyer F. Metagenomics-A guide from sampling to data analysis . Microbial Informatics and Experimentation, 2012, 2: 3-14. DOI:10.1186/2042-5783-2-3
(  0) 0) |
| [14] |
DeSantis T Z, Hugenholtz P, Larsen N. Greengenes, A chimera-checked 16S rRNA gene database and workbench compatible with ARB . Applied and Environmental Microbiology, 2006, 72(7): 5069-5072. DOI:10.1128/AEM.03006-05
(  0) 0) |
| [15] |
Kõljalg U, Larsson K, Abarenkov K, et al. UNITE:A database providing web-based methods for the molecular identification of ectomycorrhizal fungi . New Phytologist Trust, 2005, 166(3): 1063-1068. DOI:10.1111/j.1469-8137.2005.01376.x
(  0) 0) |
| [16] |
Badapanda C, Rani R, Sahoo G C. Advancing our understanding of the soil microbial communities using QⅡME software:A 16S data analysis pipeline . Journal of Applied Biotechnology & Bioengineering, 2017, 4(3): 610-616.
(  0) 0) |
| [17] |
Cole J R, Chai B, Farris R J, et al. The Ribosomal Database Project(RDP-Ⅱ):Sequences and tools for high-throughput rRNA analysis . Nucleic Acids Research, 2005, 33(S1): D294-D296.
(  0) 0) |
| [18] |
Kim O S, Cho Y J, Lee K, et al. Introducing EzTaxon-e:A prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species . International Journal of Systematic and Evolutionary Microbiology, 2012, 62(3): 716-721.
(  0) 0) |
| [19] |
Jensen L J, Julien P, Kuhn M, et al. eggNOG:Automated construction and annotation of orthologous groups of genes . Nucleic Acids Research, 2008, 36(S1): D250-D254.
(  0) 0) |
| [20] |
Kanehisa M, Goto S. KEGG:Kyoto Encyclopedia of Genes and Genomes . Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27
(  0) 0) |
| [21] |
亓合媛, 孙清岚, 马俊才. 微生物组大数据管理与分析. 微生物学报, 2017, 57(6): 932-941. Qi H Y, Sun Q L, Ma J C. Big data management and analysis of microbiome (In Chinese). Acta Microbiologica Sinica, 2017, 57(6): 932-941. (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China
 2019, Vol. 56
2019, Vol. 56


