土壤盐碱化是全球旱作农区最突出的生态环境问题,已成为影响粮食产量和粮食安全的重要限制因素[1]。根据联合国粮食及农业/教科文组织(FAO/ UNESCO)提供的数据(http://www.fao.org/ag/agl/agll/spush/intro.htm),全球约有8.3亿hm2的土地受到盐胁迫的威胁,其中约4.3亿hm2的土地为碱土。我国北方地区土壤含水量较低,土壤多呈碱性,不合理的地下水灌溉,加剧土壤的碱化[2]。目前植物耐盐碱的研究主要集中于盐胁迫上,而对碱胁迫特别是高pH的研究较少。研究表明,碱胁迫对植物的伤害较盐胁迫更严重、更复杂[3]。因此,研究碱胁迫下相关基因的表达及调控网络,将是培育耐碱新品种和减少农作物损失的重要途径之一。
高pH会严重影响土壤结构,增加土壤的硝化作用[4],还会打破植物细胞内的电荷平衡和pH内稳态,阻碍植物对水和营养物质的吸收,造成渗透胁迫,诱导植物细胞产生活性氧,破坏细胞结构完整性,最终破坏根的结构和功能,抑制根发育[5-7]。因此,维持胞内的pH稳态和调节根外pH是植物抗碱的关键所在。植物调节胞内pH稳态主要依赖于质子泵H+-ATPase[8]。定位于质膜上的H+-ATPase将胞质中的H+运输至细胞膜外;定位于液泡上的V型H+-ATPase会将胞质中的H+运输至液泡内;此外,质子共转运体或反向转运体(H+-Symporter和H+-Antiporter)也会改变胞质内的pH[9-10]。蛋白激酶PKS5(Salt overly sensitive 2-like protein kinase 5)是质膜H+-ATPase的负调控因子,pks5功能缺失突变体由于可以泵出更多的H+至胞外,从而表现出更加耐高pH的表型[11]。
近几年,研究人员利用高通量测序技术细致研究了小麦、水稻以及大豆等在碱胁迫(高浓度NaHCO3处理)条件下的转录表达谱[12-14]。然而,高pH胁迫下植物的转录应答机制,仍然知之甚少。本研究旨在对植物耐碱机制进行初步研究,通过本实验室建立的pH表型观察系统,结合新一代高通量测序手段-转录组测序(RNA-Seq),以期获得高pH胁迫响应基因以及转录调控网络,为植物耐碱分子机理的研究以及耐碱品种的培育提供强有力的理论依据。
1 材料与方法 1.1 供试材料实验所用拟南芥(Arabidopsis thaliana)为哥伦比亚(Col-0)生态型,为本实验室保存的种子。
1.2 基于固体培养基的pH实验体系预培养基的配制:称取0.866 g MS(Murashige & Skoog,Caisson labs公司),4 g蔗糖(Sigma-Aldrich公司),加入超纯水完全溶解后,定容至400 mL,NaOH调节pH至6.0,随后加入3.2 g琼脂(Sigma-Aldrich公司)摇匀,高压灭菌。培养基冷却至55℃,超净工作台内于灭菌方皿(10 cm × 10 cm)中加入培养基50 mL,现用现配。
对照培养基的配制:称取0.866 g MS(Murashige & Skoog,Caisson labs公司),加入超纯水完全溶解后,定容至400 mL,NaOH调节pH至6.0,随后加入3.2 g琼脂(Sigma-Aldrich公司)摇匀,高压灭菌。培养基冷却至55℃,超净工作台内于灭菌方皿(10 cm × 10 cm)中加入培养基50 mL,现用现配。
高pH培养基的配制:称取0.866 g MS(Murashige & Skoog,Caisson labs公司),加入超纯水完全溶解后,定容至400 mL,NaOH调节pH至8.0,随后加入3.2 g琼脂(Sigma-Aldrich公司)摇匀,高压灭菌。培养基冷却至55℃,超净工作台内于灭菌方皿(10 cm × 10 cm)中加入培养基50 mL,现用现配。
以上培养基灭菌采用湿热灭菌法,于全自动高压锅(MLS-3781L,松下,日本)中121℃灭菌20 min。
高pH胁迫处理:拟南芥Col-0种子氯气法灭菌后,播种至预培养基上[1/2 MS,1%(w/v)蔗糖,0.8%(w/v)琼脂,pH 6.0];4℃黑暗春化2 d,随后转移至光照培养箱继续生长5 d;挑选长势一致的幼苗分别转移至对照培养基[1/2 MS,0.8%(w/v)琼脂,pH 6.0]和高pH培养基[1/2 MS,0.8%(w/v)琼脂,pH 8.0],光照培养箱内生长7 d,观察表型、统计根长或取根部组织进行转录组学分析。培养条件:14 h光照/10 h黑暗,湿度60%~70%,温度23℃/21℃。
1.3 RNA提取、质控和建库RNA提取与质控:称取对照培养基和高pH培养基上处理7 d的拟南芥根组织0.1 g,TRIzol法提取组织总RNA[15],每个处理组三个生物学重复。随后用琼脂糖凝胶电泳和超微量分光光度计(Nanophotometer N60,德国IMPLEN公司)检测RNA浓度和OD260/OD280、OD260/OD230的比值。
建库:将符合标准的RNA利用Oligo dT的磁珠对mRNA进行富集,加入片段化缓冲液(Fragmentation buffer)将富集的mRNA片段化;用随机引物反转合成第一条cDNA链,随后加入缓冲液、dNTPs、RNase H和DNA polymerase I合成第二条cDNA链;利用核酸纯化试剂盒(AMPure XP beads,Beckman公司)纯化双链cDNA,再进行末端修复、加A并连接测序接头,经片段大小选择后(300~400 bp),PCR富集获得cDNA测序文库,华大测序平台(BGISEQ)完成测序。
1.4 数据处理基因表达量的计算:将测序得到的原始数据(Raw data)进行数据过滤(去除测序接头、重复冗余序列以及低质量的序列),获得高质量的干净数据(Clean data)。利用软件TopHat2将各个样品的干净数据(Clean data)与拟南芥参考基因组进行比对,并计算获得各个基因的片段数(Reads count)以及每百万转录本的每百万个映射片段碎片FPKM(Fragments Per Kilobase of transcript per Million mapped fragments)值。
差异基因表达的筛选:通过使用DESeq自带的标准化方法对原始数据进行标准化处理。使用负二项分布法对片段数(Reads count)的分布进行估计,计算完P值后,对P值进行多重假设检验校正。差异基因筛选标准为:| log2(差异倍数,Fold Change)| > 1且校正的P值(Padj) < 0.001。
GO基因功能注释分析:利用https://www.Arabidopsis.org/tools/bulk/go/index.jsp网站在线工具,对差异表达基因进行基因本体论GO(Gene ontology)注释和功能分类。
转录调控网络预测分析:利用Biomart网站获取GO亚分类中运输(Transport)、细胞组分组成(Cellular component organization)、蛋白质代谢(Protein metabolism)、根发育(Root development)、信号传导(Signal transduction)和应答非生物刺激(Response to abiotic stimulus)相关差异表达基因上游约1 kb的启动子序列。使用植物转录调控预测网站http://plantregmap.cbi.pku.edu.cn/对上述启动子序列与差异表达转录因子的转录调控关系进行预测,Cytoscape软件绘制转录调控网络图。
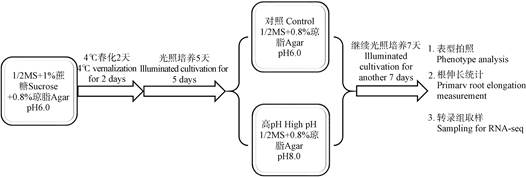
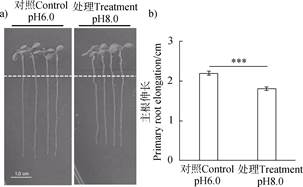
2 结 果 2.1 pH实验体系的确立为研究高pH胁迫对植物发育的影响,首先确立了pH表型观察系统。综合前人研究[16]与前期实验摸索,基础培养基选择1/2 MS培养基,pH调节剂选择NaOH,凝固剂选择琼脂。灭菌的拟南芥种子4℃黑暗春化后,在添加1%(w/v)蔗糖、初始pH(添加琼脂和灭菌前的培养基pH)为6.0的预培养基上培养5 d,转移至不添加蔗糖、初始pH 6.0的对照培养基以及初始pH 8.0的高pH培养基继续光照培养7 d,观察表型以及统计该七天主根的伸长长度(图 1)。预实验发现,不同实验批次或者同一批次相同条件的不同培养皿之间表型有时会存在较大变异,猜测可能与每次配制培养基体积以及每个培养皿中培养基体积存在差异有关。因此,本研究固定了培养基的配制体系,即单次培养基配制体积为400 mL,每皿(10 cm × 10 cm)培养基的体积为50 mL,现用现配。图 2表示萌发后五天的拟南芥幼苗经对照(pH 6.0)和高pH(pH 8.0)培养基处理七天后的表型图(图 2a))和主根伸长长度统计图(图 2b))。从图中可以看出,高pH胁迫下,拟南芥主根生长发育受到显著抑制,导致主根长度显著短于对照组。
|
图 1 pH实验体系的建立 Fig. 1 Establishment of a pH experiment system |
|
注:a)高pH胁迫处理7 d后拟南芥幼苗表型, 虚线表示处理前主根长度, 标尺为1 cm; b)高pH胁迫处理7 d后拟南芥主根伸长长度统计图, ***表示P < 0.01差异极显著(学生t检验, n = 30)。 Note: a)Phenotype of Arabidopsis seedling under high pH stress for 7 days, the red dotted line represents length of the primary root before treatment, bar = 1 cm; b)Length of the primary root increased after 7 days of high pH stress, *** indicates a significant difference at P < 0.01(student's t test, n = 30). 图 2 高pH胁迫下拟南芥表型 Fig. 2 Phenotype of Arabidopsis under high pH stress |
为确定高pH胁迫对拟南芥根转录水平的影响,将萌发后五天的拟南芥幼苗经对照(pH 6.0)和高pH(pH 8.0)培养基处理七天,分别取拟南芥总根进行转录组测序分析,每组三个生物学重复。对照组和高pH处理组测序的原始序列均为21.94 M;过滤掉接头和低质量的序列,对照组干净序列为21.86 M、21.87 M、21.87 M,高pH处理组干净序列为21.87 M、21.86 M、21.88 M;经序列拼接比对到参考基因组,其比对率分别为98.26%、98.3%、98.3%、98.28%、98.26%、98.27%(表 1)。以上数据表明转录组测序质量较高,符合进一步生物信息学分析。
|
|
表 1 各个样品转录组测序数据统计 Table 1 Statistics of RNA-seq data of samples |
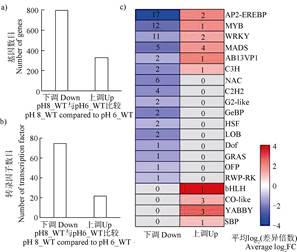
利用DESeq软件包对对照组和高pH处理组的基因进行差异表达分析,以差异表达倍数大于两倍或小于0.5倍即|log2(差异倍数Fold Change)| > 1和校正的P < 0.001为标准,共筛选获得根部差异表达基因1 129个,其中800个基因表达下调,329个基因表达上调(图 3a))。这表明高pH胁迫下,拟南芥根下调的基因显著多于上调的基因。研究表明,转录因子在基因表达中起着至关重要的作用,本研究着重分析了高pH胁迫下差异表达的转录因子。如图 3b)所示,相比对照组,高pH胁迫处理后22个转录因子表达上调,75个转录因子表达下调。将差异表达转录因子按照转录因子家族分类发现,有些转录因子家族bHLH(basic Helix-Loop-Helix)、CO-like(CONSTANS-like)、YABBY等仅存在表达上调的基因,如YABBY家族中3个成员表达上调,无表达下调的基因;有些转录因子家族NAC、C2H2、HSF(Heat stress transcription factors)等仅具有表达下调的基因,如NAC家族中6个成员表达下调,无表达上调的基因;有些转录因子家族AP2-EREBP、MYB、WRKY等既有表达上调也有表达下调的基因,如AP2-EREBP家族中17个成员表达下调,2个成员表达上调(图 3c))。
|
注:a)高pH胁迫下根组织上调和下调基因的数目;b)高pH胁迫下根组织上调和下调的转录因子的数目;c)差异表达的转录因子家族分析,每个方格颜色表示该家族差异表达转录因子的平均log2(差异倍数),方格内数值表示该家族差异表达转录因子的个数。 Note:a)Number of differentially expressed genes of Arabidopsis under high pH stress; b)Number of differentially expressed TFs(transcription factors)of Arabidopsis under high pH stress; c)Differentially expressed TFs were categorized by the TF family, the color of each square represents the value of average log2FC(Fold Change)and the number in each square represents the number of differentially expressed TFs in the corresponding TF family. 图 3 高pH胁迫下拟南芥根差异表达基因 Fig. 3 Differential expression genes of Arabidopsis root under high pH stress |
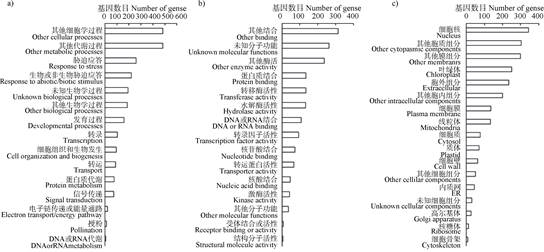
为了确定高pH胁迫响应基因的功能,对差异表达基因进行GO功能注释。如图 4a)所示,在GO类目生物学过程中,注释分类到非生物胁迫应答、发育过程、转录、细胞组织和生物发生、转运、蛋白质代谢和信号转导等GO亚类目中的差异表达基因所占比例较高;在GO类目分子功能中(图 4b)),差异表达基因主要分类到蛋白质结合、转移酶活性、水解酶活性、转录因子活性、转运蛋白活性以及激酶活性等GO亚类目中;在GO类目细胞组分中(图 4c)),差异表达基因主要分布在胞外组分、细胞膜、线粒体、细胞质和质体等部位。以上结果表明,高pH胁迫会导致拟南芥根部基因转录、离子运输、细胞组织与发生、信号转导、发育等过程发生显著变化。下面,本研究将对这些生物学过程的基因进行更加细致的分析。
|
图 4 高pH胁迫下根组织差异表达基因的基因本体论(GO)功能注释(a)生物学过程,b)分子功能,c)细胞组分) Fig. 4 Gene ontology(GO)functional annotation of differentially expressed genes under high pH condition(a)Biological process, b)Molecular function, c)Cellular component) |
离子运输是维持细胞内pH稳态的一个重要因素,也是植物应答高pH胁迫的重要途径[8]。GO功能注释显示,约51个差异表达基因被分类到GO亚类目(GO:0006811)离子运输中,其中,编码钾离子转运蛋白的基因SKOR、编码阴离子/质子转运蛋白的基因CHX20和编码锌离子转运蛋白的基因ZIP5的表达在高pH胁迫下显著上调,而编码硫酸盐转运蛋白的基因SULTR1;1、SULTR2;1和SULTR3;1以及编码HCO3-转运蛋白的基因BOR5的表达在高pH胁迫下显著下调(图 5a))。这表明,拟南芥根部编码K+、Zn2+、Ca2+以及HCO3-转运蛋白的基因参与了拟南芥高pH胁迫响应。

|
图 5 高pH胁迫响应基因差异表达分析(a)离子运输,b)细胞壁合成,c)Ca2+信号,d)植物激素) Fig. 5 Differential expression analysis of high pH responsive genes(a)Ion transport, b)Cell wall organization, c)Ca2+ signaling, d)Phytohormone) |
细胞内外pH的变化对于细胞壁的合成起着至关重要的作用。GO功能注释显示,约48个基因被分类到GO亚类目细胞壁组织和生物发生过程(GO:0071554)。在高pH胁迫下,编码扩张蛋白的基因EXPA17表达上调,而EXPA6、EXPA8、EXPB2和EXLA3等基因表达下调,编码纤维素合成酶的基因CSLG1表达下调(图 5b))。此外,编码果胶裂解酶的基因表达量在高pH胁迫下也发生了显著变化(图 5b))。这表明高pH胁迫会对拟南芥根细胞壁形成造成伤害。
Ca2+作为第二信使在许多植物发育过程中起着至关重要的作用[17]。不同刺激下细胞内Ca2+的动态变化与胞内pH的变化存在紧密联系[18]。转录组结果显示,编码钙调蛋白的基因CML23、CML37和CML38在高pH胁迫下表达量显著降低(图 5c))。这表明,高pH胁迫会减弱细胞内的Ca2+信号传递水平。此外,高pH胁迫还会影响植物激素合成运输以及信号传递相关基因的表达,本研究共筛选出10个差异表达的生长素合成、运输和信号传递相关基因以及5个细胞分裂素合成与代谢相关基因(图 5d))。
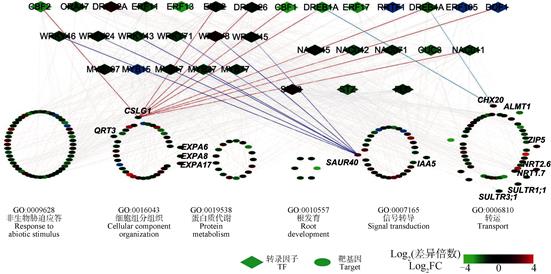
2.6 高pH胁迫转录调控网络的构建转录因子在植物非生物胁迫应答中起着至关重要的作用,本研究结果显示高pH胁迫下差异表达的转录因子占总差异表达基因的8.6%。利用转录调控关系预测网站,对参与高pH胁迫响应的非生物胁迫应答(GO:0009628)、细胞组分组织(GO:0016043)、蛋白质代谢(GO:0019538)、根发育(GO:0048364)、信号转导(GO:0007165)和转运(GO:0006810)等生物学过程相关基因与差异表达的转录因子之间的转录调控关系进行了预测。
根据预测结果,发现约有33个差异表达转录因子与高pH胁迫响应基因存在转录调控关系,且大部分转录因子在高pH胁迫下表达量显著下调(图 6)。根据转录因子家族分类,CBF1、CBF2、DDF1、DREB1A、RRTF1等14个AP2/EREBP家族成员与高pH胁迫应答基因存在密切调控关系;WRKY家族WRKY8、WRKY24、WRKY43、WRKY45、WRKY46和WRKY71等六个成员可能调控高pH胁迫响应基因的表达;NAC011、NAC042、NAC045、NAC071和CUC3等5个NAC转录因子以及MYB15、MYB17、MYB27、MYB77和MYB107等5个MYB转录因子家族也与高pH胁迫响应基因存在密切调控关系(图 6)。此外,参与不同生物学过程的高pH胁迫响应基因具有的转录因子调控位点也可能存在差异。例如,编码细胞壁纤维素合成关键酶的基因CSLG1的启动子区存在AP2/EREBP和NAC家族成员的结合位点;生长素信号响应基因SAUR40的启动子区主要具有WRKY家族转录因子的调控位点;而阴离子/质子转运基因CHX20启动子区仅存在DREB1A和DREB4A的结合位点(图 6)。
|
图 6 高pH胁迫响应基因的转录调控网络 Fig. 6 Transcriptional regulatory network of high pH responsive genes |
碱胁迫由于其高pH通常对植物的生长抑制效应远大于盐胁迫[19]。高pH处理下,拟南芥的根生长受到抑制,这种抑制可能与细胞壁酸性环境缺失、离子稳态失衡、活性氧积累、细胞壁完整性破坏、蛋白质变性等密切相关[20]。本研究结果显示,高pH胁迫处理七天后,离子转运基因的表达量发生了显著变化,如负责铜离子、硫酸盐离子、硝酸盐离子、钾离子、硼离子、碳酸氢根离子和铝离子等转运的基因均参与高pH胁迫应答(图 5a))。研究表明,细胞质中低Na+高K+水平是许多酶促反应维持所必须的,在碱胁迫下水稻根细胞中Na+积累异常,而K+、NO3-、H2PO4-、Cl-水平会显著下降,从而产生离子毒害抑制水稻根的发育[5]。本研究也发现,编码K+离子通道的基因HAK5以及NO3-离子通道的基因NRT1.7的表达量在高pH胁迫下显著下降(图 5a))。进一步,转录调控预测分析表明,HAK5和NRT1.7基因启动子区分别具有MYB15和NAC011转录因子结合位点。NAC和MYB家族成员参与多种非生物胁迫响应过程[21-22],高pH胁迫下MYB15和NAC011基因的表达也显著降低,暗示两者可能通过调控离子转运基因维持离子稳态来参与高pH的胁迫应答过程。
植物细胞的细胞壁是由纤维素和果胶组成,其中还包含一些其他酶类如伸展蛋白和扩张蛋白等[23]。细胞壁组成相关基因的异常会改变植物对非生物胁迫的抗性[23],例如,拟南芥果胶合成基因AtCSLD5功能缺失突变体sos6(Salt overly sensitive 6)表现出渗透、盐和干旱胁迫敏感的表型[24]。本研究结果表明,高pH胁迫下编码纤维素合成酶的基因CSLG1和编码扩张蛋白的基因EXPA6和EXPA8的表达量显著降低(图 5b)),这可能是拟南芥根生长受到抑制的原因之一。转录调控预测分析发现,CSLG1启动子区存在多个AP2/EREBP和NAC类转录因子的结合位点,他们的表达量均发生显著下调。这暗示提高相关转录因子的表达量可能会提高植物对高pH的耐受性。
研究表明,Ca2+信号以及植物激素在非生物胁迫应答过程中起着重要作用[25]。Yang等[26]发现钙离子感受器SCaBP3/CBL7一方面可以与质膜质子泵AHA2的C末端互作,促进AHA2的自抑制,另一方面还可以与PKS5互作,稳定PKS5与AHA2的相互作用,进而促进PKS5对AHA2的抑制,调控胞内pH稳态。此外,大豆GsCML27作为Ca2+结合蛋白在植物对碱胁迫、盐胁迫和渗透胁迫的反应中起至关重要的作用[27]。同样地,本研究结果也发现高pH胁迫会导致Ca2+信号组分相关基因表达显著降低,如编码钙调蛋白的基因CML23、CML37、CML38和编码TSK相关蛋白的基因TSA1等(图 5c))。因此,调控Ca2+信号组分的表达水平也是植物响应高pH胁迫的主要途径之一。
3.2 AP2/EREBP、WRKY、MYB以及NAC等转录因子在高pH胁迫应答网络中发挥重要作用转录因子参与调控植物代谢与发育相关基因的表达,且在环境胁迫应答等过程中具有重要作用。研究表明,转录因子在盐碱胁迫应答中也具有重要作用。An等[28]通过对盐碱处理1 d和7 d的紫花苜蓿的幼苗进行转录组测序发现,109个转录因子在盐碱处理1 d后表达发生显著变化,而96个转录因子在盐碱处理7 d后表达发生显著变化,其中它们大多数属于非生物胁迫应答转录因子家族,例如MYB、WRKY、NAC、AP2/EREBP、bHLH和bZIP等。同样地,2018年Li等[13]通过转录组分析的方法比较了水稻耐碱品种WD20342和碱敏感品种Caidao在正常和碱胁迫处理下的差异表达基因,经差异表达分析发现,一共有576个转录因子的表达存在差异,这些转录因子分别属于MYB、WRKY、NAC、AP2-EREBP、bHLH和bZIP等69个转录因子家族。与以上研究结果一致,本研究的转录组分析结果同样证实,高pH胁迫处理七天后拟南芥根部一共有97个转录因子的表达发生了显著变化,它们分别属于AP2-EREBP、MYB、WRKY、MADS、NAC、bHLH等转录因子家族(图 3b)和图 3c))。
进一步地,本研究对差异表达转录因子与碱胁迫应答关键基因之间的调控网络关系进行了预测,结果证实14个AP2/EREBP家族成员、6个WRKY家族成员、5个NAC家族成员和5个MYB家族成员与碱胁迫应答关键基因如离子运输、蛋白质代谢以及非生物胁迫应答等相关基因存在着密切的转录调控关系(图 6)。这些转录因子大多在非生物胁迫应答过程起着重要的作用。例如,AP2/EREBP家族成员CBF1和CBF2主要参与植物抗冻胁迫[29];DREB1A参与干旱和低温胁迫应答[30];WRKY家族成员WKRY8、WRKY46和STZ参与拟南芥盐胁迫应答[31-33];此外,过表达拟南芥RRTF1会导致根与茎中活性氧的积累[34]。转录调控网络预测发现,高pH胁迫响应基因启动子区存在上述转录因子的结合位点,且它们的表达也会受到高pH的影响,暗示它们在高pH胁迫应答中也具有重要的作用。因此,CBF1、DREB1A以及RRTF1等转录因子可以作为耐碱品种培育的关键候选基因进行深入研究。
4 结 论本研究通过前期建立的pH表型观察系统,结合高通RNA-seq技术将对照组(pH 6.0)和高pH处理组(pH 8.0)的拟南芥根部进行了转录组测序。分析了高pH胁迫下拟南芥根的差异表达基因,GO功能注释表明转录、离子运输、细胞组织与发生、蛋白质代谢、发育过程、非生物胁迫应答以及信号传递等生物学过程相关基因受到高pH胁迫的影响。关键基因分析表明,拟南芥通过改变离子运输、Ca2+信号和植物激素信号等相关基因的表达参与高pH胁迫应答。转录调控网络预测证明AP2/ERBEP、MYB、WRKY以及NAC家族转录因子可能在高pH胁迫应答的转录变化中起着核心作用。该研究为进一步探讨拟南芥高pH胁迫应答与耐碱品种的培育奠定了理论基础。
| [1] |
Singh A. Soil salinization and waterlogging:A threat to environment and agricultural sustainability . Ecological Indicators, 2015, 57: 128-130. DOI:10.1016/j.ecolind.2015.04.027
(  0) 0) |
| [2] |
孟昭甫, 俞仁培, 王遵亲. 碱性低矿化地下水灌溉对土壤碱化的影响. 土壤学报, 1984, 21(1): 79-86. Meng Z F, Yu R P, Wang Z Q. The effect of alkaline ground water with low salinity on soil alkalization (In Chinese). Acta Pedologica Sinica, 1984, 21(1): 79-86. (  0) 0) |
| [3] |
Guo R, Shi L X, Ding X M, et al. Effects of saline and alkaline stress on germination, seedling growth, and ion balance in wheat . Agronomy Journal, 2010, 102(4): 1252-1260. DOI:10.2134/agronj2010.0022
(  0) 0) |
| [4] |
曹彦强, 闫小娟, 罗红燕, 等. 不同酸碱性紫色土的硝化活性及微生物群落组成. 土壤学报, 2018, 55(1): 194-202. Cao Y Q, Yan X J, Luo H Y, et al. Nitrification activity and microbial community structure in purple soils with different pH (In Chinese). Acta Pedologica Sinica, 2018, 55(1): 194-202. (  0) 0) |
| [5] |
Wang H, Wu Z, Chen Y, et al. Effects of salt and alkali stresses on growth and ion balance in rice(Oryza sativa L.) . Plant, Soil and Environment, 2011, 57(6): 286-294. DOI:10.17221/36/2011-PSE
(  0) 0) |
| [6] |
Zhang P P, Fu J M, Hu L X. Effects of alkali stress on growth, free amino acids and carbohydrates metabolism in Kentucky bluegrass(Poa pratensis) . Ecotoxicology, 2012, 21(7): 1911-1918. DOI:10.1007/s10646-012-0924-1
(  0) 0) |
| [7] |
颜路明, 郭祥泉. 盐碱胁迫对香樟幼苗离子吸收与分配的影响. 土壤, 2015, 47(6): 1176-1180. Yan L M, Guo X Q. Effects of saline-alkali stress on ion absorption and distribution of camphor seedling (In Chinese). Soils, 2015, 47(6): 1176-1180. (  0) 0) |
| [8] |
Sze H, Li X H, Palmgren M G. Energization of plant cell membranes by H+-pumping ATPases:Regulation and biosynthesis . The Plant Cell, 1999, 11(4): 677-689.
(  0) 0) |
| [9] |
Maathuis F J, Sanders D. Mechanism of high-affinity potassium uptake in roots of Arabidopsis thaliana . Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(20): 9272-9276. DOI:10.1073/pnas.91.20.9272
(  0) 0) |
| [10] |
Shi H Z, Lee B H, Wu S J, et al. Overexpression of a plasma membrane Na+/H+ antiporter gene improves salt tolerance in Arabidopsis thaliana . Nature Biotechnology, 2003, 21(1): 81-85. DOI:10.1038/nbt766
(  0) 0) |
| [11] |
Fuglsang A T, Guo Y, Cuin T A, et al. Arabidopsis protein kinase PKS5 inhibits the plasma membrane H+-ATPase by preventing interaction with 14-3-3 protein . The Plant Cell, 2007, 19(5): 1617-1634. DOI:10.1105/tpc.105.035626
(  0) 0) |
| [12] |
Fan X D, Wang J Q, Yang N, et al. Gene expression profiling of soybean leaves and roots under salt, saline-alkali and drought stress by high-throughput illumina sequencing . Gene, 2013, 512(2): 392-402. DOI:10.1016/j.gene.2012.09.100
(  0) 0) |
| [13] |
Li N, Liu H L, Sun J, et al. Transcriptome analysis of two contrasting rice cultivars during alkaline stress . Scientific Reports, 2018, 8(1): 9586. DOI:10.1038/s41598-018-27940-x
(  0) 0) |
| [14] |
Meng C, Quan T Y, Li Z Y, et al. Transcriptome profiling reveals the genetic basis of alkalinity tolerance in wheat . BMC Genomics, 2017, 18(1): 24-37. DOI:10.1186/s12864-016-3421-8
(  0) 0) |
| [15] |
Connolly M A, Clausen P A, Lazar J G. Preparation of RNA from plant tissue using trizol . Cold Spring Harbor Protocols, 2006(1): pdb.prot4105.
(  0) 0) |
| [16] |
Gujas B, Alonso-Blanco C, Hardtke C S. Natural Arabidopsis brx loss-of-function alleles confer root adaptation to acidic soil . Current Biology, 2012, 22(20): 1962-1968. DOI:10.1016/j.cub.2012.08.026
(  0) 0) |
| [17] |
Clapham D E. Calcium signaling . Cell, 1995, 80(2): 259-268. DOI:10.1016/0092-8674(95)90408-5
(  0) 0) |
| [18] |
Behera S, Xu Z L, Luoni L, et al. Cellular Ca2+ signals generate defined pH signatures in plants . The Plant Cell, 2018, 30(11): 2704-2719. DOI:10.1105/tpc.18.00655
(  0) 0) |
| [19] |
Yang C W, Wang P, Li C Y, et al. Comparison of effects of salt and alkali stresses on the growth and photosynthesis of wheat . Photosynthetica, 2008, 46(1): 107-114.
(  0) 0) |
| [20] |
Yang C W, Shi D C, Wang D L. Comparative effects of salt and alkali stresses on growth, osmotic adjustment and ionic balance of an alkali-resistant halophyte Suaeda glauca(Bge.) . Plant Growth Regulation, 2008, 56(2): 179-190. DOI:10.1007/s10725-008-9299-y
(  0) 0) |
| [21] |
Wang L Q, Wang C, Wang D Y, et al. Molecular characterization and transcript profiling of NAC genes in response to abiotic stress in Tamarix hispida . Tree Genetics & Genomes, 2014, 10(1): 157-171.
(  0) 0) |
| [22] |
Yang A, Dai X Y, Zhang W H. A R2R3-type MYB gene, OsMYB2, is involved in salt, cold, and dehydration tolerance in rice . Journal of Experimental Botany, 2012, 63(7): 2541-2556. DOI:10.1093/jxb/err431
(  0) 0) |
| [23] |
Tenhaken R. Cell wall remodeling under abiotic stress . Frontiers in Plant Science, 2015, 5: Article 771.
(  0) 0) |
| [24] |
Zhu J H, Lee B H, Dellinger M, et al. A cellulose synthase-like protein is required for osmotic stress tolerance in Arabidopsis . The Plant Journal, 2010, 63(1): 128-140.
(  0) 0) |
| [25] |
Zhu J K. Abiotic stress signaling and responses in plants . Cell, 2016, 167(2): 313-324. DOI:10.1016/j.cell.2016.08.029
(  0) 0) |
| [26] |
Yang Y Q, Wu Y J, Ma L, et al. The Ca2+ sensor SCaBP3/CBL7 fine tunes Arabidopsis alkali tolerance and modulats plasma membrane H+-ATPase activity . The Plant Cell, 2019, 31(6): 1367-1384. DOI:10.1105/tpc.18.00568
(  0) 0) |
| [27] |
Chen C, Sun X L, Duanmu H Z, et al. GsCML27, a gene encoding a calcium-binding EF-hand protein from Glycine soja, plays differential roles in plant responses to bicarbonate, salt and osmotic stresses . PLoS One, 2015, 10(11): e0141888. DOI:10.1371/journal.pone.0141888
(  0) 0) |
| [28] |
An Y M, Song L L, Liu Y R, et al. De novo transcriptional analysis of alfalfa in response to saline-alkaline stress . Frontiers in Plant Science, 2016, 7: Article 931.
(  0) 0) |
| [29] |
Lee C M, Thomashow M F. Photoperiodic regulation of the C-repeat binding factor(CBF)cold acclimation pathway and freezing tolerance in Arabidopsis thaliana . Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(37): 15054-15059. DOI:10.1073/pnas.1211295109
(  0) 0) |
| [30] |
Liu Q, Kasuga M, Sakuma Y, et al. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought- and low-temperature-responsive gene expression, respectively, in Arabidopsis . The Plant Cell, 1998, 10(8): 1391-1406. DOI:10.1105/tpc.10.8.1391
(  0) 0) |
| [31] |
Ding Z J, Yan J Y, Li C X, et al. Transcription factor WRKY46 modulates the development of Arabidopsis lateral roots in osmotic/salt stress conditions via regulation of ABA signaling and auxin homeostasis . The Plant Journal, 2015, 84(1): 56-69. DOI:10.1111/tpj.12958
(  0) 0) |
| [32] |
Hu Y R, Chen L G, Wang H P, et al. Arabidopsis transcription factor WRKY8 functions antagonistically with its interacting partner VQ9 to modulate salinity stress tolerance . The Plant Journal, 2013, 74(5): 730-745. DOI:10.1111/tpj.12159
(  0) 0) |
| [33] |
Xie Y J, Mao Y, Lai D W, et al. H2 enhances Arabidopsis salt tolerance by manipulating ZAT10/12-mediated antioxidant defence and controlling sodium exclusion . PLoS One, 2012, 7(11): e49800. DOI:10.1371/journal.pone.0049800
(  0) 0) |
| [34] |
Matsuo M, Johnson J M, Hieno A, et al. High REDOX RESPONSIVE TRANSCRIPTION FACTOR1 levels result in accumulation of reactive oxygen species in Arabidopsis thaliana shoots and roots . Molecular Plant, 2015, 8(8): 1253-1273. DOI:10.1016/j.molp.2015.03.011
(  0) 0) |
 2020, Vol. 57
2020, Vol. 57


