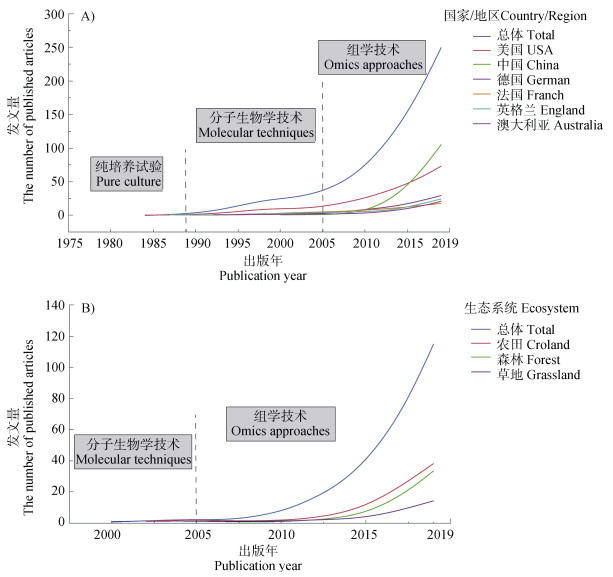
土壤微生物驱动着陆地生态系统中几乎所有已知的生态学过程,是联系大气圈、水圈、岩石圈及生物圈物质循环与能量流动的重要纽带,维系着生态系统的可持续发展[1]。土壤微生物生物地理学旨在研究土壤微生物多样性、群落组成和功能属性在时空尺度下的分布格局,其研究有助于更好地理解微生物多样性的产生和维持机制,认识微生物在调节关键生态系统过程中的重要作用。1934年,荷兰微生物学家Baas-Becking首次提出了微生物生物地理学假说:“Everything is everywhere,but,the environment selects”[2]。然而,由于绝大多数微生物不可培养,土壤微生物生物地理学研究的发展十分缓慢。20世纪90年代以前,土壤微生物生物地理学研究主要基于纯培养技术(图 1A),该方法可能大大低估了土壤微生物多样性。1990年以后,PCR-DGGE等DNA指纹图谱技术的进步极大推动了土壤微生物生物地理学研究,然而,DNA指纹图谱技术只能检测微生物中的优势类群,对稀有类群的检测略显乏力,同样无法准确评估土壤中微生物的多样性。进入21世纪以来,随着高通量测序技术的突破和生物信息学的发展,土壤微生物生物地理学研究得到了空前发展,其已成为土壤微生物学和生态学等领域的研究热点[3-4](图 1A)。土壤微生物生物地理学研究已从土壤微生物的空间分布及其驱动机制,拓展到微生物的群落构建过程和共存网络,以及全球变化背景下微生物分布的模型预测等方面。此外,土壤微生物分布与生态系统功能(植物生产力、土壤养分循环和生态系统多功能性等)的关联也得到了越来越多的关注。值得注意的是,尽管美国在土壤微生物生物地理学研究领域一直处于领先地位,近年来,在中国科学院战略性先导科技专项(B类)“土壤-微生物系统功能及其调控”等重大项目的推动下,我国土壤微生物生物地理学研究呈现快速发展态势[5]。2016年,我国在土壤微生物地理学研究方向发文量首次超越了美国(图 1A),研究主要集中在农田、森林和草地生态系统中,分别占发文总量的32.39%、20.56%和12.39%(图 1B)。本文将重点介绍我国土壤微生物生物地理学的重大研究进展以及国际研究前沿,并展望未来土壤微生物生物地理学的发展趋势及挑战。
|
A)不同国家/地区文章发表数量随时间的变化趋势the number of published articles across time in different countries or regions;B)我国不同生态系统类型文章发表数量随时间的变化趋势the number of published articles across time in different ecosystems in China. 检索方式(所有数据来源于Web of Science Core Collection),检索文章时间跨度:1900-2019;检索论文库:SCI-EXPANDED,CPCI-S,CCR-EXPANDED,IC。 A:国家/地区主题:((soil* and(“microorganism*” or “microbe*” or “bacter*” or “archae*” or “fung*”)and(“spatial distribution*” or “biogeograph*”or “geographic distance”))) B:全部生态系统主题:((soil* and(“microorganism*” or “microbe*” or “bacter*” or “archae*” or “fung*”)and(“spatial distribution*” or “biogeograph*” or “geographic distance”))) 农田生态系统主题:((soil* and(“microorganism*” or “microbe*” or “bacter*” or “archae*” or “fung*”)and(“spatial distribution*” or “biogeograph*” or “geographic distance”)and(“cropland*” or “farm*” or “field*” or “wheat*” or “paddy*” or “maize*” or “rice*” or “agricultur*”))) 森林生态系统主题:((soil* and(“microorganism*” or “microbe*” or “bacter*” or “archae*” or “fung*”)and(“spatial distribution*” or “biogeograph*” or “geographic distance”)and(“forest*” or “wood*”))) 草地生态系统主题:((soil* and(“microorganism*” or “microbe*” or “bacter*” or “archae*” or “sfung*”)and(“spatial distribution*” or “biogeograph*” or “geographic distance”)and(“grassland*” or “prairie*” or “steppe*”))) 图 1 土壤微生物生物地理学发文量 Fig. 1 The number of published articles in soil microbial biogeography |
在森林生态系统中,对于土壤微生物群落水平空间分布的研究主要集中在我国东部森林。例如,基于从南到北典型森林的115份土壤,Xia等[6]研究了土壤细菌多样性和群落组成的空间分布,发现土壤细菌多样性沿纬度梯度呈单峰模式,峰值出现在pH近中性、高碳氮含量的暖温带土壤中,环境因子(21.1%)较地理扩散限制(15.9%)对土壤细菌群落空间分异有更高的解释率。此外,通过采集自然森林四种植被样带的110份土壤,Ma等[7]解析了土壤细菌、真菌和古菌的分布模式,发现细菌、真菌和古菌的空间分布与土壤理化性质有关,地理扩散限制和环境因素分别解释细菌和真菌群落的距离衰减模式,而古菌群落在空间上则表现出同质性,该研究揭示了自然森林土壤中不同微生物类群具有不同的空间分布模式。在此基础上,Ma等[8]整合分析了细菌、真菌和古菌的共存网络,比较了不同气候类型下土壤微生物共存网络拓扑性质的空间分布格局,结果表明北部森林土壤微生物群落相较于南部森林有更高的度中心性(Degree centrality)和更低的中介中心性(Betweenness centrality),古菌在共存网络中表现出随机互作模式,中性过程可能起到重要作用,而细菌和真菌则呈现出非随机互作模式,表明确定性过程可能更占优势,此研究为土壤微生物群落的空间分布提供了新思路。Ma等[9]进一步采用宏基因组测序,发现功能基因网络中有7 191个基因呈正向连接,123个基因呈反向连接,总的网络由27个簇组成,这些簇富含特定功能的基因集,此研究揭示了功能基因网络的层级结构,并在宏基因组水平提出了预测基因和未知功能域的新方法。此外,Yang等[10]采集了长白山、东灵山、神农架、天目山和古田山共58种木本植物的根际土,发现植物系统发育、环境因素(包括土壤属性和气候因子)和空间距离分别解释了11.4%、24.1%和7.2%的土壤真菌群落的变化,且活体营养型的外生菌根真菌和植物病原菌群落受木本植物系统发育的影响显著大于自由生活的土壤腐生真菌群落,该研究首次在较大空间尺度下揭示了植物系统发育特性对土壤真菌群落空间分布的影响,为认识森林土壤真菌群落与木本植物间的进化规律提供全新视角。然而,目前对于森林生态系统土壤微生物群落空间分布的研究主要集中在东部季风区,针对西南和西北等地林区的土壤微生物群落空间分布的研究相对匮乏(参见[11])。
对于森林土壤微生物群落垂直空间分布的研究主要在我国典型自然山脉展开。例如,Shen等[12]发现土壤细菌群落沿长白山530~2 200 m海拔存在显著分异,细菌群落组成和多样性与土壤pH显著相关,该研究首次揭示了土壤pH是驱动森林土壤细菌群落垂直分布的关键因子。随后,Shen等[13]进一步研究了与土壤碳氮循环相关的疣微菌门的垂直分布,发现其垂直分布主要受土壤pH和C:N影响,揭示了优势属DA101在土壤中可能占有不同的生态位并执行寡营养生活史参与元素循环的策略。此外,Shen等[14]将土壤真核微生物群落的垂直分布与相同海拔梯度下的土壤细菌及植物群落的分布特征进行比较,发现植物多样性随海拔升高而降低,而真核微生物群落多样性与海拔无显著相关性,该研究率先探究了土壤真核微生物群落的海拔分布规律,并将微生物与宏观生物(植物)的海拔分布进行耦联,验证了生物个体大小影响海拔分布模式的假说。Shen等[15]采用GeoChip技术研究了长白山森林土壤功能基因的垂直分布,发现功能基因在林线交错带具有更高的多样性,结果可为预测气候变化背景下森林土壤微生物功能的变化提供重要依据。坐落在青藏高原边界的贡嘎山同样是研究微生物垂直分布的“热土”。Li等[16]沿贡嘎山1 800~4 100 m海拔梯度研究了该地区森林土壤细菌群落的垂直分布,研究结果表明土壤细菌多样性随海拔升高而下降,环境选择作用在低海拔(1 800~2 600 m)的细菌群落构建过程中占主导,而地理扩散限制在高海拔(2 800~4 100 m)的细菌群落构建过程中有明显优势。Wang等[17]基于同一土壤样品进行固氮微生物的垂直分布研究,发现固氮菌多样性在1 800~2 800 m无显著变化,而在2 800~4 100 m随海拔升高而降低,确定性过程在低和高海拔地区占主导,而随机性过程在中海拔地区占主导。Tian等[18]进一步研究了1 600~3 900 m海拔的土壤真菌群落垂直分布,发现真菌多样性随海拔升高而降低,海拔和温度是真菌多样性的主要驱动因子。以上研究表明不同微生物类群具有不同的海拔分布模式及驱动机制。目前,森林土壤微生物垂直分布研究仍停留在多样性海拔分布模式上,共存网络和群落构建分析可能是未来深入解析土壤微生物群落垂直分布及其驱动机制的有力工具之一。
总而言之,尽管森林土壤微生物群落沿水平和垂直空间的分布规律已取得部分成果,如何整合研究成果,多维度分析土壤微生物群落的分布格局将是未来研究工作的重点和难点。此外,植物多样性、生产力与土壤微生物群落空间分布的潜在作用机制仍需进一步探究,这将有助于深入理解土壤微生物群落的空间分布规律,揭示土壤微生物群落与生态系统功能的耦联机制,并预测生态系统对气候变化的响应和反馈。
1.2 草地生态系统在北方温带草地生态系统中,通过对新疆和内蒙古地区横跨3 700 km的草原样带调查,Wang等[19]研究了气候、土壤和地理距离等对土壤细菌群落的影响,发现干旱程度是决定北方草地土壤细菌多样性、群落组成和关键类群丰度的重要因素,地理距离能够解释36.02%的细菌群落变异,高于环境因子24.06%的解释量,该研究揭示了地理距离和环境因素在驱动北方草地土壤微生物群落空间分布上的重要性。Wang等[20]进一步发现土壤细菌群落的空间分布格局及地理距离和环境因素对其空间分布的相对重要性均具有明显的生境特异性,且这种生境特异性即使在相似的空间尺度下依然存在。随后,Wang等[21]对该地区土壤微生物共存网络的空间分布格局进行了研究,发现在大区域尺度下微生物共存网络主要受年均降雨量影响,而在小生境尺度下主要受当地土壤和植物因素影响。此外,Zhang等[22]从群落构建角度对新疆典型荒漠草地土壤细菌群落空间分布进行了研究,发现细菌群落在低盐度土壤中具有系统发育聚集的倾向,确定性过程在细菌群落构建中占主导,该研究首次揭示了土壤盐度是影响荒漠草地土壤细菌群落空间分布的关键因子。在北方七省的干旱半干旱气候类型的温带草地中,Chen等[23]发现植物群落组成能够解释土壤真菌群落9.0%的变异,结构方程模型揭示了植物物种丰富度对土壤真菌多样性的重要影响。在内蒙古草甸草原、典型草原和荒漠草原中,Wang等[24]研究了不同类型草地土壤真菌群落的空间分异,发现在干旱时土壤真菌群落将会转向由子囊菌门主导的群落组成,该研究进一步验证了北方草地土壤微生物空间分布受降水量影响。在全球气候变化和人为过度开垦、放牧的背景下,我国北方草地生态系统出现明显“三化”现象,加强草地土壤微生物群落空间分布的研究将有助于深入理解草地生态系统演替和恢复的微生物学机制,对草地生态重建和资源保护有深远意义。
在青藏高原高寒草地,通过对青藏高原东北部和中部样带进行土壤采集和植被调查,Yang等[25]发现土壤真菌α和β多样性分别与地上植物α和β多样性呈现出极强的耦合关系,揭示了植物多样性对土壤真菌多样性的驱动作用。Shi等[26]在青藏高原东部样带的研究发现,草地土壤古菌群落的空间分布主要受含水量和C:N影响,且地理距离对古菌群落空间变异的解释率低于环境因子的解释率。此外,Shi等[27]基于共存网络,发现古菌在共存网络中起到稳定网络鲁棒性的关键作用,揭示了古菌在高寒草地生态系统微生物共存网络中可能扮演着重要角色。基于青藏高原西北部草地样带,Chu等[28]同时研究了土壤细菌群落在水平空间和表层/次表层的分布规律,结果表明不同土层的土壤细菌群落差异极大,可与相间若干公里的细菌群落差异相当,土壤总碳含量和C:N共同驱动表层和次表层土壤细菌群落的空间分布。类似地,Yang等[29]发现表层土壤真菌群落空间分异不受土壤性质影响,反之,土壤、气候和地理距离可以解释次表层土壤真菌群落27%的变异。在先前研究基础上,Ladau等[30]利用物种-分布模型预测了气候变化背景下青藏高原土壤原核微生物多样性的变化,发现过去的气候(50年前)能更好地预测现在的土壤微生物多样性,进一步利用现在的气候预测未来的微生物多样性,发现青藏高原大多数地区在未来几十年后土壤微生物多样性将增加。除真菌、古菌和细菌外,基于青藏高原东北部草地样带,Yang等[31]对微生物功能基因多样性沿海拔梯度的空间分异也进行了研究,发现微生物功能基因多样性在不同海拔差异明显,土壤pH、NH4+、温度和植被多样性能够解释微生物功能基因81.4%的空间变异。此外,Deng等[32]对甲烷氧化微生物的空间分布也进行了研究,发现高亲和力的大气甲烷氧化菌是青藏高原草地土壤中主要甲烷氧化菌类群。Che等[33]对高原草地固氮微生物空间分布进行了研究,发现自养共生固氮菌是优势类群。未来研究应充分利用青藏高原气候敏感区等特点,从土壤微生物群落角度阐述生态功能及其对气候变化的响应和反馈。
微生物在草地生态系统土壤中扮演着重要角色,对维持和修复草地生态系统具有深远意义。目前的研究主要在小尺度范围内进行,未来仍需更全面、广泛地研究我国草地土壤微生物群落的空间分布格局(如南方人工草地土壤微生物群落分布)。此外,微生物是土壤碳、氮、磷、硫等元素周转的重要参与者,土壤微生物群落与宏观生态效应(如碳降解速率、固氮潜势等)的研究相结合,也是未来草地生态系统宏观生态学和微生物生态学研究的热点。
1.3 农田生态系统农田生态系统根据水分管理措施可分为旱地(偶尔灌溉)和水田(持续淹水)两种。我国旱地土壤微生物群落空间分布的研究主要集中在华北平原小麦地、东部大豆地和东北地区农田。中国科学院南京土壤研究所褚海燕课题组研究了我国华北平原小麦地土壤微生物群落的空间分布。例如,Shi等[34]通过采集华北平原小麦地243份土壤,发现土壤细菌群落23.9%的空间变异能够被环境变量(解释率19.7%)和空间变量(解释率4.1%)所解释,其中环境变量中起主要作用的为土壤pH。利用已知的华北平原小麦地土壤中细菌多样性与环境变量间的相关性,Shi等[35]进一步绘制了土壤细菌多样性的空间分布图谱,发现华北平原中部土壤细菌多样性最高,且多样性的空间变异主要由放线菌和α-变形菌引起。通过采集华北平原小麦根际与非根际各45份土壤,褚海燕课题组进一步研究了小麦根际土壤微生物群落的空间分布及其驱动机制,发现由于根际的筛选作用,离根距离越近,土壤细菌多样性逐渐降低,而由于菌根际效应,真菌在根际中仍然保持较高的多样性;当代环境因素对垄间土壤微生物群落分布起主要作用,而地理空间距离对根际微生物群落分布有更大的贡献[36-37]。此外,Fan等[38]通过共存网络分析研究了大尺度下华北平原小麦根际土壤古菌、细菌和真菌之间的关系,发现与非根际土壤相比,根际微生物网络结构相对简单却更加稳定,土壤pH与微生物多样性对网络的大小(size)和连通性(connectivity)均有正反馈作用;网络中的核心菌群具有相对灵活的代谢特征,并且根际中核心菌群受环境因子的扰动作用较小;对于土壤固氮菌来说,根际固氮菌群落结构显著区别于非根际,且根际土壤中固氮菌网络结构相对简单却更加稳定;确定性过程主导了麦田土壤固氮菌的群落构建,而在根际中,随机性过程的贡献率增加;在中性pH(6.5~7.5)下,固氮菌群落构建由随机性过程占主导,而在酸性pH(4.5~6.5)或碱性pH(7.5~8.5)下,确定性过程占主导[39]。以上研究阐明了我国华北平原麦田微生物群落的分布规律、驱动机制和群落构建过程,揭示了根际微生物共存网络的高效性(high transitivity),对深入挖掘农田土壤根际微生物资源、发挥其潜在功能提供了重要的理论依据。此外,西北农林科技大学韦革宏课题组进行了我国东部大豆根际和非根际土壤微生物生物地理学研究,发现细菌和真菌的多样性在根际和非根际土壤中差异显著,且离根系距离接近,细菌和真菌的α多样性降低;影响根际和非根际土壤微生物群落空间分布的驱动机制不同,细菌的多样性和群落周转更易受环境因素的影响,而真菌群落更易受地理距离的影响[40-41]。Zhang等[42]进一步分析了大豆根际和非根际土壤中细菌-真菌的共存网络及其在南方和北方土壤中的差异,研究结果表明细菌-真菌网络结构和组成在根际和非根际土壤中差异显著,与非根际土壤相比,根际土壤细菌-真菌间连接较少,模块性更低;南方根际和非根际土壤微生物网络细菌-真菌间的连接更多,此外,细菌在根际和非根际土壤中的子网络主要受土壤pH调节,而真菌在根际和非根际的子网络主要受土壤锰含量调节。此外,中国科学院东北地理与农业生态研究所王光华课题组采集了沿纬度分布的东北黑土带内26份土壤,研究了土壤细菌、酸杆菌、真菌、古菌和功能菌群(氨氧化古菌(AOA)和氨氧化细菌(AOB))的空间分布,发现土壤细菌的多样性随纬度增加而降低,其空间分布主要受土壤pH驱动[43];与土壤细菌类似,土壤酸杆菌的多样性和群落组成主要受土壤pH影响(解释率25%),而空间距离对酸杆菌群落空间变异的解释率仅5%[44];土壤真菌多样性也表现出与细菌类似的在较低纬度地区较高的趋势,但土壤真菌群落的空间差异主要由土壤碳含量驱动,环境变量解释了土壤真菌群落空间变异的35%,地理距离解释了土壤真菌群落空间变异的20%[45];对于古菌群落,其群落的空间变异主要受土壤pH和纬度影响,土壤性质能够解释古菌群落32%的空间变异,其中土壤pH的解释率占到了21%,空间地理距离能够解释古菌群落19%的空间变异[43]。Liu等[46]进一步研究了土壤中功能微生物AOA和AOB的空间分布规律,发现二者存在显著差异,AOA的丰度显著高于AOB,AOA的丰度与土壤碳含量显著正相关,而AOB的丰度与土壤pH显著正相关;AOA的α多样性与土壤性质无显著相关,而AOB的α多样性受土壤pH、全磷、全碳等的影响;AOA群落的空间变异主要受纬度影响,而AOB群落的空间变异主要受土壤pH影响。此外,Feng等[47]研究了我国东北地区单一作物类型(玉米)农田土壤细菌群落构建过程,发现东北玉米地土壤细菌群落的空间周转在大空间尺度上由异质性选择占主导。以上研究结果表明,旱地土壤微生物群落的空间分布、群落构建和共存网络受环境因素和地理距离等共同影响,二者相对作用大小会随着微生物类群、研究区域和作物类型的不同而有所差异。
对于水田土壤微生物群落的空间分布研究主要集中在我国东部从南到北纬度梯度上的水稻田、沿长江流域分布的水稻田以及南方多个长期水稻田。在南北纬度梯度上,Jiang等[48]发现不同耕作方式下土壤细菌和真菌的多样性和群落组成具有显著差异,细菌群落的空间分布更易受到环境异质性的影响,而真菌群落空间分布受地理距离影响更大,具有更大的空间周转率,二者的多样性和群落组成主要受土壤pH影响,此外,共存网络分析表明不同耕作方式下共有的Rhizobiales细菌和Hypocreals真菌起到了关键物种的作用。Jiao等[49]研究了我国东部相邻分布的玉米和水稻田土壤古菌群落的分布,发现水作和旱作对土壤古菌群落的空间分布具有显著影响,玉米地土壤古菌群落(主要为广古菌门)的空间分布受土壤pH驱动,而水稻田土壤古菌群落(主要为奇古菌门)的空间分布受年均温驱动,且玉米地土壤古菌多样性更高。Jiao等[50]进一步研究发现,当微生物群落构建过程受扩散限制主导时,不同物种间有更高的共存关系,阐明了物种选择和扩散限制的平衡介导了土壤微生物群落的物种共存。此外,Gao等[51]研究了长江流域水稻田土壤细菌和固氮菌群落的空间分布规律及驱动机制,发现细菌和固氮菌的空间分布具有尺度依赖性,即不同空间尺度下土壤微生物的空间周转率不同,且确定性因素在较小尺度下对微生物群落的空间分布影响较大。Zhao等[52]进一步研究了该地区土壤真菌群落的空间分布,发现真菌群落空间分布的驱动机制同样具有尺度依赖性,但与细菌和固氮菌不同,真菌群落的空间分布在较小空间尺度下主要由随机性过程主导。除了在不同空间尺度下研究水田土壤微生物群落的空间分布,在不同地点分布的长期水稻实验站上土壤微生物群落的空间分布也得到了研究。例如,Chen等[53]研究了我国南方6个长期施肥定位实验站土壤中细菌群落的空间分布规律,发现长期施肥会降低土壤细菌群落的空间周转率,使其在空间上更加匀质。以上研究表明,环境因素、地理距离等能够影响水田土壤微生物群落的空间分布,二者相对作用大小随着微生物类群和空间距离的不同而不同。
综上,尽管旱地和水田土壤微生物群落的空间分布规律有所不同,但二者均受环境因素和地理距离的影响,且环境因素和地理距离对土壤微生物群落空间分布规律的影响均随微生物类群的不同(主要为细菌和真菌)而不同。未来对于农田土壤微生物群落空间分布的研究应重点关注有重要生态功能的微生物(如丛枝菌根)的空间分布;还应关注我国其他地区(如西南水田和西北旱地)农田土壤微生物群落的空间分布,从而绘制我国农田土壤微生物群落的空间分布图谱;并研究土壤微生物群落的空间分布规律与产量、病害和土壤功能的关联,以便更好地将理论成果与实际生产相结合。
2 国际研究前沿 2.1 土壤微生物群落的空间分布及其驱动因素在国际上,土壤微生物群落在较大空间尺度下的分布受到广泛关注,尤其是微生物随纬度与海拔的分布规律。例如,Delgado-Baquerizo等[54]发现在南半球,土壤细菌多样性随纬度升高而降低,然而,北半球并无此规律[55-56]。此外,Bahram等[57]发现全球表层土壤真菌和细菌多样性沿纬度梯度呈现不同的分布模式:细菌,而非真菌,在温带地区的物种多样性最高。有研究发现土壤微生物的多样性随海拔升高而降低,这与植物多样性表现出相似的海拔分布模式[54, 58]。然而,大多数研究并未发现微生物多样性与海拔之间的负相关关系[12, 14, 59-62]。此外,不同微生物类群呈现出不同的海拔分布规律。例如,Singh等[61]发现日本富士山土壤细菌多样性在海拔梯度上呈现单峰模式,而古菌多样性则表现出双峰模式。然而,目前大多数研究集中在特定的山脉和地区(如北美或澳大利亚),并专注于单一类群(如细菌),因此我们对于微生物多样性沿纬度和海拔梯度的变化规律的认识仍十分有限。
已有研究表明,环境因素驱动了土壤微生物群落在全球尺度上的分布。Fierer和Jackson[55]发现土壤pH是驱动细菌多样性和群落组成的关键因子。Delgado-Baquerizo和Eldridge[63]进一步确定了植被类型和土壤碳含量作为全球范围内土壤细菌多样性的重要预测因子。Tedersoo等[64]首次进行了土壤真菌的全球分布研究,发现气候是驱动土壤真菌多样性全球分布的主要环境因子,其他被证实的细菌和真菌多样性的全球驱动因素包括古气候因素和生物因素[57, 65]。Bahram等[57]还发现土壤细菌和真菌多样性对年降雨量和土壤pH的响应差异导致了其在全球尺度上的生态位分化。此外,地上-地下的相互作用是大空间尺度下土壤微生物多样性的主要驱动因素。例如,地上植物分类和功能属性有助于解释微生物生物量、多样性和群落组成的分布[10, 25, 66-68]。然而,地下植物性状如何影响土壤微生物群落的分布鲜有研究。最近,Ramirez等[69]提出,植物物种扩张可能能够预测地下微生物群落的组成,这可能与植物的地下特征有关(如根)。此外,动物行为同样会影响土壤微生物群落。研究发现蚁群定殖和哺乳动物觅食坑在很大程度上改变了澳大利亚东部的微生物多样性和群落组成[70-71]。除了当代环境因素(如气候、土壤、植物和动物)之外,历史因素(如古气候特征)和微生物本身特征(如体型、附着和定殖能力)对土壤微生物群落分布的影响也十分重要。
2.2 土壤微生物群落构建与共存网络微生物生物地理学研究的另一个核心问题是阐明影响微生物群落构建的潜在机制,这曾被认为是微生物生态学研究的重要挑战之一[72]。微生物群落构建通常被划分为确定性过程(deterministic processes)和随机性过程(stochastic processes)[73],它们又可进一步划分为五个类别,即异质性选择(variable selection)、匀质性选择(homogeneous selection)、扩散限制(dispersal limitation)、匀质性扩散(homogenizing dispersal)和非主导(undominated)[74-75]。目前关于大尺度下土壤微生物群落构建机制的研究主要集中于细菌。例如,Feng等[47]发现随着空间尺度的增大,影响我国东北玉米田土壤细菌群落构建过程由最初的匀质性扩散转变为异质性选择,这一现象可用于解释土壤微生物群落空间分布中普遍存在的距离衰减模式。Tripathi等[76]基于荟萃分析(meta-analysis),研究了土壤细菌群落构建过程,发现确定性过程在极端酸性或碱性pH条件下占主导,而随机过程在中性pH条件中占主导。此外,Feng等[77]发现环境条件的初始状态和变化程度共同决定了区域范围内不同生态过程对群落构建的相对贡献。目前,我们对于微生物群落构建过程的了解仍然有限,进一步研究不同空间和时间尺度下不同微生物类群的群落构建过程十分必要。
微生物物种在微生物食物网中相互依存[78],包括拮抗、竞争、共生和捕食-被捕食等关系[79]。这些多营养级微生物食物网是能量流动[80-81]和土壤养分循环(如碳、氮、磷、硫等元素循环)的主要驱动者,且微生物食物网内相互作用类型的丢失可能导致生态系统功能损失(如土壤多功能性的下降)[82],这或将发生在物种多样性丢失之前[79-83]。然而,我们仍然缺乏对于食物网内多个微生物类群共存或潜在相互作用模式的驱动机制的研究,以及它们与生态系统功能间的关联。微生物生态学中相关性网络的应用[84]提高了我们对于不同环境梯度下[82]微生物群落构建驱动机制的理解(如土壤碳、pH和植被类型)[85-87],量化了微生物间潜在的相互作用,并确定高度相关的微生物类群和关键物种[88-89]。微生物共存网络研究是一个相对较新的领域,应利用多年应用于动植物群落研究的经验来继续发展这方面研究[89-91]。此外,纯培养方法、实验室芯片技术(lab-on-chip)、宏基因组学、单细胞测序(single-cell sequencing)、iChip和培养组学(culturomics)等技术与微宇宙实验联用,将进一步促进我们在小尺度下研究土壤食物网中存在的生物互作关系[92]。
2.3 土壤微生物生物地理学与生态系统功能的关联土壤微生物在生态功能和生态系统服务中发挥着积极的调节作用[93-94]。土壤微生物生物地理学研究的核心目标之一是将微生物群落分布与其生态功能联系起来,包括单一功能(如养分循环、植物生产力和公共卫生等)[95-98]和多种功能(如生态系统多功能性)[94, 99]。例如,运用宏基因组测序,Fierer等[100]对不同生态系统土壤微生物的多种功能进行了研究,发现土壤多功能性与土壤细菌分类学和系统发育多样性密切相关。同样,细菌和真菌的丰富度驱动陆地生态系统在区域尺度[101-102]和全球尺度[98]上的多功能性(如养分循环、有机质分解和植物生产力等)。此外,Delgado-Baquerizo等[103]发现全球干旱地区土壤微生物群落组成能够调节生态系统多功能性对全球变化的抵抗力。Jing等[101]发现青藏高原高寒草地土壤微生物多样性与生态系统多功能性之间显著相关,且这种关系受到气候等因素的调节。这些研究表明土壤微生物群落与生态系统功能之间存在着密切关系。
目前,土壤微生物群落与各种生态功能之间的联系主要基于相关性分析,缺乏微生物类群与功能之间联系的直接证据[102]。未来研究应侧重于土壤微生物类群的分离和培养,以达到通过实验方法和全基因组测序等手段将特定功能与特定微生物类群相联系的目的。这对于解释当下土壤微生物的分类也至关重要,因为大多数的微生物仍未得到注释。未来应在全球范围内支持研究者们致力于培养和分离微生物类群,这是推动微生物生态学领域向前发展的一项基础工作。
2.4 全球变化背景下土壤微生物群落的预测土壤微生物生物地理学研究的另一个主要挑战是对微生物群落进行预测。土壤微生物群落受其周围环境的强烈影响,因此,可基于土壤微生物群落的分布规律和相关的环境因素来预测微生物群落在空间和时间上的变化。生物气候模型被广泛用于预测微生物群落的分布,包括包络模型(envelop models)、物种-面积分布模型、生态位模型和人工神经网络等[104]。例如,Fierer等[105]研究美国农业社会前的草原土壤,利用物种-分布模型,生成了微生物多样性和功能基因的图谱。同样,地质统计学方法被用来预测法国20个最具代表性的微生物门类的空间异质性和结构分布[106]。目前,已经出现的全球土壤微生物多样性地图谱,包括细菌[4, 107]、真菌[64]、优势真菌[108]、线虫[109]、菌根真菌和固氮生物[110]和未知的分类群[63]。越来越多的国家开始在构建自己的微生物分布图谱。例如,欧盟的第一张细菌生物多样性图谱是根据现有的欧盟土壤pH数据绘制的[111];第一张法国土壤细菌丰富度的图谱是基于大约1 800份土壤的测序结果绘制的[112]。此外,通过利用当前细菌分布与历史气候之间的联系,Ladau等[30]预测了未来在区域和大陆尺度上土壤微生物群落的分布,并预测如果细菌群落与现有气候条件相平衡,青藏高原和北美北部的大部分地区(约75%)土壤细菌多样性将增加。这种区域高分辨率图谱的建立对于更大尺度上微生物图谱的稳健性和普适性的提高非常有必要,有助于全球变化背景下土壤生物多样性的保护和管理。今后,还需利用更详细的土壤、植物和气候等信息,获得更高分辨率的土壤微生物图谱,在更大空间尺度下对核心微生物组或关键物种进行预测。
3 展望尽管近年来土壤微生物生物地理学研究取得了一系列进展,包括一些全球性研究项目,如地球微生物组计划[113-114],但未来研究仍面临诸多挑战。本文提出了未来土壤微生物生物地理学研究领域有待回答的科学问题和主要挑战。土壤微生物生物地理学研究的发展仍然受到其研究手段和方法的制约。例如,不同研究中所采用的数据采集方法不同使得在区域和全球范围内的整合分析十分困难。此外,目前在较大空间和时间尺度上的数据集仍十分有限[115],这限制了我们在空间和时间上预测土壤微生物群落分布的能力。我国幅员辽阔,不同地区气候、植被和土壤类型差异极大,人为干扰程度不同,这为研究不同尺度下土壤微生物生物地理分布提供了理想平台。未来我国土壤微生物生物地理学研究应重点关注土壤微生物组在较大时空尺度下的动态变化,尤其是在一些特殊生境(如全球变化敏感区、农田生态系统和海陆交错带等),使其在农业可持续发展和全球气候变化等关键领域发挥重要作用。本文进一步提出对于推动土壤微生物生物地理学研究发展至关重要的方向,包括需要更清晰的微生物物种定义,提高未来全球变化背景下对土壤微生物预测的能力,研究土壤食物网中的复杂结构,以及通过往常被忽视的纯培养和分离方法来确定微生物功能特征。
3.1 微生物物种的明确定义微生物物种的定义对于生物地理学研究是必不可少的。高通量测序技术的发展使得基于谱系、基因组和表型一致性的微生物物种定义得到快速发展[116]。然而,关于“微生物物种”的概念尚未达成明确共识。基于PCR(种系型)的微生物物种的定义导致了特殊的物种种群,这限制了我们对于新物种及其生态偏好能力的识别。因此,未来基于非PCR方法,如高通量培养和鉴定技术可用于扩大参考数据库(如GenBank,GreenGene,Silva),并填补微生物分类的空缺[117]。最近,基因组分类数据库(GTDB)已被开发出来,其可基于序列距离提供更实用/客观的物种水平的定义[118],这是收集用于系统发育分析的序列从而获得物种及其他分类水平信息的开始。物种明确定义将会大大促进微生物生物地理学的发展,将更有利于微生物多样性的研究,为微生物资源开发提供科学依据。
3.2 微生物群落的时间分布特征尽管微生物群落具有较大的时间变异,但在较大的空间尺度上获得有关微生物群落时间动态(如季节,年份或更长的时间尺度)仍是难点之一[116]。基于时空置换方法(如土壤时间序列、生态系统的演替和干扰后恢复时间),已发现土壤微生物群落随时间发生可预测的变化[75, 119-122]。然而,DNA测序方法能同时检测到死亡和活体生物,这降低了对于土壤微生物时间动态研究的分辨率,并且我们缺乏多年的、全球多个位点的微生物群落相关信息的收集。因此,将样品中的遗迹DNA作为混杂因素进行考虑是使用DNA方法检测微生物群落时间动态的关键因素[123]。未来应致力于在全球尺度下监测土壤微生物群落的时间变化,包括利用现有的全球合作平台(如NEON,LUCA和NUTNET),并在一些重要生态系统(如极地和高原地区)建立长期监测站点。这些长期积累的样本和数据将帮助我们更好地理解全球变化背景下土壤微生物群落随时间和空间的变化。
3.3 多组学技术结合与合成生物学在微生物生物地理学研究中的应用宏基因组测序和单细胞测序技术等[124]已被用于研究微生物分布与生态功能之间的联系,此外,包括宏转录组学、宏蛋白质组学和宏代谢组学在内的其他组学方法也能够被应用于建立大尺度下微生物生物地理分布与生态功能间的联系。尽管我们仍然很少知道具体微生物物种发挥的具体功能,以及它们对生态系统功能的具体贡献,但合成生物学方法和工程微生物已被用于促进生态系统的恢复,提高作物产量和病虫害控制[125-126],以及应对全球环境变化[127-128],甚至应用于月球和其他行星的地形改造,使它们与地球更加相似[129-130]。它们的加入有助于在大的时空尺度下研究土壤微生物生物地理学,这也是未来研究的热点。
3.4 准确预测全球变化背景下的土壤微生物群落提高模型的预测精度对于微生物图谱的绘制至关重要。首先,必须尽可能多地收集土壤样本,更好地涵盖所有生境类型,以丰富数据集。其次,土壤微生物群落是随时间动态变化的,因此有必要了解不同时间尺度下的微生物群落特征。第三,不仅需要考虑微生物多样性和群落组成,还需要考虑其他微生物群落特征,如微生物间的相互作用,微生物与植物的互作关系,并将其整合到预测模型中。高精度的模型可进一步用于预测全球变化背景下土壤微生物群落的时空动态,这将有助于土壤微生物多样性的保护、资源开发及其生态功能的发挥。
| [1] |
朱永官, 沈仁芳, 贺纪正, 等. 中国土壤微生物组:进展与展望. 中国科学院院刊, 2017, 32(6): 554-565. Zhu Y G, Shen R F, He J Z, et al. China soil microbiome initiative:Progress and perspective (In Chinese). Bulletin of Chinese Academy of Sciences, 2017, 32(6): 554-565. (  0) 0) |
| [2] |
Baas-Becking L G M. Geobiologie of inleiding tot de milieukunde(in Dutch) . Hague, Netherlands: W P Van Stockum & Zoon N V, 1934.
(  0) 0) |
| [3] |
Fierer N. Embracing the unknown:Disentangling the complexities of the soil microbiome . Nature Reviews Microbiology, 2017, 15(10): 579-590. DOI:10.1038/nrmicro.2017.87
(  0) 0) |
| [4] |
Delgado-Baquerizo M, Oliverio A M, Brewer T E, et al. A global atlas of the dominant bacteria found in soil . Science, 2018, 359(6373): 320-325. DOI:10.1126/science.aap9516
(  0) 0) |
| [5] |
褚海燕, 王艳芬, 时玉, 等. 土壤微生物生物地理学研究现状与发展态势. 中国科学院院刊, 2017, 32(6): 585-592. Chu H Y, Wang Y F, Shi Y, et al. Current status and development trend of soil microbial biogeography (In Chinese). Bulletin of Chinese Academy of Sciences, 2017, 32(6): 585-592. (  0) 0) |
| [6] |
Xia Z W, Bai E, Wang Q K, et al. Biogeographic distribution patterns of bacteria in typical Chinese forest soils . Frontiers in Microbiology, 2016, 7: 1106. DOI:10.3389/fmicb.2016.01106
(  0) 0) |
| [7] |
Ma B, Dai Z M, Wang H Z, et al. Distinct biogeographic patterns for Archaea, bacteria, and fungi along the vegetation gradient at the continental scale in Eastern China . mSystems, 2017. DOI:10.1128/msystems.00174-16
(  0) 0) |
| [8] |
Ma B, Wang H Z, Dsouza M, et al. Geographic patterns of co-occurrence network topological features for soil microbiota at continental scale in Eastern China . The ISME Journal, 2016, 10(8): 1891-1901. DOI:10.1038/ismej.2015.261
(  0) 0) |
| [9] |
Ma B, Zhao K K, Lv X, et al. Genetic correlation network prediction of forest soil microbial functional organization . The ISME Journal, 2018, 12(10): 2492-2505. DOI:10.1038/s41396-018-0232-8
(  0) 0) |
| [10] |
Yang T, Tedersoo L, Soltis P S, et al. Phylogenetic imprint of woody plants on the soil mycobiome in natural mountain forests of Eastern China . The ISME Journal, 2019, 13(3): 686-697. DOI:10.1038/s41396-018-0303-x
(  0) 0) |
| [11] |
Shi L L, Mortimer P E, Ferry Slik J W, et al. Variation in forest soil fungal diversity along a latitudinal gradient . Fungal Diversity, 2014, 64(1): 305-315. DOI:10.1007/s13225-013-0270-5
(  0) 0) |
| [12] |
Shen C C, Xiong J B, Zhang H Y, et al. Soil pH drives the spatial distribution of bacterial communities along elevation on Changbai Mountain . Soil Biology & Biochemistry, 2013, 57: 204-211.
(  0) 0) |
| [13] |
Shen C C, Ge Y, Yang T, et al. Verrucomicrobial elevational distribution was strongly influenced by soil pH and carbon/nitrogen ratio . Journal of Soils and Sediments, 2017, 17(10): 2449-2456. DOI:10.1007/s11368-017-1680-x
(  0) 0) |
| [14] |
Shen C C, Liang W J, Shi Y, et al. Contrasting elevational diversity patterns between eukaryotic soil microbes and plants . Ecology, 2014, 95(11): 3190-3202. DOI:10.1890/14-0310.1
(  0) 0) |
| [15] |
Shen C C, Shi Y, Ni Y Y, et al. Dramatic increases of soil microbial functional gene diversity at the treeline ecotone of Changbai mountain . Frontiers in Microbiology, 2016, 7: 1184. DOI:10.3389/fmicb.2016.01184
(  0) 0) |
| [16] |
Li J B, Shen Z H, Li C N, et al. Stair-step pattern of soil bacterial diversity mainly driven by pH and vegetation types along the elevational gradients of Gongga Mountain, China . Frontiers in Microbiology, 2018, 9: 569. DOI:10.3389/fmicb.2018.00569
(  0) 0) |
| [17] |
Wang Y S, Li C N, Shen Z H, et al. Community assemblage of free-living diazotrophs along the elevational gradient of Mount Gongga . Soil Ecology Letters, 2019, 1(3/4): 136-146.
(  0) 0) |
| [18] |
Tian J Q, Wu B, Chen H, et al. Patterns and drivers of fungal diversity along an altitudinal gradient on Mount Gongga, China . Journal of Soils and Sediments, 2017, 17(12): 2856-2865. DOI:10.1007/s11368-017-1701-9
(  0) 0) |
| [19] |
Wang X B, van Nostrand J D, Deng Y, et al. Scale-dependent effects of climate and geographic distance on bacterial diversity patterns across Northern China's grasslands . FEMS Microbiology Ecology, 2015, fiv133. DOI:10.1093/femsec/fiv133
(  0) 0) |
| [20] |
Wang X B, Lü X T, Yao J, et al. Habitat-specific patterns and drivers of bacterial β-diversity in China's drylands . The ISME Journal, 2017, 11(6): 1345-1358. DOI:10.1038/ismej.2017.11
(  0) 0) |
| [21] |
Wang S, Wang X B, Han X G, et al. Higher precipitation strengthens the microbial interactions in semi-arid grassland soils . Global Ecology and Biogeography, 2018, 27(5): 570-580. DOI:10.1111/geb.12718
(  0) 0) |
| [22] |
Zhang K P, Shi Y, Cui X Q, et al. Salinity is a key determinant for soil microbial communities in a desert ecosystem . mSystems, 2019. DOI:10.1128/msystems.00225-18
(  0) 0) |
| [23] |
Chen Y L, Xu T L, Veresoglou S D, et al. Plant diversity represents the prevalent determinant of soil fungal community structure across temperate grasslands in Northern China . Soil Biology & Biochemistry, 2017, 110: 12-21.
(  0) 0) |
| [24] |
Wang D, Rui Y C, Ding K, et al. Precipitation drives the biogeographic distribution of soil fungal community in Inner Mongolian temperate grasslands . Journal of Soils and Sediments, 2018, 18(1): 222-228. DOI:10.1007/s11368-017-1727-z
(  0) 0) |
| [25] |
Yang T, Adams J M, Shi Y, et al. Soil fungal diversity in natural grasslands of the Tibetan Plateau:Associations with plant diversity and productivity . New Phytologist, 2017, 215(2): 756-765. DOI:10.1111/nph.14606
(  0) 0) |
| [26] |
Shi Y, Adams J M, Ni Y Y, et al. The biogeography of soil archaeal communities on the eastern Tibetan Plateau . Scientific Reports, 2016, 6: 38893. DOI:10.1038/srep38893
(  0) 0) |
| [27] |
Shi Y, Fan K K, Li Y T, et al. Archaea enhance the robustness of microbial Co-occurrence networks in Tibetan Plateau soils . Soil Science Society of America Journal, 2019, 83(4): 1093. DOI:10.2136/sssaj2018.11.0426
(  0) 0) |
| [28] |
Chu H Y, Sun H B, Tripathi B M, et al. Bacterial community dissimilarity between the surface and subsurface soils equals horizontal differences over several kilometers in the western Tibetan Plateau . Environmental Microbiology, 2016, 18(5): 1523-1533. DOI:10.1111/1462-2920.13236
(  0) 0) |
| [29] |
Yang T, Adams J M, Shi Y, et al. Fungal community assemblages in a high elevation desert environment:Absence of dispersal limitation and edaphic effects in surface soil . Soil Biology & Biochemistry, 2017, 115: 393-402.
(  0) 0) |
| [30] |
Ladau J, Shi Y, Jing X, et al. Existing climate change will lead to pronounced shifts in the diversity of soil prokaryotes . mSystems, 2018. DOI:10.1128/msystems.00167-18
(  0) 0) |
| [31] |
Yang Y F, Gao Y, Wang S P, et al. The microbial gene diversity along an elevation gradient of the Tibetan grassland . The ISME Journal, 2014, 8(2): 430-440. DOI:10.1038/ismej.2013.146
(  0) 0) |
| [32] |
Deng Y C, Che R X, Wang F, et al. Upland Soil Cluster Gamma dominates methanotrophic communities in upland grassland soils . Science of the Total Environment, 2019, 670: 826-836. DOI:10.1016/j.scitotenv.2019.03.299
(  0) 0) |
| [33] |
Che R X, Deng Y C, Wang F, et al. Autotrophic and symbiotic diazotrophs dominate nitrogen-fixing communities in Tibetan grassland soils . Science of the Total Environment, 2018, 639: 997-1006. DOI:10.1016/j.scitotenv.2018.05.238
(  0) 0) |
| [34] |
Shi Y, Li Y T, Xiang X J, et al. Spatial scale affects the relative role of stochasticity versus determinism in soil bacterial communities in wheat fields across the North China Plain . Microbiome, 2018, 6: 27. DOI:10.1186/s40168-018-0409-4
(  0) 0) |
| [35] |
Shi Y, Li Y T, Yuan M Q, et al. A biogeographic map of soil bacterial communities in wheats field of the North China Plain . Soil Ecology Letters, 2019, 1(1/2): 50-58.
(  0) 0) |
| [36] |
Fan K K, Cardona C, Li Y T, et al. Rhizosphere- associated bacterial network structure and spatial distribution differ significantly from bulk soil in wheat crop fields . Soil Biology & Biochemistry, 2017, 113: 275-284.
(  0) 0) |
| [37] |
Zhang K P, Adams J M, Shi Y, et al. Environment and geographic distance differ in relative importance for determining fungal community of rhizosphere and bulk soil . Environmental Microbiology, 2017, 19(9): 3649-3659. DOI:10.1111/1462-2920.13865
(  0) 0) |
| [38] |
Fan K K, Weisenhorn P, Gilbert J A, et al. Wheat rhizosphere harbors a less complex and more stable microbial co-occurrence pattern than bulk soil . Soil Biology & Biochemistry, 2018, 125: 251-260.
(  0) 0) |
| [39] |
Fan K K, Weisenhorn P, Gilbert J A, et al. Soil pH correlates with the co-occurrence and assemblage process of diazotrophic communities in rhizosphere and bulk soils of wheat fields . Soil Biology & Biochemistry, 2018, 121: 185-192.
(  0) 0) |
| [40] |
Zhang B G, Zhang J, Liu Y, et al. Biogeography and ecological processes affecting root-associated bacterial communities in soybean fields across China . Science of the Total Environment, 2018, 627: 20-27. DOI:10.1016/j.scitotenv.2018.01.230
(  0) 0) |
| [41] |
Zhang J, Zhang B G, Liu Y, et al. Distinct large-scale biogeographic patterns of fungal communities in bulk soil and soybean rhizosphere in China . Science of the Total Environment, 2018, 644: 791-800. DOI:10.1016/j.scitotenv.2018.07.016
(  0) 0) |
| [42] |
Zhang B G, Zhang J, Liu Y, et al. Co-occurrence patterns of soybean rhizosphere microbiome at a continental scale . Soil Biology & Biochemistry, 2018, 118: 178-186.
(  0) 0) |
| [43] |
Liu J J, Sui Y Y, Yu Z H, et al. High throughput sequencing analysis of biogeographical distribution of bacterial communities in the black soils of northeast China . Soil Biology & Biochemistry, 2014, 70: 113-122.
(  0) 0) |
| [44] |
Liu J J, Yu Z H, Yao Q, et al. Ammonia-oxidizing Archaea show more distinct biogeographic distribution patterns than ammonia-oxidizing bacteria across the black soil zone of northeast China . Frontiers in Microbiology, 2018, 9: 171. DOI:10.3389/fmicb.2018.00171
(  0) 0) |
| [45] |
Liu J J, Sui Y Y, Yu Z H, et al. Soil carbon content drives the biogeographical distribution of fungal communities in the black soil zone of northeast China . Soil Biology & Biochemistry, 2015, 83: 29-39.
(  0) 0) |
| [46] |
Liu J J, Yu Z H, Yao Q, et al. Biogeographic distribution patterns of the archaeal communities across the black soil zone of northeast China . Frontiers in Microbiology, 2019, 10: 23. DOI:10.3389/fmicb.2019.00023
(  0) 0) |
| [47] |
Feng M M, Tripathi B M, Shi Y, et al. Interpreting distance-decay pattern of soil bacteria via quantifying the assembly processes at multiple spatial scales . MicrobiologyOpen, 2019, 8(9): e00851. DOI:10.1002/mbo3.851
(  0) 0) |
| [48] |
Jiang Y J, Liang Y T, Li C M, et al. Crop rotations alter bacterial and fungal diversity in paddy soils across East Asia . Soil Biology & Biochemistry, 2016, 95: 250-261.
(  0) 0) |
| [49] |
Jiao S, Xu Y Q, Zhang J, et al. Environmental filtering drives distinct continental atlases of soil Archaea between dryland and wetland agricultural ecosystems . Microbiome, 2019, 7: 15. DOI:10.1186/s40168-019-0630-9
(  0) 0) |
| [50] |
Jiao S, Yang Y F, Xu Y Q, et al. Balance between community assembly processes mediates species coexistence in agricultural soil microbiomes across Eastern China . The ISME Journal, 2020, 14(1): 202-216. DOI:10.1038/s41396-019-0522-9
(  0) 0) |
| [51] |
Gao Q, Yang Y F, Feng J J, et al. The spatial scale dependence of diazotrophic and bacterial community assembly in paddy soil . Global Ecology and Biogeography, 2019, 28(8): 1093-1105.
(  0) 0) |
| [52] |
Zhao J S, Gao Q, Zhou J Z, et al. The scale dependence of fungal community distribution in paddy soil driven by stochastic and deterministic processes . Fungal Ecology, 2019, 42: 100856. DOI:10.1016/j.funeco.2019.07.010
(  0) 0) |
| [53] |
Chen R R, Zhong L H, Jing Z W, et al. Fertilization decreases compositional variation of paddy bacterial community across geographical gradient . Soil Biology & Biochemistry, 2017, 114: 181-188.
(  0) 0) |
| [54] |
Delgado-Baquerizo M, Maestre F T, Reich P B, et al. Carbon content and climate variability drive global soil bacterial diversity patterns . Ecological Monographs, 2016, 86(3): 373-390. DOI:10.1002/ecm.1216
(  0) 0) |
| [55] |
Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities . Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(3): 626-631. DOI:10.1073/pnas.0507535103
(  0) 0) |
| [56] |
Chu H Y, Fierer N, Lauber C L, et al. Soil bacterial diversity in the Arctic is not fundamentally different from that found in other biomes . Environmental Microbiology, 2010, 12(11): 2998-3006. DOI:10.1111/j.1462-2920.2010.02277.x
(  0) 0) |
| [57] |
Bahram M, Hildebrand F, Forslund S K, et al. Structure and function of the global topsoil microbiome . Nature, 2018, 560(7717): 233-237. DOI:10.1038/s41586-018-0386-6
(  0) 0) |
| [58] |
Nottingham A T, Fierer N, Turner B L, et al. Microbes follow Humboldt:Temperature drives plant and soil microbial diversity patterns from the Amazon to the Andes . Ecology, 2018, 99(11): 2455-2466. DOI:10.1002/ecy.2482
(  0) 0) |
| [59] |
Fierer N, McCain C M, Meir P, et al. Microbes do not follow the elevational diversity patterns of plants and animals . Ecology, 2011, 92(4): 797-804. DOI:10.1890/10-1170.1
(  0) 0) |
| [60] |
Singh D, Takahashi K, Adams J M. Elevational patterns in archaeal diversity on Mt. fuji . PLoS One, 2012, 7(9): e44494. DOI:10.1371/journal.pone.0044494
(  0) 0) |
| [61] |
Singh D, Takahashi K, Kim M, et al. A hump-backed trend in bacterial diversity with elevation on Mount Fuji, Japan . Microbial Ecology, 2012, 63(2): 429-437. DOI:10.1007/s00248-011-9900-1
(  0) 0) |
| [62] |
Singh D, Takahashi K, Park J, et al. Similarities and contrasts in the archaeal community of two Japanese mountains:Mt. Norikura compared to Mt. Fuji . Microbial Ecology, 2016, 71(2): 428-441. DOI:10.1007/s00248-015-0681-9
(  0) 0) |
| [63] |
Delgado-Baquerizo M. Obscure soil microbes and where to find them . The ISME Journal, 2019, 13(8): 2120-2124. DOI:10.1038/s41396-019-0405-0
(  0) 0) |
| [64] |
Tedersoo L, Bahram M, Polme S, et al. Global diversity and geography of soil fungi . Science, 2014, 346(6213): 1256688. DOI:10.1126/science.1256688
(  0) 0) |
| [65] |
Delgado-Baquerizo M, Bissett A, Eldridge D J, et al. Palaeoclimate explains a unique proportion of the global variation in soil bacterial communities . Nature Ecology & Evolution, 2017, 1(9): 1339-1347.
(  0) 0) |
| [66] |
de Vries F T, Manning P, Tallowin J R B, et al. Abiotic drivers and plant traits explain landscape-scale patterns in soil microbial communities . Ecology Letters, 2012, 15(11): 1230-1239. DOI:10.1111/j.1461-0248.2012.01844.x
(  0) 0) |
| [67] |
Prober S M, Leff J W, Bates S T, et al. Plant diversity predicts beta but not alpha diversity of soil microbes across grasslands worldwide . Ecology Letters, 2015, 18(1): 85-95. DOI:10.1111/ele.12381
(  0) 0) |
| [68] |
Delgado-Baquerizo M, Fry E L, Eldridge D J, et al. Plant attributes explain the distribution of soil microbial communities in two contrasting regions of the globe . New Phytologist, 2018, 219(2): 574-587. DOI:10.1111/nph.15161
(  0) 0) |
| [69] |
Ramirez K S, Snoek L B, Koorem K, et al. Range-expansion effects on the belowground plant microbiome . Nature Ecology & Evolution, 2019, 3(4): 604-611.
(  0) 0) |
| [70] |
Eldridge D J, Delgado-Baquerizo M, Woodhouse J N, et al. Mammalian engineers drive soil microbial communities and ecosystem functions across a disturbance gradient . Journal of Animal Ecology, 2016, 85(6): 1636-1646. DOI:10.1111/1365-2656.12574
(  0) 0) |
| [71] |
Delgado-Baquerizo M, Eldridge D J, Hamonts K, et al. Ant colonies promote the diversity of soil microbial communities . The ISME Journal, 2019, 13(4): 1114-1118. DOI:10.1038/s41396-018-0335-2
(  0) 0) |
| [72] |
Nemergut D R, Schmidt S K, Fukami T, et al. Patterns and processes of microbial community assembly . Microbiology and Molecular Biology Reviews, 2013, 77(3): 342-356. DOI:10.1128/MMBR.00051-12
(  0) 0) |
| [73] |
Stegen J C, Lin X J, Konopka A E, et al. Stochastic and deterministic assembly processes in subsurface microbial communities . The ISME Journal, 2012, 6(9): 1653-1664. DOI:10.1038/ismej.2012.22
(  0) 0) |
| [74] |
Stegen J C, Lin X J, Fredrickson J K, et al. Quantifying community assembly processes and identifying features that impose them . The ISME Journal, 2013, 7(11): 2069-2079. DOI:10.1038/ismej.2013.93
(  0) 0) |
| [75] |
Dini-Andreote F, Stegen J C, van Elsas J D, et al. Disentangling mechanisms that mediate the balance between stochastic and deterministic processes in microbial succession . Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): 1326-1332. DOI:10.1073/pnas.1414261112
(  0) 0) |
| [76] |
Tripathi B M, Stegen J C, Kim M, et al. Soil pH mediates the balance between stochastic and deterministic assembly of bacteria . The ISME Journal, 2018, 12(4): 1072-1083. DOI:10.1038/s41396-018-0082-4
(  0) 0) |
| [77] |
Feng Y Z, Chen R R, Stegen J C, et al. Two key features influencing community assembly processes at regional scale:Initial state and degree of change in environmental conditions . Molecular Ecology, 2018, 27(24): 5238-5251. DOI:10.1111/mec.14914
(  0) 0) |
| [78] |
Graham E B, Knelman J E, Schindlbacher A, et al. Microbes as engines of ecosystem function:When does community structure enhance predictions of ecosystem processes? . Frontiers in Microbiology, 2016, 7: 214. DOI:10.3389/fmicb.2016.00214
(  0) 0) |
| [79] |
Mougi A, Kondoh M. Diversity of interaction types and ecological community stability . Science, 2012, 337(6092): 349-351. DOI:10.1126/science.1220529
(  0) 0) |
| [80] |
Gessner M O, Swan C M, Dang C K, et al. Diversity meets decomposition . Trends in Ecology & Evolution, 2010, 25(6): 372-380.
(  0) 0) |
| [81] |
Nielsen U N, Ayres E, Wall D H, et al. Soil biodiversity and carbon cycling:A review and synthesis of studies examining diversity-function relationships . European Journal of Soil Science, 2011, 62(1): 105-116. DOI:10.1111/j.1365-2389.2010.01314.x
(  0) 0) |
| [82] |
Hines J, van der Putten W H, de Deyn G B, et al. Chapter four-Towards an integration of biodiversity-ecosystem functioning and food web theory to evaluate relationships between multiple ecosystem services . Advances in Ecological Research, 2015, 53: 161-199. DOI:10.1016/bs.aecr.2015.09.001
(  0) 0) |
| [83] |
Valiente-Banuet A, Aizen M A, Alcántara J M, et al. Beyond species loss:The extinction of ecological interactions in a changing world . Functional Ecology, 2015, 29(3): 299-307.
(  0) 0) |
| [84] |
Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities . The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119
(  0) 0) |
| [85] |
de Vries F T, Griffiths R I, Bailey M, et al. Soil bacterial networks are less stable under drought than fungal networks . Nature Communications, 2018, 9: 3033. DOI:10.1038/s41467-018-05516-7
(  0) 0) |
| [86] |
He D, Shen W J, Eberwein J, et al. Diversity and co-occurrence network of soil fungi are more responsive than those of bacteria to shifts in precipitation seasonality in a subtropical forest . Soil Biology & Biochemistry, 2017, 115: 499-510.
(  0) 0) |
| [87] |
Delgado-Baquerizo M, Reith F, Dennis P G, et al. Ecological drivers of soil microbial diversity and soil biological networks in the Southern Hemisphere . Ecology, 2018, 99(3): 583-596. DOI:10.1002/ecy.2137
(  0) 0) |
| [88] |
Banerjee S, Schlaeppi K, van der Heijden M G A. Reply to 'Can we predict microbial keystones?' . Nature Reviews Microbiology, 2019, 17(3): 194. DOI:10.1038/s41579-018-0133-x
(  0) 0) |
| [89] |
Röttjers L, Faust K. Can we predict keystones? . Nature Reviews Microbiology, 2019, 17(3): 193. DOI:10.1038/s41579-018-0132-y
(  0) 0) |
| [90] |
Thebault E, Fontaine C. Stability of ecological communities and the architecture of mutualistic and trophic networks . Science, 2010, 329(5993): 853-856. DOI:10.1126/science.1188321
(  0) 0) |
| [91] |
Trøjelsgaard K, Olesen J M. Macroecology of pollination networks . Global Ecology and Biogeography, 2013, 22(2): 149-162. DOI:10.1111/j.1466-8238.2012.00777.x
(  0) 0) |
| [92] |
Lv X, Zhao K K, Xue R, et al. Strengthening insights in microbial ecological networks from theory to applications . mSystems, 2019. DOI:10.1128/msystems.00124-19
(  0) 0) |
| [93] |
Philippot L, Spor A, Hénault C, et al. Loss in microbial diversity affects nitrogen cycling in soil . The ISME Journal, 2013, 7(8): 1609-1619. DOI:10.1038/ismej.2013.34
(  0) 0) |
| [94] |
Wagg C, Bender S F, Widmer F, et al. Soil biodiversity and soil community composition determine ecosystem multifunctionality . Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(14): 5266-5270. DOI:10.1073/pnas.1320054111
(  0) 0) |
| [95] |
Schnitzer S A, Klironomos J N, HilleRisLambers J, et al. Soil microbes drive the classic plant diversity- productivity pattern . Ecology, 2011, 92(2): 296-303. DOI:10.1890/10-0773.1
(  0) 0) |
| [96] |
Kanchiswamy C N, Malnoy M, Maffei M E. Chemical diversity of microbial volatiles and their potential for plant growth and productivity . Frontiers in Plant Science, 2015, 6: 151.
(  0) 0) |
| [97] |
Limmathurotsakul D, Golding N, Dance D A B, et al. Predicted global distribution of Burkholderia pseudomallei and burden of melioidosis . Nature Microbiology, 2016, 1: 15008. DOI:10.1038/nmicrobiol.2015.8
(  0) 0) |
| [98] |
Nelson M B, Martiny A C, Martiny J B H. Global biogeography of microbial nitrogen-cycling traits in soil . Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8033-8040. DOI:10.1073/pnas.1601070113
(  0) 0) |
| [99] |
Delgado-Baquerizo M, Maestre F T, Reich P B, et al. Microbial diversity drives multifunctionality in terrestrial ecosystems . Nature Communications, 2016, 7: 10541. DOI:10.1038/ncomms10541
(  0) 0) |
| [100] |
Fierer N, Leff J W, Adams B J, et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes . Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(52): 21390-21395. DOI:10.1073/pnas.1215210110
(  0) 0) |
| [101] |
Jing X, Sanders N J, Shi Y, et al. The links between ecosystem multifunctionality and above-and belowground biodiversity are mediated by climate . Nature Communications, 2015, 6: 8159. DOI:10.1038/ncomms9159
(  0) 0) |
| [102] |
Delgado-Baquerizo M, Trivedi P, Trivedi C, et al. Microbial richness and composition independently drive soil multifunctionality . Functional Ecology, 2017, 31(12): 2330-2343. DOI:10.1111/1365-2435.12924
(  0) 0) |
| [103] |
Delgado-Baquerizo M, Eldridge D J, Ochoa V, et al. Soil microbial communities drive the resistance of ecosystem multifunctionality to global change in drylands across the globe . Ecology Letters, 2017, 20(10): 1295-1305. DOI:10.1111/ele.12826
(  0) 0) |
| [104] |
Larsen P, Hamada Y, Gilbert J. Modeling microbial communities:Current, developing, and future technologies for predicting microbial community interaction . Journal of Biotechnology, 2012, 160(1/2): 17-24.
(  0) 0) |
| [105] |
Fierer N, Ladau J, Clemente J C, et al. Reconstructing the microbial diversity and function of pre-agricultural tallgrass prairie soils in the United States . Science, 2013, 342(6158): 621-624. DOI:10.1126/science.1243768
(  0) 0) |
| [106] |
Karimi B, Terrat S, Dequiedt S, et al. Biogeography of soil bacteria and archaea across France . Science Advances, 2018, 4(7): eaat1808. DOI:10.1126/sciadv.aat1808
(  0) 0) |
| [107] |
Delgado-Baquerizo M, Eldridge D J. Cross-biome drivers of soil bacterial alpha diversity on a worldwide scale . Ecosystems, 2019, 22(6): 1220-1231. DOI:10.1007/s10021-018-0333-2
(  0) 0) |
| [108] |
Egidi E, Delgado-Baquerizo M, Plett J M, et al. A few Ascomycota taxa dominate soil fungal communities worldwide . Nature Communications, 2019, 10: 2369. DOI:10.1038/s41467-019-10373-z
(  0) 0) |
| [109] |
van den Hoogen J, Geisen S, Routh D, et al. Soil nematode abundance and functional group composition at a global scale . Nature, 2019, 572(7768): 194-198. DOI:10.1038/s41586-019-1418-6
(  0) 0) |
| [110] |
Steidinger B S, Consortium G, Crowther T W, et al. Climatic controls of decomposition drive the global biogeography of forest-tree symbioses . Nature, 2019, 569(7756): 404-408. DOI:10.1038/s41586-019-1128-0
(  0) 0) |
| [111] |
Griffiths R I, Thomson B C, Plassart P, et al. Mapping and validating predictions of soil bacterial biodiversity using European and national scale datasets . Applied Soil Ecology, 2016, 97: 61-68. DOI:10.1016/j.apsoil.2015.06.018
(  0) 0) |
| [112] |
Terrat S, Horrigue W, Dequietd S, et al. Mapping and predictive variations of soil bacterial richness across France . PLoS One, 2017, 12(10): e0186766. DOI:10.1371/journal.pone.0186766
(  0) 0) |
| [113] |
Thompson L R, Sanders J G, McDonald D, et al. A communal catalogue reveals Earth's multiscale microbial diversity . Nature, 2017, 551(7681): 457-463. DOI:10.1038/nature24621
(  0) 0) |
| [114] |
Gilbert J A, Jansson J K, Knight R. Earth microbiome project and global systems biology . mSystems, 2018, 3: e00217. DOI:10.1128/msystems.00217-17
(  0) 0) |
| [115] |
Cameron E K, Martins I S, Lavelle P, et al. Global gaps in soil biodiversity data . Nature Ecology & Evolution, 2018, 2(7): 1042-1043.
(  0) 0) |
| [116] |
Achtman M, Wagner M. Microbial diversity and the genetic nature of microbial species . Nature Reviews Microbiology, 2008, 6(6): 431-440. DOI:10.1038/nrmicro1872
(  0) 0) |
| [117] |
Pham V H T, Kim J. Cultivation of unculturable soil bacteria . Trends in Biotechnology, 2012, 30(9): 475-484. DOI:10.1016/j.tibtech.2012.05.007
(  0) 0) |
| [118] |
York A. Next-generation bacterial taxonomy . Nature Reviews Microbiology, 2018, 16(10): 583.
(  0) 0) |
| [119] |
Shade A, Gregory Caporaso J, Handelsman J, et al. A meta-analysis of changes in bacterial and archaeal communities with time . The ISME Journal, 2013, 7(8): 1493-1506. DOI:10.1038/ismej.2013.54
(  0) 0) |
| [120] |
Delgado-Baquerizo M, Bardgett R D, Vitousek P M, et al. Changes in belowground biodiversity during ecosystem development . Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(14): 6891-6896. DOI:10.1073/pnas.1818400116
(  0) 0) |
| [121] |
Nemergut D R, Anderson S P, Cleveland C C, et al. Microbial community succession in an unvegetated, recently deglaciated soil . Microbial Ecology, 2007, 53(1): 110-122. DOI:10.1007/s00248-006-9144-7
(  0) 0) |
| [122] |
Ortiz-Álvarez R, Fierer N, de los Ríos A, et al. Consistent changes in the taxonomic structure and functional attributes of bacterial communities during primary succession . The ISME Journal, 2018, 12(7): 1658-1667. DOI:10.1038/s41396-018-0076-2
(  0) 0) |
| [123] |
Carini P, Delgado-Baquerizo M, Hinckley E L S, et al. Unraveling the effects of spatial variability and relic DNA on the temporal dynamics of soil microbial communities . bioRxiv, 2018, 12: 43.
(  0) 0) |
| [124] |
Huang G, Li Y, Su Y G. Effects of increasing precipitation on soil microbial community composition and soil respiration in a temperate desert, Northwestern China . Soil Biology & Biochemistry, 2015, 83: 52-56.
(  0) 0) |
| [125] |
Marian M, Shimizu M. Improving performance of microbial biocontrol agents against plant diseases . Journal of General Plant Pathology, 2019, 85(5): 329-336. DOI:10.1007/s10327-019-00866-6
(  0) 0) |
| [126] |
van Oppen M J H, Blackall L L. Coral microbiome dynamics, functions and design in a changing world . Nature Reviews Microbiology, 2019, 17(9): 557-567. DOI:10.1038/s41579-019-0223-4
(  0) 0) |
| [127] |
Epstein H E, Smith H A, Torda G, et al. Microbiome engineering:Enhancing climate resilience in corals . Frontiers in Ecology and the Environment, 2019, 17(2): 100-108. DOI:10.1002/fee.2001
(  0) 0) |
| [128] |
Petipas R H, González J B, Palmer T M, et al. Habitat-specific AMF symbioses enhance drought tolerance of a native Kenyan grass . Acta Oecologica, 2017, 78: 71-78. DOI:10.1016/j.actao.2016.12.005
(  0) 0) |
| [129] |
Solé R V, Montañez R, Duran-Nebreda S. Synthetic circuit designs for earth terraformation . Biology Direct, 2015, 10: 37. DOI:10.1186/s13062-015-0064-7
(  0) 0) |
| [130] |
Solé R V, Montañez R, Duran-Nebreda S, et al. Population dynamics of synthetic terraformation motifs . Royal Society Open Science, 2018, 5(7): 180121. DOI:10.1098/rsos.180121
(  0) 0) |
 2020, Vol. 57
2020, Vol. 57

