2. 中国科学院大学, 北京 100049;
3. 南京农业大学资源与环境科学学院, 土壤生态学实验室, 南京 210095;
4. 中国科学院北京生命科学研究院, 北京 100101;
5. 中国科学院深圳先进技术研究院, 深圳 518055;
6. 中国科学院生态环境研究中心, 北京 100085;
7. 南京师范大学环境学院, 南京 210023
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Soil Ecology Lab, College of Resources and Environmental Sciences, Nanjing Agricultural University, Nanjing 210095, China;
4. Beijing Institutes of Life Sciences, Chinese Academy of Sciences, Beijing 100101, China;
5. Shenzhen Institutes of Advanced Technology Chinese Academy of Sciences, Shenzhen 518055, China;
6. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
7. School of Environment, Nanjing Normal University, Nanjing 210023, China
病毒是地球上数量最多的生命体,广泛存在于地球各种环境中[1]。土壤病毒在调控微生物群落组成、影响元素循环利用、促进生物进化等方面发挥重要作用[2-4]。近年来,海洋环境中病毒生态学研究进展迅速,虽然学术界逐渐意识到病毒在土壤环境中发挥着重要作用,但由于病毒缺少通用的标记基因,以及受到土壤异质性、多样性的限制,陆地系统,特别是土壤环境中病毒研究进展相对缓慢[5-6],土壤病毒学研究远落后于海洋等水体环境[2]。噬菌体是寄生在细菌、古菌等原核生物里的病毒,也是土壤中最主要的病毒类群。单株噬菌体的全基因组测序有助于了解噬菌体的基因组结构及功能特征;而随着测序技术的发展,宏病毒组学研究日益受到关注,它摆脱了以往微生物分离纯培养的限制,以环境样品为研究对象,直接从土壤样品中富集和提取病毒基因组后进行测序和生物信息学分析[7-8],为土壤病毒研究提供了新的技术手段[9]。
由此,本文主要介绍了土壤研究方法及病毒组研究进展,指出了土壤病毒研究当前面临的困境,并对未来的研究方向和发展趋势进行了展望,以期为后续土壤宏病毒研究提供科学参考。
1 土壤宏病毒组的研究方法 1.1 土壤病毒核酸提取大部分土壤病毒被土壤颗粒吸附固定,对土壤病毒研究的关键是建立精准、专性、高效的提取方法,获得吸附于土壤颗粒表面及内部的病毒粒子[10]。但土壤环境复杂且微生物多样性高,在提取病毒宏基因组核酸时易混入细菌、真菌等其他微生物的核酸序列,从而影响后续基因序列分析、功能基因注释的准确性。因此,土壤病毒提取方法的选择(如滤膜、提取剂等)对后续病毒核酸纯度、功能基因注释的准确性具有较大影响。Göller等[6]分别使用0.22 μm和0.45 μm滤膜过滤病毒,发现0.22 μm滤膜可去除更多的细菌DNA,且不影响土壤病毒多样性,而通过0.45 μm孔径过滤的病毒体中不仅细菌污染程度增加,病毒多样性也有所下降。Williamson等[11]比较了10%(w/v)的牛肉膏、250 mmol·L–1甘氨酸溶液、10 mmol·L–1焦磷酸钠和1%(w/v)柠檬酸钾溶液四种提取剂对粉砂壤土和砂壤土中病毒的提取效果。通过活菌计数法(也称间接计数法,仅测活菌总数)计算提取率,发现250 mmol·L–1甘氨酸溶液提取率可达28.9%;其次是10%(w/v)牛肉膏,提取率为26.0%;而1%(w/v)柠檬酸钾溶液和10 mmol·L–1焦磷酸钠提取率较低,分别为16.9%和15.0%。后续荧光显微镜计数时,发现牛肉膏提取物、粉砂壤土中焦磷酸钠提取物和砂壤土中的甘氨酸提取物均无法计数类病毒颗粒(virus-like particles,VLPs),故该研究认为1%(w/v)柠檬酸钾溶液是最佳的土壤病毒提取剂。与此同时,土壤pH、土壤阳离子交换量、温度、含水量等环境因子均会影响土壤病毒粒子的提取效率[10]。此外,当病毒遗传物质含量较低时,会影响后续宏病毒组分析,虽可经多重置换扩增(multiple displacement amplification,MDA)或引物延伸预扩增(primer extension preamplification,PEP)等全基因组扩增技术增加病毒核酸浓度,但这些技术在扩增产物上可能存在偏好性。为保证结果的准确性,建议提高原始土壤病毒样品富集量,再进行宏病毒组基因测序[2]。
1.2 测序与病毒序列鉴定第二代测序技术具有通量高、速度快、成本低的优点[12-13],使得宏病毒组研究发生巨大变化。其中Illumina目前拥有MiSeq、HiSeq、NovaSeq等多个技术平台,并有多种读长和通量模式供选择。但总体而言,第二代测序技术仍存在序列读长短的局限性。近年来,第三代测序技术的快速发展在宏病毒组研究中显示出巨大潜力[14],它无需进行PCR扩增,且具有读长长的优点。随着第三代测序技术的不断成熟及测序成本的降低,预计它的普及程度将不断提升。
在含有病毒和宿主的混合基因组数据中,鉴定病毒序列是解析病毒信息的关键[15],会直接影响后续病毒功能基因注释的效果。目前,识别完整微生物基因组中原噬菌体的工具主要有Phage_Finder[16],Prophinder[17]和PHAST。PHAST速度快、准确性高的优势使其成为极具吸引力的工具,后续由于序列数据库规模的扩大及用户数量的增加,PHAST发行了新版本PHASTER[18]。与PHAST相比,新版本的突出优势在于可以识别宏基因组拼接产物的原噬菌体[19]。尽管PHASTER与PHAST是在细菌基因组中鉴别原噬菌体的两个广泛使用的工具,但值得注意的是,对预测的原噬菌体精准程度,如融合位点的位置仍存在一些不确定性[19]。此外,这些软件大多没有设计专门的算法用于相对较短的重叠群或支架,且无法在短时间内同时处理大量的序列,因此并不适合从宏基因组数据中鉴别病毒序列[15]。2015年Roux等[20]开发了一款工具—VirSorter,不仅可识别完整微生物基因组中的原噬菌体,还可用于检测拼接宏基因组数据中的病毒序列。VirSorter虽然在很大程度上依赖于对已有病毒基因组的相似性搜索,但它却使用了一个自定义的病毒参考基因组数据库,增加了从淡水、海水、人体肠道、肺和唾液中取样的宏病毒组序列[15]。2017年Ren等[15]开发了一款基于k-mer的工具VirFinder,用于从宏基因组数据中识别原核病毒序列。与基于基因的病毒分类工具VirSorter相比,VirFinder在识别病毒序列方面明显优于VirSorter。在模拟数据集中,VirFinder从1、3和5 kb序列中识别病毒序列的真阳性率分别较VirSorter高78倍、2.4倍和1.8倍;其假阳性率与VirSorter相同,表明VirFinder真阳性更高、对短片段序列的识别效果更好[15]。但这两款软件也有其局限性,VirSorter和VirFinder为检测细菌和古细菌病毒而优化设计,不能很好地检测真核病毒;且两者在微生物群落中对病毒分析的功能相对有限,在鉴定出病毒序列后,没有进一步分析病毒与宿主的对应关系。相比之下,VirMiner是一款病毒预测与分析宏病毒组样本的综合工具[21],能够捕获到高丰度的噬菌体序列,这些噬菌体在感染细菌和影响微生物群落动态方面起着关键作用;更重要的是,VirMiner提供了更全面的噬菌体分析流程,包括宏基因组原始读段处理、功能注释、噬菌体序列鉴定、噬菌体-宿主侵染关系预测;此外,当宏基因组序列包括不同条件的数据(如处理组和对照组)时,还可支持不同组之间的统计比较。
1.3 病毒功能基因注释病毒功能基因注释是将预测出的编码基因通过与相关数据库的参考序列进行比对,在与现有病毒进行同源性搜索的基础上,获取该基因的功能信息。通过对病毒功能基因注释,不仅为深入认识病毒个体生命过程提供理论基础;还有助于了解病毒群落的生态过程及与宿主群落的生态互作关系,从而阐释病毒与宿主、环境间复杂的相互作用机制。本文重点从单个噬菌体的全基因组测序及土壤宏病毒组两方面阐释了土壤病毒的功能基因注释。
单个土壤噬菌体的全基因组注释流程,首先从土壤筛选分离得到噬菌体,经纯化、浓缩后采用透射电子显微镜观察其形态特征;随后提取噬菌体核酸,进行全基因组测序,在过滤掉低质量序列后进行全基因组序列组装,并通过注释软件等在线工具,对噬菌体全基因组序列进行功能注释[22]。
土壤宏病毒组的功能基因注释由土壤样品制备与宏病毒组分析两个主要部分组成(图 1),具体步骤包括:(1)根据研究目的从相应的土壤中采集样本,并根据病毒类型、土壤理化性质等选择合适的缓冲液进行土壤病毒提取,随后对提取液进行过滤,去除细菌等其他潜在宿主,进而浓缩富集、纯化病毒;(2)通过病毒核酸提取试剂盒或手工提取的方式获取病毒核酸、构建测序文库、并通过测序平台进行宏病毒组测序;(3)对测序得到的原始数据进行质量控制,基于重叠区(overlap)将高质量测序读段(reads)拼接为重叠群(contigs),进一步组装成支架(scaffolds);(4)通过病毒序列识别软件在重叠群或支架中判别、筛选出病毒序列;(5)使用基因预测软件对病毒基因组ORF进行预测,再通过注释工具将ORF与多个数据库比对进行功能基因注释;(6)自动注释后,为保证结果准确性,可手动修正自动注释结果并进行适当补充。
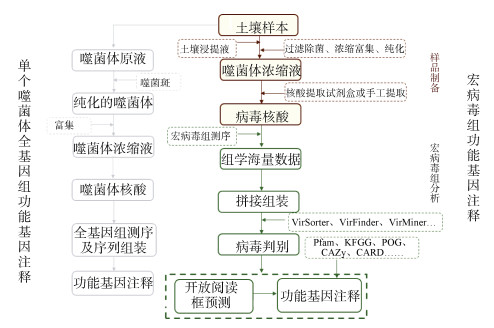
|
图 1 病毒功能基因注释流程图 Fig. 1 Flowcharts of functional gene annotation for virus |
目前,普遍采用与各种数据库进行蛋白质序列比对的方法,对样本中的基因功能进行注释分析。在注释过程中,研究人员通常根据自身需求选择合适的功能数据库。其中Pfam数据库是一个基于多序列比对和隐马尔可夫模型的蛋白质结构域和家族数据库,可提供蛋白质家族和结构域的完整准确分类,被广泛用于查询蛋白家族或蛋白结构域的注释,结构及多序列比对信息,在基因功能注释上可用性较强[23-24]。它有A和B两个质量级别的家族数据库,Pfam A通过比对人工校正过的种子序列,并使用隐马尔可夫模型进行选择,数据质量较高;Pfam B为算法自动生成,虽可靠性降低,但也可以被用于鉴别功能保守的区域。GO(gene ontology)数据库分别从细胞学组件、分子功能、生物学途径对基因表达产物进行简单注释。经GO数据库注释后,可得到基因在不同类别中注释的具体情况。而KEGG(kyoto encyclopedia of genes and genomes)是一个系统分析基因功能的知识库,核心为KEGG PATHWAY数据库。利用KEGG进行注释后,能清楚地反映出基因与相关代谢的关系[25]。COG(clusters of orthologous groups)数据库是由NCBI创建并维护的蛋白数据库[26]。随着测序技术的发展,COG陆续在不同物种中建立相关的同源蛋白簇。基于完整噬菌体基因组中的编码蛋白系统进化关系,构建而成的POG(phage orthologous groups)数据库便是其中的一个分支[27]。通过比对,可将某个特定蛋白序列注释到一个由直系同源序列构成的POG中,从而推测该序列的功能。此外,POG数据库包含了进化过程中基因得失信息,还可用于系统发育的统计推断和祖先基因组的重建[27]。CAZy(carbohydrate-active enzyme)数据库则针对性较强,是一类与合成或分解复杂碳水化合物和糖复合物酶类有关的数据库,可提供碳水化合物酶类物种来源、酶功能分类、基因序列、蛋白质序列及其结构等信息[28]。还有一些小众数据库,如抗性基因数据库CARD(comprehensive antibiotic resistance database)在细菌耐药性的分子基础上,提供了参考DNA和蛋白质序列、检测模型和生物信息学工具[29]。ARO(antibiotic resistance ontology)是该数据库的核心,包含了与抗生素抗性基因、抗性机制、抗生素相关的条目。通过与该数据库进行比对,可得到与耐药基因相关的注释信息。
2 土壤噬菌体基因组研究进展噬菌体是土壤中最主要的病毒类群,对单个噬菌体进行全基因组测序和功能基因注释,有助于探明该噬菌体的基因信息和功能特征,进而挖掘该噬菌体在环境修复、疾病治疗等实际应用中的巨大潜力。如近年来,噬菌体疗法作为一种可以高效靶向追踪灭活土壤体系中致病细菌的有效手段而日益受到关注[30],故分离筛选出新型烈性噬菌体菌株资源对于噬菌体疗法具有重要意义。但由于一些噬菌体携带毒力基因等原因,导致噬菌体防治存在“双刃剑”风险[31]。因此,从土壤中分离单株噬菌体,并对其进行全基因组测序、功能基因注释有助于我们更好的了解噬菌体的基因组结构及功能特征。
2.1 全基因组测序下的土壤噬菌体研究趋势借助Web of Science核心合集以“complete genome sequence”和“phage”为关键词进行检索,在2000年-2020年时间段内共有1 508篇相关文献。发现近20年该领域发文量呈现整体上升趋势(图 2a)。随后利用VOSviewer[32]可视化分析软件对关键词进行聚类分析(图 2b)。图中节点大小表示关键词出现的频率,颜色反映不同时间的研究热点,其中黄色部分代表了较为前沿的关键词。总体而言,“开放阅读框open reading frame”、“原噬菌体prophage”、“病原体pathogen”、“表征characterization”、“作用role”是出现频率较高的词,表明对噬菌体的研究主要聚焦于其形态和功能。值得注意的是,“土壤soil”、“抗生素抗性antibiotic resistance”、“噬菌体疗法phage therapy”是近几年出现的关键词。“土壤soil”的出现表明土壤噬菌体领域的研究将日益受到重视;此外,由于近年来土壤中抗生素抗性基因的增多、超级细菌的出现,噬菌体疗法可能成为未来土壤中病原细菌灭活的重要手段。因此,研发对病原细菌具有广谱性的混合噬菌体鸡尾酒制剂,探明噬菌体疗法对于土壤微生物群落结构、功能及养分循环的影响可能是未来土壤噬菌体领域的研究热点。
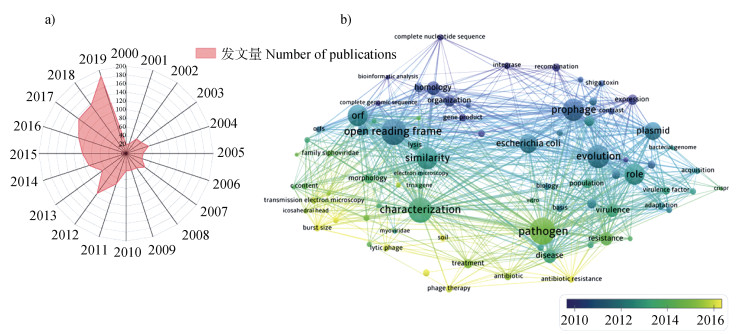
|
图 2 噬菌体全基因组测序领域年发表论文数量(a)及关键词(b)共现网络图谱 Fig. 2 Number of publications(a)and keyword(b)co-occurrence network map in the field of phage genome sequencing |
苏靖芳等人[31]以引起烟草以及多种茄科植物产生萎蔫症的青枯雷尔氏菌(Ralstonia solanacearum)为宿主,采用双层平板法从烟田土壤中分离出一株烈性噬菌体RS-PII-1。随后对噬菌体RS-PII-1进行全基因组测序,并通过RAST在线软件对噬菌体全基因组序列进行功能注释,发现该基因组含有46个ORF,其中25个ORF可被注释为相应的功能蛋白、结构蛋白,但仍存在21个功能未知的ORF假想蛋白(hypothetical protein)。通过与已知噬菌体全基因组序列比较分析以及噬菌体RS-PII-1系统进化树的构建,发现噬菌体RSJ2、RSB1与RS-PII-1相似度最高,但噬菌体RS-PII-1基因组中功能蛋白和假想蛋白区域与两株噬菌体仍存在明显差异,表明RS-PII-1是一株全新的青枯雷尔氏菌烈性噬菌体,这对防控由青枯雷尔氏菌引起的细菌性病害具有较强的应用意义。蜡状芽孢杆菌(Bacillus cereus)可导致人体腹泻、呕吐等食源性中毒,Hock等[33]从土壤中分离出一株感染蜡状芽孢杆菌的长尾病毒科噬菌体Deep‑Purple,随后对该噬菌体进行全基因组测序、预测潜在编码序列(coding sequence,CDS),并对CDS进行功能注释。结果表明,CDS可分为结构相关蛋白(structural related proteins)、DNA复制转录(DNA replication and transcription)、DNA包装(DNA packaging)及宿主裂解(host lysis)四个功能组。同时发现,该噬菌体不存在编码潜在毒力因子的基因,且具有热稳定性、pH稳定性的特点,研究人员认为噬菌体Deep‑Purple可作为治疗由蜡状芽孢杆菌引起的食物中毒的潜在药物。综上所述,全基因组测序下的土壤噬菌体研究不仅有助于了解土壤中新型噬菌体的结构和功能,在致病菌引起的细菌性病害治疗方面也具有积极意义。
3 土壤宏病毒基因组研究进展 3.1 土壤环境中的宏病毒组研究宏病毒组主要对种群结构、基因功能活性、病毒与宿主的协作关系以及与环境之间的联系进行探究,这为土壤环境微生物群落的研究提供了有力支撑[9,34]。病毒识别及其功能基因注释是其中一个重要环节,在完成数据质控、基因组装、基因预测等过程后,将预测的病毒编码基因与COG、eggNOG[35]等数据库比对,获得病毒功能信息。表 1对近年来土壤病毒组研究中具有代表性的功能基因进行总结,以揭示病毒与宿主群落的相互作用机制及其在元素生物地球化学循环中的作用机理。
|
|
表 1 典型土壤病毒基因组功能基因及作用 Table 1 Typical functional genes of soil viral genome |
Jin等[36]在中国广西和海南三个红树林生境采集样本,研究了红树林土壤病毒功能多样性。通过将预测的病毒ORF与eggNOG数据库进行比对,发现大多数ORF无法获得注释信息,但有138个ORF可以注释到与碳水化合物活性酶(carbohydrate-active enzyme,CAZyme)相关的基因。随后,经过CAZy数据库的进一步注释,显示糖苷水解酶类(glycoside hydrolases)ORF最为丰富,其次是糖基转移酶类(glycosyl transferases)、碳水化合物结合模块(carbohydrate-binding modules)等。这表明病毒可通过复杂多糖的生物分解直接调控碳循环,揭示了病毒在有机碳分解中的重要作用。Bi等[37]在中国西南部地区采集了4个玉米根际土壤及4个非根际土壤样本,探究农业生态系统中病毒的多样性及其对潜在生物地球化学循环的影响机制。通过对土壤病毒组进行测序,将测序读段组装成237个长度为10kb以上的重叠群,并通过VirSorter软件鉴定病毒序列。对这些土壤病毒进行功能基因注释,共有40个基因被鉴定为溶菌酶或几丁质酶,可用于降解宿主细胞壁。此外,共鉴定出了48个ORF与碳水化合物活性酶相关,包括碳水化合物结合模块、碳水化合物酯酶(carbohydrate esterases)及糖苷水解酶。表明病毒可能编码改变宿主活性的辅助代谢基因,间接参与土壤碳元素的生物地球化学循环。值得注意的是,研究人员发现该农业土壤中病毒的大多数CAZyme基因与红树林土壤中的病毒并不同[36],认为病毒编码的酶可能具有环境特异性。Segobola等[38]通过宏病毒组技术对灌木土壤的病毒群落进行探究。经过病毒基因组测序、组装及功能基因注释,发现组装后最长的重叠群近乎一个完整的噬菌体基因组。该基因组的基因15和基因16分别对应末端酶大亚基和小亚基基因,参与了噬菌体双链DNA的裂解和包装;基因34的翻译产物被识别为假定的ERF超家族蛋白,可参与噬菌体基因组的重组;基因41的翻译产物被鉴定为gp77蛋白,与分枝杆菌(Mycobacterium)噬菌体Che9d编码的同源物有95%的相似性,该蛋白在噬菌体复制的早期起到关闭基因(shut-off genes)的作用。通过KEGG Orthology(KO)数据库对宏病毒组进行分析,发现相关代谢蛋白(如碳水化合物代谢、氨基酸代谢和核苷酸代谢)识别率最高,表明土壤病毒很可能干扰宿主的新陈代谢。Liang等[39]从美国东南部农业区老成土(美国土壤分类系统的一个土纲)中提取病毒。通过对土壤表层(0~16 cm)和亚表层(55~92 cm)病毒组基因序列进行组装、分类,随后采用VIROME对ORF进行功能基因注释,发现大量病毒组序列功能未知,50.3%的预测蛋白在所比对的数据库中没有显著同源性;有35.4%~38.7%的ORF被注释为与宿主代谢途径相关,如细胞信号(cell signaling)、氧化磷酸化(oxidative phosphorylation)、遗传信息处理(genetic information processing)和磷、蛋白质及碳水化合物的代谢(metabolisms of phosphorous,protein,and carbohydrates)等。此外,研究人员还在病毒组中发现了丰富的碳水化合物代谢(carbohydrate metabolism)基因,表明病毒可能参与土壤碳循环的调节。值得注意的是,在亚表层土壤病毒组中检测到的功能蛋白(除参与氧化磷酸化的蛋白质)编码基因丰度比表层土壤病毒体高16倍,表明亚表层土壤病毒虽密度较低,但可能与微生物介导的过程密切相关;与噬菌体感染循环(即噬菌体溶源、裂解循环和原噬菌体诱导)和噬菌体结构成分(如噬菌体衣壳)相关的蛋白标准化丰度(normalized abundances)在亚表层土壤病毒组中也更高,这可能与病毒宿主在贫瘠营养条件下的协同进化有关。由此可得,病毒在调控土壤中营养元素生物地球化学循环,调节宿主新陈代谢及微生物群落结构方面发挥重要作用。
3.2 特殊及极端陆地环境中宏病毒组研究Gao等[40]于广东某铅锌矿尾矿库采集样本,研究高度分层硫化尾矿中病毒群落组成和功能特征。通过将预测的病毒蛋白与eggNOG数据库比对,进行了病毒基因组直系同源基因簇COG的注释分析。发现地表尾矿由于存在大量古菌和古菌病毒,导致大多数COG注释困难;反之,以细菌为主的深层病毒群落存在着大量与同化硫酸盐还原、转座酶、DNA复制、噬菌体整合酶和重组酶相关的COG。随后研究者试图确定病毒编码的辅助代谢基因,发现深层病毒群落含有丰富的同化硫酸盐还原辅助代谢基因,这有利于宿主利用硫酸盐,进而促进病毒的复制和繁殖。Daly等[41]对水力压裂井中病毒与宿主的相互作用动力学进行探究,施加应激源对菌株(Halanaerobium. congolense WG8)进行原噬菌体诱导实验,并对病毒核酸进行纯化、测序、基因注释。结果显示,病毒基因组中存在与整合酶(integrase)、切除酶(excisionase)、假想蛋白以及转座酶(transposase)相关的基因;同时发现,其中一个基因被注释为重叠感染排斥蛋白(superinfection exclusion protein)基因,推测该基因的存在可能有助于维持宿主容纳原噬菌体与细胞裂解的平衡。Bezuidt等[42]在南极土壤群落中使用VirSorter工具从宏基因组序列数据中组装了793个重叠群。通过病毒基因组数据库对其进行分类注释,有645个contigs被定义为病毒,且560个被进一步划分为有尾噬菌体目。随后使用eggNOG数据库进行功能分析,发现噬菌体具有促进宿主感染的基因,如编码几丁质酶的基因,可参与宿主生物膜的降解。此外,eggNOG功能分析也揭示了增强噬菌体毒性基因的存在,其中编码甲基转移酶的基因最为丰富,该基因编码的酶有利于噬菌体规避宿主的限制修饰(restriction-modification,RM)系统。这一结果表明,噬菌体可能在面临进化压力时发展出对宿主的规避机制。Adriaenssens等[43]对纳米比亚沙漠岩石下生物宏病毒组进行探究,发现大多数病毒为有尾噬菌体目,其中长尾病毒科是最常见的病毒型。通过功能基因注释,有3%的基因被MG-RAST分类为“毒力、疾病、防御”(virulence,disease and defense)子系统,相应的编码序列被鉴定为来自致病细菌的假想蛋白或噬菌体相关蛋白,如整合酶和复制蛋白(replication proteins)。此外,研究人员还发现噬菌体中存在与磷酸盐调节相关的辅助代谢基因phoH,并通过MetaVir鉴定出18条完整的phoH基因和23条部分phoH序列。而在用极大或然法构建的系统发生树却显示大部分沙漠岩石下病毒phoH氨基酸序列与NCBI数据库中的完整噬菌体基因组序列关系较远,且海洋和沙漠岩石下病毒phoH基因分布在不同的进化枝上,表明纳米比亚沙漠岩石中存在独特的噬菌体phoH基因序列。值得注意的是,该病毒样本中没有发现与光合作用或营养胁迫相关的宿主衍生基因,推测此生境中phoH基因较其他辅助代谢基因发挥更重要的作用。Emerson等[44]在瑞典斯托达伦沼泽地的泥炭岩芯处,采集了三个不同栖息地的病毒样本,从中获得了53个vOTUs(viral operational taxonomic units),发现仅有约30%的基因可被注释,该结果可以佐证土壤是大量未知病毒遗传多样性的储存库[45]。此外,在13个vOTUs中鉴定出多个参与多糖结合(polysaccharide binding)、多糖降解(polysaccharide degradation)、中心碳代谢(central C metabolism)及孢子形成(sporulation)的辅助代谢基因。病毒的中心碳代谢基因可能在感染宿主期间增加核苷酸和能量产生;调节内孢子形成过程中的两个辅助代谢基因spoVS和whiB分别有助于形成隔膜和外套,从而提高孢子的耐热性。这揭示了病毒在介导碳代谢、土壤有机质降解、多糖结合和孢子形成过程中的调控作用。
上述案例揭示病毒不仅在农业土壤中与宿主、环境之间存在复杂的相互作用联系,而且在极端、特殊的陆地环境中对调控微生物群落组成、影响生物地球化学循环、促进生物协同进化等方面也具有巨大潜能。
4 土壤宏病毒基因组的前沿与展望现阶段学术界对土壤病毒及其功能基因的科学认知依然十分有限。基于病毒组学领域的发展趋势,今后土壤病毒组研究方向主要聚焦在以下方面:
1)土壤病毒主要通过微孔滤膜过滤的方式进行富集,该方法易将较大的病毒类型(如最近发现的巨型噬菌体)屏蔽在外[46-47],从而缺乏对此类病毒功能基因的认识;此外,目前的研究主要聚焦于DNA病毒,对RNA病毒研究较少。未来需研发针对巨型病毒的提取和富集技术、关注土壤RNA病毒的基因功能,这有助于学者探明土壤整体病毒的生态功能及作用机制。
2)现阶段土壤病毒的提取、宏病毒组分析等缺乏统一技术规范。开发宏病毒组研究独有的新方法,逐步规范技术流程,制定可以广泛通用于土壤宏病毒组分析的技术导则和标准十分必要。
3)目前病毒功能基因注释较为费时费力,即使通过自动注释也因其准确性不足,需后期人工注释进行修正,因此未来需开发更加高效、准确的生物信息学工具,识别病毒并注释其基因组中的基因功能。
4)土壤病毒学的研究还处于起步阶段,病毒基因组测序的数量远落后于相应宿主细菌的基因组测序数量,通过同源蛋白对土壤病毒基因组上的蛋白功能进行注释时,由于病毒ORF更短、进化更快,以及全球病毒取样的有限性、数据库较小等原因,导致部分ORF找不到匹配的功能注释。故仍需大力发展全基因组扩增技术和测序技术,不断完善土壤病毒资源库,为病毒功能基因注释提供有力支撑。
5)关注土壤病毒群落与宿主菌群的生态关系,进一步探究土壤病毒群落在元素生物地球化学循环中的直接与间接调控作用,深入探明土壤病毒与污染物的响应机制。
致谢 衷心感谢美国Rice大学土木与环境工程系俞萍锋博士在本文撰写和修改过程中给予的学术指导和建议。
| [1] |
Cobián Güemes A G, Youle M, Cantú V A, et al. Viruses as winners in the game of life[J]. Annual Review of Virology, 2016, 3(1): 197-214. DOI:10.1146/annurev-virology-100114-054952
(  0) 0) |
| [2] |
Wang G H, Liu J J, Zhu D, et al. A review of researches on viruses in soil-Advancement and challenges (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(6): 1319-1332. [王光华, 刘俊杰, 朱冬, 等. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319-1332.]
(  0) 0) |
| [3] |
Emerson J B. Soil viruses: A new hope[J]. mSystems, 2019, 4(3): e00120-19.
(  0) 0) |
| [4] |
Kuzyakov Y, Mason-Jones K. Viruses in soil: Nano-scale undead drivers of microbial life, biogeochemical turnover and ecosystem functions[J]. Soil Biology & Biochemistry, 2018, 127: 305-317.
(  0) 0) |
| [5] |
Wang G H. Lift mysterious veil of soil virus: 'Dark Matter' of soil biota (In Chinese)[J]. Bulletin of Chinese Academy of Sciences, 2017, 32(6): 575-584. [王光华. 掀开土壤生物"暗物质"——土壤病毒的神秘面纱[J]. 中国科学院院刊, 2017, 32(6): 575-584.]
(  0) 0) |
| [6] |
Göller P C, Haro-Moreno J M, Rodriguez-Valera F, et al. Uncovering a hidden diversity: Optimized protocols for the extraction of dsDNA bacteriophages from soil[J]. Microbiome, 2020, 8: 17. DOI:10.1186/s40168-020-0795-2
(  0) 0) |
| [7] |
Garza D R, Dutilh B E. From cultured to uncultured genome sequences: Metagenomics and modeling microbial ecosystems[J]. Cellular and Molecular Life Sciences, 2015, 72(22): 4287-4308. DOI:10.1007/s00018-015-2004-1
(  0) 0) |
| [8] |
Xu Z W, Wei Y L, Ji X L. Advances in viral metagenomics (In Chinese)[J]. Microbiology China, 2020, 47(8): 2560-2570. [徐志伟, 魏云林, 季秀玲. 病毒宏基因组学研究进展[J]. 微生物学通报, 2020, 47(8): 2560-2570.]
(  0) 0) |
| [9] |
Munson-Mcgee J H, Peng S Y, Dewerff S, et al. A virus or more in(nearly) every cell: Ubiquitous networks of virus-host interactions in extreme environments[J]. The ISME Journal, 2018, 12(7): 1706-1714. DOI:10.1038/s41396-018-0071-7
(  0) 0) |
| [10] |
Han L L, Yu D T, He J Z. Research methods for soil viral ecology (In Chinese)[J]. Acta Ecologica Sinica, 2017, 37(6): 1749-1756. [韩丽丽, 于丹婷, 贺纪正. 土壤病毒生态学研究方法[J]. 生态学报, 2017, 37(6): 1749-1756.]
(  0) 0) |
| [11] |
Williamson K E, Wommack K E, Radosevich M. Sampling natural viral communities from soil for culture-independent analyses[J]. Applied and Environmental Microbiology, 2003, 69(11): 6628-6633. DOI:10.1128/AEM.69.11.6628-6633.2003
(  0) 0) |
| [12] |
Shendure J, Ji H. Next-generation DNA sequencing[J]. Nature Biotechnology, 2008, 26(10): 1135-1145. DOI:10.1038/nbt1486
(  0) 0) |
| [13] |
He J Z, Yuan C L, Shen J P, et al. Methods for and progress in research on soil metagenomics (In Chinese)[J]. Acta Pedologica Sinica, 2012, 49(1): 155-164. [贺纪正, 袁超磊, 沈菊培, 等. 土壤宏基因组学研究方法与进展[J]. 土壤学报, 2012, 49(1): 155-164.]
(  0) 0) |
| [14] |
van Dijk E L, Jaszczyszyn Y, Naquin D, et al. The third revolution in sequencing technology[J]. Trends in Genetics, 2018, 34(9): 666-681. DOI:10.1016/j.tig.2018.05.008
(  0) 0) |
| [15] |
Ren J, Ahlgren N A, Lu Y Y, et al. VirFinder: A novel k-mer based tool for identifying viral sequences from assembled metagenomic data[J]. Microbiome, 2017, 5: 69. DOI:10.1186/s40168-017-0283-5
(  0) 0) |
| [16] |
Fouts D E. Phage_Finder: Automated identification and classification of prophage regions in complete bacterial genome sequences[J]. Nucleic Acids Research, 2006, 34(20): 5839-5851. DOI:10.1093/nar/gkl732
(  0) 0) |
| [17] |
Lima-Mendez G, van Helden J, Toussaint A, et al. Prophinder: A computational tool for prophage prediction in prokaryotic genomes[J]. Bioinformatics, 2008, 24(6): 863-865. DOI:10.1093/bioinformatics/btn043
(  0) 0) |
| [18] |
Arndt D, Grant J R, Marcu A, et al. PHASTER: A better, faster version of the PHAST phage search tool[J]. Nucleic Acids Research, 2016, 44(W1): W16-W21. DOI:10.1093/nar/gkw387
(  0) 0) |
| [19] |
Arndt D, Marcu A, Liang Y J, et al. PHAST, PHASTER and PHASTEST: Tools for finding prophage in bacterial genomes[J]. Briefings in Bioinformatics, 2019, 20(4): 1560-1567. DOI:10.1093/bib/bbx121
(  0) 0) |
| [20] |
Roux S, Enault F, Hurwitz B L, et al. VirSorter: Mining viral signal from microbial genomic data[J]. PeerJ, 2015, 3: e985. DOI:10.7717/peerj.985
(  0) 0) |
| [21] |
Zheng T T, Li J, Ni Y Q, et al. Mining, analyzing, and integrating viral signals from metagenomic data[J]. Microbiome, 2019, 7: 42. DOI:10.1186/s40168-019-0657-y
(  0) 0) |
| [22] |
McNair K, Aziz R K, Pusch G D, et al. Phage genome annotation using the RAST pipeline . New York: Springer New York, 2017: 231-238.
(  0) 0) |
| [23] |
El-Gebali S, Mistry J, Bateman A, et al. The Pfam protein families database in 2019[J]. Nucleic Acids Research, 2019, 47(D1): D427-D432. DOI:10.1093/nar/gky995
(  0) 0) |
| [24] |
Triant D A, Pearson W R. Most partial domains in proteins are alignment and annotation artifacts[J]. Genome Biology, 2015, 16: 99. DOI:10.1186/s13059-015-0656-7
(  0) 0) |
| [25] |
Kanehisa M, Furumichi M, Tanabe M, et al. KEGG: New perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092
(  0) 0) |
| [26] |
Galperin M Y, Makarova K S, Wolf Y I, et al. Expanded microbial genome coverage and improved protein family annotation in the COG database[J]. Nucleic Acids Research, 2015, 43(D1): D261-D269. DOI:10.1093/nar/gku1223
(  0) 0) |
| [27] |
Liu J, Glazko G, Mushegian A. Protein repertoire of double-stranded DNA bacteriophages[J]. Virus Research, 2006, 117(1): 68-80. DOI:10.1016/j.virusres.2006.01.015
(  0) 0) |
| [28] |
Huang L, Zhang H, Wu P Z, et al. DbCAN-seq: A database of carbohydrate-active enzyme(CAZyme) sequence and annotation[J]. Nucleic Acids Research, 2018, 46(D1): D516-D521. DOI:10.1093/nar/gkx894
(  0) 0) |
| [29] |
Alcock B P, Raphenya A R, Lau T T Y, et al. CARD 2020:Antibiotic resistome surveillance with the comprehensive antibiotic resistance database[J]. Nucleic Acids Research, 2020, 48(D1): D517-D525.
(  0) 0) |
| [30] |
Ye M, Sun M M, Huang D, et al. A review of bacteriophage therapy for pathogenic bacteria inactivation in the soil environment[J]. Environment International, 2019, 129: 488-496. DOI:10.1016/j.envint.2019.05.062
(  0) 0) |
| [31] |
Su J F, Liu J J, Yu H, et al. Isolation and whole genome sequencing of a novel lytic bacteriophage RS-PⅡ-1 infecting Ralstonia solanacearum (In Chinese)[J]. Chinese Journal of Virology, 2017, 33(3): 441-449. [苏靖芳, 刘俊杰, 于浩, 等. 一株烟草青枯雷尔氏菌烈性噬菌体RS-PⅡ-1的分离及全基因组分析[J]. 病毒学报, 2017, 33(3): 441-449.]
(  0) 0) |
| [32] |
van Eck N J, Waltman L. Citation-based clustering of publications using CitNetExplorer and VOSviewer[J]. Scientometrics, 2017, 111(2): 1053-1070. DOI:10.1007/s11192-017-2300-7
(  0) 0) |
| [33] |
Hock L, Gillis A, Mahillon J. Complete genome sequence of bacteriophage Deep-Purple, a novel member of the family Siphoviridae infecting Bacillus cereus[J]. Archives of Virology, 2018, 163(9): 2555-2559. DOI:10.1007/s00705-018-3865-z
(  0) 0) |
| [34] |
Sutton T D S, Clooney A G, Ryan F J, et al. Choice of assembly software has a critical impact on virome characterisation[J]. Microbiome, 2019, 7: 12. DOI:10.1186/s40168-019-0626-5
(  0) 0) |
| [35] |
Huerta-Cepas J, Szklarczyk D, Heller D, et al. EggNOG 5.0:A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses[J]. Nucleic Acids Research, 2019, 47(D1): D309-D314. DOI:10.1093/nar/gky1085
(  0) 0) |
| [36] |
Jin M, Guo X, Zhang R, et al. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7: 58. DOI:10.1186/s40168-019-0675-9
(  0) 0) |
| [37] |
Bi L, Yu D T, Du S, et al. Diversity and potential biogeochemical impacts of viruses in bulk and rhizosphere soils[J]. Environmental Microbiology, 2020.
(  0) 0) |
| [38] |
Segobola J, Adriaenssens E, Tsekoa T, et al. Exploring viral diversity in a unique south African soil habitat[J]. Scientific Reports, 2018, 8: 111. DOI:10.1038/s41598-017-18461-0
(  0) 0) |
| [39] |
Liang X L, Wagner R E, Zhuang J, et al. Viral abundance and diversity vary with depth in a southeastern United States agricultural ultisol[J]. Soil Biology & Biochemistry, 2019, 137: 107546.
(  0) 0) |
| [40] |
Gao S M, Schippers A, Chen N, et al. Depth-related variability in viral communities in highly stratified sulfidic mine tailings[J]. Microbiome, 2020, 8: 89. DOI:10.1186/s40168-020-00848-3
(  0) 0) |
| [41] |
Daly R A, Roux S, Borton M A, et al. Viruses control dominant bacteria colonizing the terrestrial deep biosphere after hydraulic fracturing[J]. Nature Microbiology, 2019, 4(2): 352-361. DOI:10.1038/s41564-018-0312-6
(  0) 0) |
| [42] |
Bezuidt O, Lebre P H, Pierneef R A, et al. Phages actively challenge niche communities in Antarctic soils[J]. mSystems, 2020. DOI:10.1128/mSystems.00234-20
(  0) 0) |
| [43] |
Adriaenssens E M, van Zyl L, de Maayer P, et al. Metagenomic analysis of the viral community in Namib Desert hypoliths[J]. Environmental Microbiology, 2015, 17(2): 480-495. DOI:10.1111/1462-2920.12528
(  0) 0) |
| [44] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880. DOI:10.1038/s41564-018-0190-y
(  0) 0) |
| [45] |
Williamson K E, Fuhrmann J J, Wommack K E, et al. Viruses in soil ecosystems: An unknown quantity within an unexplored territory[J]. Annual Review of Virology, 2017, 4(1): 201-219. DOI:10.1146/annurev-virology-101416-041639
(  0) 0) |
| [46] |
Schulz F, Alteio L, Goudeau D, et al. Hidden diversity of soil giant viruses[J]. Nature Communications, 2018, 9: 4881. DOI:10.1038/s41467-018-07335-2
(  0) 0) |
| [47] |
Al-Shayeb B, Sachdeva R, Chen L X, et al. Clades of huge phages from across Earth's ecosystems[J]. Nature, 2020, 578(7795): 425-431. DOI:10.1038/s41586-020-2007-4
(  0) 0) |
 2021, Vol. 58
2021, Vol. 58


