2. 中国科学院遗传与发育生物学研究所植物基因组学国家重点实验室, 北京 100101;
3. 中国科学院-英国约翰英纳斯中心植物和微生物科学联合研究中心, 北京 100101;
4. 南京农业大学资源与环境科学学院, 江苏省固体有机废弃物资源化高技术研究重点实验室, 国家有机类肥料工程技术研究中心, 南京 210095
2. State Key Laboratory of Plant Genomics, Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101;
3. Chinese Academy of Sciences-UK John Innes Centre Joint Research Centre for Plant and Microbial Sciences, Beijing 100101;
4. College of Resources and Environmental Sciences, Jiangsu Provincial Key Lab for Organic Solid Waste Utilization, National Engineering Research Center for Organic-based Fertilizers, Nanjing Agricultural University, Nanjing 210095, China
粮食安全和环境保护是一个国家赖以生存与发展的根本。全球新的需求与挑战将国家粮食安全、生态环境健康等问题推到前所未有的战略高度。当前,中国既要将饭碗牢牢地端在自己手中,又要大力推进生态文明建设,就必须依靠科技,大幅度提高作物产量的同时,保护生态环境,促进农业绿色发展。化肥作为粮食增产的关键保障,对农业生产做出了巨大贡献。我国化肥年生产和消费量已超过6 000万t,占全球三分之一多。化肥的大量施用虽然支撑了国家粮食安全,但也引起了土壤酸化、水体富营养化等一系列环境问题[1-4]。如何协同粮食安全、资源高效和环境保护已成为我国乃至全球农业可持续发展面临的重大挑战,也是未来农业科技革命的突破口和多学科交叉创新的科学前沿。
作为植物营养研究的核心目标,养分高效同时可以协同作物产量、资源效率和环境安全,成为破解可持续发展难题的金钥匙。近年来,国际上植物营养学科发展迅速,尤其在植物营养分子生物学、微生物组学等方面取得了一系列重要突破。我国科学家在植物养分高效分子机理、植物-微生物互作、根际微生态调控、根际营养与农田养分管理等方面取得重要进展,使我国在该领域跃居国际先进水平。本文系统总结了植物营养相关领域的新进展,剖析了协调资源、环境和粮食安全的学术思路,提出了交叉创新研究领域-根际生命共同体的学术观点、理论框架、关键科学问题和未来研究重点,以期引发思考与讨论,推动相关学科发展。
1 植物营养研究的主要进展与突破 1.1 植物养分高效生物学机制研究进展与突破随着生物学新理论与新技术的不断涌现,尤其是分子生物学的快速发展,植物营养基础研究全面进入分子时代。传统研究无法破译的植物营养机理被逐步阐明,如转运蛋白介导的植物养分吸收与运输机制、植物感知养分信号途径与调控等[5-8]。我国科学家在植物氮、磷、钾营养信号转导与分子调控机理研究上做出了一系列开创性工作[9-11]。不同于欧美科学家专注于模式植物拟南芥的基础性研究,我国科学家坚持围绕作物开展基础与应用研究,克隆了多个具有重要育种价值的关键基因,如NRT1.1B、GRF4、OsNR2、ARE1、NGR5、NRT2.3b、OsTCP19,并阐明了其调控机制[12-18]。在水稻氮营养信号通路与氮高效遗传改良方面,克隆了籼稻中氮高效基因NRT1.1B,发现该基因单碱基变异是导致籼粳稻间氮肥利用效率差异的主要原因,并将籼型NRT1.1B导入粳稻,大幅度提高了作物氮利用效率[12];克隆了NRT1.1A和Ef-cd养分高效基因,可在显著缩短作物生育期的情况下,大幅提高作物产量和氮肥利用效率,使培育兼具氮高效、高产与早熟作物品种成为可能[15, 19];阐明了硝酸盐信号从细胞膜受体感知到细胞核内响应的主信号通路,揭示了硝酸盐信号驱动植物氮磷营养平衡的分子机制[11]。研究成果不仅为遗传改良作物养分高效性状提供了创新思路,也为养分高效品种培育提供了优异的基因与种质资源。然而,尽管在作物自身改良提高养分效率的理论和应用研究方面取得了长足进步,但缺乏对优良品种与根际微生物和土壤互作以及养分调控的全局性理解,限制了高效植物基因型生物学潜力的发挥。
1.2 根际营养研究主要进展与挑战:从根际过程到根际生物互作 1.2.1 根际过程与养分高效养分的高效利用不仅取决于植物本身,而且在很大程度上也取决于其所处的根际环境。自20世纪80年代美国科学家Barber[20-21]提出土壤养分生物有效性概念以来,根系的养分活化作用得到了广泛重视;德国植物营养学家Marschner[22]首次揭示植物养分高效的根际作用机理,掀起了根际营养研究的热潮;以养分高效吸收转运的分子机理和理想根构型为代表的关键性突破[23-25],使植物营养研究进入了新阶段。我国科学家在专一性根分泌物活化根际养分[26-27]、菌根菌丝拓展根际范围[28]、菌丝际生物互作促进养分活化利用[29-30]等方面取得了创新性进展。尤其是深入生产一线,发现禾本科与豆科作物间作使豆科作物结瘤固氮能力提高近2倍,认识到玉米吸氮多解除蚕豆“氮阻遏”、玉米分泌的染料木素促进蚕豆结瘤固氮,从而首次揭示了间套作体系根际互作增产和氮磷养分高效利用的机理[31-32];证明根分泌物可在土壤中形成增强难溶性养分活化的特殊微域,并能改善根际土壤结构等物理环境,增强根-土的连接性和养分的传输,拓展了根系-根际-养分互作的空间维度[27, 33];发现根际局部养分调控可以进一步强化根际互作,促进根系增生和根际酸化,从而改变根系的趋肥性和竞争策略[34-35];提出可持续作物系统需要适应异质性根际的学术观点,阐述了根际科学与可持续农业发展的关系,提出根际物理、化学和生物互作过程及其调控机制的概念模型[36];利用菌根真菌双重培养体系,结合13C-DNA-SIP示踪植物光合碳流技术,阐明菌丝际互作活化土壤难溶性磷的机理:发现菌丝分泌物能快速被菌丝际解磷细菌利用,同时作为碳源和信号刺激菌丝际解磷细菌的解磷功能,增强土壤有机磷活化[30];阐明根系形态、根分泌物、菌根之间养分获取策略的协同和权衡机制,发现粗根植物更依赖于菌根或根分泌物,而细根植物更依赖于根系形态变化[37]。尽管我国在根际营养研究上取得显著成就,但这些研究主要集中在专一性根分泌物和根际化学过程的养分活化机理方面,对数量巨大的非专一性根分泌物的多样化功能、以及植物-土壤系统多界面互作增效的潜力仍知之甚少,成为进一步破解养分高效难题的瓶颈。
1.2.2 根际微生物组与养分高效根际富集了大量各种各样的微生物,这些微生物作为一个整体统称为根际微生物组[38]。根际微生物组是植物功能发挥不可或缺的组成部分,且基因数量远大于植物基因组,被称为植物的第二基因组。多年来,在根瘤菌、菌根真菌、解磷细菌提高养分效率的作用与机理研究方面取得了一系列新突破[39-42],但这些微生物只是根际微生物组中的很小一部分[43-45]。近年来,微生物组测序、高通量培养、人工重组和功能研究体系等方法的建立极大地推动了根际微生物组功能研究[46-47]。拟南芥、玉米、水稻、小麦等植物根际微生物组构成被解析[43, 48-50]。根际微生物组及菌群互作关系促进植物生长、适应逆境、维持植物健康以及帮助植物高效获取养分的机理也被逐步揭示[51-53]。例如,香豆素参与调控假单胞菌在拟南芥根系定殖,促进植物对难溶性铁的吸收利用[54]。根际微生物组不仅受土壤类型、养分供应等因素的影响,而且还受植物基因型的调控[43-44, 55-56]。拟南芥三萜合成途径显著调控根际微生物组组成,52%的拟南芥根系特异细菌受三萜合成基因调控。通过分离培养的细菌与化合物的共培养试验,揭示了三萜化合物直接并选择性调控根际细菌生长,表明三萜化合物参与植物与根际微生物组的共进化[57]。籼稻根系较粳稻富集了更多与土壤氮循环相关的细菌,表明籼稻根系微环境的氮循环更活跃,发现籼稻型NRT1.1B基因在促进氮循环相关细菌在水稻根系富集、提高有机氮利用方面起重要作用[57]。因此,植物与其第二基因组的融合为揭示根际生物互作、实现养分高效提供了新的途径。然而,植物养分高效的根际微生物组功能研究才刚刚起步,参与养分高效的核心微生物及其养分高效的机理、核心微生物与其他微生物互作机制、核心微生物与植物互作机制等仍知之甚少,限制了植物与其第二基因组-微生物组互作增效潜力的发挥。
2 根际生命共同体的理论框架与学术思路 2.1 根际生命共同体理论的提出养分高效是一个从土壤到植物、从微生物到农业生产多界面互作的系统过程。任何单一过程的研究都无法破解整个系统的运行机制,也就难以从整体上大幅度提高植物养分效率。根系作为植物吸收养分的主要器官,把地上和地下连为一体;根际则是控制植物-土壤系统物质、能量流动和信息交换的枢纽;菌丝际及其他微生物极大地拓展了植物吸收利用土壤养分的范围与功能。在“植物-根系-根际-菌丝际-土体及其微生物”系统中,植物将光合产物输送到地下,驱动了地下多样化生命过程的运转。光合产物一部分用于支撑根系生长及其生理活动,另一部分以根分泌物的形式释放到根际环境中,极大地改变了根际土壤的物理、化学和生物学性质,招募和供养微生物,使根际成为植物-微生物、微生物-微生物互作最活跃的热点区域,成倍提升植物对土壤养分的活化与利用效率。根外菌丝和其他土壤微生物又将根际范围扩展上百倍,并能从距离根系很远的土体、以及根系难于进入的微团聚体内将养分捕获并传输给植物。这种自上而下的能量流与自下而上的养分流形成一个完整的闭环系统,将“植物-根系-根际-菌丝际-土体及其微生物”连接成一个以根际互作为核心,“牵一发而动全身”的根际生命共同体(Rhizobiont)(图 1),将植物与其第二基因组--微生物组完美地结合在一起,控制着植物对养分的活化、吸收和利用。
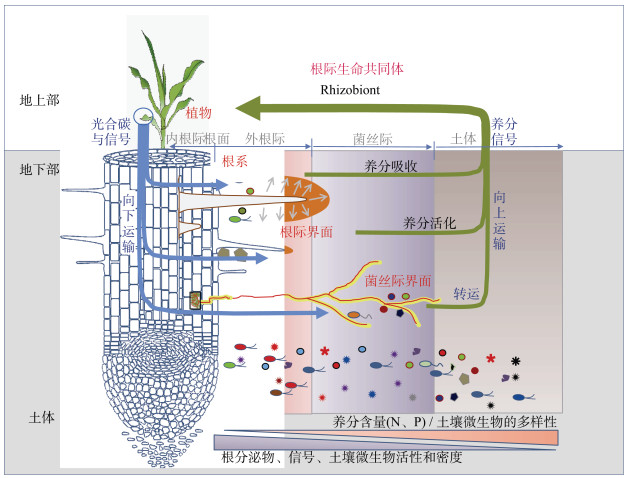
|
图 1 根际生命共同体结构与运行机制模式图 Fig. 1 Model diagram of the structure and operation mechanism of rhizobiont |
长期以来,化肥显著的增产效应,使人们在生产实践中往往只重视肥料养分的投入,却忽视了植物-土壤系统巨大的生物学潜力,尤其是没有认识到根际生命共同体多界面互作增效提高养分效率的作用,因此常常施肥过量,导致根系生长受阻、根际过程减弱、植物-微生物互作紊乱、根际微生态环境恶化,从而严重制约了养分效率的提高。因此,深入揭示根际生命共同体互作增效机制,建立生物学调控新途径,提高养分效率,从根本上改变高投入、高污染的农业生产方式,既是国内外农业科技创新的交叉前沿,又是破解我国农业绿色转型的关键突破口。
2.2 根际生命共同体的学术思路通过植物营养学、土壤学、微生物学、农学、生态学、分子生物学和生物信息学等多学科交叉创新,改变以往过度依赖外部投入的传统思路,提出根际生命共同体理论模型与学术思路,从系统角度阐明共同体运行机制,破解多界面互作提高养分效率的科学难题。首先,通过碳和养分的流动轨迹,确定根际生命共同体的整体框架和内部脉络;通过定量各个界面碳流与养分流的通量,揭示分泌物驱动的界面互作控制物质流动的规律及碳与养分效率的耦合机制,解析共同体系统活化、吸收与利用养分的功能;通过比较植物基因型、匹配核心微生物、及调控关键环境因子对系统物质和能量流动及其信号交换的影响,解析共同体运行与调控机制,建立理论模式。其次,针对植物-微生物互作界面,筛选鉴定养分高效的根际核心微生物,阐明植物与核心微生物相互感知、微生物定殖与调控植物养分效率的机制;针对微生物-微生物互作界面,发掘增强核心微生物定殖和功能的专性物质、靶向抑制有害微生物的有益微生物,揭示微生物-微生物协同和竞争影响养分效率的机制。在以上两个关键界面互作机制取得突破的基础上,阐明从微生物菌群互作到核心微生物-植物互作的级联放大作用及其撬动多界面互作增效的联动机制,全面提升共同体系统功能与运行效率。最后,在共同体理论创新的基础上,从养分高效植物基因型、与其匹配的根际核心微生物、营造理想根际微生态环境最大化植物-核心微生物互作潜能的新技术三个视角协同突破根际互作调控瓶颈问题;再从地下根际互作调控、地上高产群体构建和农田养分管理三个方面集成创建根际生命共同体互作增效的生物学调控新途径,从根本上提高养分效率,推动农业绿色发展。
3 根际生命共同体的研究内容与主要创新提高养分效率,从根本上改变高投入、高污染的农业生产方式,是我国农业绿色转型的迫切需求。以往研究多关注单一过程的解剖,无法从整体上解决植物-土壤系统,尤其是以根际为核心的多组分、多界面的互作增效问题,难以从整体上提高养分效率。围绕“根际互作与养分高效”这一研究主题,通过多学科交叉创新,聚焦根际生命共同体(图 2)系统研究,阐明根际生命共同体结构、功能、及其在养分活化、吸收与利用中的作用,重点突破植物-微生物、微生物-微生物关键界面互作机制,阐明根际生命共同体运行规律,创建养分高效的生物学调控途径,从根本上提高养分效率,支撑我国农业绿色转型。

|
图 2 根际生命共同体研究思路及四大研究内容 Fig. 2 Research pathways and four research contents of rhizobiont |
根际互作过程在养分高效中发挥关键作用。当前研究主要针对单一根际过程,无法破解整个系统的运行机制,难以从整体上提高养分效率。构建“植物-根系-根际-菌丝际-土体及其微生物”根际生命共同体理论模式,研究共同体多界面碳流与养分效率的耦合机制,从系统角度深入揭示根际生命共同体关键界面的互作过程与对话机制。主要包括:(1)构建养分高效的根际生命共同体模式;(2)阐明植物根分泌物介导的养分高效根际对话机制;(3)阐明菌丝分泌物介导的菌丝际菌根真菌-细菌互作机制及效应。
3.2 根际微生物对植物养分高效的作用机制植物根系从土壤选择性招募并供养大量微生物,根际微生物组与作物养分高效密切相关。然而,当前研究主要关注根瘤菌和菌根真菌为代表的少数微生物种类,对参与作物养分高效的其他大量微生物种类与功能缺乏系统性研究,它们与植物的互作机理更不清楚。未来应着重聚焦植物与根际微生物互作过程,筛选鉴定养分高效的根际核心微生物,揭示微生物定殖、调控植物养分利用通路的机制,以及植物感知与调控微生物的机理,阐明植物与根际微生物协同互作提高养分效率的机理,为理解根际生命共同体互作增效机制、创建生物学调控途径提供理论支撑。主要包括:(1)植物养分高效的根际核心微生物种类和功能;(2)根际微生物调控植物吸收养分的机制;(3)植物对根际微生物的感知和调控机理。
3.3 养分高效的根际微生态调控机制根际微生态对养分高效至关重要。高投入的农业生产方式造成了根际微生态失衡,削弱了根际菌群促进养分周转、植物生长和抑制土传病害的功能,制约着作物养分高效。当前根际菌群与养分高效的研究主要集中在促生微生物资源筛选、产品开发及应用阶段,忽视了根际微生态与养分高效关系的系统研究。未来应围绕根际生命共同体中微生物-微生物以及微生物-环境生态互作,阐明根际微生态与养分高效的定量关系,建立根际微生态定向调控途径。主要包括:(1)阐明典型根际菌群失衡特征与装配机制以及与作物养分效率的关系;(2)探明根际有益菌群多营养级生态互作与作物养分效率的关系;(3)建立根际微生态定向调控技术体系,实现作物促生、抑病与养分高效。
3.4 养分高效的地上地下互作调控机制调控根际生命共同体的核心是高效协同地上地下系统物质、能量流动和信息交换。在植株水平,冠根交流能够协同地上地下生理过程,调控植物生长发育与养分效率。在群体水平,植物-土壤系统地上地下紧密的互作反馈过程决定了整个体系的生产力与养分效率。通过生物学调控可以发掘地上地下互作增效潜力,在作物个体、群体与系统水平提高养分效率,实现可持续集约化农业生产。未来的研究应重点围绕养分高效的地上地下互作机制与调控途径开展研究,在植株水平上揭示地上地下高效协同的生理与分子机制;在土壤-作物系统水平上阐明地上地下生物多样性互作的增产增效机制;依据根际生命共同体理论及其互作增效机制新认识,建立养分高效的生物学调控途径。
4 未来交叉创新方向与展望综上所述,在当前植物营养学、微生物学、分子生物学、土壤学、农学、可持续发展科学等快速发展与交叉融合的大背景下,新的学科生长点不断涌现,根际生命共同体理论体系的构建是多学科交叉创新的典型案例。这一学术观点突破了以往研究多关注单一过程的解剖,无法从整体上解决植物-土壤系统,尤其是以根际为核心的多组分、多界面的互作增效问题。围绕“根际生命共同体与养分高效利用”这一焦点,开展以植物营养学为核心的多学科交叉创新,系统深入研究“植物-根系-根际-菌丝际-土体及其微生物”根际生命共同体的结构、功能、及其在养分活化、吸收与利用中的作用,无疑将为新时代植物营养学科的快速发展提供了新机遇。
未来根际生命共同体交叉创新研究方向主要集中在:
1)创建“植物-根系-根际-菌丝际-土体及其微生物”根际生命共同体理论体系。植物在生长过程中将光合碳分配到地下,驱动“植物-根系-根际-菌丝际-土体及其微生物”根际生命共同体运行。自上而下的碳流和自下而上的养分流之间的关系与耦合机制是什么?植物又是如何通过分泌物及信号物质来调控这一系统过程?通过融合多学科理论与技术,有望突破根际生命共同体的结构、功能及其物质、能量和信息交换机制,阐明根际生命共同体的运行规律及多界面互作增效机制,破解传统单一过程研究无法系统提高养分效率难题,开创植物-土壤-微生物互作增效的交叉创新领域。
2)破解植物第一基因组与第二基因组(微生物组)协同提高养分效率的理论难题。植物-微生物、微生物-微生物互作在植物养分高效中发挥着重要作用。哪些核心微生物参与植物养分高效,如何与植物交换养分,怎样影响植物吸收利用养分通路?植物如何感知和调控这些根际核心微生物?同时,根际微生物-微生物竞争与协同互作如何影响养分效率,如何实现定向调控?回答这些科学问题,有望阐明植物-微生物信号感知、核心微生物根际定殖、以及与植物互作增效的分子机制,揭示根际微生物-微生物协同与竞争影响养分效率的机制,并将植物-微生物互作从当前定性描述提升到功能解析和系统定量水平。
3)建立根际生命共同体互作增效提高养分效率的生物学调控途径。在根际生命共同体理论创新基础上,从植物基因型、根际核心微生物和根际微生态定向调控三个视角协同突破根际互作调控瓶颈问题,再从地下根际互作调控、地上高产群体构建和农田养分管理三个方面集成创建根际生命共同体互作增效的生物学调控新途径,大幅度提高作物养分利用效率。
全球农业发展面临巨大挑战,过度依赖外部化学品大量投入难以持续[58]。创新根际生命共同体界面互作增效理论与生物学调控途径,以根际互作为支点,撬动整个植物-土壤系统养分效率的提升,达到“四两拨千斤”的效果,从根本上实现粮食安全、资源高效和环境保护多目标协同,将为我国农业绿色转型、乃至解决全球可持续发展难题提供新路径。
| [1] |
Jiao X Q, Lyu Y, Wu X B, et al. Grain production versus resource and environmental costs: Towards increasing sustainability of nutrient use in China[J]. Journal of Experimental Botany, 2016, 67(17): 4935-4949. DOI:10.1093/jxb/erw282
(  0) 0) |
| [2] |
Cui Z L, Zhang H Y, Chen X P, et al. Pursuing sustainable productivity with millions of smallholder farmers[J]. Nature, 2018, 555(7696): 363-366. DOI:10.1038/nature25785
(  0) 0) |
| [3] |
Shen J B, Zhang F S, Siddique K H M. Sustainable resource use in enhancing agricultural development in China[J]. Engineering, 2018, 4(5): 588-589. DOI:10.1016/j.eng.2018.08.007
(  0) 0) |
| [4] |
Chen X P, Cui Z L, Fan M S, et al. Producing more grain with lower environmental costs[J]. Nature, 2014, 514(7523): 486-489. DOI:10.1038/nature13609
(  0) 0) |
| [5] |
Ho A Y Y, Day D A, Brown M H, et al. Arabidopsis phospholipase Dδ as an initiator of cytoskeleton-mediated signalling to fundamental cellular processes[J]. Functional Plant Biology, 2009, 36: 190-198. DOI:10.1071/FP08222
(  0) 0) |
| [6] |
Yamaji N, Xia J X, Mitani-Ueno N, et al. Preferential delivery of zinc to developing tissues in rice is mediated by P-type heavy metal ATPase OsHMA2[J]. Plant Physiology, 2013, 162(2): 927-939. DOI:10.1104/pp.113.216564
(  0) 0) |
| [7] |
Wild R, Gerasimaite R, Jung J Y, et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains[J]. Science, 2016, 352(6288): 986-990. DOI:10.1126/science.aad9858
(  0) 0) |
| [8] |
Liu K H, Niu Y J, Konishi M, et al. Discovery of nitrate-CPK-NLP signalling in central nutrient-growth networks[J]. Nature, 2017, 545(7654): 311-316. DOI:10.1038/nature22077
(  0) 0) |
| [9] |
Xu J, Li H, Chen L, et al. A protein kinase, interacting with two calcineurin B-like proteins, regulates K+ transporter AKT1 in Arabidopsis[J]. Cell, 2006, 125: 1347-1360. DOI:10.1016/j.cell.2006.06.011
(  0) 0) |
| [10] |
Lv Q, Zhong Y J, Wang Y G, et al. SPX4 negatively regulates phosphate signaling and homeostasis through its interaction with PHR2 in rice[J]. Plant Cell, 2014, 26(4): 1586-1597. DOI:10.1105/tpc.114.123208
(  0) 0) |
| [11] |
Hu B, Jiang Z M, Wang W, et al. Nitrate-NRT1.1B-SPX4 cascade integrates nitrogen and phosphorus signalling networks in plants[J]. Nature Plants, 2019, 5(4): 401-413. DOI:10.1038/s41477-019-0384-1
(  0) 0) |
| [12] |
Hu B, Wang W, Ou S J, et al. Variation in NRT1.1B contributes to nitrate-use divergence between rice subspecies[J]. Nature Genetics, 2015, 47(7): 834-838. DOI:10.1038/ng.3337
(  0) 0) |
| [13] |
Fan X R, Tang Z, Tan Y W, et al. Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(26): 7118-7123. DOI:10.1073/pnas.1525184113
(  0) 0) |
| [14] |
Li S, Tian Y H, Wu K, et al. Modulating plant growth-metabolism coordination for sustainable agriculture[J]. Nature, 2018, 560(7720): 595-600. DOI:10.1038/s41586-018-0415-5
(  0) 0) |
| [15] |
Wang W, Hu B, Yuan D Y, et al. Expression of the nitrate transporter gene OsNRT1.1A/OsNPF6.3 confers high yield and early maturation in rice[J]. Plant Cell, 2018, 30(3): 638-651. DOI:10.1105/tpc.17.00809
(  0) 0) |
| [16] |
Gao Z Y, Wang Y F, Chen G, et al. The indica nitrate reductase gene OsNR2 allele enhances rice yield potential and nitrogen use efficiency[J]. Nature Communications, 2019, 10(1): 5207. DOI:10.1038/s41467-019-13110-8
(  0) 0) |
| [17] |
Wu K, Wang S, Song W, et al. Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice[J]. Science, 2020, 367(6478): eaaz2046. DOI:10.1126/science.aaz2046
(  0) 0) |
| [18] |
Liu Y Q, Wang H R, Jiang Z M, et al. Genomic basis of geographical adaptation to soil nitrogen in rice[J]. Nature, 2021.
(  0) 0) |
| [19] |
Fang J, Zhang F T, Wang H R, et al. Ef-cd locus shortens rice maturity duration without yield penalty[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(37): 18717-18722. DOI:10.1073/pnas.1815030116
(  0) 0) |
| [20] |
Barber S A. Soil nutrient bioavailability: A mechanistic approach . New York: John Wiley and Son, 1984.
(  0) 0) |
| [21] |
Barber S A. Soil Nutrient Bioavailability: A Mechanistic Approach . 2nd edition[M]. New York: John Wiley and Son, 1995.
(  0) 0) |
| [22] |
Marschner H. Preface to first edition[M]//Mineral nutrition of higher plants. Amsterdam: Elsevier, 1995.
(  0) 0) |
| [23] |
Ma J F, Tamai K, Yamaji N, et al. A silicon transporter in rice[J]. Nature, 2006, 440(7084): 688-691. DOI:10.1038/nature04590
(  0) 0) |
| [24] |
Ma J F, Yamaji N, Mitani N, et al. An efflux transporter of silicon in rice[J]. Nature, 2007, 448(7150): 209-212. DOI:10.1038/nature05964
(  0) 0) |
| [25] |
Lynch J P. Steep, cheap and deep: An ideotype to optimize water and N acquisition by maize root systems[J]. Annals of Botany, 2013, 112(2): 347-357. DOI:10.1093/aob/mcs293
(  0) 0) |
| [26] |
Zhang F S, Romheld V, Marschner H. Release of zinc mobilizing root exudates in different plant species as affected by zinc nutritional status[J]. Journal of Plant Nutrition, 1991, 14(7): 675-686. DOI:10.1080/01904169109364234
(  0) 0) |
| [27] |
Zhang D S, Zhang C C, Tang X Y, et al. Increased soil phosphorus availability induced by Faba bean root exudation stimulates root growth and phosphorus uptake in neighbouring maize[J]. New Phytologist, 2016, 209(2): 823-831. DOI:10.1111/nph.13613
(  0) 0) |
| [28] |
Li X L, George E, Marschner H. Phosphorus depletion and pH decrease at the root-soil and hyphae-soil interfaces of VA mycorrhizal white clover fertilized with ammonium[J]. New Phytologist, 1991, 119(3): 397-404. DOI:10.1111/j.1469-8137.1991.tb00039.x
(  0) 0) |
| [29] |
Zhang L, Xu M G, Liu Y, et al. Carbon and phosphorus exchange may enable cooperation between an arbuscular mycorrhizal fungus and a phosphate-solubilizing bacterium[J]. New Phytologist, 2016, 210(3): 1022-1032. DOI:10.1111/nph.13838
(  0) 0) |
| [30] |
Zhang L, Feng G, Declerck S. Signal beyond nutrient, fructose, exuded by an arbuscular mycorrhizal fungus triggers phytate mineralization by a phosphate solubilizing bacterium[J]. The ISME Journal, 2018, 12(10): 2339-2351. DOI:10.1038/s41396-018-0171-4
(  0) 0) |
| [31] |
Li L, Li S, Sun J, et al. Diversity enhances agricultural productivity via rhizosphere phosphorus facilitation on phosphorus-deficient soils[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(27): 11192-11196. DOI:10.1073/pnas.0704591104
(  0) 0) |
| [32] |
Li B, Li Y, Wu H, et al. Root exudates drive interspecific facilitation by enhancing nodulation and N2 fixation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(23): 6496-6501. DOI:10.1073/pnas.1523580113
(  0) 0) |
| [33] |
Jin K M, White P J, Whalley W R, et al. Shaping an optimal soil by root-soil interaction[J]. Trends in Plant Science, 2017, 22(10): 823-829. DOI:10.1016/j.tplants.2017.07.008
(  0) 0) |
| [34] |
Shen J B, Yuan L X, Zhang J L, et al. Phosphorus dynamics: From soil to plant[J]. Plant Physiology, 2011, 156(3): 997-1005. DOI:10.1104/pp.111.175232
(  0) 0) |
| [35] |
Zhang D S, Lyu Y, Li H B, et al. Neighbouring plants modify maize root foraging for phosphorus: Coupling nutrients and neighbours for improved nutrient-use efficiency[J]. New Phytologist, 2020, 226(1): 244-253. DOI:10.1111/nph.16206
(  0) 0) |
| [36] |
Wang X, Whalley W R, Miller A J, et al. Sustainable cropping requires adaptation to a heterogeneous rhizosphere[J]. Trends in Plant Science, 2020, 25(12): 1194-1202. DOI:10.1016/j.tplants.2020.07.006
(  0) 0) |
| [37] |
Wen Z H, Li H B, Shen Q, et al. Tradeoffs among root morphology, exudation and mycorrhizal symbioses for phosphorus-acquisition strategies of 16 crop species[J]. New Phytologist, 2019, 223(2): 882-895. DOI:10.1111/nph.15833
(  0) 0) |
| [38] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health[J]. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001
(  0) 0) |
| [39] |
Oldroyd G E D, Murray J D, Poole P S, et al. The rules of engagement in the legume-rhizobial symbiosis[J]. Annual Review of Genetics, 2011, 45: 119-144. DOI:10.1146/annurev-genet-110410-132549
(  0) 0) |
| [40] |
Hacquard S, Spaepen S, Garrido-Oter R, et al. Interplay between innate immunity and the plant microbiota[J]. Annual Review of Phytopathology, 2017, 55: 565-589. DOI:10.1146/annurev-phyto-080516-035623
(  0) 0) |
| [41] |
Martin F M, Uroz S, Barker D G. Ancestral alliances: Plant mutualistic symbioses with fungi and bacteria[J]. Science, 2017, 356(6340): eaad4501. DOI:10.1126/science.aad4501
(  0) 0) |
| [42] |
Oldroyd G E D, Leyser O. A plant's diet, surviving in a variable nutrient environment[J]. Science, 2020, 368(6486): eaba0196. DOI:10.1126/science.aba0196
(  0) 0) |
| [43] |
Bulgarelli D, Rott M, Schlaeppi K, et al. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota[J]. Nature, 2012, 488(7409): 91-95. DOI:10.1038/nature11336
(  0) 0) |
| [44] |
Lundberg D S, Lebeis S L, Paredes S H, et al. Defining the core Arabidopsis thaliana root microbiome[J]. Nature, 2012, 488(7409): 86-90. DOI:10.1038/nature11237
(  0) 0) |
| [45] |
Müller D B, Vogel C, Bai Y, et al. The plant microbiota: Systems-level insights and perspectives[J]. Annual Review of Genetics, 2016, 50: 211-234. DOI:10.1146/annurev-genet-120215-034952
(  0) 0) |
| [46] |
Bai Y, Müller D B, Srinivas G, et al. Functional overlap of the Arabidopsis leaf and root microbiota[J]. Nature, 2015, 528(7582): 364-369. DOI:10.1038/nature16192
(  0) 0) |
| [47] |
Li M, Wei Z, Wang J N, et al. Facilitation promotes invasions in plant-associated microbial communities[J]. Ecology Letters, 2019, 22(1): 149-158. DOI:10.1111/ele.13177
(  0) 0) |
| [48] |
Peiffer J A, Spor A, Koren O, et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(16): 6548-6553. DOI:10.1073/pnas.1302837110
(  0) 0) |
| [49] |
Edwards J, Johnson C, Santos-Medellín C, et al. Structure, variation, and assembly of the root-associated microbiomes of rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8): E911-E920. DOI:10.1073/pnas.1414592112
(  0) 0) |
| [50] |
Chen S M, Waghmode T R, Sun R B, et al. Root-associated microbiomes of wheat under the combined effect of plant development and nitrogen fertilization[J]. Microbiome, 2019, 7(1): 136. DOI:10.1186/s40168-019-0750-2
(  0) 0) |
| [51] |
Hiruma K, Gerlach N, Sacristán S, et al. Root endophyte Colletotrichum tofieldiae confers plant fitness benefits that are phosphate status dependent[J]. Cell, 2016, 165(2): 464-474. DOI:10.1016/j.cell.2016.02.028
(  0) 0) |
| [52] |
Wang G Z, Schultz P, Tipton A, et al. Soil microbiome mediates positive plant diversity-productivity relationships in late successional grassland species[J]. Ecology Letters, 2019, 22(8): 1221-1232.
(  0) 0) |
| [53] |
Zhang C L, Zhang Y M, Ding Z J, et al. Contribution of microbial inter-kingdom balance to plant health[J]. Molecular Plant, 2019, 12(2): 148-149. DOI:10.1016/j.molp.2019.01.016
(  0) 0) |
| [54] |
Voges M J E E E, Bai Y, Schulze-Lefert P, et al. Plant-derived coumarins shape the composition of an Arabidopsis synthetic root microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(25): 12558-12565. DOI:10.1073/pnas.1820691116
(  0) 0) |
| [55] |
Castrillo G, Teixeira P J P L, Paredes S H, et al. Root microbiota drive direct integration of phosphate stress and immunity[J]. Nature, 2017, 543(7646): 513-518. DOI:10.1038/nature21417
(  0) 0) |
| [56] |
Zhang J Y, Liu Y X, Zhang N, et al. NRT1.1B is associated with root microbiota composition and nitrogen use in field-grown rice[J]. Nature Biotechnology, 2019, 37(6): 676-684. DOI:10.1038/s41587-019-0104-4
(  0) 0) |
| [57] |
Huang A, Jiang T, Liu Y, et al. A specialized metabolic network selectively modulates Arabidopsis root microbiota[J]. Science, 2019, 364(6640): eaau6389.
(  0) 0) |
| [58] |
Zhang W F, Cao G X, Li X L, et al. Closing yield gaps in China by empowering smallholder farmers[J]. Nature, 2016, 537(7622): 671-674. DOI:10.1038/nature19368
(  0) 0) |
 2021, Vol. 58
2021, Vol. 58


