土壤有机复合污染是亟需解决的重要环境问题之一[1-2]。生物修复是解决有机复合污染土壤的一种绿色、高效、低成本的修复方法,具有广阔的应用前景[3-4]。土壤微生物具有丰富的功能多样性,不仅是元素生物地球化学循环过程的引擎,而且是土壤污染物消纳的净化器和核心资源[5]。近年来,在Nature、Science等期刊上已发表多篇论文,从全球和局域尺度研究土壤微生物对陆地生物地球化学循环过程的驱动机制[6-7]、污染土壤中微生物资源分布模式及功能多样性[8-9],阐明土壤微生物对陆地生态系统元素周转、污染物转化过程中的贡献。在实际污染土壤中,有机复合污染物降解由微生物菌群(微生物组)介导,通过群落个体间代谢互补性、互养作用完成污染物的去除[10-11]。因此,基于微生物组的原位修复已成为国际土壤污染与生物修复领域的研究热点与趋势。
1 国内外研究现状及趋势近年来,污染土壤微生物修复研究关注焦点逐渐从微生物个体向微生物群体、从单一微生物种群向跨物种互作、从宏基因组学向宏表型组学、从单一污染物转化过程向交叉互补与协同污染物转化网络过渡,从而多维度揭示微生物组协同修复污染土壤的作用机制[12-13]。目前,在土壤单一有机污染物微生物组降解方面研究已日益深入[11-14],而有机复合污染土壤中微生物组对复合污染物的响应和演替规律、共代谢过程和代谢机制、修复型合成微生物组构建及原位耦合强化修复机制等基础理论和方法学探索也逐渐成为国内外研究前沿和热点。
1.1 有机复合污染土壤微生物群落生态网络结构与适应机制解析有机复合污染土壤微生物群落的生态网络结构与适应机制是微生物组修复的基础。土壤中有机污染物的去除是一个长期且复杂的过程。土壤微生物类群多样,适应有害环境的微生物会自然富集,并降解有机污染物。国内外已针对单一有机污染物对土壤微生物群落结构的影响展开了广泛的研究。如在多环芳烃、多氯联苯等有机污染物胁迫下,土壤微生物群落组装、演替规律、响应过程及生理代谢会受到显著影响,进而降低土壤微生态体系的稳定性和功能性[15-17]。然而,在实际污染土壤中,复合污染物在多介质界面间存在复杂的相互作用,使得对土壤微生物组结构、功能及互作生态网络产生更为复杂的协同影响效应[18]。自2005年,国内期刊发表研究论文,研究者采用稀释平板培养法、变性梯度凝胶电泳(DGGE)、磷脂脂肪酸(PLFA)分析等方法,发现多氯联苯复合污染、重金属复合污染显著改变土壤微生物群落结构及遗传多样性[19-20]。随着高通量测序技术的发展,研究者能够更精细地刻画复合污染状态下微生物群落组装和演替特性,以解析微生物在共代谢污染物过程中的功能[21]。
土壤微生物组的活性和功能不仅受到时空和环境梯度的限制[22],而且取决于复杂的微生物菌群网络结构。微生物分子生态网络是微生物群落结构的重要特征,且是国际微生物生态领域的研究前沿。已有研究[12-23]表明,土壤中微生物通过协同互作、互养等形式相互通讯,形成特定的微生物群落间生态互作模式、稳定空间排布,以共代谢污染物。在解析微生物群落网络通讯、互作的过程中,可进一步筛选出对外界环境梯度或特定生态功能有响应的核心微生物类群(keystone taxa),其在微生物组群落构建、维持种群生态功能中可能发挥关键作用[14]。网络生态模型、稳定性同位素示踪、多组学等先进手段方法的不断涌现,使得分析微生物共存网络模式、关键微生物类群与环境修复功能关联性成为可能,为多层次深入研究污染土壤中微生物的适应机制提供有力的方法支撑[14-24]。近年来,研究者结合共发生网络分析(co-occurrence networks)、典范对应分析(canonical correspondence analysis)等生物信息学分析方法,对多环芳烃、多氯联苯等污染土壤中微生物群落生物地理分布特征及微生物生态网络特征进行了系统深入分析,且多为国内研究者工作[14-25]。但是,此方面研究多针对单一污染物存在的环境,仅少量报道研究重金属复合、有机-重金属复合污染土壤中微生物群落的共发生生态网络及核心关键物种的生态功能。在砷、锑复合污染农田土壤中,研究者鉴定参与砷、锑生物地球化学氧化还原反应的关键微生物类群[26],并发现相比锑而言,砷对微生物群落的影响更强[27],并且砷转化基因丰度与锑组分含量之间存在显著相关性[28]。厘清有机复合污染土壤中微生物群落生态网络结构与适应机制,可为优选高效修复手段、定位修复靶点提供理论基础。近年来的国际研究方向,也逐渐转向研究不同修复措施处理后土壤中微生物群落网络拓扑特性及核心关键物种的功能[29]。
1.2 土壤中有机复合污染物的微生物组转化过程与分子机制深刻认识土壤中有机复合污染物的微生物组转化过程与分子机制是微生物组修复的关键。有机污染物的微生物降解过程复杂,一般需要多步反应才能实现完全矿化。污染土壤中有机污染物的转化过程大多是由微生物群落介导的,而并非单个菌株独立完成[22-30]。不同微生物对有机污染物的代谢具有多样性,而土壤微生物群落通常会自组装形成特定的代谢功能网络(如通过形成生物膜、群体感应效应等),以实现对有机污染物的高效、协同降解[13, 31]。研究[32]发现,通过对4株Bacillus spp.(芽孢杆菌属)菌株构建微生物组进行石油降解实验,发现Bacillus spp.属内亲缘性近的菌株之间对石油降解主要表现为协同促进作用;然而,并非所有降解菌株的组合均会提高对有机污染物的降解效率。比如,由Bacillus sp. S2和Bacillus sp. S3构成的微生物组F4对烷烃和芳烃的降解率分别较单菌降解率降低了55.3%和39.0%,表现出同菌属微生物种间对石油降解的拮抗抑制作用。此外,降解菌株和其他功能的辅助菌株之间的互养关系也可显著提高降解效率[33]。新加坡国立大学Nagarajan和Loh[34]发现恶臭假单胞菌(Pseudomonas putida F1)和施氏假单胞菌(Pseudomonas stutzeri OX1)能分别降解乙基苯和二甲苯,通过构建两者的混菌体系可实现对混合苯系化合物的高效降解。分析、量化微生物之间的相互作用有助于加深对复合有机污染物降解机理的认识。
污染物的降解效率不仅取决于微生物间的互作关系,也受共存于环境中其他污染物的影响。研究表明,不同污染物的降解过程是互相关联的,结构相似的复合污染物能够竞争性结合控制降解代谢途径的调控蛋白,从而抑制微生物降解功能的发挥[35]。作为代谢互作网络中的重要节点,关键降解酶的组成、丰度、酶学性质以及产物对酶的反馈调节等直接决定了微生物对污染物的降解能力与代谢途径[36-37]。前人在降解酶基因的发掘、鉴定、表达方面已有相关研究基础,但是目前对蛋白水平上降解酶行使生物学功能的分子机制认识不足。近年来,快速发展的宏基因组、宏转录组及宏代谢组等组学技术,结合新兴的基于代谢模型的计算机模拟技术,可以追踪和定量预测土壤微生物信号分子、代谢物的交换过程[38],为深入理解原位复杂环境中微生物群落对有机复合污染物的共代谢过程、代谢互作机制提供了可能。稳定性同位素核酸探针技术DNA-SIP(DNA-based stable isotope probing)联合扩增子测序技术与宏组学技术,已经成为研究微生物群落结构与功能相结合的最有力的工具,前者能够将自然环境中的微生物与其功能直接相联系,清晰地识别出复杂环境中的功能微生物;后者则能同时获得微生物的物种分类和功能基因分布信息,在发掘污染物降解基因方面具有较大的优势。目前国内外研究者已利用这两种技术预测污染环境中菌株间的互作关系,进而揭示微生物对单一污染物的协同降解机制。研究[11]发现,具有降解功能的菌株与其他菌株之间的协同代谢(代谢物质交换)显著提高了除草剂阿特拉津的降解效率,揭示了阿特拉津降解菌群的组成、功能与相互作用关系。以双酚A降解菌群为研究模型,研究者综合利用宏基因组学、宏转录组学和目标代谢组学技术,通过纯菌分离验证,破译了菌群中微生物个体在双酚A降解不同时期中的作用及多个菌株之间的相互关系[13]。总体而言,现有研究仍然主要集中于纯培养条件下单一降解菌的代谢途径与降解机制。尽管目前关于微生物菌群高效降解有机污染物的研究日益引起关注,但关于菌群内的共代谢过程及机制尚处于起步阶段,仅有的少数研究一般局限于人工构建的简单微生物菌群。目前人们对实际有机复合污染土壤中微生物群落对污染物的共代谢过程、不同菌株间的代谢互作关系、关键酶的催化机制的认识更是十分匮乏,尚需进一步研究。
1.3 高效降解合成微生物组的设计与构建高效降解合成微生物组(synthetic microbial communities,SynComs)的设计与构建是实现有机复合污染土壤微生物组修复的重要手段。合成微生物组是基于合成生物学方法,以代谢通路模块化为核心特征,构建具有特定功能的菌群,从而实现多个菌株的分工与合作,在生物技术、环境修复等多领域具有广阔前景[33, 39-40]。合成微生物组基于自下而上的“网络设计-组学构建-功能验证-参数学习循环”策略,其设计和构建主要分为三个阶段:首先,针对研究目的,运用合成生物学的方法设计模块化代谢通路;其次,为每一个代谢模块匹配具有相应功能的菌株;最后,建立人工合成的多菌株共存体系,并实现对微生物代谢网络的精确调控,如合成特定化合物、生产生物燃料、协助植物抗病、降解有机污染物[11, 39-41]。与单一菌株体系相比,合成微生物组具有可定义性/模块化、高效性、鲁棒性(相互作用的微生物对于外界环境的扰动具有一定的弹性及抗性)、可预测性、可伸缩性、稳定性等特征[39]。明确菌群中微生物菌株之间的交互作用关系(如共生、竞争、捕食等)及代谢功能网络,是实现多菌株代谢分工合作、构建合成微生物体系的基础。例如,群体感应效应(quorum sensing,QS)是交链微生物生态系统中细胞间交流的重要机制,涉及信号分子(多肽、小分子和天然产物等)的产生和感应。微生物菌株之间通过分泌、扩散和交换上述信号分子,判断菌群密度和周围环境变化,当菌群数达到一定的阈值后,启动相应一系列基因的调节表达,激活细胞信号级联应答,以在群落水平上调节微生物群体行为[42]。在污染土壤中,研究者基于已知土壤中微生物群落结构、生态网络及功能等信息定位关键控制节点,通过种群动态模拟、污染物转化代谢模型、空间模型等对合成微生物体系的降解功能微生物群落进行生态建模,设计并预测合成菌群的代谢途径网络和相互作用,从而构建具有特定环境修复功能的微生物群落[43]。虽然研究者已认识到合成微生物组在污染环境修复领域中具有巨大的潜力[10, 43],但是国内外应用合成微生物组修复污染土壤方面的报道仍然非常有限[11]。Romero等[44]采用可培养和宏基因组方法,明确尾矿修复植物根际微生物群落特征和互作特性,构建不同组合的合成细菌菌群,并研究其促进植物生长、提高根际修复效率的作用机制。Xu等[11]首次基于污染土壤的微生物菌群动态,通过多菌株代谢模拟,从头合成能高效降解阿特拉津的微生物“队伍”,阐明合成微生物种群之间协同去除土壤中阿特拉津的机制。可控的实验室模拟生态系统可用来验证合成微生物组效果,并结合机器学习算法、代谢通量分析、生态系统建模等计算方法以进一步明确影响合成微生物组互作和功能的主要驱动因素,从而反馈优化合成微生物组设计。
由于环境中存在大量难培养微生物,因此,仅依赖于传统微生物富集培养法筛选获得的降解微生物资源十分有限[45]。为了高效获得丰富、有降解活性的合成微生物菌种库资源,磁性纳米颗粒分选、微流控芯片微滴技术、kChip等表型筛选方法不断涌现,具有高效、快速、灵敏和准确等特点[46-48]。磁性纳米颗粒具有较好的吸附性能和超顺磁性,因此可作为可控性生物标记物,从原油污染土壤、水体等环境样品中有效分离具有特定降解功能的微生物[49-50]。磁性纳米颗粒分选具有不依赖微生物培养、直接从环境样品中原位分离出目标微生物、分离过程温和而不影响微生物活性及功能等优点[49]。微流控芯片微滴技术是在微米尺度下,对生物样本或者环境样本进行精确、高度均一的操控(制备、混匀、融合、分割、孵育、检测、分选等),从而由复杂体系中筛选目标物,具有超高通量、高灵敏度、定量准确等特点,为合成生物学自动化、定向、高通量筛选提供了重要的方法学基础[51]。Jiang等[47]利用微流控高通量单细胞培养板从多环芳烃污染土壤中分离出多株高效降解微生物菌株。然而,目前关于高通量筛选降解菌群的研究尚处于起步阶段,未来研究应将该技术与人工合成微生物组相结合,应用于有机污染土壤的生物修复研究中。
1.4 有机复合污染土壤合成微生物组原位修复过程、调控原理及生态安全性评价阐明合成微生物组原位修复过程与调控原理是发展有机复合污染土壤微生物组原位修复方法的理论依据。原位条件下,污染物的微生物转化过程受水文地质条件、土壤理化性质等多因素的制约,修复效率难以预测。因此,提高原位生物修复效率及稳定性是污染土壤生物修复领域亟须解决的关键问题。相比单一菌株修复方法,合成微生物组以微生物群体的代谢协同性及功能互补性为切入点[52],对复合污染土壤环境具有更强适应性、高效性、模块化、可控性、稳定性等特点[43, 48, 53]。目前关于合成微生物组对土壤污染修复研究方面尚处于实验室阶段,对合成微生物组的原位修复效果、生态网络作用原理及定向调控机制研究较少。发展原位耦合强化生物修复方法,提高微生物转化效率,成为定向调控原位修复污染土壤的绿色、高效和可持续治理的新策略。关于微生物修复强化研究目前主要针对单一污染物,如通过调控碳、氮、硫、磷等生源要素物质循环,协同植物根际微生物组,调节环境条件(如土壤水分、温度、通透性)等方法[4, 54]。植物根际圈是污染物生物地球化学循环和微生物代谢的重要界面微环境。植物根部可通过根际分泌物、挥发性物质通讯等方式,募集并组装特定的根际功能菌群,增加微生物功能冗余、增强种间互作、重叠基因表达等方式以响应污染等环境扰动,维持污染条件下植物和土壤系统的功能性及稳定性[54-55]。因此,根际微生物组已成为土壤污染生物修复领域的国际研究前沿,而国内此方面研究与国际水平差距较大。研究者采用柳树、杨树等木本植物作为修复载体,外源接种降解功能内生细菌或转基因菌株,有效促进原位污染土壤中多环芳烃、三氯乙烯的去除[56-58]。在模式修复植物柳树的研究中,宏基因组、宏转录组等研究结果显示植物通过根际分泌物改变根际微生物组组成、群落丰度及转录模式(相互作用组,interactome),从而促进修复过程[59-60]。Thomas等[54]采用稳定性同位素标记和宏基因组方法,揭示在污染土壤中种植修复植物对根际不可培养型菲降解微生物组的影响,揭示污染土壤中根际微生物组降解污染物的复杂功能互作过程。未来研究亦需针对复合污染土壤情况,拓展、加深合成微生物组修复与多调控方法的耦合、协同强化机制等方面的研究。
此外,系统研究生物修复技术的生态安全性及关键影响因子,是科学评估污染土壤修复技术绿色可持续性的重要环节。当多种污染物复合存在时,会对土壤微生态造成更为复杂的生态效应。近年来,生态风险评估效应研究已逐渐从物种水平转向更为全面、系统的种群和生态系统水平分析[61]。当引入的外源微生物具有高适合度并能在生态系统中扩散时,易于造成土著种群更替并产生复杂生态效应,如影响污染物转化和营养循环过程。相比实验室条件下,原位自然环境中环境参数复杂多变,如气候因素、水文地质条件、土壤理化性质、微生物因素等,上述差异通常使得研究者难以预测外源微生物菌剂的添加对生态系统结构及功能的影响[3]。因此,探究原位生物修复过程中外源微生物组引入对土著微生物区系(数量、多样性、群落结构)和生态系统多功能性的影响并辨析关键影响因子[4],是生物修复技术生态安全性评估的重要组成部分[62]。相比单纯的基于风险评估的方法,近年来研究者逐渐青睐采用基于修复过程中全生命周期的可持续评价策略[63-64]。该方法在满足地块环境功能、使用功能和风险控制的基础上,综合地评价环境成本与收益,从而优化修复方案并使修复净效益最大化[65-66]。因此,未来研究需要构建有机复合污染土壤合成微生物组修复过程的生命周期评估框架程序,为该技术的生态安全性评估及修复方案优化提供科学依据。
综上所述,当前土壤污染物微生物转化与修复方法研究主要集中于微生物类群对单一污染物的响应、适应机制及微生物的单一转化途径等方面,而针对土壤中有机复合污染物的微生物组转化机制与调控原理等领域研究基础较薄弱。未来研究应加强以具有代谢协同性及功能互补性的微生物组为切入点,通过宏基因组、宏转录组、稳定性同位素分子探针、纳米二次离子质谱技术、计算机模拟(菌群代谢模型、化学计量矩阵)等先进手段,系统深入研究有机复合污染土壤中微生物组对复合污染物的响应和演替规律,解析土壤微生物组对复合污染物的共代谢过程和分子机制,构建并设计修复型合成微生物组,揭示合成微生物组原位修复过程及耦合强化修复机制,构建污染土壤合成微生物组修复过程的生态安全性评估体系,为发展复合污染土壤绿色可持续原位生物修复技术提供科学依据。
2 研究展望针对我国重点区域污染土壤中复合污染突出、修复效率不高、调控缺乏科学基础等问题,以我国重点区域(长江经济带、京津冀等场地污染土壤;珠三角、黄河流域、华北典型污灌区等农田污染土壤)典型有机复合污染土壤为研究对象,选择典型有机复合污染物(场地污染物:多环芳烃-氯代烃、多环芳烃-多氯联苯等;农田污染物:多环芳烃-多氯联苯、农药-邻苯二甲酸酯、有机氯农药-多环芳烃等)为对象,建议围绕有机复合污染土壤微生物群落适应机制-微生物组转化过程-合成微生物组设计构建-原位微生物组修复开展系统研究(图 1),应重点开展以下研究:
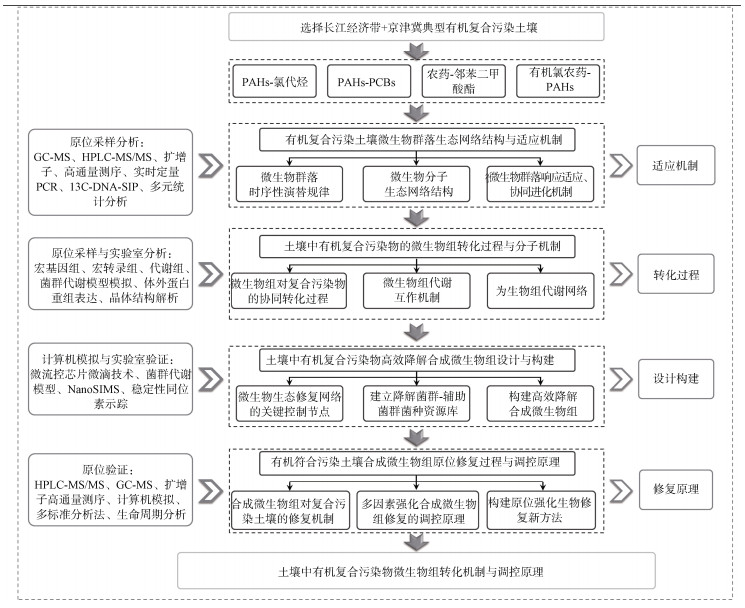
|
注:PAHs:多环芳烃;PCBs:多氯联苯;GC-MS:气相色谱-质谱联用法;HPLC-MS/MS:高效液相色谱-串联质谱法;DNA-SIP:稳定性同位素核酸探针技术;NanoSIMS:纳米二次离子质谱技术。 Note: PAHs: Polycyclic aromatic hydrocarbons; PCBs: Polychlorinated biphenyls; GC-MS: Gas chromatography-mass spectrometer; HPLC-MS/MS: Liquid chromatography-mass spectrometry; DNA-SIP: DNA Stable-isotope probing; NanoSIMS: Nano-scale secondary ion mass spectrometry. 图 1 土壤中有机复合污染物微生物组转化机制与调控原理研究框架体系 Fig. 1 Research framework of microbiome-mediated transformation mechanism and regulation principle of organic pollutants in co-contaminated soils |
(1)研究典型区有机复合污染土壤微生物群落组成、结构与多样性,探明不同类型有机复合污染土壤微生物群落时序性演替规律;研究有机复合污染土壤中微生物分子生态网络结构及拓扑特性,鉴定微生物群落互作中关键微生物类群,并明确主要环境影响因子;识别适应于有机复合污染土壤的指示菌种,阐明不同功能微生物对不同有机复合污染物的响应适应与协同进化机制。
(2)研究土壤中有机复合污染物微生物降解代谢的功能基因、关键酶及代谢途径,探明土壤微生物组对复合污染物的共代谢过程;构建有机复合污染土壤微生物群落代谢模型,分析微生物类群间物质交流、能量交换、信息通讯,揭示菌群代谢互作机制;解析关键降解酶的酶学性质、底物识别与催化机制,阐明关键酶行使降解转化功能的分子机制,重构有机复合污染物的高效转化途径,揭示其微生物组代谢网络。
(3)针对有机复合污染土壤的土壤条件和污染物类型,基于复合污染土壤中微生物分子网络结构及污染物转化的原理,明确微生物生态修复网络的关键控制节点;定向富集、分离污染土壤中高效降解菌、核心功能微生物,建立降解菌群-辅助菌群菌种资源库;结合基因组-关键酶-代谢特性网络关系设计并构建降解性合成微生物组;研究合成微生物组对有机复合污染物的降解特性和转化机制,优化微生物组结构,构建有机复合污染物高效降解合成微生物组。
(4)针对不同类型典型复合污染土壤污染特征及微生物特性,研究合成微生物组对土壤界面有机复合污染物原位转化生态网络关键控制节点的定向调控过程,明确合成微生物组参与污染物转化过程的主要影响因素,揭示合成微生物组对土壤中复合污染物的修复机制;阐明不同调控措施(环境条件、生源要素及根际强化等)对合成微生物组修复的协同强化机制,评估合成微生物组强化修复方法的稳定性和修复效率,建立污染土壤合成微生物组修复过程的生态安全性评估体系,构建复合污染土壤原位强化生物修复新方法。
基于上述关键科学问题,未来研究应着重阐明土壤中有机复合污染物的微生物组转化机制与调控原理,阐明不同复合污染条件下微生物群落对复合污染物的响应适应、协同进化机制,明确土壤中有机复合污染物的微生物组转化过程与分子机制,设计并构建针对有机复合污染物的修复型合成微生物组,揭示多因素强化合成微生物组修复的调控原理,构建有机复合污染土壤的微生物组修复理论,为土壤有机复合污染过程研究与生物修复技术发展提供新理论和新原理。
| [1] |
Liu Z P, Liu S J. Development of bioremediation in China-A review (In Chinese)[J]. Chinese Journal of Biotechnology, 2015, 31(6): 901-916. [刘志培, 刘双江. 我国污染土壤生物修复技术的发展及现状[J]. 生物工程学报, 2015, 31(6): 901-916.]
(  0) 0) |
| [2] |
Luo Y M, Teng Y. Regional difference in soil pollution and strategy of soil zonal governance and remediation in China (In Chinese)[J]. Bulletin of Chinese Academy of Sciences, 2018, 33(2): 145-152. [骆永明, 滕应. 我国土壤污染的区域差异与分区治理修复策略[J]. 中国科学院院刊, 2018, 33(2): 145-152.]
(  0) 0) |
| [3] |
Atashgahi S, Sánchez-Andrea I, Heipieper H J, et al. Prospects for harnessing biocide resistance for bioremediation and detoxification[J]. Science, 2018, 360(6390): 743-746. DOI:10.1126/science.aar3778
(  0) 0) |
| [4] |
Davoodi S M, Miri S B, Taheran M, et al. Bioremediation of unconventional oil contaminated ecosystems under natural and assisted conditions: A review[J]. Environmental Science & Technology, 2020, 54(4): 2054-2067.
(  0) 0) |
| [5] |
Zhu Y G, Shen R F, He J Z, et al. China soil microbiome initiative: Progress and perspective (In Chinese)[J]. Bulletin of the Chinese Academy of Sciences, 2017, 32(6): 554-565. [朱永官, 沈仁芳, 贺纪正, 等. 中国土壤微生物组: 进展与展望[J]. 中国科学院院刊, 2017, 32(6): 554-565.]
(  0) 0) |
| [6] |
Bahram M, Hildebrand F, Forslund S K, et al. Structure and function of the global topsoil microbiome[J]. Nature, 2018, 560(7717): 233-237. DOI:10.1038/s41586-018-0386-6
(  0) 0) |
| [7] |
Crowther T W, van den Hoogen J, Wan J, et al. The global soil community and its influence on biogeochemistry[J]. Science, 2019, 365(6455): eaav0550. DOI:10.1126/science.aav0550
(  0) 0) |
| [8] |
Vila J, Tauler M, Grifoll M. Bacterial PAH degradation in marine and terrestrial habitats[J]. Current Opinion in Biotechnology, 2015, 33: 95-102. DOI:10.1016/j.copbio.2015.01.006
(  0) 0) |
| [9] |
Podar M, Gilmour C C, Brandt C C, et al. Global prevalence and distribution of genes and microorganisms involved in mercury methylation[J]. Science Advances, 2015, 1(9): e1500675. DOI:10.1126/sciadv.1500675
(  0) 0) |
| [10] |
Dvořák P, Nikel P I, Damborský J, et al. Bioremediation 3.0:Engineering pollutant-removing bacteria in the times of systemic biology[J]. Biotechnology Advances, 2017, 35(7): 845-866.
(  0) 0) |
| [11] |
Xu X H, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5
(  0) 0) |
| [12] |
Worrich A, Musat N, Harms H. Associational effects in the microbial neighborhood[J]. The ISME Journal, 2019, 13(9): 2143-2149. DOI:10.1038/s41396-019-0444-6
(  0) 0) |
| [13] |
Yu K, Yi S, Li B, et al. An integrated meta-omics approach reveals substrates involved in synergistic interactions in a bisphenol A(BPA)-degrading microbial community[J]. Microbiome, 2019, 7(1): 16. DOI:10.1186/s40168-019-0634-5
(  0) 0) |
| [14] |
Jiao S, Chen W M, Wei G H. Resilience and assemblage of soil microbiome in response to chemical contamination combined with plant growth[J]. Applied and Environmental Microbiology, 2019, 85(6): e02523-02518. DOI:10.1128/aem.02523-18
(  0) 0) |
| [15] |
Ren G D, Teng Y, Ren W J, et al. Pyrene dissipation potential varies with soil type and associated bacterial community changes[J]. Soil Biology and Biochemistry, 2016, 103: 71-85. DOI:10.1016/j.soilbio.2016.08.007
(  0) 0) |
| [16] |
Zenteno-Rojas A, Martínez-Romero E, Castañeda- Valbuena D, et al. Structure and diversity of native bacterial communities in soils contaminated with polychlorinated biphenyls[J]. AMB Express, 2020, 10(1): 124. DOI:10.1186/s13568-020-01058-8
(  0) 0) |
| [17] |
Jiang L F, Luo C L, Zhang D Y, et al. Biphenyl-metabolizing microbial community and a functional operon revealed in E-waste-contaminated soil[J]. Environmental Science & Technology, 2018, 52(15): 8558-8567.
(  0) 0) |
| [18] |
Teng Y, Luo Y M, Shen R F, et al. Research progress and perspective of the multi-medium interface process and regulation principle of pollutants in site soil-groundwater (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(6): 1333-1340. [滕应, 骆永明, 沈仁芳, 等. 场地土壤-地下水污染物多介质界面过程与调控研究进展与展望[J]. 土壤学报, 2020, 57(6): 1333-1340.]
(  0) 0) |
| [19] |
Teng Y, Xu L, Zou D X, et al. Changes of soil microbial structural diversity in polychlorinated biphenyls mixed polluted soils (In Chinese)[J]. Ecology and Environmnet, 2007(6): 1688-1693. DOI:10.3969/j.issn.1674-5906.2007.06.021 [滕应, 徐莉, 邹德勋, 等. 多氯联苯复合污染土壤的微生物群落结构多样性变化[J]. 生态环境, 2007(6): 1688-1693.]
(  0) 0) |
| [20] |
Li M Z, Liao Q, Dong Y P, et al. Effects of copper and sulfadiazine combined pollution on soil enzyme activity and metabolic function diversity of microbial dommunity (In Chinese)[J]. Soils, 2020, 52(5): 987-993. [李明珠, 廖强, 董远鹏, 等. 铜和磺胺嘧啶复合污染对土壤酶活性及微生物群落功能多样性的影响[J]. 土壤, 2020, 52(5): 987-993.]
(  0) 0) |
| [21] |
Wang J W, Liu T, Sun W L, et al. Bioavailable metal(loid)s and physicochemical features co-mediating microbial communities at combined metal(loid)pollution sites[J]. Chemosphere, 2020, 260: 127619. DOI:10.1016/j.chemosphere.2020.127619
(  0) 0) |
| [22] |
Jansson J K, Hofmockel K S. Soil microbiomes and climate change[J]. Nature Reviews. Microbiology, 2020, 18(1): 35-46. DOI:10.1038/s41579-019-0265-7
(  0) 0) |
| [23] |
Xun W B, Li W, Xiong W, et al. Diversity-triggered deterministic bacterial assembly constrains community functions[J]. Nature Communications, 2019, 10(1): 3833.
(  0) 0) |
| [24] |
Angulo M T, Moog C H, Liu Y Y. A theoretical framework for controlling complex microbial communities[J]. Nature Communications, 2019, 10(1): 1045. DOI:10.1038/s41467-019-08890-y
(  0) 0) |
| [25] |
Jiao S, Chen W M, Wei G H. Biogeography and ecological diversity patterns of rare and abundant bacteria in oil-contaminated soils[J]. Molecular Ecology, 2017, 26(19): 5305-5317.
(  0) 0) |
| [26] |
Sun W M, Xiao E Z, Xiao T F, et al. Response of soil microbial communities to elevated antimony and arsenic contamination indicates the relationship between the innate microbiota and contaminant fractions[J]. Environmental Science & Technology, 2017, 51(16): 9165-9175.
(  0) 0) |
| [27] |
Sun X X, Kong T L, Xu R, et al. Comparative characterization of microbial communities that inhabit arsenic-rich and antimony-rich contaminated sites: Responses to two different contamination conditions[J]. Environmental Pollution, 2020, 260: 114052. DOI:10.1016/j.envpol.2020.114052
(  0) 0) |
| [28] |
Xiao E Z, Krumins V, Xiao T F, et al. Depth-resolved microbial community analyses in two contrasting soil cores contaminated by antimony and arsenic[J]. Environmental Pollution, 2017, 221: 244-255.
(  0) 0) |
| [29] |
Cai X X, Yuan Y, Yu L P, et al. Biochar enhances bioelectrochemical remediation of pentachlorophenol-contaminated soils via long-distance electron transfer[J]. Journal of Hazardous Materials, 2020, 391: 122213. DOI:10.1016/j.jhazmat.2020.122213
(  0) 0) |
| [30] |
Louca S, Polz M F, Mazel F, et al. Function and functional redundancy in microbial systems[J]. Nature Ecology & Evolution, 2018, 2(6): 936-943.
(  0) 0) |
| [31] |
Niehaus L, Boland I, Liu M H, et al. Microbial coexistence through chemical-mediated interactions[J]. Nature Communications, 2019, 10(1): 2052.
(  0) 0) |
| [32] |
Wang J N, Shi Y Y, Zheng L Y, et al. Isolation and identification of petroleum degradation bacteria and interspecific interactions among four Bacillus strains (In Chinese)[J]. Chinese Journal of Environmental Science, 2015, 36(6): 2245-2251. [王佳楠, 石妍云, 郑力燕, 等. 石油降解菌的分离鉴定及4株芽胞杆菌种间效应[J]. 环境科学, 2015, 36(6): 2245-2251.]
(  0) 0) |
| [33] |
Kenny D J, Balskus E P. Engineering chemical interactions in microbial communities[J]. Chemical Society Reviews, 2018, 47(5): 1705-1729.
(  0) 0) |
| [34] |
Nagarajan K, Loh K C. Formulation of microbial cocktails for BTEX biodegradation[J]. Biodegradation, 2015, 26(1): 51-63.
(  0) 0) |
| [35] |
Busch A, Lacal J, Martos A, et al. Bacterial sensor kinase TodS interacts with agonistic and antagonistic signals[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(34): 13774-13779.
(  0) 0) |
| [36] |
Yang Z G, Jiang W K, Wang X H, et al. An amidase gene, ipaH, is responsible for the initial step in the iprodione degradation pathway of Paenarthrobacter sp. strain YJN-5[J]. Applied and Environmental Microbiology, 2018, 84(19): e01150-01118.
(  0) 0) |
| [37] |
Yang H X, Hu S L, Wang X, et al. Erratum for Yang et al., "Pigmentiphaga sp. strain D-2 uses a novel amidase to initiate the catabolism of the neonicotinoid insecticide acetamiprid"[J]. Applied and Environmental Microbiology, 2020, 86(12): e02425-02419.
(  0) 0) |
| [38] |
Crits-Christoph A, Diamond S, Butterfield C N, et al. Novel soil bacteria possess diverse genes for secondary metabolite biosynthesis[J]. Nature, 2018, 558(7710): 440-444.
(  0) 0) |
| [39] |
Zhu T, Wu B. Synthetic microbiome: When "synthetic biology" meets "microbiomics" (In Chinese)[J]. Chinese Science Bulletin, 2019, 64: 1791-1798. [朱彤, 吴边. 合成微生物组: 当"合成生物学"遇见"微生物组学"[J]. 科学通报, 2019, 64(17): 1791-1798.]
(  0) 0) |
| [40] |
Lawson C E, Harcombe W R, Hatzenpichler R, et al. Common principles and best practices for engineering microbiomes[J]. Nature Reviews. Microbiology, 2019, 17(12): 725-741.
(  0) 0) |
| [41] |
Feng J, Li R F, Zhang S S, et al. Bioretrosynthesis of functionalized N-heterocycles from glucose via one-pot tandem collaborations of designed microbes[J]. Advanced Science, 2020, 7(17): 2001188.
(  0) 0) |
| [42] |
Trivedi P, Leach J E, Tringe S G, et al. Plant-microbiome interactions: From community assembly to plant health[J]. Nature Reviews Microbiology, 2020, 18(11): 607-621.
(  0) 0) |
| [43] |
de Lorenzo V, Prather K L J, Chen G Q, et al. The power of synthetic biology for bioproduction, remediation and pollution control: The un's sustainable development goals will inevitably require the application of molecular biology and biotechnology on a global scale[J]. EMBO Reports, 2018, 19(4): e45658.
(  0) 0) |
| [44] |
Romero M, Gallego D, Blaz J, et al. Rhizosphere metagenomics of mine tailings colonizing plants: Assembling and selecting synthetic bacterial communities to enhance in situ bioremediation[J]. bioRxiv, 2019. DOI:10.1101/664805
(  0) 0) |
| [45] |
Mu D S, Liang Q Y, Wang X M, et al. Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing[J]. Microbiome, 2018, 6(1): 230. DOI:10.1186/s40168-018-0613-2/fulltext.html
(  0) 0) |
| [46] |
Zhang D Y, Berry J P, Zhu D, et al. Magnetic nanoparticle-mediated isolation of functional bacteria in a complex microbial community[J]. The ISME Journal, 2015, 9(3): 603-614.
(  0) 0) |
| [47] |
Jiang C Y, Dong L B, Zhao J K, et al. High-throughput single-cell cultivation on microfluidic streak plates[J]. Applied and Environmental Microbiology, 2016, 82(7): 2210-2218.
(  0) 0) |
| [48] |
Kehe J, Kulesa A, Ortiz A, et al. Massively parallel screening of synthetic microbial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(26): 12804-12809.
(  0) 0) |
| [49] |
Li J B, Luo C L, Zhang G, et al. Coupling magnetic-nanoparticle mediated isolation(MMI)and stable isotope probing(SIP)for identifying and isolating the active microbes involved in phenanthrene degradation in wastewater with higher resolution and accuracy[J]. Water Research, 2018, 144: 226-234.
(  0) 0) |
| [50] |
Wang X Z, Zhao X H, Li H B, et al. Separating and characterizing functional alkane degraders from crude-oil-contaminated sites via magnetic nanoparticle-mediated isolation[J]. Research in Microbiology, 2016, 167(9/10): 731-744.
(  0) 0) |
| [51] |
Ma F Q, Yang G Y. Ultra-high-throughput screening system based on droplet microfluidics and its applications in synthetic biology (In Chinese)[J]. Biotechnology Bulletin, 2017(1): 83-92. [马富强, 杨广宇. 基于液滴微流控技术的超高通量筛选体系及其在合成生物学中的应用[J]. 生物技术通报, 2017(1): 83-92.]
(  0) 0) |
| [52] |
Yin J, Ma A Z, Song M Y, et al. Research progress in synthetic microbial systems (In Chinese)[J]. Microbiology China, 2020, 47(2): 583-593. [尹珺, 马安周, 宋茂勇, 等. 合成微生物体系研究进展[J]. 微生物学通报, 2020, 47(2): 583-593.]
(  0) 0) |
| [53] |
Wang B B, Teng Y, Xu Y F, et al. Effect of mixed soil microbiomes on pyrene removal and the response of the soil microorganisms[J]. Science of the Total Environment, 2018, 640/641: 9-17.
(  0) 0) |
| [54] |
Thomas F, Corre E, Cébron A. Stable isotope probing and metagenomics highlight the effect of plants on uncultured phenanthrene-degrading bacterial consortium in polluted soil[J]. The ISME Journal, 2019, 13(7): 1814-1830.
(  0) 0) |
| [55] |
Gonzalez E, Pitre F E, Pagé A P, et al. Trees, fungi and bacteria: Tripartite metatranscriptomics of a root microbiome responding to soil contamination[J]. Microbiome, 2018, 6(1): 53.
(  0) 0) |
| [56] |
Khan Z, Roman D, Kintz T, et al. Degradation, phytoprotection and phytoremediation of phenanthrene by endophyte Pseudomonas putida, PD1[J]. Environmental Science & Technology, 2014, 48(20): 12221-12228.
(  0) 0) |
| [57] |
Doty S L, Freeman J L, Cohu C M, et al. Enhanced degradation of TCE on a superfund site using endophyte-assisted poplar tree phytoremediation[J]. Environmental Science & Technology, 2017, 51(17): 10050-10058.
(  0) 0) |
| [58] |
Legault E K, James C A, Stewart K, et al. A field trial of TCE phytoremediation by genetically modified poplars expressing cytochrome P4502E1[J]. Environmental Science & Technology, 2017, 51(11): 6090-6099.
(  0) 0) |
| [59] |
Yergeau E, Sanschagrin S, Maynard C, et al. Microbial expression profiles in the rhizosphere of willows depend on soil contamination[J]. The ISME Journal, 2014, 8(2): 344-358.
(  0) 0) |
| [60] |
Yergeau E, Tremblay J, Joly S, et al. Soil contamination alters the willow root and rhizosphere metatranscriptome and the root-rhizosphere interactome[J]. The ISME Journal, 2018, 12(3): 869-884.
(  0) 0) |
| [61] |
Wang K L, Miao F, Shi K, et al. Research advances in eco-toxicological diagnosis methods of soil pollution (In Chinese)[J]. Soils, 2019, 51(5): 854-863. [王开来, 苗峰, 史柯, 等. 土壤污染生态毒理诊断方法研究进展[J]. 土壤, 2019, 51(5): 854-863.]
(  0) 0) |
| [62] |
Fang H, Deng Y F, Ge Q Q, et al. Biodegradability and ecological safety assessment of Stenotrophomonas sp. DDT-1 in the DDT-contaminated soil[J]. Ecotoxicology and Environmental Safety, 2018, 158: 145-153.
(  0) 0) |
| [63] |
Lemming G, Hauschild M Z, Chambon J, et al. Environmental impacts of remediation of a trichloroethene-contaminated site: Life cycle assessment of remediation alternatives[J]. Environmental Science & Technology, 2010, 44(23): 9163-9169.
(  0) 0) |
| [64] |
Dong J Q, Zhang H Z, Lei Q S, et al. Review of LCA procedure and models for contaminated site remediation (In Chinese)[J]. Environmental Pollution & Control, 2016, 38(12): 89-95. [董璟琦, 张红振, 雷秋霜, 等. 污染场地修复生命周期评估程序与模型的研究进展[J]. 环境污染与防治, 2016, 38(12): 89-95.]
(  0) 0) |
| [65] |
Hou D Y, Li G H. Green and sustainable remediation of contaminated soil in China: Core elements and development direction (In Chinese)[J]. Environmental Protection, 2016, 44(20): 16-19. [侯德义, 李广贺. 污染土壤绿色可持续修复的内涵与发展方向分析[J]. 环境保护, 2016, 44(20): 16-19.]
(  0) 0) |
| [66] |
Hu X T, Zhu J X, Ding Q. Environmental life-cycle comparisons of two polychlorinated biphenyl remediation technologies: Incineration and base catalyzed decomposition[J]. Journal of Hazardous Materials, 2011, 191(1/2/3): 258-268.
(  0) 0) |
 2021, Vol. 58
2021, Vol. 58

