硝化作用是全球氮循环的中心环节,是微生物在有氧条件下将铵态氮(NH4+-N)转变为硝态氮(
硝化作用由氨氧化微生物和亚硝酸盐氧化微生物(NOB)共同催化完成。由于氨氧化是硝化作用第一步且是限速步骤,因此,氨氧化微生物成为全球氮循环的研究热点。氨氧化微生物主要包括氨氧化古菌(AOA)、细菌(AOB)和新发现的全程氨氧化细菌(Comammox)[3]。土壤中AOA数量通常高于AOB[4]。然而,AOA和AOB生理活性受土壤pH、氮的来源和浓度等环境因素的影响。其中,AOA倾向于贫营养环境生长,在酸性土壤中主导硝化过程[5],而AOB则更加适应铵态氮浓度较高的环境,如中性-碱性土壤[6]。Comammox可以进行一步硝化,挑战了传统分步硝化理论。已培养的Comammox具有贫营养生活方式[7],但最近Li等[8]发现Comammox在施用氮肥的农田土壤中也具有硝化活性,表明Comammox在氮循环中的生态作用仍需深入评估。
硝化抑制剂主要通过影响氨单加氧酶AMO活性、抑制硝化细菌呼吸、改变土壤微域环境或影响土壤氮矿化和固持过程等方式,抑制硝化作用[9]。硝化抑制剂与氮肥配施已被证明是一项现实有效的保氮减排措施。其中,双氰胺(DCD)的应用最为广泛,在较多田间试验中表现出显著抑制硝酸盐积累,并降低N2O排放的效果[10]。烯丙基硫脲(ATU)在众多液体培养基和污泥反应器研究中显示出优越的抑制细菌硝化的性能[11],表明其在调控AOB主导的土壤硝化过程中具有应用潜力。一些研究表明ATU可以显著抑制土壤AOB生长和硝酸盐积累[12-13],也有研究表明ATU不能有效抑制土壤硝化作用[14],以上结果差异可能与ATU施用量和土壤类型有关。张苗苗等[9]还指出ATU可能存在一定化学毒性不宜推广,但可能引发何种生态效应未见报道。因此,系统探究ATU在土壤中的有效抑制浓度和微生物机理,有利于明确ATU施用的生态环境后果。
本研究以DCD为参考,采用室内微宇宙培养法,评价ATU施用量对土壤氮素转化及N2O和CO2排放的影响,进一步结合分子生物学手段,明确不同土壤微生物类型对ATU的响应机制。该研究将加深碳氮循环微生物机理的理解,为指导农业生产和缓解气候变化提供理论依据。
1 材料与方法 1.1 供试土壤土壤取自安徽省巢湖市栏杆集镇(117°50'E,31°59'N),土壤类型为黄棕壤,种植作物为玉米,采集于2019年10月玉米收获后。采用五点交叉法进行采样,用土钻采集耕层0~20 cm土壤混合组成代表土样。将土样去除石块、植物残体,过2 mm筛并混合均匀,4 ℃保存待用。土壤基本理化性质为:pH(H2O)5.99;硝态氮9.20 mg·kg–1;铵态氮1.18 mg·kg–1;全碳7.41 g·kg–1;全氮0.75 g·kg–1;碳氮比9.92;含水量5.88%;最大持水量(SWHC)61.5%。
1.2 微宇宙培养培养设8个处理:(1)CK处理:以等体积去离子水代替其他底物加入土壤;(2)N处理:氮肥选用硫酸铵,以N 200 μg·g–1施入土壤,其他施氮处理的氮肥形式和用量与此相同;(3)1-ATU+N处理:ATU用量为施氮量的1%,按2 μg·g–1施入土壤;(4)5-ATU+N处理:ATU用量为施氮量的5%,按10 μg·g–1施入土壤;(5)10-ATU+N处理:ATU用量为施氮量的10%,按20 μg·g–1施入土壤;(6)15-ATU+N处理:ATU用量为施氮量的15%,按30 μg·g–1施入土壤;(7)20-ATU+N处理:ATU用量为施氮量的20%,按40 μg·g–1施入土壤;(8)10-DCD+N处理:DCD用量为施氮量的10%,按20 μg·g–1施入土壤。
具体实验操作[15]如下:称取5.29 g新鲜土壤(相当于5.0 g干土),均匀平铺于120 mL玻璃培养瓶瓶底,用微量注射器逐滴均匀向土壤中加入灭菌去离子水,调节土壤水分至最大持水量40%,用橡胶塞密封,于28℃恒温培养箱中,避光预培养3 d,以恢复土壤微生物活性。预培养结束后,将培养瓶取出,打开橡胶塞,更新培养瓶上部空气,用微量注射器逐滴均匀向土壤加入对应底物,使土壤水分达最大持水量60%,塞紧橡胶塞,重新置于28℃恒温培养箱,避光培养21 d。每个处理设置3个重复。
1.3 温室气体CO2、N2O和CH4的排放动态在0、7、14、21 d分别采集培养瓶上部气体,用气相色谱仪(Agilent7890)分别对CO2、N2O和CH4的浓度进行检测。采完气后,将培养瓶橡胶塞打开,更新瓶内空气,再塞紧橡胶塞,于28℃恒温培养箱中继续培养。每种气体的累积排放量以其周排放量之和表示。经检测,CH4排放量低于检测限,故仅以图表展示CO2和N2O动态排放情况。
N2O的增温潜势是100年时间尺度上单位质量CO2的298倍。由CO2和N2O的累积排放量,可计算综合温室效应(Global warming potentials,GWP),公式如下:
| ${\rm{GWP}} = {\rm{GW}}{{\rm{P}}_{{\rm{C}}{{\rm{O}}_2}}} + {\rm{GW}}{{\rm{P}}_{{{\rm{N}}_2}{\rm{O}}}} \times 298$ | (1) |
在7、14、21 d进行破坏性采样。从每个培养瓶中各取1.5 g土壤置于无菌离心管,保存于-20℃,以备后续分子生物学分析。再取出1.5 g于10 mL离心管中,以1:5(w/v)加入1 mol·L–1 KCl溶液,充分震荡并静置30 min,用玻璃电极测定土壤pH[16];剩余土壤(约3.8 g)以1:5(w/v)加入2 mol·L–1 KCl溶液,以200 r·min–1震荡60 min,过滤后将滤液经流动分析仪(Skalar,Inc.,Breda,Netherlands)测定土壤铵态氮和硝态氮含量。
1.5 土壤微生物DNA提取和实时荧光定量PCR监测利用FastDNA® Spin Kit for Soil(MP Biomedicals)试剂盒提取土壤微生物总DNA,并溶解于100 μL无菌水。通过Nano Drop® ND-2000微量紫外分光光度计,测定土壤总DNA浓度和纯度,确保大部分DNA样品OD260/OD280值在1.8~2.0之间以保证DNA质量。最后将DNA稀释10倍后,保存于–20℃。
实时荧光定量PCR的引物和反应条件,见表 1。PCR扩增反应体系为:10 μL的TB GreenTM Premix Ex TaqTM II(Takara),上、下游引物(20 μmol·L–1)各1.0 μL,1.0 μL稀释10倍的DNA模板,补充灭菌双蒸水至20 μL。采用无菌水代替DNA作为严格的阴性对照。定量PCR反应在CFX96 Optical Real-Time Detection System(Bio-Rad)上进行。获得扩增产物后,通过2.0%琼脂糖凝胶电泳检测PCR产物的特异性。
|
|
表 1 荧光实时定量PCR引物和反应条件 Table 1 Primers and conditions used in this study |
由上海派森诺生物科技股份有限公司(Shanghai Personal Biotechnology Co.,Ltd)对土壤样品中微生物16S rRNA基因V4区进行扩增(引物515F/907R)和建库,最后在Illumina NovaSeq PE250测序系统上进行双端测序。下机数据先根据barcode信息进行样本划分,去除barcode序列。再通过FLASH[17]软件进行双端合并(-m 15 -M 300 -x 0.2 -p 33 -t 6)。最后利用QIIME2[18]流程进行ASV(100%相似度聚类)分析,主要步骤为:使用dada2 denoise-single插件[19]进行质控和特征序列ASV提取(--p-trim-left 0 --p-trunc-len 350),获取ASVs丰度表;再将ASVs用classify-consensus-blast插件,按照默认参数[20],以silva138数据库[21]为参考进行物种注释。最后,在各分类水平统计样本的群落组成和各分类单元的相对丰度。相对丰度,以各样本中每个分类单元的ASV序列除以总序列条数的值表示。所测序列已提交至NCBI SRA(Sequence Read Archive),序列号PRJNA699396。
1.7 统计分析采用SPSS 16.0进行统计分析。采用单因素方差分析检验同一时刻不同处理样品间的差异显著性(P < 0.05)。采用Pearson双尾t检验分析土壤微生物指标与温室气体排放量以及土壤理化性质的相关性。
2 结果 2.1 土壤无机氮转化强度土壤硝化强度,以培养期内土壤
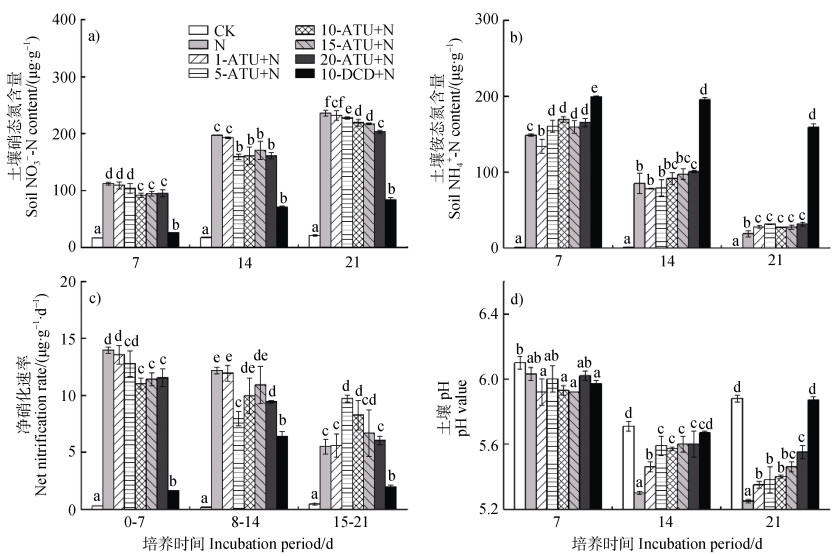
|
图 1 培养期间各处理土壤硝态氮含量(a)、铵态氮含量(b)、净硝化速率(c)以及pH(d)的变化 Fig. 1 Change of soil |
DCD和ATU均能通过抑制硝化,减缓土壤酸化。与CK相比,N处理显著降低了土壤pH,在14和21 d分别降低0.41和0.63。ATU和DCD在14和21 d土壤pH则均显著高于N处理。10-DCD+N处理在14和21 d土壤pH与CK处理无显著差异。
2.2 N2O和CO2通量变化及综合温室效应N2O和CO2累积排放量随培养时间延长而增加,但处理间存在明显差异(图 2)。N2O的排放情况,如图 2a所示。在21 d培养期内,CK处理N2O累积排放量最低,仅为0.04 µg·g–1。而单独施氮(N处理)强烈刺激了N2O排放,N2O累积排放量在所有处理中最高,达3.15 µg·g–1,较CK处理提高87.6倍。ATU与N配施显著抑制了N2O排放,但不同ATU用量间差异不显著。ATU+N处理中N2O累积排放量较N处理降低了60.3%~68.2%。与ATU+N相比,10-DCD+N处理对N2O抑制作用更为强烈,N2O累积排放量仅为N处理的6.7%。各处理CO2累积排放量(图 2b)均高于N2O,是N2O的118.7倍以上。培养21 d后,1-ATU+N与5-ATU+N处理CO2累积排放量显著低于N处理,其他施氮处理间无显著差异。

|
图 2 培养期间各处理N2O(a)和CO2(b)的排放动态 Fig. 2 Dynamics of greenhouse gas N2O(a) and CO2 emission(b) during the incubation |
各处理产生的综合温室效应(GWP),如表 2所示,总体而言,N > ATU+N > 10-DCD+N≈CK,且不同ATU施用量差异不显著。N处理产生的综合温室效应最高,约为1 314.4 µg·g–1(以CO2当量计),较CK处理增加227.9%,而ATU+N处理GWP较CK处理增加53.4%~81.9%。CK处理CO2排放对总GWP的贡献高达97.3%。单独施N促进N2O的排放,使N2O对GWP的贡献由2. 7%上升至71.5%。硝化抑制剂ATU降低了N2O的排放,从而使N2O对GWP的贡献下降到48.6%~55.5%,DCD使N2O的贡献降至更低水平(13.8%)。
|
|
表 2 培养期间CO2和N2O的累积排放量和温室效应 Table 2 The emission and global warming potential of CO2 and N2O during the incubation |
通过荧光定量PCR评估了土壤不同微生物类群对氮肥及硝化抑制剂的响应。研究表明土壤总体微生物16S rRNA基因拷贝数约为(3.65~5.10)×109 g–1,在施氮处理间变化不显著(图 3a)。但是,AOB、AOA以及Comammox这三类硝化微生物对不同处理的响应规律不同(图 3b-图 3d)。单施氮肥促进AOB生长,培养21 d后AOB amoA基因达到3.59×108 g–1,是CK处理1.60倍。DCD+N能够显著抑制AOB增长,与CK相比无显著变化。ATU对AOB的影响表现为:低浓度促进,高浓度抑制。1-ATU+N处理中AOB amoA基因数量最多(3.88×108 g–1),是N处理1.08倍,20-ATU+N处理AOB amoA基因最低(2.56×108 g–1),仅为N处理的0.71倍。ATU+N对AOA和Comammox特征amoA基因的拷贝数无显著影响。而DCD+N显著刺激了AOA和Comammox生长,其amoA基因拷贝数分别为2.56×108 g–1和3.45×108 g–1,是N处理的1.58倍和1.22倍。

|
注:柱形图上方标注的不同小写字母表示处理间差异显著(P < 0.05)。 Note: Bars with different letters above indicate a significant difference (P < 0.05) among treatments. 图 3 培养21 d后各处理土壤原核微生物16S rRNA基因拷贝数(a)、硝化微生物amoA基因拷贝数(b,c,d)和16S rRNA基因相对丰度(e,f,g) Fig. 3 Copy number of 16S rRNA genes of soil prokaryotic microbes(a) and amoA genes of soil AOB(b), AOA(c) and Comammox(d), and the relative abundance of soil AOB(e), AOA(f) and NOB(g) based on 16S rRNA gene sequencing in different treatments after a 21-day incubation |
通过高通量测序技术对培养21 d后土壤整体微生物群落组成进行分析可知,不同处理AOB的16S rRNA基因相对丰度与其amoA基因丰度的变化趋势较为一致(图 3e)。N处理AOB相对丰度增加到0.179%,是CK处理4.34倍。与N处理相比,高浓度ATU(20-ATU+N)显著抑制了AOB的增加,AOB相对丰度仅为N处理的0.53倍。10-DCD+N处理AOB相对丰度与CK处理无显著差异,进一步证明DCD能够通过抑制AOB生长而较大程度抑制硝化作用。N处理对AOA相对丰度的影响不显著(图 3f)。相比于单施氮肥,中等浓度ATU(5-ATU+N、10-ATU+N和15-ATU+N)和DCD与氮肥配施使AOA相对丰度有所提高,但彼此之间无显著差异。氮肥及与硝化抑制剂配施处理均检测到大量NOB生长(图 3g),NOB相对丰度(0.840~1.061%)均显著高于CK处理(0.454%)。
总体而言,土壤硝化作用直接导致了土壤pH降低和N2O排放。土壤
|
|
表 3 土壤硝化微生物的丰度与温室气体N2O、CO2累计排放量以及土壤理化性质的相关分析 Table 3 Correlation between the relative abundance of different nitrifying guilds and greenhouse gas (N2O and CO2) emission and soil properties |
培养21 d后,土壤AOB群落组成在处理间存在明显差异(图 4a),而AOA和NOB群落组成在处理间较为相似(图 4b,图 4c)。高通量测序结果显示Nitrosospira属在AOB群落中占主导地位(> 93%),远远多于Nitrosomonas属AOB。在分类种水平可将Nitrosospira属AOB分为四个类型,其中,CK处理AOB群落中,unclassified Nitrosospira占比最大,其次为Nitrosovibrio tenuis。Nitrosovibrio tenuis类AOB在氮肥刺激下快速生长,但受到DCD强烈抑制,表明Nitrosovibrio tenuis类AOB主导土壤硝化作用和高水平N2O的产生。ATU对Nitrosospira sp. Nsp65类AOB抑制作用随ATU用量增加而增强,可能与N2O排放的降低有关。AOA在共检测到5个属,但各处理土壤AOA群落组成基本保持一致,其中,unclassified Nitrososphaeraceae所占比例最大(86%~90%),其次为Nitrososphaera(10%~14%)。NOB群落共检测到两个属,其中Nitrospira的生长占据绝对优势(97%~99%)。
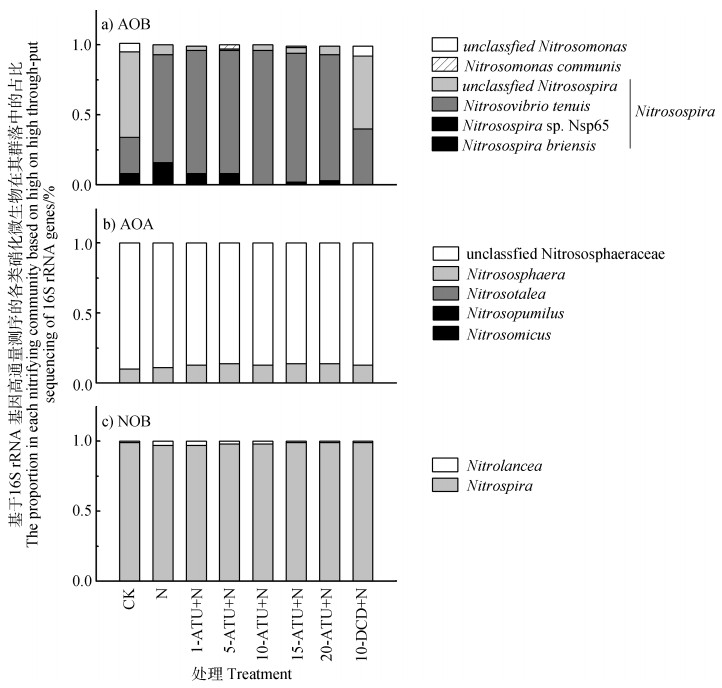
|
注:以各类型硝化微生物的相对丰度在其群落总相对丰度中的百分比表示。 Note: The value was calculated by the relative abundance of each taxonomic group in the total relative abundance of each nitrifying community. 图 4 培养21 d后各处理土壤AOB(a)、AOA(b)和NOB(c)的群落结构变化 Fig. 4 Community change of soil AOB(a), AOA(b) and NOB(c) in different treatments after 21-day incubation |
高通量测序结果的生物信息分析显示,在分类属水平,特定微生物在处理间存在显著差异。如图 5所示,N与CK处理相比、20-ATU+N与N处理相比、以及10-DCD+N与N处理相比,分别存在22个、25个以及27个显著改变的微生物属。其中,除AOB Nitrosospira属外,在三组比较中存在6个共有微生物属发生显著改变,分别为Patulibacter、Aeromicrobium、Actinomycetospora、Defluviicoccus、Acidipila和Cupriavidus。单独施N促进前5个属微生物相对丰度显著增加,其中Patulibacter和Aeromicrobium增加最多。同时单独施N还对Cupriavidus产生了一定抑制,其相对丰度低于CK。ATU和DCD与N配施对这6个属微生物的影响趋势与N处理相反,且Patulibacter和Aeromicrobium降幅最大,一定程度上缓解了施N引起的微生物变化。此外,单独施N显著刺激了Bacillus生长,而ATU和DCD均未对其产生明显作用;ATU最大程度促进了Fictibacillus的增加,而DCD对MND1有最强的刺激作用。
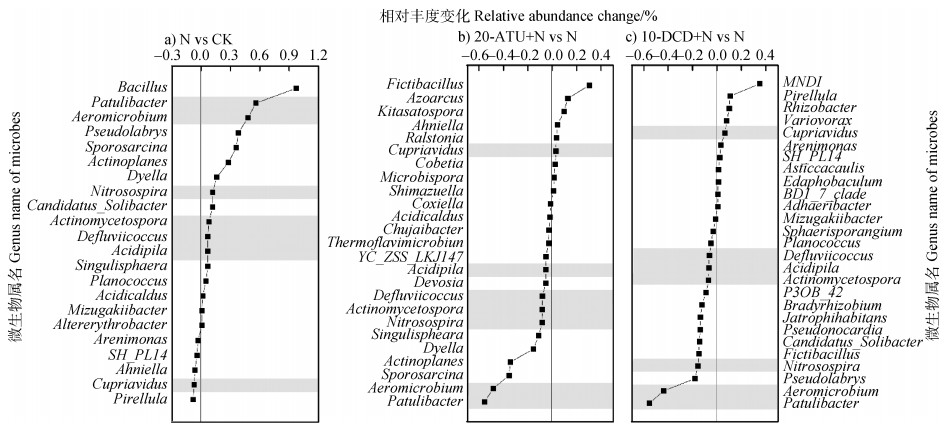
|
注:图中列出了处理间发生显著变化的微生物属(P < 0.05)。(a)表示N与CK处理之间比较;(b)表示20-ATU+N与N处理之间比较;(c)表示10-DCD+N与N处理之间比较。以平均相对丰度之差表示对应微生物属的相对丰度变化。标注灰色的属为在三组(a,b,c)比较中均显著变化的微生物类型。 Note: The genera which were influenced significantly were shown above (P < 0.05).(a) represents the comparisons between N and CK treatments: (b) represents the comparison between 20-ATU+N and N treatments : and (c) represents the comparison between 10-DCD+N and N treatments. The change of relative abundance was expressed by the difference of average relative abundance of each genus in treatments. The genus marked with the grey color changed significantly in all three comparisons (a, b, c). 图 5 不同微生物属在处理间的相对丰度变化 Fig. 5 Relative abundance change of different microbial genus between treatments |
硝化抑制剂与氮肥配施可以抑制硝化作用,减少N2O排放,从而降低温室效应。本研究通过室内培养试验模拟了抑制剂ATU和DCD与氮肥配合施入农田土壤的过程,发现单独施N产生的综合温室效应最高,ATU+N处理次之,DCD+N处理最低。在21 d培养期内,DCD使土壤
AOA和AOB对ATU的响应并不一致。多数AOB菌株对ATU敏感,小于100 μmol·L–1 ATU即对其产生强烈抑制[23]。而AOA对ATU敏感范围较广,100 μmol·L–1ATU能够完全抑制一些嗜酸Nitrosotalea类AOA,但中性Nitrososphaera类AOA则需要在更高ATU浓度(> 500 μmol·L–1)才有较强抑制[23-24]。本研究设置了一系列ATU与氮肥的施用配比,发现20% ATU(40 μg·g–1)与氮肥配施对土壤硝化作用的抑制最强此时土壤溶液中ATU浓度约为500 μmol·L–1,显著抑制了AOB生长并改变了AOB群落结构。Nitrosospira sp.Nsp65类AOB在群落中的占比明显下降,表明其对ATU较为敏感,与几株代表性AOB菌株的动力学特征较为相似[23,25],而Nitrosovibrio tenuis类AOB在群落中的占比增加,表明其具有较强ATU耐受能力,与上述类型有所不同,表明AOB群落具有较高的生理多样性。主导供试土壤AOA群落的中性Nitrososphaera类和unclassfied Nitrososphaeraceae AOA未受明显影响,表明其对ATU耐受能力较强,与先前研究较为一致[23]。
ATU在土壤中的施用效果差异较大。如Li等[12]发现14% ATU(14 μg·g–1)在草地土壤中显著降低了AOB和Comammox的数量,但在中性农田土壤中却明显刺激了Comammox的生长。另一些农田或草地土壤中观测到100 μmol·L–1 ATU降低了AOB的生长活性[26-27],但在冰岛草地土壤中相同浓度ATU似乎未对AOB和AOA生长产生影响[14]。由此可见,硝化微生物对ATU的响应规律因土壤类型而异。硝化微生物群落组成差异、微生物的相互作用以及土壤本身的高度异质性等因素,都可能使土壤硝化微生物对ATU的响应机制更加复杂。
Comammox对陆地生态系统氮循环的贡献正在重新评估。已培养的Comammox代表菌株Nitrospira inopinata的Km(NH3)仅为63 nmol·L–1[7]。供试黄棕壤原位NH3浓度与该值较为接近,并检测到了丰富的Comammox amoA基因,证明该土壤在无外源氮输入情况下能够为Comammox生长提供适宜底物条件。而施入氮肥后土壤游离NH3的浓度高于220 nmol·L–1,不利于Comammox生长,同时Comammox amoA基因拷贝数与土壤
本研究施用DCD处理中Comammox和AOA amoA基因显著增加,表明硝化群落中部分氨氧化菌对DCD不敏感,生长活性不受DCD抑制,具有区别于典型氨氧化菌的生理特点。Palomo等[29]已经证实Comammox Nitrospira Clade B可能通过非氨氧化途径产能,并还原三羧酸循环进行CO2固定,为其适应更多生境提供了能量代谢基础。未来在纯培养体系中展开更硝化菌株生理代谢和基因水平研究,可以为揭示复杂环境中硝化微生物生态功能多样性提供理论依据。
此外,ATU和DCD可以促进多种有益微生物或植物促生菌(PGPR)的生长。例如,ATU处理中Azoarcus属和DCD处理中Rhizobacter属均显著提高,可以固氮或促进植物根系氮吸收[30-31],提高作物产量和品质。ATU和DCD还刺激了Cupriavidus属的长,而已知Cupriavidus属多个代表菌株均具有固氮结瘤潜力[32]。DCD处理中Variovorax属也显著增加,而其能够改变植物激素水平,解除根系生长抑制[33]。具有降解有机物能力的微生物属也在加入抑制剂后迅速增加,如Arenimonas,Ralstonia等。此外,硫脲类化合物具有一定杀菌作用,本研究发现ATU对16个微生物属有显著抑制作用(图 5),其中,降幅最大的两个属(Patulibacter和Aeromicrobium)均属于放线菌门。DCD也具有相似的抑菌效果。鉴于目前在更低分类水平的微生物功能划分未有准确认知,它们对土壤生态功能的影响以及对植物生长的调节作用有待进一步研究。
4 结论单施氮素提高了土壤硝化强度和N2O排放,导致土壤pH下降。低浓度烯丙基硫脲对硝化作用的抑制不明显,随烯丙基硫脲浓度提高,抑制效果增强。与烯丙基硫脲相比,双氰胺能更大程度抑制土壤硝化作用和N2O排放。Nitrsospira属AOB是黄棕壤农田土壤硝化过程的主要驱动者,而AOA和NOB群落对施肥措施无明显响应。除硝化微生物外,一些特定微生物属对ATU和DCD产生相似响应规律。
| [1] |
Meinhardt K A, Stopnisek N, Pannu M W, et al. Ammonia-oxidizing bacteria are the primary N2O producers in an ammonia-oxidizing archaea dominated alkaline agricultural soil[J]. Environmental Microbiology, 2018, 20(6): 2195-2206. DOI:10.1111/1462-2920.14246
(  0) 0) |
| [2] |
Yan X Y, Zhou W. Groundwater nitrate removal through denitrification under farmland in Yangtze River Delta (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(2): 350-362. [颜晓元, 周伟. 长江三角洲农田地下水反硝化对硝酸盐的去除作用[J]. 土壤学报, 2019, 56(2): 350-362.]
(  0) 0) |
| [3] |
Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461
(  0) 0) |
| [4] |
Leininger S, Urich T, Schloter M, et al. Archaea predominate among ammonia-oxidizing prokaryotes in soils[J]. Nature, 2006, 442(7104): 806-809. DOI:10.1038/nature04983
(  0) 0) |
| [5] |
Lu L, Jia Z J. Urease gene-containing archaea dominate autotrophic ammonia oxidation in two acid soils[J]. Environmental Microbiology, 2013, 15(6): 1795-1809. DOI:10.1111/1462-2920.12071
(  0) 0) |
| [6] |
Xia W W, Zhang C X, Zeng X W, et al. Autotrophic growth of nitrifying community in an agricultural soil[J]. The ISME Journal, 2011, 5(7): 1226-1236. DOI:10.1038/ismej.2011.5
(  0) 0) |
| [7] |
Kits K D, Sedlacek C J, Lebedeva E V, et al. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature, 2017, 549(7671): 269-272. DOI:10.1038/nature23679
(  0) 0) |
| [8] |
Li C Y, Hu H W, Chen Q L, et al. Comammox Nitrospira play an active role in nitrification of agricultural soils amended with nitrogen fertilizers[J]. Soil Biology and Biochemistry, 2019, 138: 107609. DOI:10.1016/j.soilbio.2019.107609
(  0) 0) |
| [9] |
Zhang M M, Shen J P, He J Z, et al. Microbial mechanisms of nitrification inhibitors and their application (In Chinese)[J]. Journal of Agro-Environment Science, 2014, 33(11): 2077-2083. DOI:10.11654/jaes.2014.11.001 [张苗苗, 沈菊培, 贺纪正, 等. 硝化抑制剂的微生物抑制机理及其应用[J]. 农业环境科学学报, 2014, 33(11): 2077-2083.]
(  0) 0) |
| [10] |
Zhang H Q, Zhao X Q, Zhang L Y, et al. Effects of liming and dicyandiamide (DCD) application on soil pH and nitrification of acidic red soil (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(1): 169-179. [张昊青, 赵学强, 张玲玉, 等. 石灰和双氰胺对红壤酸化和硝化作用的影响及其机制[J]. 土壤学报, 2021, 58(1): 169-179.]
(  0) 0) |
| [11] |
He X J, Ji G D. Responses of AOA and AOB activity and DNA/cDNA community structure to allylthiourea exposure in the water level fluctuation zone soil[J]. Environmental Science and Pollution Research, 2020, 27(13): 15233-15244. DOI:10.1007/s11356-020-07952-9
(  0) 0) |
| [12] |
Li C Y, Hu H W, Chen Q L, et al. Growth of comammox Nitrospira is inhibited by nitrification inhibitors in agricultural soils[J]. Journal of Soils and Sediments, 2020, 20(2): 621-628. DOI:10.1007/s11368-019-02442-z
(  0) 0) |
| [13] |
Taylor A E, Zeglin L H, Dooley S, et al. Evidence for different contributions of archaea and bacteria to the ammonia-oxidizing potential of diverse Oregon soils[J]. Applied and Environmental Microbiology, 2010, 76(23): 7691-7698. DOI:10.1128/AEM.01324-10
(  0) 0) |
| [14] |
Daebeler A, Bodelier P L E, Hefting M M, et al. Ammonia-limited conditions cause of Thaumarchaeal dominance in volcanic grassland soil[J]. FEMS Microbiology Ecology, 2015, 91(3): fiv014.
(  0) 0) |
| [15] |
Zhou X, Wang S W, Ma S T, et al. Effects of commonly used nitrification inhibitors-dicyandiamide(DCD), 3, 4-dimethylpyrazole phosphate(DMPP), and nitrapyrin-on soil nitrogen dynamics and nitrifiers in three typical paddy soils[J]. Geoderma, 2020, 380: 114637. DOI:10.1016/j.geoderma.2020.114637
(  0) 0) |
| [16] |
Zhang X P, Zhong Z K, Bian F Y, et al. Effects of composted bamboo residue amendments on soil microbial communities in an intensively managed bamboo(Phyllostachys praecox) plantation[J]. Applied Soil Ecology, 2019, 136: 178-183. DOI:10.1016/j.apsoil.2018.12.025
(  0) 0) |
| [17] |
Magoč T, Salzberg S L. FLASH: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507
(  0) 0) |
| [18] |
Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J]. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9
(  0) 0) |
| [19] |
Callahan B J, McMurdie P J, Rosen M J, et al. DADA2:High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869
(  0) 0) |
| [20] |
Bokulich N A, Kaehler B D, Rideout J R, et al. Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2's q2-feature-classifier plugin[J]. Microbiome, 2018, 6: 90. DOI:10.1186/s40168-018-0470-z
(  0) 0) |
| [21] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D596.
(  0) 0) |
| [22] |
Dai Y, He J Z, Shen J P. Effects and influence factors of dicyandiamide(DCD) application in agricultural ecosystem (In Chinese)[J]. Chinese Journal of Applied Ecology, 2014, 25(1): 279-286. [戴宇, 贺纪正, 沈菊培. 双氰胺在农业生态系统中的应用效果及其影响因素[J]. 应用生态学报, 2014, 25(1): 279-286.]
(  0) 0) |
| [23] |
Shen T L, Stieglmeier M, Dai J L, et al. Responses of the terrestrial ammonia-oxidizing archaeon Ca. Nitrososphaera viennensis and the ammonia-oxidizing bacterium Nitrosospira multiformis to nitrification inhibitors[J]. FEMS Microbiology Letters, 2013, 344(2): 121-129. DOI:10.1111/1574-6968.12164
(  0) 0) |
| [24] |
Taylor A E, Vajrala N, Giguere A T, et al. Use of aliphatic n-alkynes to discriminate soil nitrification activities of ammonia-oxidizing thaumarchaea and bacteria[J]. Applied and Environmental Microbiology, 2013, 79(21): 6544-6551. DOI:10.1128/AEM.01928-13
(  0) 0) |
| [25] |
Ginestet P, Audic J M, Urbain V, et al. Estimation of nitrifying bacterial activities by measuring oxygen uptake in the presence of the metabolic inhibitors allylthiourea and azide[J]. Applied and Environmental Microbiology, 1998, 64: 2266-2268. DOI:10.1128/AEM.64.6.2266-2268.1998
(  0) 0) |
| [26] |
Wang J C, Wang J L, Rhodes G, et al. Adaptive responses of comammox Nitrospira and canonical ammonia oxidizers to long-term fertilizations: Implications for the relative contributions of different ammonia oxidizers to soil nitrogen cycling[J]. Science of the Total Environment, 2019, 668: 224-233. DOI:10.1016/j.scitotenv.2019.02.427
(  0) 0) |
| [27] |
Liu T L, Ren J Q, Wang T Y, et al. Relative contribution of bacteria and archaea to soil nitrification in neutral paddy soil (In Chinese)[J]. Chinese Journal of Soil Science, 2018, 49(5): 1091-1096. [刘天琳, 任佳琪, 王天佑, 等. 中性紫色水稻土硝化作用中细菌和古菌的相对贡献[J]. 土壤通报, 2018, 49(5): 1091-1096.]
(  0) 0) |
| [28] |
Shi X Z, Hu H W, Wang J Q, et al. Niche separation of comammox Nitrospira and canonical ammonia oxidizers in an acidic subtropical forest soil under long-term nitrogen deposition[J]. Soil Biology & Biochemistry, 2018, 126: 114-122.
(  0) 0) |
| [29] |
Koch H, van Kessel M A H J, Lücker S. Complete nitrification: Insights into the ecophysiology of comammox Nitrospira[J]. Applied Microbiology and Biotechnology, 2019, 103(1): 177-189. DOI:10.1007/s00253-018-9486-3
(  0) 0) |
| [30] |
Hurek T, Reinhold-Hurek B. Azoarcus sp. strain BH72 as a model for nitrogen-fixing grass endophytes[J]. Journal of Biotechnology, 2003, 106(2/3): 169-178.
(  0) 0) |
| [31] |
Sun Z, Zheng L, Qiu H B. Research advances on colonization of plant growth-promoting rhizobacteria (In Chinese)[J]. Biotechnology Bulletin, 2017, 33(2): 8-15. [孙真, 郑亮, 邱浩斌. 植物根际促生细菌定殖研究进展[J]. 生物技术通报, 2017, 33(2): 8-15.]
(  0) 0) |
| [32] |
Checcucci A, DiCenzo G C, Perrin E, et al. Genomic diversity and evolution of rhizobia[M]//Microbial diversity in the genomic era. Amsterdam: Elsevier, 2019: 37-46.
(  0) 0) |
| [33] |
Finkel O M, Salas-González I, Castrillo G, et al. A single bacterial genus maintains root growth in a complex microbiome[J]. Nature, 2020, 587(7832): 103-108. DOI:10.1038/s41586-020-2778-7
(  0) 0) |
 2021, Vol. 58
2021, Vol. 58


