驯化是指人们对野生物种某些可利用的性状进行选择、修饰和利用的过程[1]。它以食物可持续供应为目的,推动了人类文明进步[2]。植物驯化始于约12 000年前,包括水稻、小麦、大麦、马铃薯和番茄在内的353种粮食作物[3]。伴随着栖息地的迁移和农田管理措施的改良,作物产量不断提高[4],表型性状也在不断进化,包括种子体积更大、扩散机制丧失和生长期缩短、可食用部分的苦味物质减少以及光周期敏感性变化等[2]。作物的根系形态和次生代谢产物也随之改变[5]。相比于人工驯化后的栽培作物,野生植物具有广泛的生境范围,可适应各种极端恶劣的生长环境[6]。对基因性状的研究表明,栽培种积累了更多有害和不利的突变[7-8]。并且作物育种阻碍了作物与当地环境的适应,特别是与土著微生物群落的相互适应[9]。在人为选择栽培种的过程中,野生种的部分生理和遗传特性逐渐丢失,导致栽培作物的遗传多样性显著降低,这种现象被称为驯化综合症(domestication syndrome)[10]。菜豆[11]、水稻[12]和小麦[13]栽培种均出现该现象。由于野生植物物种可能包含有益于作物育种的遗传多样性,因此,加大对野生种质资源的保护对于农业的长期发展十分重要。
驯化过程是植物-微生物共同进化的结果。植物-微生物的相互作用对陆地植物进化起到重要作用[14]。如丛枝菌根真菌和根瘤菌,与植物有着漫长而持久的共同进化史[15]。植物与病原菌之间也存在共进化的关系,植物通过对自身性状的改变和与其他有益微生物的协同进化,以抵御病原菌侵染[16]。同时,微生物-微生物互作也是驱动共进化的重要因素:微生物群落中产生的有益突变可通过基因水平转移进行扩散,改变微生物与微生物之间的相互作用,形成新的群落结构,进而改变微生物与植物的互作关系[17]。例如,为了促进高产并保护驯化作物免受生物和非生物胁迫因素的影响,植物驯化过程通常伴随着植物栖息地的变化和杀虫剂、肥料的过度使用[4]。长期施氮导致弱互生根瘤菌的进化,降低了豆类寄主的生长效益,表现为土壤呼吸受抑制,微生物生物量减少[18]。
根际微生物组具有促进植物养分吸收,提高植物抗逆性,抵御土传病害、改良土壤和调节植物免疫应答的有益性状[4, 19-20]。因此,植物健康和生长发育依赖于根际微生物的功能和性状。此前人们更多关注驯化后植物地上部产量及表型的变化,却忽略了驯化对地下微生物的影响以及微生物对作物的反馈作用。植物与微生物组协同进化,微生物组的稳定性对植物的适应性至关重要[9]。微生物群落失衡会对植物的适应性产生负面影响[21]。对不同植物的微生物组研究表明,寄主种类、地理位置、土壤环境、寄生部位均能够影响微生物群落的组成和功能,但是每个因素在多大程度上发挥作用,不同研究之间不尽相同。为此,本文综述了近年来植物驯化过程中的自身因素(基因型、根外代谢)和环境因素(栖息地)的变化对根际和种际微生物组的结构和功能的影响以及重要作用,以期从微生物组角度提高对驯化的利用程度(图 1)。
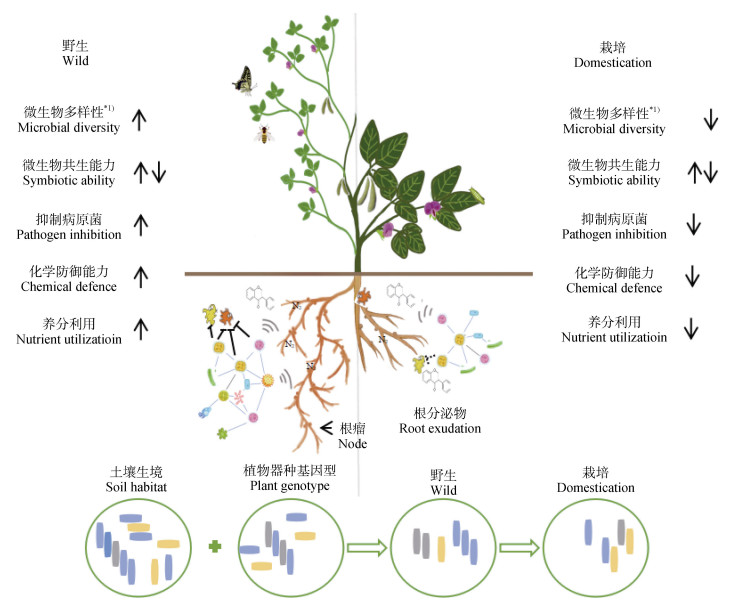
|
注:箭头向上代表多样性和相应功能增加或提升。箭头向下代表多样性和相应功能较少或降低。*代表例外情况。1)微生物多样性,大部分研究,驯化导致了根际微生物群落多样性的降低,但也有反例,例如向日葵。2)现代抗病育种是例外,无意中选择了更多具有抑病潜能的有益微生物。 Note: The upward arrows indicate improvement or enhancement of diversity and the corresponding function, and vice versa. * representing execptions.1)Microbial diversity in most cases, decreases as a result of domestication, but exceptions do exist, like sunflower. 2)The modern plant breeding for disease resistance is an exception, unintentionally attracting more beneficial microbes that possess disease inhibiting potentials. 图 1 植物驯化对其微生物组结构和功能影响的模式图 Fig. 1 Schematic graph of the impacts of plant domestication on composition and functions of the microbiome |
驯化改变了植物的表型性状,提高了植物产量,但同时减少了植物遗传多样性。研究发现,不同植株种类会招募特定的根际微生物,调节根际微生物群落;同种植物驯化过程中产生的不同基因型其根际微生物组成也不同[22]。对于种际微生物(种子内生菌)而言,受胁迫时植物会调节种际微生物群落,以确保其最有益微生物群的后代传递,从而促进形成植物基因型特异性种子内生菌群[23-24]。同时,植物驯化也影响着微生物组的功能,对于生长需要特定共生微生物的作物,作物驯化可导致与其共生微生物间关系发生变化[25]。目前已有很多学者就植物驯化是如何影响植物的微生物组这一问题进行了研究,逐渐揭示驯化过程影响微生物组的组装和功能的潜在机制。
1.1 驯化过程中植物品种和基因型对微生物组群落结构的影响Kim等[26]在研究水稻基因型对微生物群落结构的影响时发现,基因型解释了总方差的24.2%(细菌)和20.3%(真菌),野生稻真菌多样性高于栽培稻,更易受驯化影响。并且作者认为驯化过程是影响种际微生物群落结构的主要因素,野生型和驯化水稻种子微生物群落(细菌和真菌)显著分离,真菌群落似乎较细菌群落更易受到驯化的影响。对驯化水稻种际微生物的网络分析表明,鞘氨醇单胞菌为中心枢纽,而野生型水稻的中心为一种细菌操作分类单元(OTU)(Pantoea)和两种真菌OTUs(Penicillium和一种未分类真菌)。Hassani等[27]发现野生小麦和栽培小麦根际微生物群落组成不同。其中,厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)在野生小麦中更丰富,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)在栽培型中占比更多。同时,野生小麦根际细菌群落组成更多样,而真菌差异不明显。在大麦中,Bulgarelli等[28]的研究发现,宿主基因型对根际相关细菌群落的多样性影响较小但显著,单独的宿主基因型可解释5.7%的变异。在门水平,野生型作物根际招募更多的拟杆菌(Bacteroidetes),而放线菌(Actinobacteria)、变形菌(Proteobacteria)在栽培型作物中比例上升。在豆科作物的研究中,野生大豆和栽培大豆根际细菌群落显著分离:芽单胞菌门(Gemmatimonadetes)、酸性细菌门(Acidobacteria)、慢生根瘤菌门(Bradyrhizobium)和芽孢杆菌门(Bacillus)在野生型中富集程度更高。说明基因型是影响其根际细菌群落组成的一个重要因素[29]。同时,对于菜豆的研究发现,拟杆菌在野生种根系中具有较高的丰富度,而放线菌和变形菌在栽培种根系中更为丰富[30]。Pérez-Jaramillo等[31]推测拟杆菌门在野生种根上富集可能是由于它们能够降解根分泌物中的高分子量有机物。向日葵不同部位(种子、根内、根际)真菌和细菌多样性差异显著(两者均P < 0.001),种子内真菌和细菌多样性低于根内和根际群落,与根内和根际样品相比,拟诺卡菌科(Nocardiopsaceae)、肠杆菌科(Enterobacteriaceae)和鞘脂单胞菌科(Sphingomonadaceae)的相对丰度较高。驯化对根际真菌群落组成具有显著影响,现代栽培种(Unclassified Pleosporales,Preussia spp.,unclassified Thelebolaceae,Fusarium spp.,Conocybe spp.)相对丰度较高且病原真菌丰度相比野生型更低[32]。总体而言,大部分植物物种的栽培和野生品种之间细菌和真菌群落多样性具有显著差异。与栽培品种相比,野生种具有更多样的根际微生物。然而,也在向日葵的研究中观察到相反的结果[32],这说明需要对大量的植物品种进行分析。
1.2 驯化过程中植物品种和基因型对共生微生物关系的影响伴随驯化过程,作物会产生不同的植物-微生物共进化结果,可能是有利的正向共进化,也可能不利于现代栽培种的负向共进化。Hetrick等[33]发现,野生六倍体小麦品种较现代品种更能受益于菌根共生。此外,在两种不同磷浓度(低磷和适量磷)处理下,对4个玉米地方品种和1个现代杂交种对丛枝菌根真菌(AMF,arbuscular mycorrhizal fungi)的响应时发现,与现代杂交种相比,两个地方品种更容易被AMF定殖,并在幼苗中获得更多的磷[34]。Martín-Robles等[35]测定了驯化对27种作物及其野生型菌根响应性的影响,结果表明,栽培作物仅在低磷条件下才能从菌根中获益,而野生种在高磷或低磷条件下均可获益;在高磷条件下,栽培作物对AMF的响应显著降低,这可能是由于高产品种为增加地上部的生物量,碳分配的改变引起的;作物根部的碳量减少,从而降低与AMF的共生程度。在与根瘤菌的共生关系中,Kiers等[36]的研究发现,与老品种相比,新品种大豆的种子产量较低,老品种中有效根瘤菌占比更高。然而,驯化也可能选择更有效的共生关系,根瘤菌接种后,栽培大豆根瘤菌共生性能指标(组织质量、总根瘤菌数、总氮、总脲积累)均优于野生大豆[37]。因此,同种植株也会因为品种不同,而产生不同驯化结果。与野生型相比,共生真菌(主要是球囊菌门Glomeromycota)在栽培型的向日葵根中被检测到的比例更大[32]。以马铃薯为例,Weinert等[38]对三个不同的马铃薯栽培种检测到的OTUs发现其具有物种特异性,在不同土壤检测到的2 432个OTUs中,65% OTUs在栽培种根际中均可发现,并且发现链霉菌具有品种特异性,对栽培品种响应强烈;相似的品种特异性效应也出现在马铃薯幼嫩植株的根际微生物群落中,在栽培种中放线菌和变形杆菌是最丰富的类群,高淀粉品种对α-变形菌(Alphaproteobacteria)在各个生长阶段均表现出积极的根际效应,而低淀粉块茎品种仅在衰老阶段表现出这种效应[39]。
1.3 驯化过程中植物品种和基因型对微生物组功能的影响对玉米种际微生物的研究表明,从野生玉米驯化为10个不同的现代栽培玉米品种的过程中,保留的一个内生菌核心菌群可促进根系生长及根茎生物量,同时具有分泌铁载体、抗生素类物质等多种与促生相关的特性[40]。通过宏基因组对大麦根相关的微生物组基因的潜在功能进行分析,发现存在大量与宿主-病原体相互作用(Ⅲ型分泌系统T3SS、毒力调节、侵袭和细胞内抵抗),微生物-微生物相互作用(VI型分泌系统T6SS),微生物-噬菌体相互作用(转座元件、噬菌体整合)和养分活化相关的功能基因[28]。栽培水稻的根际微生物对病原菌(Magnaporthe oryzae)的入侵更敏感,更容易受到干扰。与栽培稻相比,入侵的病原菌在野生稻根际的丰度明显较低,而有益菌和真菌的相对丰度较高。在致病和非致病条件下,对水稻微生物组和根转录组的分析发现,球囊菌门(Glomeromycota)有利于促进野生稻中苯丙烷和木质素的合成,对抵抗稻瘟病菌具有重要作用[41]。Leff等[32]对野生型和栽培型向日葵进行了研究,发现向日葵基因型对根际微生物(细菌、真菌)群落多样性无影响。但是,野生向日葵与栽培种根际真菌群落组成仍存在较大差异,尤其是现代栽培向日葵品种的致病真菌相对丰度较低。刘昭军等[42]从野生大豆分离出可降解大豆化感物质的菌种,均可拮抗大豆立枯丝核菌,初步认为可缓解大豆的连作障碍。Zachow等[43]对野生型和栽培型的甜菜根际可培养细菌进行分析,与驯化甜菜相比,野生种根际土壤中对植物病原菌具有拮抗作用的菌株比例较低。但经过培养基接种病原菌离体培养发现,从野生甜菜根际分离的假单胞菌和狭养单胞菌具有更强的抵抗病害特性。并且上述来自野生甜菜的细菌更利于作物应对非生物(水分、盐分、活性氧)胁迫。
此外,研究表明,抗性育种会影响根际微生物群落结构和功能,抗病品种较感病品种的根际有益微生物的相对丰度更高[44-47]。Mendes等[48]对菜豆的不同抗病品种(抗病原菌Fusarium oxysporum)展开研究,网络分析表明在抗病品种的根际,存在更为复杂和高度相连的细菌群落,假单胞菌科(Pseudomonadaceae)、芽胞杆菌科(Bacillaceae)、橄榄枝科(Solibacteraceae)和噬纤维菌科(Cytophagaceae)丰富度更高,对于属水平的微生物群落分析显示假单胞菌属和芽孢杆菌属更为活跃。随后,通过宏基因组对其功能进行探究,抗病品种根际菌群富含与蛋白分泌系统、抗真菌物质非那嗪和鼠李糖醇类生物合成等相关的功能基因。宏转录组对此现象进行进一步的机制解析表明,抗病品种的微生物组中,与营养代谢、运动性、趋化性以及抗真菌化合物非那嗪和大肠杆菌素V的生物合成相关的基因表达显著提高。证实抗病品种的根际土壤富含有益菌群,帮助植物抵御病原体[49]。因此,作者认为育种中抗性的培育无意中选择了有益的根际菌群结构和功能,使特定根际活性的细菌类群丰度增加,提供对根系真菌感染相应的保护[46]。
上述研究表明,驯化过程中产生的多种基因型,不仅影响微生物群落结构,同时对微生物-植物之间的共生关系以及微生物组的功能,也产生影响。细菌丰度的变化在不同类型的植物中规律不同(表 1),真菌与植物联系更紧密,更易受驯化作用的影响(表 2)。高产量基因型作物弱化了与绝大多数微生物的共生关系。野生植物根际微生物群落有利于维持植物的正常生长,尤其在抗病、养分活化方面效用突出。然而抗性育种得到的抗病植株可能无意选择了更多的有益微生物,从而进一步提高自身的抗病能力。因此在驯化过程中,需要进一步解析作物类型、基因型与根际微生物组的互作机理,为以微生物组为指导的育种提供理论依据,达到增产、提质、减肥的效果。
|
|
表 1 不同种类作物驯化中招募细菌类型差异 Table 1 Species of bacteria attracted by the plant during domestication relative to species of the plant |
|
|
表 2 不同种类植物驯化中招募真菌类型差异 Table 2 Species of fungi attracted by the plant during domestication relative to species of the plant |
由于驯化,野生植物产生重大遗传变化、生存环境变化,影响了根外代谢产物(根系分泌物和挥发性有机化合物),进而对微生物群落产生重要影响[4, 50-52]。而不同驯化程度下有机化合物产量的差异可能是生长速率与生物和非生物胁迫防御之间权衡的结果[53]。
首先,驯化作用对根系分泌物的影响导致细菌、真菌重要的碳源发生改变,其中含有驱赶病菌或者吸引有益菌的物质[52],如蔗糖代谢对微生物细胞的生长、基因表达调控、应激反应和信号通路有直接和间接的影响[54]。根系分泌物可通过农业生态系统的选择或直接的人为干预而得到大幅度的改变。以小麦为例,在无菌砂培养的野生二粒小麦中果糖含量高,而栽培型硬粒小麦根际代谢产物中最丰富的是甘露醇和山梨醇[50]。而根代谢物的改变,可能是决定微生物组组装的重要因素[55]。与植物的共同进化驱动了微生物基因组的进化,与土壤中提取的枯草芽孢杆菌菌群相比,根际枯草芽孢杆菌菌群的核心基因组中,与中间代谢和次生代谢产物生物合成相关的基因更为丰富,显示出对根系分泌物的积极响应;同时,根际芽孢杆菌的核心基因组中,具有更多可利用源于植物多糖和合成抗生素的特异性基因[56]。
其次,驯化影响到植物共生信号。如作物通过释放独脚金内酯来启动菌根的定殖[57],从而影响了与植物共生的微生物群落。对于植物和固氮菌之间的共生关系而言,寄主植物根系通过分泌黄酮类化合物,激活负责结瘤过程的根瘤菌基因[58],进而吸引根瘤菌[59]。而通过研究豌豆、蚕豆和鹰嘴豆发现,驯化降低了豆科植物与许多根瘤菌种群相联系的能力,导致与相关野生物种相比,共生根瘤菌的多样性降低[60]。然而,其他研究发现,在驯化品种中菌根的定殖能力并不一定低,如玉米[61],新品种较野生型对菌根反应更敏感[62]。这说明了驯化过程中根系分泌物的改变,不一定会造成不利影响,还需要针对不同作物进行研究。因此,驯化过程中共生关系破坏可能是有害的,也可能是对植物性能的适应,主要取决于当地条件[25]。
同时,驯化导致作物对虫害抵抗力下降。化学防御是植物防御食草昆虫的重要机制,研究发现,与野生型相比,栽培型作物通常对昆虫的抵抗力更低,包括玉米、蔓越莓和羽扇豆[63-65]。这可能是由于育种过程直接选择去除具有保护性但是不受欢迎的食物特性,如苦味、毒性、韧性或多毛,也可能因为增产所采取的育种措施,导致防御机制的丢失[66-67]。Chen等[68]也强调,与野生植物相比,驯化导致栽培植物挥发性物质释放减少,影响对天敌的吸引力。如野生型欧洲玉米可释放倍半萜烯(E)-β-石竹烯,吸引一种昆虫病原线虫,该线虫以甲虫幼虫为食,从而减轻虫害。然而,大多数北美玉米品种不会释放该化学信号,因此更容易受到甲虫攻击;这种特性可能在育种过程中丢失[69]。
植物的野生种提供了巨大的遗传材料和信息来源。与现代栽培种相比,野生种的根外代谢物的数量和组成可能有所不同,并具有整合至现代育种中的理想特性(如抗虫害、增强共生能力等)。利用上述特征,为提高农业产量、减少环境影响特别是减少对农药和肥料的依赖、提高农业可持续性发展提供了可能性。
3 驯化中土壤生境对微生物组的影响微生物群落在根际的聚集受非生物和生物因素的控制。即便作物驯化带来了对根际微生物组的特定选择能力,但生态环境制约其最终结构形成。一般地,野生生境通常蕴含着独特的微生物功能类群[70]。诸多研究表明,土壤对根际细菌和菌根真菌群落的聚集有显著的影响。土壤理化性质是影响细菌群落组成的重要因素之一[71-73]。向日葵根际真菌群落组成与驯化程度有关,这可能是由驯化对土壤特性的间接影响、通过根系性状的差异所驱动的[32]。Chang等[72]认为大豆根际真菌群落的多样性和结构主要受土壤类型影响而不是大豆的品种,驯化导致野生大豆和栽培大豆招募到不同的根际真菌。类球囊霉菌(Paraglomus)在仅有野生大豆生长的黑土中丰度最高,根内球囊霉(Rhizophagus)、镰胞菌(Fusarium)则是红壤显著聚集,丝核菌(Ascorhizoctoni)在盐碱土相对丰度最高。同时,与自然生态系统相比,农业土壤中AMF的多样性和丰度较低[74]。土壤对草莓根际菌根定殖程度和AMF组装结构有显著影响,不同土壤中AMF定殖根长不同,在未经过化肥农药处理过的耕作土壤中,AMF定殖根长更长,且以无梗囊霉(Acaulospora scrobiculata、Acaulospora laevis、Acaulospora undulata)居多,而不同草莓品种间无明显差异[75]。目前为止,诸多研究均指出在栽培作物中微生物多样性较低,生长在原生土壤中的植株具有更多样化的根际群落[76]。Coleman-Derr等[76]发现栽培龙舌兰的根际和根内生细菌和真菌多样性低于原生环境龙舌兰(Agave salmiana)和沙漠龙舌兰(Agave deserti)。Zachow等[43]发现与现代栽培品种相比,在自然生境采集的原生土壤中,野生甜菜具有更多样化的根际微生物群落,仅在原生土壤培养的植物根际中才能检测到酸性细菌门(Acidobacteria)、绿弯菌门(Chloroflexi)和浮霉菌门(Planctomycetes);并且通过试验证明野生型作物及原生土壤分离出的微生物群落具有抗菌活性,可保护寄主抵御胁迫(如假单胞菌和狭养单胞菌)。Cassman等[77]研究也发现了向日葵和真菌群落组成之间的重要关系:在肥沃的土壤中,植物与真菌群落较与细菌群落的联系更紧密。通过上述研究可发现,驯化过程中的土壤因素改变会影响作物微生物群落结构,原生土壤生境具有更多样和独特的微生物组成。如何利用这些“丢失的微生物和性状”,以充分发挥野生植物中的有益微生物功能有待进一步研究。
4 结论与展望驯化过程中植物种类和基因型影响了根际微生物组的组成。细菌丰富度的差异主要体现在拟杆菌、放线菌、厚壁菌、变形菌、酸性细菌等种类。栽培作物中,由于人为提供大量养分,导致富营养性微生物富集;同时,驯化对于共生关系(根瘤菌、菌根真菌等)影响较大,共生作用的改变会因作物种类不同而产生不同的驯化结果。在禾本科作物中,栽培种与微生物的共生作用明显减弱,而豆科栽培种的共生能力反而有所改善。目前大部分的研究表明,驯化导致了根际微生物群落多样性的降低,但也有反例,因此,尚需对大量植物品种进行挖掘分析。根际微生物群落不同,其潜在功能也受到影响,根据对野生型和栽培型的微生物功能分析,保存下的种内菌群对作物-微生物共进化具有重要作用。在野生型根际发现很多有益微生物,具有抵御病原菌、活化养分的作用;而抗性育种无意中选择的有益微生物,对根系提供了保护作用,抗病品种中的有益菌属与野生植物根际菌群出现重叠,如芽孢杆菌等菌属,因此,需要进一步挖掘野生植物根际富集的有益菌及其功能。
其次,根外代谢物在驯化过程中的改变,影响了微生物的养分供应,减弱了作物的化学防御能力。第三,植物微生物群落的组成离不开土壤环境的贡献,相对于农田土壤,原生土壤生境具有更多样和独特的微生物组成。因此,驯化过程中的微生物组结构和功能是由植物种类基因型、根外代谢物、土壤生境等多因素作用的结果。在自然系统中,植物从有益微生物的共生关系中获取的资源较投入要多;但在农业系统中,由于植物可从土壤和肥料中容易地获得养分资源,减少了植物从微生物共生体中获得养分的依赖[25]。因此,农业措施(如耕作、作物轮作或对土壤施用化学物质等)造成有益微生物减少,抑制共生或减弱共生关系。由于共生微生物丰度降低,植物从共生中获益有限,植物便会弱化从共生关系中获取资源,从而导致了弱共生作物基因型的进化[78]。
迄今为止,植物驯化微生物组的大部分研究停留在对微生物种群类别的分析与描述。这些研究利用细菌16S rRNA和真菌18S rRNA或内部转录间隔区(ITS)区域的高通量测序进行分析。在此类实验中,微生物群落分析主要集中在植物驯化下微生物分类组成的潜在变化上,而对于野生种和驯化品种之间的微生物组的功能装配及其产生的特征和机理的探索尚很有限。少数研究通过宏基因组和宏转录组学分析,揭示了微生物组与宿主相互作用的功能基因类别和潜能。在未来的研究中,应更加重视微生物组的代谢途径等功能,以目标性状作为研究目标,利用基因编辑及微生物组学等技术深入理解驯化过程中菌植互作,以阐明驯化中植物-微生物组互作的机理,对现代作物抗病增产特性进行改良,制定合理的、基于微生物组改良的植物和土壤策略,以提高农田系统的生产力与可持续性,实现农业绿色发展。因此未来的研究重点和方向包括:(1)在驯化过程中和原生土壤生境中,丢失的微生物是否构成植物微生物组结构和功能的重要组成部分有待探讨;同时,结合培养组和组学的手段,解析微生物组的性状和功能。(2)需要对微生物群落组装和适应性机理深入探究,充分理解驯化中微生物组对植物性状的影响;阐明驯化过程中植物和微生物组共进化的关系,以解析植物和微生物组的互作特征。(3)需要更全面地研究种子相关微生物组的组成、稳定性和可遗传性,从而理解种际微生物短期和长期的动态和协同适应性。(4)由于土壤环境的异质性和复杂性,根外代谢产物的原位检测和研究仍处于起步阶段,未来应注重探索土壤原位检测根外代谢产物的方法和途径。
| [1] |
Zamir D. Improving plant breeding with exotic genetic libraries[J]. Nature Reviews. Genetics, 2001, 2(12): 983-989. DOI:10.1038/35103590
(  0) 0) |
| [2] |
Purugganan M D, Fuller D Q. The nature of selection during plant domestication[J]. Nature, 2009, 457(7231): 843-848. DOI:10.1038/nature07895
(  0) 0) |
| [3] |
Meyer R S, Purugganan M D. Evolution of crop species: Genetics of domestication and diversification[J]. Nature Reviews Genetics, 2013, 14(12): 840-852. DOI:10.1038/nrg3605
(  0) 0) |
| [4] |
Pérez-Jaramillo J E, Mendes R, Raaijmakers J M. Impact of plant domestication on rhizosphere microbiome assembly and functions[J]. Plant Molecular Biology, 2016, 90(6): 635-644. DOI:10.1007/s11103-015-0337-7
(  0) 0) |
| [5] |
Meyer R S, DuVal A E, Jensen H R. Patterns and processes in crop domestication: An historical review and quantitative analysis of 203 global food crops[J]. New Phytologist, 2012, 196(1): 29-48. DOI:10.1111/j.1469-8137.2012.04253.x
(  0) 0) |
| [6] |
Chang C L, Ma L N, Tian C J. A Selecting effect of crop domestication on rhizomicrobiome (In Chinese)[J]. Soils and Crops, 2018, 7(2): 236-241. [常春玲, 马丽娜, 田春杰. 作物驯化对根际微生物组的选择效应[J]. 土壤与作物, 2018, 7(2): 236-241.]
(  0) 0) |
| [7] |
Lu J, Tang T, Tang H, et al. The accumulation of deleterious mutations in rice genomes: A hypothesis on the cost of domestication[J]. Trends in Genetics, 2006, 22(3): 126-131. DOI:10.1016/j.tig.2006.01.004
(  0) 0) |
| [8] |
Renaut S, Rieseberg L H. The accumulation of deleterious mutations as a consequence of domestication and improvement in sunflowers and other compositae crops[J]. Molecular Biology and Evolution, 2015, 32(9): 2273-2283. DOI:10.1093/molbev/msv106
(  0) 0) |
| [9] |
Hassani M A, Özkurt E, Seybold H, et al. Interactions and coadaptation in plant metaorganisms[J]. Annual Review of Phytopathology, 2019, 57(1): 483-503. DOI:10.1146/annurev-phyto-082718-100008
(  0) 0) |
| [10] |
Doebley J F, Gaut B S, Smith B D. The molecular genetics of crop domestication[J]. Cell, 2006, 127(7): 1309-1321. DOI:10.1016/j.cell.2006.12.006
(  0) 0) |
| [11] |
Bitocchi E, Nanni L, Bellucci E, et al. Mesoamerican origin of the common bean(Phaseolus vulgaris L.) is revealed by sequence data[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(14): E788-E796. DOI:10.1073/pnas.1108973109
(  0) 0) |
| [12] |
Ram S G, Thiruvengadam V, Vinod K K. Genetic diversity among cultivars, landraces and wild relatives of rice as revealed by microsatellite markers[J]. Journal of Applied Genetics, 2007, 48(4): 337-345. DOI:10.1007/BF03195230
(  0) 0) |
| [13] |
Haudry A, Cenci A, Ravel C, et al. Grinding up wheat: A massive loss of nucleotide diversity since domestication[J]. Molecular Biology and Evolution, 2007, 24(7): 1506-1517. DOI:10.1093/molbev/msm077
(  0) 0) |
| [14] |
Graham L E, Graham J M, Wilcox L W, et al. Evolutionary roots of plant microbiomes and biogeochemical impacts of nonvascular autotroph-microbiome systems over deep time[J]. International Journal of Plant Sciences, 2018, 179(7): 505-522. DOI:10.1086/698709
(  0) 0) |
| [15] |
Fitzpatrick C R, Salas-González I, Conway J M, et al. The plant microbiome: From ecology to reductionism and beyond[J]. Annual Review of Microbiology, 2020, 74(1): 81-100. DOI:10.1146/annurev-micro-022620-014327
(  0) 0) |
| [16] |
Turner T R, James E K, Poole P S. The plant microbiome[J]. Genome Biology, 2013, 14(6): 209. DOI:10.1186/gb-2013-14-6-209
(  0) 0) |
| [17] |
Miller E T, Svanbäck R, Bohannan B J M. Microbiomes as metacommunities: Understanding host-associated microbes through metacommunity ecology[J]. Trends in Ecology & Evolution, 2018, 33(12): 926-935.
(  0) 0) |
| [18] |
Weese D J, Heath K D, Dentinger B T M, et al. Long-term nitrogen addition causes the evolution of less-cooperative mutualists[J]. Evolution, 2015, 69(3): 631-642. DOI:10.1111/evo.12594
(  0) 0) |
| [19] |
Qin H, Bai J F, Xu Q F, et al. Effects of arbuscular mycorrhizal fungal hyphae on soil microbial community composition and polychlorinated biphenyls degradation (In Chinese)[J]. Soils, 2015, 47(4): 704-710. [秦华, 白建峰, 徐秋芳, 等. 丛枝菌根真菌菌丝对土壤微生物群落结构及多氯联苯降解的影响[J]. 土壤, 2015, 47(4): 704-710.]
(  0) 0) |
| [20] |
Cao Y F, Shen Z Z, Liu S S, et al. Evaluation of effect of Trichoderma controlling fusarium wilt disease and its influencing factors with meta-analysis in China (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(3): 716-727. [操一凡, 沈宗专, 刘珊珊, 等. Meta分析评估中国木霉对枯萎病防控效果及其影响因素[J]. 土壤学报, 2019, 56(3): 716-727.]
(  0) 0) |
| [21] |
Bartoli C, Frachon L, Barret M, et al. In situ relationships between microbiota and potential pathobiota in Arabidopsis thaliana[J]. The ISME Journal, 2018, 12(8): 2024-2038. DOI:10.1038/s41396-018-0152-7
(  0) 0) |
| [22] |
Khan A L, Asaf S, Al-Rawahi A, et al. Rhizospheric microbial communities associated with wild and cultivated frankincense producing Boswellia sacra tree[J]. PLoS One, 2017, 12(10): e0186939. DOI:10.1371/journal.pone.0186939
(  0) 0) |
| [23] |
Ewald P W. Transmission modes and evolution of the parasitism-mutualism continuum[J]. Annals of the New York Academy of Sciences, 1987, 503: 295-306. DOI:10.1111/j.1749-6632.1987.tb40616.x
(  0) 0) |
| [24] |
Truyens S, Weyens N, Cuypers A, et al. Bacterial seed endophytes: Genera, vertical transmission and interaction with plants[J]. Environmental Microbiology Reports, 2015, 7(1): 40-50. DOI:10.1111/1758-2229.12181
(  0) 0) |
| [25] |
Porter S S, Sachs J L. Agriculture and the disruption of plant-microbial symbiosis[J]. Trends in Ecology & Evolution, 2020, 35(5): 426-439.
(  0) 0) |
| [26] |
Kim H, Lee K K, Jeon J, et al. Domestication of Oryza species eco-evolutionarily shapes bacterial and fungal communities in rice seed[J]. Microbiome, 2020, 8(1): 20. DOI:10.1186/s40168-020-00805-0
(  0) 0) |
| [27] |
Amine Hassani M, Özkurt E, Franzenburg S, et al. Ecological assembly processes of the bacterial and fungal microbiota of wild and domesticated wheat species[J]. bioRxiv, 2020. DOI:10.1101/2020.01.07.896910
(  0) 0) |
| [28] |
Bulgarelli D, Garrido-Oter R, Münch P C, et al. Structure and function of the bacterial root microbiota in wild and domesticated barley[J]. Cell Host & Microbe, 2015, 17(3): 392-403.
(  0) 0) |
| [29] |
Ma L, Luo S, Xu S, et al. Different effects of wild and cultivated soybean on rhizosphere bacteria[J]. Microbiology, 2019, 88(6): 720-728. DOI:10.1134/S0026261719060109
(  0) 0) |
| [30] |
Pérez-Jaramillo J E, Carrión V J, Bosse M, et al. Linking rhizosphere microbiome composition of wild and domesticated Phaseolus vulgaris to genotypic and root phenotypic traits[J]. The ISME Journal, 2017, 11(10): 2244-2257. DOI:10.1038/ismej.2017.85
(  0) 0) |
| [31] |
Pérez-Jaramillo J E, Carrión V J, de Hollander M, et al. The wild side of plant microbiomes[J]. Microbiome, 2018, 6(1): 143. DOI:10.1186/s40168-018-0519-z
(  0) 0) |
| [32] |
Leff J W, Lynch R C, Kane N C, et al. Plant domestication and the assembly of bacterial and fungal communities associated with strains of the common sunflower, Helianthus annuus[J]. New Phytologist, 2017, 214(1): 412-423. DOI:10.1111/nph.14323
(  0) 0) |
| [33] |
Hetrick B A D, Wilson G W T, Cox T S. Mycorrhizal dependence of modern wheat varieties, landraces, and ancestors[J]. Canadian Journal of Botany, 1992, 70(10): 2032-2040. DOI:10.1139/b92-253
(  0) 0) |
| [34] |
Sangabriel-Conde W, Negrete-Yankelevich S, Maldonado-Mendoza I E, et al. Native maize landraces from Los Tuxtlas, Mexico show varying mycorrhizal dependency for P uptake[J]. Biology and Fertility of Soils, 2014, 50(2): 405-414. DOI:10.1007/s00374-013-0847-x
(  0) 0) |
| [35] |
Martín-Robles N, Lehmann A, Seco E, et al. Impacts of domestication on the arbuscular mycorrhizal symbiosis of 27 crop species[J]. New Phytologist, 2018, 218(1): 322-334. DOI:10.1111/nph.14962
(  0) 0) |
| [36] |
Kiers E T, Hutton M G, Denison R F. Human selection and the relaxation of legume defences against ineffective rhizobia[J]. Proceedings of the Royal Society B: Biological Sciences, 2007, 274(1629): 3119-3126. DOI:10.1098/rspb.2007.1187
(  0) 0) |
| [37] |
Muñoz N, Qi X, Li M W, et al. Improvement in nitrogen fixation capacity could be part of the domestication process in soybean[J]. Heredity, 2016, 117(2): 84-93. DOI:10.1038/hdy.2016.27
(  0) 0) |
| [38] |
Weinert N, Piceno Y, Ding G C, et al. Phylochip hybridization uncovered an enormous bacterial diversity in the rhizosphere of different potato cultivars: Many common and few cultivar-dependent taxa[J]. FEMS Microbiology Ecology, 2011, 75(3): 497-506. DOI:10.1111/j.1574-6941.2010.01025.x
(  0) 0) |
| [39] |
İnceoğlu Ö, Falcão Salles J, van Elsas J D. Soil and cultivar type shape the bacterial community in the potato rhizosphere[J]. Microbial Ecology, 2012, 63(2): 460-470. DOI:10.1007/s00248-011-9930-8
(  0) 0) |
| [40] |
Johnston-Monje D, Raizada M N. Conservation and diversity of seed associated endophytes in Zea across boundaries of evolution, ethnography and ecology[J]. PLoS One, 2011, 6(6): e20396. DOI:10.1371/journal.pone.0020396
(  0) 0) |
| [41] |
Shi S H, Tian L, Nasir F, et al. Impact of domestication on the evolution of rhizomicrobiome of rice in response to the presence of Magnaporthe oryzae[J]. Plant Physiology and Biochemistry, 2018, 132: 156-165. DOI:10.1016/j.plaphy.2018.08.023
(  0) 0) |
| [42] |
Liu Z J, Wang D G, Li T, et al. Isolation of bacteria in wild soybean rhizosphere and study on its function to reduce disadvantage of soybean continuous cropping (In Chinese)[J]. Soybean Science, 2007, 26(2): 176-180. DOI:10.3969/j.issn.1000-9841.2007.02.012 [刘昭军, 王德国, 李铁, 等. 野生大豆根际微生物的分离及其缓解大豆连作障碍的研究[J]. 大豆科学, 2007, 26(2): 176-180.]
(  0) 0) |
| [43] |
Zachow C, Muller H, Tilcher R, et al. Differences between the rhizosphere microbiome of Beta vulgaris ssp. Maritima-ancestor of all beet crops and modern sugar beets[J]. Frontiers in Microbiology, 2014, 5: 415. DOI:10.3389/fmicb.2014.00415
(  0) 0) |
| [44] |
Fernández-González A J, Cardoni M, Gómez-Lama Cabanás C, et al. Linking belowground microbial network changes to different tolerance level towards Verticillium wilt of olive[J]. Microbiome, 2020, 8(1): 11. DOI:10.1186/s40168-020-0787-2
(  0) 0) |
| [45] |
Kwak M J, Kong H G, Choi K, et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature Biotechnology, 2018, 36(11): 1100-1109. DOI:10.1038/nbt.4232
(  0) 0) |
| [46] |
Mendes L W, de Chaves M G, Fonseca M D C, et al. Resistance breeding of common bean shapes the physiology of the rhizosphere microbiome[J]. Frontiers in Microbiology, 2019, 10: 2252. DOI:10.3389/fmicb.2019.02252
(  0) 0) |
| [47] |
Yang L, Du Y X, Xu L J, et al. Effects of soil microecological environment on occurrence of 'black humor' disease in cherry tree (In Chinese)[J]. Soils, 2017, 49(2): 308-313. [杨璐, 杜岩新, 徐利娟, 等. 土壤微生态环境对樱桃树"黑疙瘩"病发生的影响[J]. 土壤, 2017, 49(2): 308-313.]
(  0) 0) |
| [48] |
Mendes L W, Raaijmakers J M, de Hollander M, et al. Influence of resistance breeding in common bean on rhizosphere microbiome composition and function[J]. The ISME Journal, 2018, 12(1): 212-224. DOI:10.1038/ismej.2017.158
(  0) 0) |
| [49] |
Mendes L W, Mendes R, Raaijmakers J M, et al. Breeding for soil-borne pathogen resistance impacts active rhizosphere microbiome of common bean[J]. The ISME Journal, 2018, 12(12): 3038-3042. DOI:10.1038/s41396-018-0234-6
(  0) 0) |
| [50] |
Iannucci A, Fragasso M, Beleggia R, et al. Evolution of the crop rhizosphere: Impact of domestication on root exudates in tetraploid wheat(Triticum turgidum L.)[J]. Frontiers in Plant Science, 2017, 8: 2124. DOI:10.3389/fpls.2017.02124
(  0) 0) |
| [51] |
Preece C, Peñuelas J. A return to the wild: Root exudates and food security[J]. Trends in Plant Science, 2020, 25(1): 14-21. DOI:10.1016/j.tplants.2019.09.010
(  0) 0) |
| [52] |
Walker T S, Bais H P, Grotewold E, et al. Root exudation and rhizosphere biology[J]. Plant Physiology, 2003, 132(1): 44-51. DOI:10.1104/pp.102.019661
(  0) 0) |
| [53] |
Mayrose M, Kane N C, Mayrose I, et al. Increased growth in sunflower correlates with reduced defences and altered gene expression in response to biotic and abiotic stress[J]. Molecular Ecology, 2011, 20(22): 4683-4694. DOI:10.1111/j.1365-294X.2011.05301.x
(  0) 0) |
| [54] |
Ruan Y L. Sucrose metabolism: Gateway to diverse carbon use and sugar signaling[J]. Annual Review of Plant Biology, 2014, 65(1): 33-67. DOI:10.1146/annurev-arplant-050213-040251
(  0) 0) |
| [55] |
Sasse J, Martinoia E, Northen T. Feed your friends: Do plant exudates shape the root microbiome?[J]. Trends in Plant Science, 2018, 23(1): 25-41. DOI:10.1016/j.tplants.2017.09.003
(  0) 0) |
| [56] |
Zhang N, Yang D Q, Kendall J R A, et al. Comparative genomic analysis of Bacillus amyloliquefaciens and Bacillus subtilis reveals evolutional traits for adaptation to plant-associated habitats[J]. Frontiers in Microbiology, 2016, 7: 2039.
(  0) 0) |
| [57] |
Biate D L, Kumari A, Annapurna K, et al. Legume root exudates: Their role in symbiotic interactions[M]//Plant microbes symbiosis: Applied facets. New Delhi: Springer India, 2014: 259-271.
(  0) 0) |
| [58] |
Peters N, Frost J, Long S. A plant flavone, luteolin, induces expression of Rhizobium meliloti nodulation genes[J]. Science, 1986, 233(4767): 977-980. DOI:10.1126/science.3738520
(  0) 0) |
| [59] |
Hassan S, Mathesius U. The role of flavonoids in root-rhizosphere signalling: Opportunities and challenges for improving plant-microbe interactions[J]. Journal of Experimental Botany, 2012, 63(9): 3429-3444. DOI:10.1093/jxb/err430
(  0) 0) |
| [60] |
Mutch L A, Young J P W. Diversity and specificity of Rhizobium leguminosarum biovar viciae on wild and cultivated legumes[J]. Molecular Ecology, 2004, 13(8): 2435-2444. DOI:10.1111/j.1365-294X.2004.02259.x
(  0) 0) |
| [61] |
An G H, Kobayashi S, Enoki H, et al. How does arbuscular mycorrhizal colonization vary with host plant genotype? An example based on maize(Zea mays) germplasms[J]. Plant and Soil, 2010, 327(1/2): 441-453.
(  0) 0) |
| [62] |
Lehmann A, Barto E K, Powell J R, et al. Mycorrhizal responsiveness trends in annual crop plants and their wild relatives-A meta-analysis on studies from 1981 to 2010[J]. Plant and Soil, 2012, 355(1/2): 231-250.
(  0) 0) |
| [63] |
Rodriguez-Saona C, Vorsa N, Singh A P, et al. Tracing the history of plant traits under domestication in cranberries: Potential consequences on anti-herbivore defences[J]. Journal of Experimental Botany, 2011, 62(8): 2633-2644. DOI:10.1093/jxb/erq466
(  0) 0) |
| [64] |
Rosenthal J P, Dirzo R. Effects of life history, domestication and agronomic selection on plant defence against insects: Evidence from maizes and wild relatives[J]. Evolutionary Ecology, 1997, 11(3): 337-355. DOI:10.1023/A:1018420504439
(  0) 0) |
| [65] |
Wink M. Plant breeding: Importance of plant secondary metabolites for protection against pathogens and herbivores[J]. Theoretical and Applied Genetics, 1988, 75(2): 225-233. DOI:10.1007/BF00303957
(  0) 0) |
| [66] |
Stenberg J A, Heil M, Åhman I, et al. Optimizing crops for biocontrol of pests and disease[J]. Trends in Plant Science, 2015, 20(11): 698-712. DOI:10.1016/j.tplants.2015.08.007
(  0) 0) |
| [67] |
Whitehead S R, Turcotte M M, Poveda K. Domestication impacts on plant-herbivore interactions: A meta-analysis[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2017, 372(1712): 20160034. DOI:10.1098/rstb.2016.0034
(  0) 0) |
| [68] |
Chen L, Xin X L, Zhang J B, et al. Soil characteristics overwhelm cultivar effects on the structure and assembly of root-associated microbiomes of modern maize[J]. Pedosphere, 2019, 29(3): 360-373. DOI:10.1016/S1002-0160(17)60370-9
(  0) 0) |
| [69] |
Köllner T G, Held M, Lenk C, et al. A maize(E)-β-caryophyllene synthase implicated in indirect defense responses against herbivores is not expressed in most American maize varieties[J]. The Plant Cell, 2008, 20(2): 482-494. DOI:10.1105/tpc.107.051672
(  0) 0) |
| [70] |
Ji L, Shi S H, Tian L, et al. A discussion on issues related to the recruit of rhizosphere microbes caused by crop domestication (In Chinese)[J]. Soils and Crops, 2019, 8(4): 368-372. [吉丽, 石绍华, 田磊, 等. 作物驯化对根际微生物组的选择[J]. 土壤与作物, 2019, 8(4): 368-372.]
(  0) 0) |
| [71] |
Berg G, Smalla K. Plant species and soil type cooperatively shape the structure and function of microbial communities in the rhizosphere[J]. FEMS Microbiology Ecology, 2009, 68(1): 1-13. DOI:10.1111/j.1574-6941.2009.00654.x
(  0) 0) |
| [72] |
Chang C L, Zhang J X, Liu T T, et al. Rhizosphere fungal communities of wild and cultivated soybeans grown in three different soil suspensions[J]. Applied Soil Ecology, 2020, 153: 103586. DOI:10.1016/j.apsoil.2020.103586
(  0) 0) |
| [73] |
Liu K, Ge Z, Xu Y D, et al. Responses of soil microbial community to drying-wetting alternation relative to tillage mode (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(1): 206-216. [刘奎, 葛壮, 徐英德, 等. 不同耕作方式下黑土微生物群落对干湿交替的响应[J]. 土壤学报, 2020, 57(1): 206-216.]
(  0) 0) |
| [74] |
Verbruggen E, Röling W F M, Gamper H A, et al. Positive effects of organic farming on below-ground mutualists: Large-scale comparison of mycorrhizal fungal communities in agricultural soils[J]. New Phytologist, 2010, 186(4): 968-979. DOI:10.1111/j.1469-8137.2010.03230.x
(  0) 0) |
| [75] |
Santos-González J C, Nallanchakravarthula S, Alström S, et al. Soil, but not cultivar, shapes the structure of arbuscular mycorrhizal fungal assemblages associated with strawberry[J]. Microbial Ecology, 2011, 62(1): 25-35. DOI:10.1007/s00248-011-9834-7
(  0) 0) |
| [76] |
Coleman-Derr D, Desgarennes D, Fonseca-Garcia C, et al. Plant compartment and biogeography affect microbiome composition in cultivated and native Agave species[J]. New Phytologist, 2016, 209(2): 798-811. DOI:10.1111/nph.13697
(  0) 0) |
| [77] |
Cassman N A, Leite M F A, Pan Y, et al. Plant and soil fungal but not soil bacterial communities are linked in long-term fertilized grassland[J]. Scientific Reports, 2016, 6: 23680. DOI:10.1038/srep23680
(  0) 0) |
| [78] |
Fox J E, Gulledge J, Engelhaupt E, et al. Pesticides reduce symbiotic efficiency of nitrogen-fixing rhizobia and host plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(24): 10282-10287. DOI:10.1073/pnas.0611710104
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


