2. 全球变化与生态预测研究中心, 上海市城市化生态过程与生态恢复重点实验室, 华东师范大学生态与环境科学学院, 上海 200241;
3. 中国科学院大学, 北京 100049;
4. 上海污染控制与生态安全研究院, 上海 200437;
5. 沈阳农田生态系统国家野外科学观测研究站, 沈阳 110016
2. Center for Global Change and Ecological Forecasting, Shanghai Key Lab for Urban Ecological Processes and Eco-Restoration, School of Ecological and Environmental Sciences, East China Normal University, Shanghai 200241, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Shanghai Institute of Pollution Control and Ecological Security, Shanghai 200437, China;
5. National Field Research Station of Shenyang Agroecosystems, Shenyang 110016, China
土壤微生物驱动并调控土壤有机碳(SOC)的形成、分解和稳定化过程[1]。但是,由于微生物数量巨大、群落组成复杂[2],不同微生物类群的底物利用策略和功能难以确定,尤其是无法鉴定参与SOC转化的活性微生物类群[3]。近年来高通量测序技术快速发展,可明确海量微生物的分子系统发育分类特征;同时,稳定性同位素示踪复杂土壤中微生物核酸DNA技术(DNA-based stable isotope probing,DNA-SIP)则可从无法计数的微生物类群中准确鉴定活性微生物。因此,耦合DNA-SIP与高通量测序技术,通过分析13C-DNA微生物的系统发育分类关系,可准确解析微生物功能类群对外源有机底物的响应和利用规律[4-5]。低分子量底物(如葡萄糖)是微生物生长过程中可利用的重要活性碳源。在利用13C-葡萄糖为底物的培养实验中,已有的研究发现土壤中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)等优先利用葡萄糖[6-7]。变形菌门多被认为是富营养菌,但是关于放线菌门的营养类型划分仍具有一定的争议[8-9]。而隶属于寡营养菌的酸杆菌门(Acidobacteria)和绿弯菌门(Chloroflexi)则未被葡萄糖来源的13C所标记,表明这些微生物类群对活性碳源的利用能力较低[10]。细菌的底物利用策略可能和细菌功能类群的内部竞争或合作动态有关,存在复杂的动态变化。然而,以往研究多关注细菌对底物利用的瞬时性响应[11],不同时间的动态响应报道较少,且细菌的底物利用策略是否与底物浓度相关仍不清楚。
真菌的物种多样性明显低于细菌,大多数研究认为真菌在降解难分解底物时发挥更重要的作用[12],如利用纤维素和木质素等进行培养时发现子囊菌门可利用这些难分解组分[13],但13C-葡萄糖为底物的培养实验中也发现,子囊菌门(Ascomycota)是活性碳的主要分解者[14]。此外,也有报道表明担子菌门(Basidiomycota)既能利用难分解有机物质(例如纤维素,半纤维素和植物残体等),也可以利用根系分泌物[13-15]。这些结果表明,真菌可能对不同的底物类型保持着广谱性利用特征,底物选择性较低。然而,在更加精细化的系统发育分子分类水平上,真菌群落对不断变化的碳底物的利用策略仍然关注较少。
据此,采用DNA–SIP和高通量测序技术,结合室内土壤微宇宙培养,通过13C-标记的葡萄糖连续添加,研究不同培养期间活性的13C-真菌和细菌类群演替规律,明确区分细菌和真菌的底物利用策略与传统营养类型的关系。
1 材料与方法 1.1 土壤样品红壤(Ultisol,0~20 cm)于2014年7月采自江西省鹰潭市余江县中国科学院红壤生态试验站(海拔65 m,北纬28°15′、东经116°55′)。土壤样品按照“Z”形取样方案,采集十点后进行混合。土壤样品过2 mm筛后备用。土壤pH 4.46(土︰水=1︰2.5),有机碳5.67 g·kg–1,全氮0.67 g·kg–1,可溶性有机碳4.27 mg·kg–1,有效磷5.25 mg·kg–1,速效钾84.65 mg·kg–1,黏粒、粉粒和砂粒含量41.2%、33.2%和25.6%[16]。
1.2 稳定性同位素示踪培养实验称取30 g土壤置于培养瓶中,加入KH2PO4溶液(每克土壤0.2 mg P,0.22 mg K)。调节含水量至土壤最大持水量的60%,置于25℃恒温培养箱中进行预培养1周,3次重复。预培养结束后,将13C标记的葡萄糖(13C-glucose)加入培养瓶中。标记底物添加培养组合为:土壤+H2O为对照处理(设置为0周),目的在于检测未添加底物时土壤初始微生物群落;土壤+葡萄糖+NH4NO3作为DNA–SIP的对照组;土壤+13C-glucose(99 atom% 13C)+NH4NO3作为DNA–SIP的实验组。添加硝酸铵目的是满足微生物对养分的基本需求,依据Paul和Clark[17]对微生物底物利用效率的推算,本培养实验底物添加量为C 1 mg·g–1土壤、N 0.1 mg·g–1土壤,底物C︰N=10︰1。碳氮底物每周加入一次,共培养8周,分别在底物加入培养1周、3周和6周后进行破坏性采样,样品置于–80℃用于提取土壤总DNA。
1.3 土壤微生物DNA的提取利用FastDNA® Spin Kit for Soil(MP Biomedicals公司)试剂盒提取土壤基因组总DNA,具体流程如下:称取0.5 g鲜土于2.0 mL离心管中,加入978 μL SPB和122 μL MT缓冲液,在核酸提取仪(Fast Prep® FP120)上以6.0 m·s–1的速度裂解微生物细胞30 s,随后离心10 min(14 000×g),上清液移至灭菌离心管(2.0 mL)中,加入250 μL PPS,震荡离心后转移上清液至15 mL灭菌离心管,加入1.0 mL Binding Matrix,震荡2 min后静止,以保证提取的DNA在硅胶上吸附并能沉淀至离心管的下部。用移液枪摒弃500 μL上清液,将剩余液体分次全部转移至SPINTM离心管滤膜上并离心1 min(14 000×g),弃滤液;滤膜上加入500 μL的SEWS-M的乙醇溶液,14 000×g离心1 min,弃滤液;然后将滤膜移至新的2.0 mL离心管中,14 000×g离心2 min;重复滤膜转移操作,室温下干燥5 min;最后将提取得到的土壤总DNA溶解于70 μL无菌TE缓冲液(10 mmol·L–1 Tris-HCl,1 mmol·L–1 EDTA,pH 8.0)。通过微量紫外分光光度计(NanoDrop® ND-1000 UV-Vis)对提取的土壤DNA测定DNA浓度和纯度(OD260/OD280和OD260/OD230)。利用TE缓冲液对土壤DNA进行10倍梯度稀释并进行PCR扩增。土壤DNA稀释10倍后保存于–20℃冰箱待用。
1.4 超高速密度梯度离心DNA和离心后DNA的分离纯化将土壤DNA(2.0 μg)与氯化铯(Cesium chloride,CsCl)溶液进行混合,密度调节为1.725 g·mL–1的离心溶液(pH 8.0;100 mmol·L–1 Tris-HCl;100 mmol·L–1 KCl;1.0 mmol L–1·EDTA),随后转移至体积为5.1 mL的超高速离心试管(贝克曼);采用贝克曼垂直转子Vti 65.2(Beckman Coulter,Inc.,Palo Alto,CA,USA)在20℃环境下以177 000×g的速度离心44 h;随后采用NE-1000固定流速泵(New Era Pump Systems,Inc.,Farmingdale,NY,USA)通过超纯水从离心管顶部置换出来,置换时的流速为0.38 mL·min–1。共收集15个等体积的DNA分层样品,每层样品体积大约380 μL;分层溶液结束后,利用AR200手持式折光仪(Reichert Inc.,Buffalo,NYUSA)测定浮力密度。
离心后DNA的分离纯化主要包括以下步骤:分层后的DNA溶液用PEG 6000在37℃沉淀1 h;再置于高速离心机13 000×g离心30 min,除去CsCl溶液;随后加入500 µL 70 %乙醇清洗DNA沉淀,离心10 min,除去上清液;重复加入乙醇以进一步去除CsCl和PEG 6000;最后将DNA沉淀物在室温下干燥15 min,溶于30 μL的TE buffer溶液中,保存于–20℃冰箱。
1.5 土壤中微生物各分层DNA同位素比值测定溶解于TE buffer溶液中的各分层DNA的13C丰度采用同位素比例质谱仪(IRMS,Delta plus XP,ThermoFinnigan,USA)耦合元素分析仪(Flash HT 2000,Thermo Fisher,USA)测定,具体方法如下,利用移液枪称取5 μL分层DNA(如上所述溶于TE buffer缓冲液内含分层DNA)置于锡舟中,于60℃烘干2 h,烘干后的样品进行质谱分析[18],δ13C(‰)值利用下列公式计算:δ13C(‰)=[(Rsample/Rstandard)–1]×1000,其中R=13C/12C,Rsample和Rstandard分别代表样品和标准物质(Pee Dee Belemnite)的同位素比值。目前,IRMS质谱分析中碳含量的最低检测限约为10~15 μg C,本实验中上机样品中包含的含碳化合物主要是DNA样本,TE缓冲液(含Tris-base和EDTA等有机试剂)或其他难以完全分离的高质量的含碳化合物,如PEG 6000等,根据实验过程中所获得的谱图可以得知本实验中上机样品可以满足最低检测限。
1.6 实时荧光定量PCR分层DNA中的细菌和真核基因拷贝数(绝对丰度)采用实时荧光定量PCR(quantitative real-time polymerase chain reaction,qPCR)。标准曲线用细菌16S rRNA gene和真核18S rRNA gene的代表性克隆重组质粒进行10倍稀释,稀释梯度为6到8个。细菌16S rRNA gene和真核18S rRNA gene标准曲线的拷贝数范围分别为4.07×102~4.07×108(R2= 0.985)和1.44×102~1.44×108(R2=0.994)。空白样品的模板为水。根据标准曲线的浓度计算出样品中的基因拷贝数,最后以每克干土重的基因拷贝数为单位进行分析。每个样品3次重复。qPCR的反应体系为20 μL,包括1 μL DNA模板、10 μL SYBR Premix Ex TaqTM(TaKaRa Biotech,Dalian,China),正向和反向引物分别为0.25 μL(50 μmol·L–1)和8.5 μL的灭菌双蒸水(引物及PCR条件见表 1)。定量PCR产物用浓度为1.2%的琼脂糖凝胶电泳检测扩增特异性。
|
|
表 1 通用引物和PCR扩增反应 Table 1 Primers used and PCR amplification reaction in this study |
对于超高速密度梯度离心获得的各浮力密度层DNA,首先利用通用引物扩增其中的土壤微生物16S rRNA gene和18S rRNA gene,将PCR产物纯化后分别将其等摩尔混合进行测序。用于测序的通用引物含有11个碱基的特异Tag标签,用以区分不同的分层样品。通用引物和PCR扩增反应见表 1。获得扩增产物后,利用Agarose Gel DNA Fragment Recovery Kit Ver. 2.0试剂盒(Takara)切胶纯化并将其溶于30 μL去离子水中。进一步通过2.0%琼脂糖凝胶电泳检测PCR产物纯化效果,利用微量紫外分光光度计(NanoDrop ND- 1000 UV-Vis)测定纯化后PCR产物的浓度。将分层样品16S rRNA gene和18S rRNA gene的PCR纯化产物分别等摩尔数混合,于Illumina Miseq测序仪(Illumina,San Diego,CA,USA)进行测序分析。
原始测序数据主要使用QIIME[19]和UPARSE[20]进行处理。主要步骤包括:去除原始数据的接头序列,利用FLASH将双端测序序列拼接成单条序列[21];根据测序特异Tag标签区分不同的样本序列并保留平均质量得分高于20的序列;样品的靶序列读数以97%的相似性聚类为操作分类单元(OTU),并使用UPARSE丢弃嵌合序列,根据OTU数据对土壤细菌和真菌进行下游分析。虽然18S rRNA gene的真菌种类分辨率有限,但本实验中测序结果表明70%以上的培养样品中真菌分类学分辨率远大于60%。16S rRNA gene和18S rRNA gene均使用Silva 123作为参考数据库,通过QIIME中的Assign_taxonomy.py工具为每个代表性序列分配分类注释[22]。原始测序数据上传至美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)(https://www.ncbi.nlm.nih.gov/)的BioProject数据库中,序列号为PRJNA487919。
1.8 数据处理及分析采用Excel(Microsoft Office 2010)处理数据,利用SAS 9.2软件对试验数据进行重复测量单因素方差分析(repeated-ANOVA)。图表主要利用Origin 9.0绘制。根据Zumsteg等[26]和Pepe-Ranney等[27]中提到的重层与轻层的“差减法”和“丰度差异”,利用13C-标记的葡萄糖和未标记葡萄糖培养的重层和轻层DNA中的不同分类单元计算了重层中微生物的富集因子(Enrichment Factors,EF)[6],计算公式如下:EF=RAh/RAl(13C)-RAh/RAl(12C),其中RAh(13C)和RAl(13C)表示13C-标记的葡萄糖处理下重层和轻层中的分类单元的相对丰度;RAh(12C)和RAl(12C)表示未标记的葡萄糖处理下重层和轻层中的分类单元的相对丰度。13C-标记的细菌或真菌门,需满足以下2点:1)13C-标记的葡萄糖处理下重层中的细菌门相对丰度大于轻层中相对丰度;2)EF > 0.5。
2 结果 2.1 土壤中细菌和真菌各分层样本DNA的同位素比值和基因拷贝数的相对分布和未添加葡萄糖处理相比,13C-葡萄糖标记处理分层DNA中16S rRNA gene和18S rRNA gene拷贝数随浮力密度的增加呈现一个明显峰值(图 1A,图 1B),说明细菌和真核基因组DNA被明显标记,并且随着底物的连续添加,标记程度不断增加。此外,13C标记后细菌和真核基因拷贝数出现的峰值主要集中在3~7层(除第一周的18S rRNA基因拷贝数外),而未标记的葡萄糖处理下的峰值主要集中在9~11层。进一步应用质谱分析技术表明土壤微生物被13C所标记,利用13C-葡萄糖培养后红壤DNA的13C丰度(δ13C)在重密度层3~5层最高,其他各浮力密度层的δ13C值均相对较低(图 1C)。在轻密度层9~11层,无论葡萄糖是否标记,各分层DNA的δ13C值均无显著差异。因此,选取3、4、5层为重层DNA的代表层,即13C-DNA,该重层DNA所代表的土壤微生物主要参与外源葡萄糖利用,是对外源物质响应最为活跃的微生物区系;而9、10、11层则为轻层DNA的代表层,即12C-DNA,该轻层DNA可能主要参与土壤原有有机碳的分解过程。
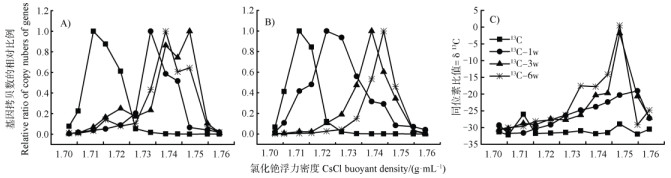
|
注:相对比值通过将每层中的基因拷贝数除以所有层次中最多的基因拷贝数得到。 Note: Relative ratio was obtained by dividing the highest number of gene copies of all layers with the number of gene copies in each layer. 图 1 各浮力密度层DNA的细菌(A)、真核(B)基因的相对分布及同位素比值(C) Fig. 1 Relative distribution of Bacterial 16S rRNA genes, Eukaryotic 18S rRNA genes and δ13C value in each buoyant density layer, bacteria(A), Eukaryota(B)and δ13C value(C) |
土壤13C-DNA中的细菌和真菌基因拷贝数均明显高于12C-DNA,且两者的比值随时间不断增加(图 2A—图 2C)。和对照处理(0 w)相应层次相比,13C-DNA中细菌基因拷贝数在培养一周后增加约2.99倍,而真菌基因拷贝数反而较对照减少45%。随着底物的不断添加,13C-DNA中细菌基因拷贝数不断下降,但真菌基因拷贝数随培养时间急剧增加(图 2A,图 2B)。因此,13C-DNA中真菌和细菌基因拷贝数的比值随培养时间不断增加(图 2D)。
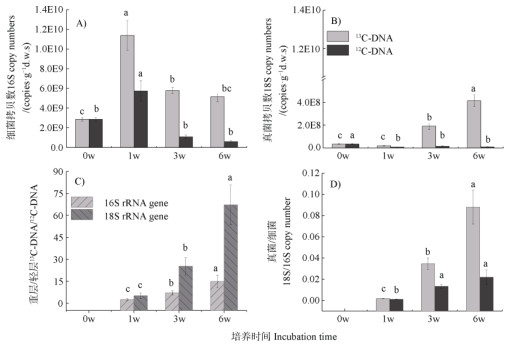
|
注:(A)细菌基因拷贝数,(B)真核基因拷贝数,(C)细菌和真核重层和轻层中基因拷贝数比值,(D)重层和轻层中真菌与细菌基因拷贝数的比值。横坐标轴中的0 w、1 w、3 w和6 w分别表示对照处理及添加底物后培养1周、3周和6周。误差棒表示标准误(n = 9)。不同字母表示重层或轻层中不同培养时间的差异显著性(P < 0.05)。 Note: (A) Bacterial 16S rRNA gene copy numbers, (B) Eukaryotic 18S rRNA gene copy numbers; (C) Ratios of the 13C-DNA and 12C-DNA in 18S rRNA and 16S rRNA genes, (D) Ratio of 18S rRNA and 16S rRNA copy numbers in 13C-DNA and 12C-DNA. 0 w, 1 w, 3 w and 6 w in X-axis indicate control treatment, incubated 1 week, 3 weeks and 6 weeks after substrate addition. Error bar represents standard errors(n = 9). Different letters indicate significant differences between samples different in incubation time in 13C-DNA or 12C-DNA(P < 0.05). 图 2 不同培养时间细菌和真菌重层轻层中基因拷贝数及比值变化 Fig. 2 Changes of Bacterial, eukaryotic 18S rRNA gene copy numbers and ratios of them in 13C-DNA and 12C-DNA under different incubation time |
培养过程中,13C-DNA中变形菌门和放线菌门的相对丰度显著均高于对照处理(图 3A)。变形菌门的相对丰度在第3周达到最高值,从初始的14%增加至59%,随后下降至49%。放线菌门初始的相对丰度约为7%,但培养1周后即达到最高值约45%,随后显著下降,在培养3周和6周后相对丰度约为20%。对照处理中厚壁菌门(Firmicutes)的相对丰度仅约为1%,但培养1周后即达到16%,然后降至不足5%。酸杆菌门和绿弯菌门在初始土壤中的相对丰度约为30%。尽管随着外源葡萄糖的不断添加,这两种门的相对丰度有所增加,但其相对丰度在整个培养期间仍显著低于对照处理。
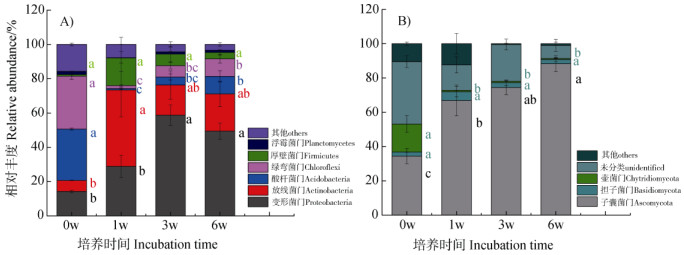
|
注:不同颜色字母表示同一细菌门在不同培养时间的差异显著性(P < 0.05)。 Note: Different colors of the letters indicate significant differences between samples in incubation time in the same bacterial phylum(P < 0.05). 图 3 不同培养时间下重层中细菌(A)和真菌(B)优势门的相对丰度 Fig. 3 Relative abundance of dominant bacterial(A)and fungal(B)phyla in 13C-DNA under different incubation time |
在添加13C-葡萄糖培养过程中,变形菌门的EF值始终大于0.5,变化范围为0.5~4.3。放线菌门的EF值约为1.1~1.5。厚壁菌门的EF值在1周和3周培时大于0.5,但培养6周后下降至零以下。尽管随着外源底物的添加酸杆菌门和绿弯菌门的EF值呈现增加趋势,但在整个培养过程中其EF值仍小于0.5,主要为负值(表 2)。
|
|
表 2 不同培养时间下细菌和真菌优势门的富集比例 Table 2 Enrichment factors(EF)of dominant bacterial and fungal phyla relative to incubation time |
子囊菌门、担子菌门和壶菌门(Chytridiomycota)是对照处理土壤中的优势门,相对丰度分别为34%、2%和16%。加入13C-标记葡萄糖后,13C-DNA中子囊菌门的相对丰度在培养期间显著增加,到第6周其相对丰度达到88%。但是,子囊菌门的EF值不断下降,从第1周的3.8逐渐降低至第6周的0.5。担子菌门在13C-DNA中的相对丰度在培养期间并未发生显著变化,平均约为3%。担子菌门的EF值在第1周为0.6,在培养后期则下降至负值。壶菌门的相对丰度在培养期间均低于1%,且其EF值均为负值(图 3B,表 2)。
3 讨论 3.1 真菌和细菌对活性底物的响应和利用特征在培养微域中连续添加13C-葡萄糖后,红壤真菌和细菌13C-DNA和12C-DNA中基因拷贝数均不断增加(图 2),说明微生物既可以利用外源葡萄糖碳,也可以利用土壤原有组分进行自身生长。但是,13C-DNA中真菌和细菌基因拷贝数明显高于12C-DNA且两者的比值随着葡萄糖的加入而不断增加,表明土壤中微生物优先利用活性底物维持生长和代谢,从而抑制了微生物对土壤中原有稳定组分的利用[28]。添加13C-葡萄糖后,真菌基因拷贝数的初始增加速率明显低于细菌,表明具有r-策略特征的细菌利用活性底物的能力高于真菌[14]。随着外源碳底物的输入,真、细菌基因拷贝数的比值不断增加,说明作为K-策略者的真菌在培养的后期显著提高了利用外源碳底物的能力,使土壤微生物的响应由细菌逐渐转为以真菌为主导,从而产生微生物群落组成的演替。
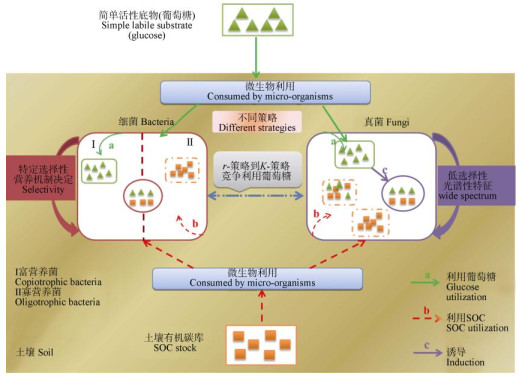
|
图 4 红壤中细菌和真菌底物利用策略概念图 Fig. 4 Concept graph of substrate utilization strategies of bacteria and fungi in Ultisol |
培养过程中13C-DNA和12C-DNA中细菌门类相对丰度和EF值的动态变化表明,不同种类细菌对外源活性碳底物的利用策略不同。变形菌门和放线菌门在13C-DNA中的相对丰度显著增加,且其EF值均大于0.5(图 3A,表 2),说明这两种细菌门对活性碳底物响应迅速,对葡萄糖碳的竞争能力强[6,10]。变形菌门多被认为是富营养菌,主要以竞争利用活性碳底物作为生存策略[28]。虽然对放线菌门的底物偏好性存在争议,但是多数研究发现放线菌门在活性底物丰富的条件下响应比较活跃[11,29]。在本培养实验中,放线菌门对于葡萄糖碳具有明显响应,表明放线菌具有较强的活性底物利用能力。
尽管变形菌门和放线菌门均可利用活性碳底物,但两种细菌门的相对丰度随培养时间的变化动态并不一致。初始加入葡萄糖后,放线菌门相对丰度的增加幅度明显高于变形菌门,表明放线菌门对活性底物的竞争能力高于变形菌门。有研究认为,和变形菌门内部种群相比,放线菌门中可能有较多的基因参与葡萄糖碳的利用[30],从而增加其竞争能力。然而,由于放线菌门中总转运蛋白数量少,对特定底物具有高亲和力,这可能导致该门类在高营养浓度下的增殖具有饱和性[30]。因此,连续添加葡萄糖培养3周后,变形菌门的相对丰度显著增加并高于放线菌门,表明变形菌门对活性底物的持续利用能力显著高于后者。随着葡萄糖持续添加,变形菌门和放线菌门的相对丰度逐渐相等,表明底物可利用性提高可减弱富营养菌对外源活性碳的竞争。
3.2.2 厚壁菌门在以往研究中,厚壁菌门多被认为是富营养菌[7,27],因此厚壁菌门对初始葡萄糖添加产生了快速响应。但是,厚壁菌门的相对丰度随着底物的添加不断下降,其EF值也逐渐降低并转为负值(图 3A,表 2),表明厚壁菌门营养机制的分类仍具有不确定性。根据厚壁菌门底物利用特征的动态变化,推测厚壁菌门可能作为一种“机会主义”细菌参与底物利用[9-10],这类细菌对底物的利用往往具有较低的竞争力,但却可以通过降低底物选择性在不同的生境中生存。
3.2.3 酸杆菌门和绿弯菌门在培养微域添加葡萄糖后,酸杆菌门和绿弯菌门在13C-DNA中的相对丰度均低于对照处理(图 3A),说明酸杆菌门和绿弯菌门对活性碳底物利用能力较低,这可能与其具有寡营养特性有关[7,10]。研究已发现,酸杆菌门在活性底物较丰富的环境中丰度较低,在营养较缺乏时含量较丰富[31]。绿弯菌门在以往的研究中也多被定义为寡营养菌[11]。随着13C-葡萄糖的添加,13C-DNA中酸杆菌门和绿弯菌门的相对丰度不断增加,但其EF值仍保持负值(表 2),进一步说明即使培养微域中存在大量活性底物,这两种菌群可能仍保持其寡营养特性,可通过利用土壤原有组分以及部分葡萄糖衍生的土壤碳进行慢速生长。造成寡营养菌这种底物利用策略的原因之一在于高浓度的可利用性底物在一定程度上也会引起寡营养细菌细胞中的生长失衡,可能作为有毒物质来阻止细胞中蛋白质的合成[32]。
综上所述,在高活性碳底物同化过程中,尽管不同的优势细菌门对底物输入的动态响应在时间上并不完全一致,但细菌的底物利用策略关键决定于细菌的寡、富营养类型。富营养菌优先利用活性碳底物,而寡营养菌对土壤原有组分利用能力较强,表明细菌群落的生理分类具有保守进化特征并保持稳定的底物代谢功能,而且这一底物利用策略并未随着底物数量的增加而发生改变。
3.3 真菌底物利用策略添加13C-葡萄糖培养1周后,13C-DNA中子囊菌门和担子菌门的相对丰度显著增加,且其EF值高于0.5(图 3B,表 2),说明真菌的优势门可优先利用活性碳源。随着葡萄糖的添加,子囊菌门和担子菌门的EF值随培养时间不断下降,说明真菌对外源碳底物的选择性逐渐降低。其原因可能在于真菌利用外源碳底物的同时可能会诱导其对土壤原有组分(SOC)的利用,从而降低真菌对外源活性底物的偏好。可见,真菌不仅可利用活性底物,同时也可以利用具有一定稳定性的土壤组分。即使有大量可利用性碳供给,真菌依然可以保持底物利用的广谱性。
和对照处理相比,子囊菌门的相对丰度增加较快而担子菌门的相对丰度无显著变化,说明子囊菌门对外源碳底物的竞争能力和响应能力均强于后者。此外,担子菌门的EF值随葡萄糖数量的增加而不断下降直至负值,说明担子菌门利用外源碳底物的能力不断降低,可能与其寡营养特性有关[33]。因此,和细菌的底物利用策略相比,真菌对底物利用的选择性较低,但是,真菌的底物利用策略在系统发育水平上也会遵循保守性特性。
4 结论本研究利用DNA–SIP技术解析了特定底物诱导的微生物响应,从而阐明真菌和细菌微生物底物利用特征具有特异性。葡萄糖添加后,细菌和真菌的增殖与其固有的r/K策略有关,作为r-策略者的细菌可以迅速响应并利用葡萄糖,而真菌作为K-策略者主要在培养的后期利用外源碳底物。无论底物的可利用性如何变化,细菌对底物利用具有特定选择性,主要由营养机制决定,微生物对外源活性碳底物的偏好利用归因于其富营养菌和寡营养菌的生活史策略。即使培养微域中外源可利用碳活性不断增加,不同门类细菌仍可维持其内在的营养机制,说明微生物在利用外源碳底物时在系统发育上具有保守性。相比之下,在外源碳底物添加下,真菌对底物的选择具有低选择性,主要归因于真菌对底物利用的广谱性特征。因此,在外源高活性底物(葡萄糖)供给条件下,活跃的微生物是土壤有机碳转化的关键驱动力,微生物对特定底物的利用特征可以在一定程度上介导土壤有机碳循环。
致谢 感谢中国科学院南京土壤研究所贾仲君研究员团队在样品测试和分析中给予的帮助,感谢中国科学院红壤生态试验站在土壤采集过程中给予的帮助。
| [1] |
Trivedi P, Delgado-Baquerizo M, Trivedi C, et al. Microbial regulation of the soil carbon cycle: Evidence from gene-enzyme relationships[J]. The ISME Journal, 2016, 10(11): 2593-2604. DOI:10.1038/ismej.2016.65
(  0) 0) |
| [2] |
Louca S, Polz M F, Mazel F, et al. Function and functional redundancy in microbial systems[J]. Nature Ecology & Evolution, 2018, 2(6): 936-943.
(  0) 0) |
| [3] |
de Nobili M, Contin M, Mondini C, et al. Soil microbial biomass is triggered into activity by trace amounts of substrate[J]. Soil Biology & Biochemistry, 2001, 33(9): 1163-1170.
(  0) 0) |
| [4] |
Jia Z J. Principle and application of DNA-based stable isotope probing-A review (In Chinese)[J]. Acta Microbiologica Sinica, 2011, 51(12): 1585-1594. [贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用[J]. 微生物学报, 2011, 51(12): 1585-1594.]
(  0) 0) |
| [5] |
Lin Z, Sun L Y, Chen H, et al. Application of stable isotope probing on in situ identification of soil functional microorganism (In Chinese)[J]. Journal of Agro-Environment Science, 2012, 31(1): 1-6. [蔺中, 孙礼勇, 陈昊, 等. 稳定性同位素探针技术在土壤功能微生物原位鉴定的应用[J]. 农业环境科学学报, 2012, 31(1): 1-6.]
(  0) 0) |
| [6] |
Kramer S, Dibbern D, Moll J, et al. Resource partitioning between bacteria, fungi, and protists in the detritusphere of an agricultural soil[J]. Frontiers in Microbiology, 2016, 7: 1524.
(  0) 0) |
| [7] |
Verastegui Y, Cheng J, Engel K, et al. Multisubstrate isotope labeling and metagenomic analysis of active soil bacterial communities[J]. mBio, 2014, 5(4): e01157-e01114.
(  0) 0) |
| [8] |
Eilers K G, Lauber C L, Knight R, et al. Shifts in bacterial community structure associated with inputs of low molecular weight carbon compounds to soil[J]. Soil Biology & Biochemistry, 2010, 42(6): 896-903.
(  0) 0) |
| [9] |
Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria[J]. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839
(  0) 0) |
| [10] |
Arcand M M, Levy-Booth D J, Helgason B L. Resource legacies of organic and conventional management differentiate soil microbial carbon use[J]. Frontiers in Microbiology, 2017, 8: 2293. DOI:10.3389/fmicb.2017.02293
(  0) 0) |
| [11] |
Ai C, Liang G Q, Sun J W, et al. Reduced dependence of rhizosphere microbiome on plant-derived carbon in 32-year long-term inorganic and organic fertilized soils[J]. Soil Biology & Biochemistry, 2015, 80: 70-78.
(  0) 0) |
| [12] |
Paterson E, Osler G, Dawson L A, et al. Labile and recalcitrant plant fractions are utilised by distinct microbial communities in soil: Independent of the presence of roots and mycorrhizal fungi[J]. Soil Biology & Biochemistry, 2008, 40(5): 1103-1113.
(  0) 0) |
| [13] |
Hannula S E, Boschker H T, de Boer W, et al. 13C pulse-labeling assessment of the community structure of active fungi in the rhizosphere of a genetically starch-modified potato(Solanum tuberosum) cultivar and its parental isoline[J]. New Phytologist, 2012, 194(3): 784-799. DOI:10.1111/j.1469-8137.2012.04089.x
(  0) 0) |
| [14] |
Kong Y L, Zhu C, Ruan Y, et al. Are the microbial communities involved in glucose assimilation in paddy soils treated with different fertilization regimes for three years similar[J]. Journal of Soils and Sediments, 2018, 18(7): 2476-2490. DOI:10.1007/s11368-018-1961-z
(  0) 0) |
| [15] |
van der Wal A, Geydan T D, Kuyper T W, et al. A thready affair: Linking fungal diversity and community dynamics to terrestrial decomposition processes[J]. FEMS Microbiology Reviews, 2013, 37(4): 477-494. DOI:10.1111/1574-6976.12001
(  0) 0) |
| [16] |
Lin Y X, Ye G P, Liu D Y, et al. Long-term application of lime or pig manure rather than plant residues suppressed diazotroph abundance and diversity and altered community structure in an acidic Ultisol[J]. Soil Biology & Biochemistry, 2018, 123: 218-228.
(  0) 0) |
| [17] |
Paul E A, Clark F E. Methods for studying soil microorganisms . San Diego: Academic Press, 1996: 184-188.
(  0) 0) |
| [18] |
Haichar F Z, Marol C, Berge O, et al. Plant host habitat and root exudates shape soil bacterial community structure[J]. The ISME Journal, 2008, 2(12): 1221-1230. DOI:10.1038/ismej.2008.80
(  0) 0) |
| [19] |
Caporaso J G, Kuczynski J, Stombaugh J, et al. Qiime allows analysis of high throughput community sequencing data[J]. Nature Methods, 2010, 7: 335-336. DOI:10.1038/nmeth.f.303
(  0) 0) |
| [20] |
Edgar R. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10: 996-998. DOI:10.1038/nmeth.2604
(  0) 0) |
| [21] |
Magoč T, Salzberg S L. FLASH: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507
(  0) 0) |
| [22] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41: D590-D596.
(  0) 0) |
| [23] |
Xia W W, Zhang C X, Zeng X W, et al. Autotrophic growth of nitrifying community in an agricultural soil[J]. The ISME Journal, 2011, 5(7): 1226-1236. DOI:10.1038/ismej.2011.5
(  0) 0) |
| [24] |
Bailly J, Fraissinet-Tachet L, Verner M C, et al. Soil eukaryotic functional diversity, a metatranscriptomic approach[J]. The ISME Journal, 2007, 1(7): 632-642. DOI:10.1038/ismej.2007.68
(  0) 0) |
| [25] |
Zhou X Q, Guo Z Y, Chen C R, et al. Soil microbial community structure and diversity are largely influenced by soil ph and nutrient quality in 78-year-old tree plantations[J]. Biogeosciences, 2017, 14(8): 2101-2111. DOI:10.5194/bg-14-2101-2017
(  0) 0) |
| [26] |
Zumsteg A, Schmutz S, Frey B. Identification of biomass utilizing bacteria in a carbon-depleted glacier forefield soil by the use of 13C DNA stable isotope probing[J]. Environmental Microbiology Reports, 2013, 5(3): 424-437. DOI:10.1111/1758-2229.12027
(  0) 0) |
| [27] |
Pepe-Ranney C, Campbell A N, Koechli C N, et al. Unearthing the ecology of soil microorganisms using a high resolution DNA-sip approach to explore cellulose and xylose metabolism in soil[J]. Frontiers in Microbiology, 2016, 7: 703.
(  0) 0) |
| [28] |
di Lonardo D P, de Boer W, Klein Gunnewiek P J A, et al. Priming of soil organic matter: Chemical structure of added compounds is more important than the energy content[J]. Soil Biology & Biochemistry, 2017, 108: 41-54.
(  0) 0) |
| [29] |
Goldfarb K C, Karaoz U, Hanson C A, et al. Differential growth responses of soil bacterial taxa to carbon substrates of varying chemical recalcitrance[J]. Frontiers in Microbiology, 2011, 2: 94.
(  0) 0) |
| [30] |
Trivedi P, Anderson I C, Singh B K. Microbial modulators of soil carbon storage: Integrating genomic and metabolic knowledge for global prediction[J]. Trends in Microbiology, 2013, 21(12): 641-651. DOI:10.1016/j.tim.2013.09.005
(  0) 0) |
| [31] |
Wang J C, Xue C, Song Y, et al. Wheat and rice growth stages and fertilization regimes alter soil bacterial community structure, but not diversity[J]. Frontiers in Microbiology, 2016, 7: 1207. DOI:10.3389/fmicb.2016.01207/pdf
(  0) 0) |
| [32] |
Koch A L. Oligotrophs versus copiotrophs[J]. Problems and Paradigms, 2001, 23: 657-661.
(  0) 0) |
| [33] |
Yao F, Yang S, Wang Z R, et al. Microbial taxa distribution is associated with ecological trophic cascades along an elevation gradient[J]. Frontiers in Microbiology, 2017, 8: 2071. DOI:10.3389/fmicb.2017.02071
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


