2. 土肥资源高效利用国家工程实验室, 山东农业大学资源与环境学院, 山东泰安 271018;
3. 郑州轻工业大学食品与生物工程学院, 郑州 450002;
4. 中国科学院南京土壤研究所, 南京 210008
2. National Engineering Laboratory for Efficient Utilization of Soil and Fertilizer Resources, College of Resources and Environment, Shandong Agricultural University, Tai'an, Shandong 271018, China;
3. College of Food and Biological Engineering, Zhengzhou University of Light Industry, Zhengzhou 450002, China;
4. Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
甲烷(CH4)和氧化亚氮(N2O)是两种重要的温室气体,其全球增温潜势分别是二氧化碳(CO2)的28倍~34倍和265倍~298倍[1-2]。旱地土壤(森林土壤、草原土壤等)是大气甲烷的有效汇,因其具有能够氧化大气浓度甲烷的高亲和力甲烷氧化单加氧酶(MMO)[3]。好氧土壤中,N2O的主要产生途径是有氧硝化作用。氨氧化过程是有氧硝化的第一步,也是限速步骤,主要由具有氨单加氧酶(AMO)的氨氧化古菌(AOA)和氨氧化细菌(AOB)完成。研究发现,MMO在进化上和AMO密切相关,它们有高度相似的氨基酸序列、相似的蛋白复合体结构、相似结构的底物(CH4和NH3)、相似的被抑制特性和相似的生态位,而且都竞争N源[4]。甲烷氧化细菌(MOB)的生长繁殖需要氮源,但其自身不具备固氮能力,因此,MOB需要获取外源氮源。鉴于以上研究背景,甲烷氧化和硝化过程有着必然的内在联系[5]。
草原生态系统是世界上最大的陆地生态系统,草地面积占我国国土面积的41%[6]。草原土壤既是CH4的汇,又是N2O的源,因此,有关草原生态系统中土壤的碳氮循环一直是研究热点问题。当前关于草原土壤系统中硝化过程和甲烷氧化过程的研究发现,Nitrosospira AOB和Methylobacter MOB分布广泛,在多种草原土壤生境中参与硝化和甲烷氧化过程[7-8]。旱地土壤类群(USC)MOB因其具有高甲烷亲和力在草原土壤中的重要性逐渐被发现[9-10]。但目前关于草原土壤中不同氮水平下甲烷氧化活性和相关活性微生物,以及甲烷氧化和硝化之间复杂的相互作用关系机制的研究较少[11-12]。因此本研究选取内蒙古锡林郭勒盟草原土壤为研究对象,结合稳定同位素核酸探针(DNA-SIP)、实时荧光定量PCR和Miseq测序等分子生物学技术,分析不同氮水平下,甲烷氧化和硝化微生物的相互关系,阐明草原生态系统CH4氧化和硝化的交互作用机制,以期为深入了解草原土壤碳氮循环、提高草原氮肥利用率、增强草原生产力、实现温室气体减排提供科学依据。
1 材料与方法 1.1 土壤样品概况土壤样品采自位于内蒙古锡林郭勒盟草原(43°26′-44°39′N,115°32′-117°12′E)的中国科学院内蒙古草原生态系统定位研究站。该地区冬季寒冷,夏季凉爽,属于半干旱大陆性季风气候,每年大约102~136 d无霜期,全年平均气温-0.4 ℃,年均降水量350 mm左右,降雨多发生在六月至八月,雨热同期,季节分布不均,与植物生长期一致。主要植被类型包括羊草(Leymus chinensis)、大针茅(Stipa grandis)和粗隐子草(Cleistogenes squarrosa)。土壤类型为暗栗钙土。该样地1996年以前自由放牧,自1996年起禁牧围封。样地大小为400 m × 60 m,包括5个小区,每个小区70 m × 60 m,相邻的两个小区之间有10 m的缓冲带便于取样。2016年8月用5 cm直径土钻按照“S”型取样法采集0~20 cm表层土,全部混匀,研磨过2 mm筛。土壤基本理化性质如下:pH 7.22,容重1.32 g·cm-3,有机质25.47 g·kg-1,全碳16.67 g·kg-1,全氮1.50 g·kg-1,硝态氮63.36 mg·kg-1,铵态氮4.45 mg·kg-1,有效磷(Olsen P)7.31 mg·kg-1,速效钾251.10 mg·kg-1。
1.2 微宇宙培养实验将相当于6.0 g干重的新鲜土壤样品转移至120 mL血清瓶,调节40%田间持水量,胶塞加铝盖封口,25 ℃黑暗中预培养,每周监测CO2浓度,当浓度降至5 000 μL·L-1时开始正式培养。
正式培养实验共设7个处理,如表 1所示,每个处理3个重复。13CH4处理(99 atom% 13C,Sigma-Aldrich,St Louis,MO,USA)用以分析甲烷氧化活性和活性甲烷氧化微生物;13CO2+Urea和12CO2+Urea处理(99 atom% 13C,上海稳定性同位素工程技术研究中心,上海,中国)用以分析硝化活性和活性硝化微生物;13CH4+13CO2+Urea和12CH4+12CO2+Urea处理用以分析甲烷氧化和硝化之间的相互作用。其中,添加尿素的处理均包括N 20 μg·g-1干土(U20)和100 μg·g-1干土(U100)两个氮素水平。试验流程如图 1所示,每周用高纯空气(20% O2,80% N2)换气1 min,保证有氧环境,同时以溶液的形式逐滴加入尿素溶液,调节60%田间持水量,血清瓶胶塞加铝盖密封后加5%的CO2。其中甲烷浓度动态监测,随时添加,保证1%浓度。在25 ℃生化培养箱中黑暗培养21 d,分别在0、7、14、21 d进行破坏性取样。所取样品分两部分,2 g左右保存在-80 ℃冰箱,用以提取土壤DNA;剩余样品用以分析硝化活性。
|
|
表 1 微宇宙培养试验处理概况 Table 1 Schedule of the treatments |
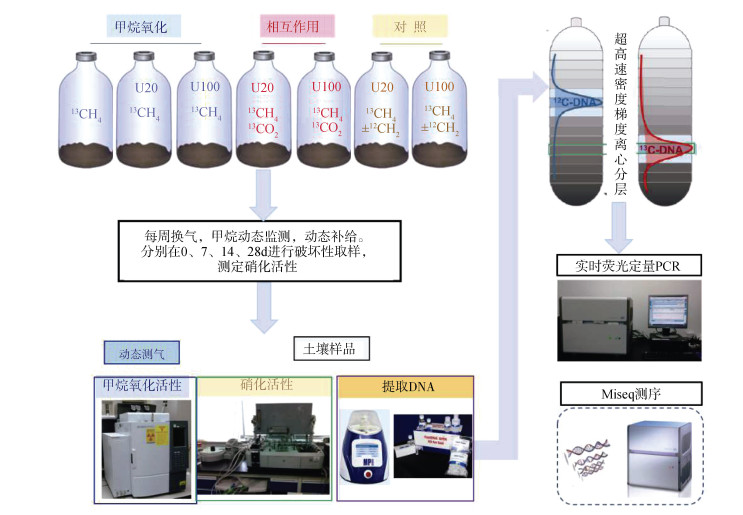
|
图 1 实验流程图 Fig. 1 The experimental procedure |
将0 d和21 d的土壤样品用FastDNA Spin Kit for Soil(MP)提取土壤基因组总DNA。用0.7%的凝胶电泳检测DNA的完整性,NanoDrop® ND-2000 UV-Vis spectrophotometer(NanoDrop Technologies,Wilmington,DE,USA)检测DNA的质量和纯度。DNA样品-20 ℃冰箱保存,用以进行超高速等密度梯度离心。
SIP分层方法参考文献[13]。具体操作方法如下:大约3 μg DNA与4.9 mL氯化铯(CsCl)溶液混合,调节浮力密度至1.725 g·mL-1。然后将混合液转移至5.1 mL超高速离心试管中,调平密封后用贝克曼Vti65.2转子(Beckman Coulter Inc.,Palo Alto,CA,USA)在20 ℃条件下以177 000×g的速度离心44 h。离心结束后,利用NE-1000注射器泵(New Era Pump Systems Inc.,Farmingdale,NY,USA)将新灭菌蒸馏水以稳定流速(0.38 mL· min-1)从上向下将溶于CsCl中的DNA置换到15个1.5 mL离心管中,即每个土壤DNA样品分层后总共得到15层DNA组分溶液,每层体积为380 μL,取65 μL用手持式AR200折光率仪(Reichert Inc.,Buffalo,NY,USA)测定折光率。将分层后的DNA组分溶液用PEG6000和乙醇纯化,溶于30 μL新灭菌蒸馏水进行后续分析。
1.4 甲烷氧化菌pmoA和氨氧化菌amoA基因的定量PCR将0 d和21 d的总DNA以及上述分层纯化后的DNA样品进行pmoA和amoA基因的定量PCR分析(LightCycler 480,Roche Applied Science),具体引物信息和PCR条件见表 2。反应体系20 μL,包括10 μL SYBR Premix Ex Taq(TaKaRa,Dalian,China)、1.5 μL DNA模板(1~10 ng)、0.79 μL milli-Q水,体系中引物终浓度0.5 μmol·L-1。最后跑溶解曲线,利用50~99 ℃的升温连续检测荧光以确认PCR产物的特异性。本实验定量PCR扩增效率在80.5%~108.3%之间,R2值在0.990~0.998之间。
|
|
表 2 本试验中引物信息及PCR条件 Table 2 Primers and conditions used in this study |
将0 d和21 d的总DNA以及上述分层纯化后的重层DNA样品进行Miseq高通量测序。利用Illumina®MiSeq测序仪(Illumina,San Diego,CA,USA)对上述DNA的16S rRNA基因的V4区进行测序,引物序列及扩增条件见表 2。本实验中,只有序列长度大于200 bp,平均质量大于25,没有模糊噪声,至少80%符合16S rRNA序列的结果才会进行后续分析。在97%序列相似度水平上进行OTU(可操作分类单元)聚类,最后参考Silva数据库对OTU进行物种注释[18]。
总DNA样品共得到1 149 320条高质量序列,平均每个样品得到47 888个高质量序列。其中属于细菌的序列有1 123 384条,占97.74%;属于古菌的序列有25 936条,占2.26%。重层DNA样品在13CH4、U20+13CO2、U100+13CO2、U20+13CH4+13CO2和U100+13CH4+13CO2处理下分别得到426 842、407 179、429 748、501 376和480 393条高质量序列。
选取属于MOB、AOA、AOB和NOB的序列,用Molecular Evolutionary Genetics Analysis(MEGA 7.0)分别进行系统发育分析。以重层MOB、AOB和NOB的OTU丰度表为原始数据,采用Spearman相关系数模型,显著性P < 0.05,通过CoNet软件构建共线性网络图,Gephi 0.9.2对网络图进行可视化分析。网络图包括节点和边,各节点代表不同的物种,以颜色区分。边代表物种之间的相互作用,红色和绿色分别代表正相关和负相关,线的粗细表示相关性的大小。将总DNA和重层DNA的测序数据上传至NCBI的Sequence Read Archive(SRA)数据库,检索号分别为PRJNA611699和PRJNA611713。
1.6 数据处理硝化活性、甲烷氧化活性、pmoA/amoA基因丰度和相对丰度的显著性差异分析通过SPSS 20.0单因素方差分析(one-way ANOVA)的Duncan多重检验完成;作图通过Origin 9.0完成;P < 0.05表示差异显著。
2 结果 2.1 甲烷氧化活性和甲基营养微生物氮素水平对甲烷氧化活性和相关微生物丰度均有显著影响(图 2),经过21d的微宇宙培养,相比不施氮(U0),甲烷氧化活性、pmoA基因丰度和甲基营养微生物相对丰度在低氮水平(U20)下显著增加,在高氮水平(U100)下显著降低。在U0、U20和U100三个处理下,甲烷氧化活性分别是37.79、45.27和16.11 μmol·g-1干土,pmoA基因丰度分别是3.08×107、4.24×107和9.50×106 copies·g-1干土。甲烷氧化活性、pmoA基因丰度以及甲基营养微生物的相对丰度均在不同氮水平下呈现出“低促高抑”的规律(图 2a)。
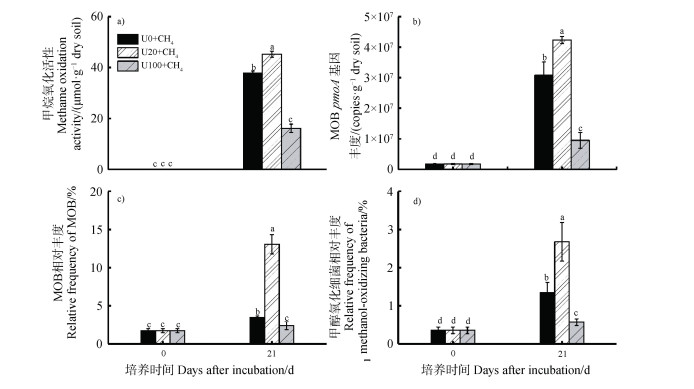
|
图 2 甲烷氧化活性(a)、甲烷氧化细菌pmoA基因丰度(b)和甲基营养微生物相对丰度(c~d)分析 Fig. 2 Changes in methane oxidation activity(a) and quantitative distribution of methylotrophs(b-d) in soil microcosms over an incubation period of 21 days |
净硝化速率、硝化微生物丰度和相对丰度如图 3所示。U20、U20+CH4、U100和U100+CH4的净硝化速率分别为51.57、34.60、163.50和165.02 μg·g-1干土(图 3a);AOB amoA基因丰度分别为1.90×108、1.23×108、6.34×108、8.04×108 copies·g-1干土(图 3b)。低氮水平下,CH4的存在显著抑制硝化活性和AOB amoA基因丰度;而高氮水平下,CH4的存在对硝化活性和AOB amoA基因丰度没有显著影响。AOB和NOB的相对丰度也符合该规律(图 3d,3f)。AOA amoA基因的丰度和相对丰度在培养21 d之后显著降低,甲烷存在与否对AOA amoA基因丰度影响不显著(图 3c,图 3e)。

|
图 3 净硝化活性(a)和氨氧化微生物amoA基因丰度(b~c),硝化微生物相对丰度(d~f)分析 Fig. 3 Net nitrification activity(a) and quantitative distribution of nitrifiers(b-f) in soil microcosms over an incubation period of 21 days |
离心后每个样品分为15层,浮力密度在1.676~1.791 g·mL-1之间。对每一层进行pmoA和amoA基因的定量PCR分析,然后将每一层的分析结果与各层次中最大的基因丰度作比,得到pmoA和amoA基因在各浮力密度梯度中的分布图(图 4a)。甲烷氧化菌在不施氮处理中被显著标记,低氮处理增加了MOB被标记的程度,而高氮处理则降低了MOB的标记程度。AOB在低氮和高氮水平下均被显著标记,尤其是在高氮水平下的标记更显著。低氮水平下,甲烷的存在抑制AOB的被标记程度。低氮和高氮水平下,AOA均没有被标记。根据定量分布结果选取重层DNA(3~12层)进行高通量测序,选择分别属于MOB、AOB和NOB的OTU绘制各浮力密度上的相对丰度分布图,结果如图 4b、图 4c。MOB和AOA的相对丰度在各浮力密度的分布趋势和上述定量分析的结果完全一致。AOB和NOB的相对丰度分布图显示低氮水平和高氮水平下,甲烷的存在对AOB和NOB的被标记程度均有抑制效果。
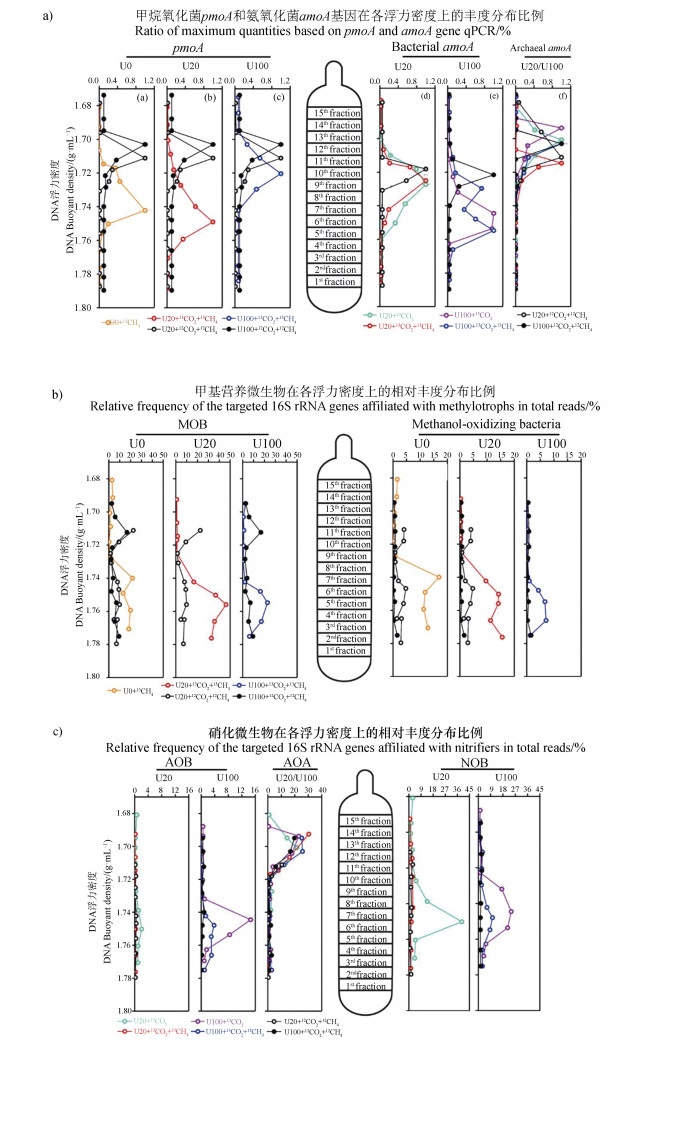
|
图 4 甲基营养微生物和硝化微生物丰度和相对丰度在不同浮力密度上的分布 Fig. 4 Quantitative distribution of methanotrophs as well as nitrifiers across the entire buoyant density gradient of the fractionated DNA from soil microcosms after incubation for 21 days |
如图 5所示,草原土壤99.7%以上的MOB属于甲基杆菌属(Methylobacter);活性MOB包括甲基杆菌属(Methylobacter)和甲基暖菌属(Methylocaldum),以Methylobacter为主,占90%以上。活性Methylobacter MOB在U0+13CH4、U20+13CH4和U100+13CH4的处理中所占的比例分别为92.62%、96.35%和91.79%,活性Methylocaldum MOB所占的比例分别为7.38%、3.65%和8.21%。硝化微生物AOB主要包括亚硝化球菌属(Nitrosococcus)、亚硝化螺旋菌属(Nitrosospira)和亚硝化单胞菌属(Nitrosomonas),分别占72.00%、26.00%和2.00%。65%以上的活性AOB为Nitrosospira,其在U20、U20+13CH4、U100、U100+13CH4处理中对AOB的占比分别为65.33%、80.23%、78.30%和82.10%;Nitrosomonas 在AOB中的占比分别为28.41%、10.13%、20.77%和16.13%;Nitrosococcus 在AOB中的占比分别为6.25%、9.64%、0.93%和1.77%。该草原土壤中的亚硝酸盐氧化细菌(NOB)均隶属于Nitrospira,包括defluvii、moscoviensis和marina三个种。活性NOB以defluvii为主,其在U20、U20+13CH4、U100、U100+13CH4处理的占比分别是:71.3%、61.9%、78.4%和78.8%;moscoviensis的占比分别为:23.6%、27.7%、16.9%和14.5%;marina的占比分别为5.1%、10.4%、4.7%和6.7%。
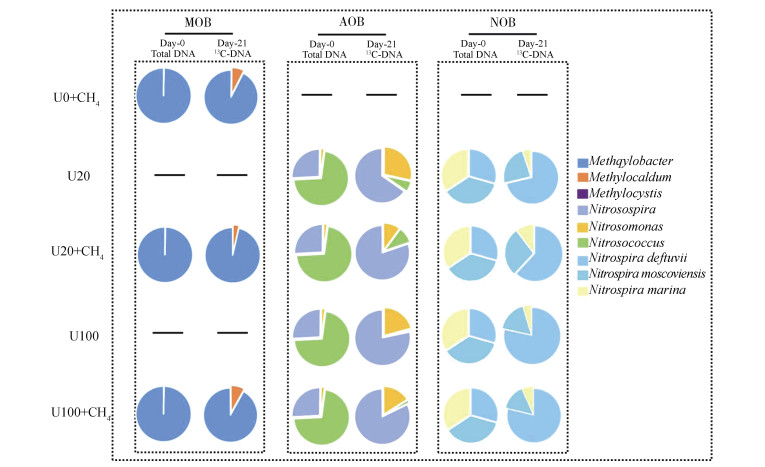
|
图 5 活性甲烷氧化微生物和硝化微生物的群落组成分析 Fig. 5 Proportional changes of active methanotrophs and nitrifying phylotypes in SIP microcosms after an incubation period of 21 days |
为明确活性甲烷氧化微生物和硝化微生物的相互作用,将重层MOB、AOB和NOB三个功能微生物的高丰度OTU构建共线性网络图(图 6)。结果发现,Methylobacter MOB与Nitrosospira AOB存在显著的负相关关系,此外,Methylobacter MOB还与Nitrospira NOB存在显著的负相关关系,Nitrosococcus AOB与Nitrospira NOB存在显著正相关关系。

|
注:红线表示正相关,绿线表示负相关;线的粗细表示相关性的大小。橙色圆代表标记的MOB(Methylobacter和Methylotenera);蓝色圆代表标记的AOB(Nitrosospira和Nitrosococcus);红色圆代表标记的NOB(Nitrospira)。 Note: Connecting lines stand for significant(P < 0.05) positive(red) or negative(green) correlations where thicker lines represent stronger correlations. Orange circles represent labeled MOB: Methylobacter and Methylotenera; Blue circles represent labeled AOB: Nitrosospira and Nitrosococcus; Red circles represent labeled NOB: Nitrospira. 图 6 活性MOB、AOB和NOB的共线性网络图 Fig. 6 Network analysis of co-occurring phylotypes of MOB, AOB and NOB in microcosms |
甲烷氧化活性分析结果表明(图 2a),U20显著刺激甲烷氧化,而U100显著抑制甲烷氧化。草原土壤中甲烷氧化能力对氮素的响应体现出“低促高抑”的趋势。对0 d和21 d总DNA的pmoA定量分析(图 2b)结果发现,和甲烷氧化活性一致,低氮处理的土壤pmoA丰度显著高于不施氮处理,而高氮处理的pmoA丰度显著低于不施氮处理。这个结果进一步说明甲烷氧化活性对氮素的“低促高抑”响应。甲烷氧化菌和甲醇氧化菌的相对丰度变化也符合该趋势(图 2c、图 2d)。SIP分析结果同样发现MOB在不施氮处理中被明显标记,在低氮处理中标记程度显著增加,而在高氮处理中仅轻微标记(图 4a)。不同浮力密度上的MOB相对丰度分布也符合以上特点(图 4b)。这可能是由于该围封草原土壤N素主要来源是氮沉降和氮固定,整体N素水平较低,加入20 μg·g-1干土的尿素刺激土壤甲烷氧化菌,导致其大量繁殖,活性增加,从而提高甲烷氧化能力。100 μg·g-1 干土的尿素态N显著刺激了硝化微生物的繁殖,抑制了甲烷氧化菌的繁殖和功能发挥,同时,氨氧化产生的中间产物羟胺(NH2OH)和亚硝酸盐(NO2-),以及硝化终产物硝酸盐(NO3-)均会对甲烷氧化菌产生毒害作用,进而抑制甲烷氧化活性。本实验用13CO2标记活性硝化微生物,13CH4标记活性甲烷氧化微生物,两者均不会以被标记的微生物体的有机碳作为碳源进行繁殖,所以即便可能存在,cross-feeding问题对本实验结果的影响会非常小。因此,本实验中利用DNA-SIP技术分析确定活性甲烷氧化微生物和硝化微生物,其结果是可靠的。
关于氮对甲烷氧化活性影响的争论由来已久。氮添加可能刺激甲烷氧化以及甲烷氧化细菌的活性,因为甲烷氧化菌生长需要氮源[4, 11-12]。但是也有研究发现氮添加会抑制甲烷氧化活性。这种抑制情况一方面是因为NH3与CH4竞争MMO活性位点导致抑制效应;另一方面氨氧化中间产物NH2OH和NO2-会对甲烷氧化菌产生毒害作用也会抑制甲烷氧化活性[4]。
本世纪初,Stein等[4]提出铵盐对MOB刺激还是抑制取决于铵态氮与甲烷态碳的比例(N-CH4)。然而,Bodelier [19]和Zheng [12]等发现N-CH4比分别为200和0.11时,尿素添加均显著刺激甲烷氧化。而本实验中U100(100 μg·g-1 干土)处理的N-CH4比仅为0.70,但是显著抑制了甲烷氧化。同样,Zhang等[20]通过四年田间试验也发现100 kg·hm-2·a-1(约158 μg·g-1 干土)的氮添加显著抑制甲烷氧化。Zhao等[21]推测氮肥对甲烷氧化的刺激或抑制作用可能取决于主要甲烷氧化菌的种系型,比如,Zheng等[12]发现水稻土中添加尿素后,活性甲烷氧化微生物以Methylobacter为主,而未施加尿素的处理以Methylosarcina为主。在本草原土的试验中,无论是否添加尿素以及添加尿素多少,均是以Methylobacter为主[22]。因此,有关氮素水平对甲烷氧化的机理尚不明确,未来关于氮素对甲烷氧化影响的研究可考虑多种类型土壤的综合分析。
3.2 甲烷对净硝化活性和活性硝化微生物的影响类似于对MOB的分析,本实验对氨氧化功能微生物在0 d和21 d的总DNA分别进行定量分析。结果显示(图 3b),低浓度和高浓度氮添加均会刺激AOB丰度的增长,而抑制AOA的繁殖,说明AOB在硝化作用过程中具有重要作用。DNA-SIP结果发现AOB在各处理中被13C标记,而AOA在各种处理中均未表现出明显的被标记迹象(图 4a),说明AOB同化了来自CO2的13C,进一步证明AOB是驱动该草原土壤硝化作用的活性硝化微生物。
不同浮力密度范围的AOB amoA分布,以及AOB和NOB相对丰度的分布进一步表明低氮水平下,甲烷添加显著抑制了活性硝化微生物同化13CO2(图 4a,图 4c)。Zheng等[12]在水稻土的研究发现,100 μg·g-1干土的尿素处理,甲烷添加显著抑制硝化活性和活性硝化微生物。而本研究发现,在草原土壤中,添加20 μg·g-1干土的尿素,甲烷即显著抑制活性硝化微生物同化13CO2,降低硝化活性,而100 μg·g-1干土的尿素处理下,硝化活性和活性硝化微生物对甲烷添加无响应。无机氮对于生物量的形成是至关重要的,尤其是对于那些缺乏固定分子态N的甲烷氧化菌来说。说明甲烷对硝化活性的抑制作用可能只出现在氨氧化底物不足时,氨氧化微生物和甲烷氧化微生物竞争N源[4],一部分N作为N源被甲烷氧化菌同化[12],但是当N源浓度较高时,甲烷氧化微生物反被抑制(图 2,4),丧失了与氨氧化微生物竞争的能力。结合上述氮水平对甲烷氧化的影响,氮素添加体现出对甲烷氧化的刺激效应时,甲烷的存在就会抑制氮素转化(硝化作用);氮素添加体现出对甲烷氧化的抑制效应时,甲烷的存在对氮素转化过程(硝化作用)没有显著影响,说明甲烷氧化和硝化过程的相互作用关系受氮素水平的调控。
3.3 活性甲烷氧化和硝化微生物及相互作用群落组成分析发现,活性MOB群落结构比较单一,以Type Ia的甲基杆菌属(Methylobacter)为主,占90%以上,还有一些Type Ib的甲基暖菌属(Methylocaldum)和极少比例的Type II型的Methylocystis。一方面,研究发现Type Ia型甲烷氧化菌在湿地、水稻土、冰下水系等多种环境中都作为主要的甲烷氧化微生物[23-24];另一方面,通过菌株分离分析发现,高浓度甲烷条件下的Methylobacter活性远高于低浓度甲烷环境[23]。此外,在本试验中,尿素施入并没有改变草原土壤中活性甲烷氧化微生物的群落组成,这一结果与Zhao等[21]在水稻土中的发现一致,说明草原土和水稻土中氮有效性不是决定甲烷氧化微生物组成的关键因子。本试验发现活性硝化微生物以Nitrosospira AOB和Nitrospira NOB为主,这一结果与之前在草原土壤[7]、水稻土壤[12]和森林土壤[25]中的研究结果一致。
尿素对甲烷氧化菌群落的低促高抑主要反应在Methylobacter MOB,Methylobacter菌株生长需要氮源,但其不具备固氮能力[26],因此,氮施入可促进Methylobacter生长繁殖。但随着施氮量增加,硝化活性增强,硝化作用产生的NH2OH和NO2-以及硝化产物NO3-对MOB均存在毒害作用;活性Nitrosospira AOB和Nitrospira NOB比例随施氮量增加而增加,导致MOB与AOB/NOB在同一生境中竞争O2、NH4+-N和生存空间,网络分析结果也发现Methylobacter与活性硝化微生物(Nitrosospira AOB和Nitrospira NOB)存在显著负相关关系。结合低氮条件下,甲烷氧化增强而硝化被抑制;高氮水平下,硝化增强而甲烷氧化被抑制,说明草原土壤中甲烷氧化和硝化之间存在氮引发的Methylobacter MOB与Nitrosospira AOB/Nitrospira NOB之间的竞争性相互作用关系。
4 结论草原土壤甲烷氧化活性及微生物功能活性在低浓度氮素水平下显著增加,而在高浓度氮素水平下显著降低,呈现出“低促高抑”现象。N对甲烷氧化的影响(刺激或抑制)可能是土壤条件和主要微生物种系型共同作用的结果。DNA-SIP结果发现,活性甲烷氧化微生物以Methylobacter为主,活性硝化微生物以Nitrosospira AOB和Nitrospira NOB为主。而网络分析结果发现Methylobacter MOB和Nitrosospira AOB/Nitrospira NOB之间存在显著负相关关系,说明甲烷氧化和硝化微生物之间存在竞争性相互作用。以上结果表明甲烷氧化和硝化过程的相互作用受氮素水平的调控。
| [1] |
Stocker T F, Qin D, Plattner G K, et al. Climate Change 2013: The physical science basis. contribution of working group I to the fifth assessment report of IPCC the intergovernmental panel on climate change[EB/OL]. 2014
(  0) 0) |
| [2] |
Shcherbak I, Millar N, Robertson G P. Global metaanalysis of the nonlinear response of soil nitrous oxide(N2O) emissions to fertilizer nitrogen[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25): 9199-9204. DOI:10.1073/pnas.1322434111
(  0) 0) |
| [3] |
Kolb S. The quest for atmospheric methane oxidizers in forest soils[J]. Environmental Microbiology Reports, 2009, 1(5): 336-346. DOI:10.1111/j.1758-2229.2009.00047.x
(  0) 0) |
| [4] |
Stein L Y, Roy R, Dunfield P F. Aerobic methanotrophy and nitrification: Processes and connections[M]/Encyclopedia of life sciences. John Wiley & Sons Inc, 2012
(  0) 0) |
| [5] |
Jia Z J, Cai Z C. Methane consumption in relation to ammonia oxidation in paddy soils (In Chinese)[J]. Rural Eco-Environment, 2003, 19(4): 40-44. [贾仲君, 蔡祖聪. 稻田甲烷氧化与铵氧化关系研究进展[J]. 农村生态环境, 2003, 19(4): 40-44.]
(  0) 0) |
| [6] |
Sala O E, Austin A T, Vivanco L. Temperate grassland and shrubland ecosystems[M]//Encyclopedia of Biodiversity. Amsterdam: Elsevier, 2001: 627-635.
(  0) 0) |
| [7] |
Pan H, Xie K X, Zhang Q C, et al. Archaea and bacteria respectively dominate nitrification in lightly and heavily grazed soil in a grassland system[J]. Biology and Fertility of Soils, 2018, 54(1): 41-54. DOI:10.1007/s00374-017-1236-7
(  0) 0) |
| [8] |
Mo Y L, Jin F, Zheng Y, et al. Succession of bacterial community and methanotrophy during lake shrinkage[J]. Journal of Soils and Sediments, 2020, 20(3): 1545-1557. DOI:10.1007/s11368-019-02465-6
(  0) 0) |
| [9] |
Deng Y C, Che R X, Wang F, et al. Upland Soil Cluster Gamma dominates methanotrophic communities in upland grassland soils[J]. Science of the Total Environment, 2019, 670: 826-836. DOI:10.1016/j.scitotenv.2019.03.299
(  0) 0) |
| [10] |
Cai Y F, Jia Z J. Research progress of atmospheric methane oxidizers in soil (In Chinese)[J]. Acta Microbiologica Sinica, 2014, 54(8): 841-853. [蔡元锋, 贾仲君. 土壤大气甲烷氧化菌研究进展[J]. 微生物学报, 2014, 54(8): 841-853.]
(  0) 0) |
| [11] |
Daebeler A, Bodelier P L, Yan Z, et al. Interactions between Thaumarchaea, Nitrospira and methanotrophs modulate autotrophic nitrification in volcanic grassland soil[J]. The ISME Journal, 2014, 8(12): 2397-2410. DOI:10.1038/ismej.2014.81
(  0) 0) |
| [12] |
Zheng Y, Huang R, Wang B Z, et al. Competitive interactions between methane-and ammonia-oxidizing bacteria modulate carbon and nitrogen cycling in paddy soil[J]. Biogeosciences, 2014, 11(12): 3353-3368. DOI:10.5194/bg-11-3353-2014
(  0) 0) |
| [13] |
Jia Z J, Conrad R. Bacteria rather than Archaea dominate microbial ammonia oxidation in an agricultural soil[J]. Environmental Microbiology, 2009, 11(7): 1658-1671. DOI:10.1111/j.1462-2920.2009.01891.x
(  0) 0) |
| [14] |
Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. DOI:10.1073/pnas.0506625102
(  0) 0) |
| [15] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/aem.63.12.4704-4712.1997
(  0) 0) |
| [16] |
Kolb S, Knief C, Stubner S, et al. Quantitative detection of methanotrophs in soil by novel pmoA-targeted real-time PCR assays[J]. Applied and Environmental Microbiology, 2003, 69(5): 2423-2429. DOI:10.1128/AEM.69.5.2423-2429.2003
(  0) 0) |
| [17] |
Stubner S. Enumeration of 16S rDNA of Desulfotomaculum lineage 1 in rice field soil by real-time PCR with SybrGreen™ detection[J]. Journal of Microbiological Methods, 2002, 50(2): 155-164. DOI:10.1016/S0167-7012(02)00024-6
(  0) 0) |
| [18] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D596.
(  0) 0) |
| [19] |
Bodelier P L E, Hahn A P, Arth I R, et al. Effects of ammonium-based fertilisation on microbial processes involved in methane emission from soils planted with rice[J]. Biogeochemistry, 2000, 51(3): 225-257. DOI:10.1023/A:1006438802362
(  0) 0) |
| [20] |
Zhang L H, Yuan F H, Bai J H, et al. Phosphorus alleviation of nitrogen-suppressed methane sink in global grasslands[J]. Ecology Letters, 2020, 23(5): 821-830. DOI:10.1111/ele.13480
(  0) 0) |
| [21] |
Zhao J, Cai Y F, Jia Z J. The pH-based ecological coherence of active canonical methanotrophs in paddy soils[J]. Biogeosciences, 2020, 17(6): 1451-1462. DOI:10.5194/bg-17-1451-2020
(  0) 0) |
| [22] |
Li Y, Liu Y W, Pan H, et al. Impact of grazing on shaping abundance and composition of active methanotrophs and methane oxidation activity in a grassland soil[J]. Biology and Fertility of Soils, 2020, 56(6): 799-810. DOI:10.1007/s00374-020-01461-0
(  0) 0) |
| [23] |
Hao Q Q, Liu F H, Zhang Y C, et al. Methylobacter accounts for strong aerobic methane oxidation in the Yellow River Delta with characteristics of a methane sink during the dry season[J]. Science of the Total Environment, 2020, 704: 135383. DOI:10.1016/j.scitotenv.2019.135383
(  0) 0) |
| [24] |
Sultana N, Zhao J, Zheng Y, et al. Stable isotope probing of active methane oxidizers in rice field soils from cold regions[J]. Biology and Fertility of Soils, 2019, 55(3): 243-250. DOI:10.1007/s00374-018-01334-7
(  0) 0) |
| [25] |
Huang X R, Zhao J, Su J, et al. Neutrophilic bacteria are responsible for autotrophic ammonia oxidation in an acidic forest soil[J]. Soil Biology & Biochemistry, 2018, 119: 83-89.
(  0) 0) |
| [26] |
Whitman WB, Rainey F, Kämpfer P, et al. Bergey's manual of systematics of Archaea and Bacteria . Hoboken, New Jersey: Wiley, 2015.
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


