2. 中国科学院大学, 北京 100049;
3. 南京市农业技术推广站, 南京 210036;
4. 南京市蔬菜花卉科学研究所, 南京 210042
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Nanjing Agricultural Technique Extension Station, Nanjing 210036, China;
4. Nanjing Vegetables Scientific Institute, Nanjing 210042, China
番茄青枯病是由茄科劳尔氏菌(Ralstonia solanacearum)侵染引起的一种典型的细菌性土传病害,其病菌能长时间存活在土壤中,危害性较强,发病范围广[1],能波及我国大多数地区,尤其以南方地区和中部地区居多[2],给我国农业生产造成了严重困扰,解决这一问题迫在眉睫。
国内外学者针对植物土传病害的发生及防治方法进行了广泛研究[3-6],抑病土壤的发现及其与土传病害的关系就是目前研究的热点之一[7-9]。研究发现,抑病土是一种对植物病害有抑制作用的特殊土壤[10],它最早于1931年由Henry[11]在观察小麦角腐病时发现。抑病土的发现为解析土传病害的发生以及宿主植物-病原菌-环境的相互关系提供了便利。大量研究结果表明,与感病土相比,在受到病原菌侵染时,抑病土壤中的宿主植物往往能在其根际募集大量微生物,其中的有益微生物通过拮抗病原菌或通过竞争养分与生态位等途径来抑制病害,从而维持植物健康[9,12]。根际作为连接宿主植物-土壤-微生物相互作用的重要桥梁,在促进植物吸收土壤养分、协调环境胁迫以及调节土壤微生物群落等方面发挥了重要作用[12-13]。因此,研究抑病土壤应对病原菌入侵后的植物根际微生物群落的变化,并对明确植物土传病害的发生机制具有重要意义。
微生物群落构建在塑造土壤微生物群落结构方面发挥着重要作用,目前普遍认为微生物群落构建过程是随机性和确定性过程共同主导,其中确定性过程包括生物因素(病害入侵等)与非生物因素(盐碱化、酸化等极端条件)施加的选择[14-15],故确定性过程在某一特定环境条件下可直接塑造微生物群落结构;而随机性过程则是自然过程的体现,包括种群出生、死亡或生态漂变等[16]。因此可以假设,随机性过程起主导的微生物群落往往对外界的扰动具有更强的适应性。微生物群落构建过程与土传病害的关系也备受关注[17-20],因此基于微生物群落构建理论研究抑病土壤中根际微生物群落对土传病害的响应可加深我们对抑病土壤中土传病害的发生机制的认识。
抑病土壤的形成与土壤微生物区系有关,包括土壤微生物生物群落多样性以及动态平衡等,然而其抑病作用机制还不太清楚,被认为是与土壤中的某类微生物的作用有关,且具有传递性[8-10]。不同地域的青枯病抑病土壤中的微生物组具有不同的特征,且具有明显的区域性[2]。而江苏地区的番茄青枯病发生普遍,且在进行长期番茄连作试验过程中也发现青枯病抑病土壤的产生。因此,明确青枯病抑病土的微生物区系特征对理解青枯病的发病过程以及通过微生物调控青枯病的发生具有重要意义。本研究以番茄青枯病为研究对象,研究该地区的感病土和抑病土中根际微生物群落的组成、结构、多样性以及群落构建过程的差异,同时探讨抑病土对番茄青枯病的抑病特性,并对其转移机制进行验证,以揭示抑病土壤中番茄根际微生物群落抵御病害的机理。
1 材料与方法 1.1 供试材料与实验设计供试抑病土(番茄青枯病的田间发病率为18.85%)和感病土(番茄青枯病的发病率为84.09%)于2019年3月采自江苏省南京市蔬菜花卉科学研究所(118°46′E,31°43′N)长期种植番茄的耕作层(0~20 cm)土壤,两个样点相距5 m左右,二者的判定方法参考Expósito等[21]。供试土壤的理化性质如表 1。
|
|
表 1 供试土壤理化性质 Table 1 Physico-chemical properties of the soils in the study |
盆栽试验于中国科学院南京土壤研究所温室中进行。供试土壤研磨过5 mm筛后进行装盆,每盆1.5 kg土。该试验包括2个处理(抑病土和感病土),每个处理10个重复。番茄种子(合作903品种)培育至三叶一心期时,选取生长健壮且长势相近的番茄幼苗进行移栽,每盆1株苗,不接种病原菌,种植周期45 d,每天对番茄进行浇水并观察番茄生长情况。于开花期时统计番茄青枯病的发病等级及其株高,每个处理去掉首尾盆栽(除去边缘效应的影响),然后选择收集中间的7盆,采集其根际土,保存至–20℃冰箱用于提取土壤DNA,并做好标记(感病土壤中的番茄根际土DRS、抑病土壤中的番茄根际土HRS)。
为验证抑病土的抑病记忆传递特性,15 d后对上述试验中DRS和HRS的7盆土(每盆1.5 kg土)单独研磨过5 mm筛后进行装回原盆,每盆1.0 kg土,多余的土壤按照等质量进行掺混,然后装盆,每盆1 kg土,最终形成3个处理:感病土D10H0、掺混土D5H5、抑病土D0H10,每个处理6个重复。三叶一心期时对长势相近的番茄幼苗进行移栽,每盆1株苗,不接种病原菌,种植周期45 d,每天浇水并观察番茄生长情况。于开花期时统计番茄青枯病的发病等级及其株高,每个处理去掉首尾盆栽,选择收集中间的4盆,采集其根际土,保存至–20℃冰箱用于土壤DNA的提取。
1.2 土壤理化性质测定土壤基本理化性质的测定均参考鲁如坤[22]的方法。土壤pH采用土/水质量比1︰2.5浸提,用pH计(SevenMulti Mettler Toledo,瑞士)测定;采用重铬酸钾氧化法测定土壤有机碳;采用凯氏定氮法测定土壤全氮;采用氢氟酸-次氯酸消煮,钼蓝比色法测定土壤全磷和火焰光度计法测定土壤全钾;采用2 mol·L–1 KCl浸提-靛酚蓝比色法测定土壤铵态氮和硝态氮;采用0.5 mol·L–1 NaHCO3提取,钼蓝比色法测定土壤有效磷;采用10 % HNO3与1 % 氢氟酸消煮、电感耦合等离子体发射光谱法测定土壤有效钾。
1.3 土壤DNA提取与16S rRNA基因扩增子高通量测序准确称取0.5 g土壤,采用土壤DNA提取试剂盒(FastDNA™ SPIN Kit for Soil,MP Biomedicals)进行根际土壤DNA的提取。提取完毕后使用NanoDrop 2000(Thermo Scientific,美国)测定DNA的浓度,并置于–20℃冰箱保存。
利用细菌特异性引物(341F:5'-CCTACGGGRB GCASCAG-3'和806R:5'-GGACTACNNGGGTATCT AAT-3')对细菌16S rRNA基因的V3~V4可变区域进行PCR扩增。PCR反应体系为50 μL,包括Premix Tap DNA聚合酶(5 U·μL–1)25 μL,正、反向引物(20 mg·L–1)各0.5 μL,DNA模板(20 mg·L–1)1 μL,加水补至50 μL。PCR反应的扩增条件如下:94℃预变性5 min,35个循环(94℃ 30 s,55℃ 30 s,72℃ 45 s),最后72℃ 10 min。所有PCR产物均通过1% 琼脂糖凝胶进行条带特异性检测。每一个样本的引物中有对应的Barcode用于数据拆分。PCR产物经Qubit仪(Qubit 2.0,Thermo Scientific,美国)测定浓度后等质量混合并纯化。最后使用建库试剂盒(TruSeq® DNA PCR-Free Sample Preparation Kit)进行文库构建,构建好的文库合格后,使用Illumina MiSeq平台进行测序。
16S rRNA基因序列使用Quantitative Insights Into Microbial Ecology(QIIME version 1.9.1)软件对拼接数据并进行过滤,过滤掉低质量碱基较多的序列(质量低于20、长度小于200 bp),并根据Barcode序列与样品进行匹配。使用cutadapt trimmomatic分别去除扩增引物和接头序列,使用Usearch(Version 2.13.2)软件按照97%的相似性对序列进行聚类并进行嵌合体的去除,将样品划分为不同的OTU,其中每个OTU中丰度最高的序列被选作代表序列。代表序列通过MUSCLE(Version 3.8.31)软件进行序列比对,进而构建所有OTU的系统进化树,物种注释选择SILVA 1.19 database(http://www.arb-silva.de/download/archive/qiime/)数据库,最后以测序数据量最小的样本为标准对所有样本的OTU丰度数据进行均一化处理。
1.4 土壤中青枯菌的定量检测采用定量PCR方法检测番茄根际土壤中青枯菌的丰度,以编码青枯菌鞭毛亚基的flic基因为特异性检测序列[23],其正向引物为:5'-GAACGCCAAC GGTGCGAACT-3',反向引物为:5'-GGCGGCCTTC AGGGAGGTC-3',定量PCR分析采用美国Applied Biosystems公司的7500 Real-Time PCR系统,采用SYBR Green I荧光染料进行。PCR反应体系为20 μL,包含SYBR@ Premix Ex TaqTM(2×)10 μL,ROX Reference Dye(50×)0.4 μL,DNA模板2 μL,正、反向引物(10 mmol·L–1)均为0.4 μL,加水补至25 μL。反应条件为:95℃ 30 s,95℃ 5 s,60℃ 30 s,72℃ 30 s,40个循环。该方案在95℃下进行15秒的熔体曲线分析,60℃下进行1 min分析,最后95℃下进行15 s分析。根据标准程序得到熔解曲线,并用于PCR产物特征峰的鉴定。
1.5 数据处理与统计分析数据处理和分析在R语言(Version 3.5.2)中完成。平均最近物种距离(between-community mean nearest taxon distance,βMNTD)和最近物种指数(between community nearest taxon index,βNTI)被用来计算样品间的系统发育多样性[24]。该过程由R软件的“picante”包中的“comdistnt”函数来完成。βNTI表示βMNTD的观测值和基于零模型计算的随机值之间的差异,βNTI的值在–2~2之间表示随机性过程,而βNTI的值< –2或 > 2则表示确定性过程。进一步,我们还利用表征Bray-Curtis距离观测值和基于零模型的随机值之间差异的Bray-Curtis-based Raup-Crick(RCbray)值来计算基于扩散影响的微生物群落构建过程[25]。确定性构建过程由同质性选择(HS,βNTI < –2)和异质性选择(VS,βNTI > 2)组成,随机性过程包括扩散限制(DL,|βNTI| < 2,RCbray > 0.95)、同质性扩散(HD,|βNTI| < 2,RCbray < –0.95)和生态漂变(UD,|βNTI| < 2,RCbray < 0.95)。
通过QIIME软件(Version 1.9.1)计算样本的多样性指数,其中基于物种水平的丰富度(richness)、Chao 1、和基于系统遗传发育水平的遗传多样性(Faith’s Phylogenetic diversity,Faith PD)用于表征群落alpha多样性。使用“vegan”包对基于Bray-Curtis距离(OTUs水平)进行主坐标轴分析(PCOA)用以展示微生物群落结构的差异,并利用置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA)来检验微生物群落结构差异的显著性。利用SPSS(Version 20.0)软件进行方差分析(ANOVA)。
2 结果 2.1 抑病土壤和感病土壤中番茄的株高与根际青枯菌密度变化感病土壤中番茄青枯病的病情指数明显高于抑病土壤(抑病土和感病土中的番茄病情指数分别为47.5和22.5)(图 1a),表明抑病土壤中番茄青枯病的发生明显得到缓解。qPCR结果表明,感病土壤和抑病土壤中的植物根际样品中均能检测到青枯菌,抑病土壤中的病原菌丰度显著低于感病土壤,其中每克感病根际土壤中log10(fliC)基因拷贝数为7.29~8.56,抑病土壤中则为6.79~6.94。与抑病土相比,感病根际样品中的病原菌的丰度增加了12.22倍(图 1b)。此外,抑病土中的健康番茄的株高显著高于感病土壤(图 1c)。
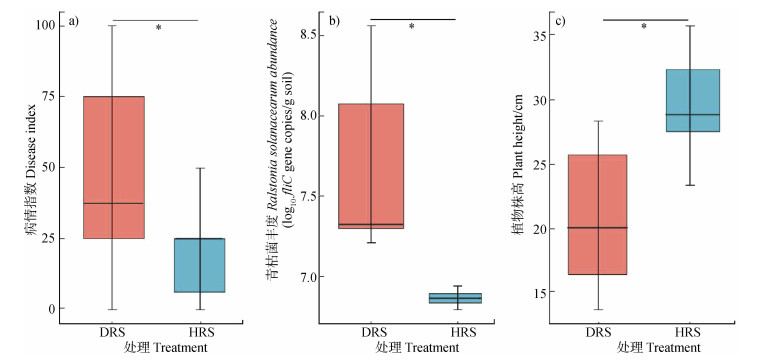
|
注:DRS:感病土壤的番茄根际土,HRS:抑病土壤的番茄根际土。下同。多组比较使用克鲁斯卡尔-沃利斯(Kruskal-Wallis)检验,成对比较使用Wilcoxon检验。“*”表示处理间差异显著(P < 0.05),n=7。 Note: DRS: rhizosphere soil of tomato in disease-conductive soil, HRS: rhizosphere soil of tomato in disease-suppressive soil. The same below.Kruskal-Wallis test was used for multi-group comparison and Wilcoxon test for paired comparison. "*"indicates significant differences at the 0.05 level, n=7. 图 1 感病土壤和抑病土壤的番茄青枯病的病情指数(a)、根际土壤青枯病菌丰度(b)、番茄株高(c) Fig. 1 The disease index of tomato wilt(a), Ralstonia solanacearum abundance of rhizospheric soil(b), and plant height(c)in disease-conductive soil and disease-suppressive soil |
抑病土壤和感病土壤根际中富集的微生物在门和科水平上的差异显著。在门水平上,变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)在根际土壤细菌微生物群落中占主导地位。与感病土壤相比,抑病土壤根际样品中的放线菌门、厚壁菌门的丰度明显高于感病土,而变形菌门和拟杆菌门的丰度则低于感病土(图 2a)。从科水平看,抑病土壤中富集了较多的芽孢杆菌科、链霉菌科等,而感病土壤中富集了较多的布克氏菌科和根瘤菌科等(图 2b)。
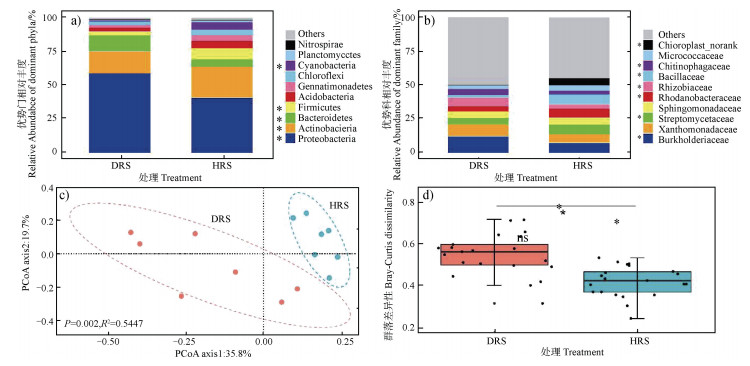
|
注:“*”表示处理间差异显著,“ns”表示处理间差异不显著(P < 0.05),n=4。 Note: "*"indicates significant differences and "ns" indicates there is no significance at the 0.05 level, n=4. 图 2 感病土壤与抑病土壤中优势细菌类群在门水平(a)和科水平(b)上的相对丰度以及细菌群落的主坐标轴分析(PCOA,c)和群落差异性分析(d) Fig. 2 Relative abundance of dominant bacterial groups at the phylum(a), family(b)level, and Principal coordinate analysis (PCOA, c)and its community dissimilarity analysis(d)in disease-conductive soil and disease-suppressive soil |
土壤细菌群落的alpha多样性通过丰富度(richness)、Chao 1以及遗传多样性(Faith PD)来表征。抑病土壤中根际细菌群落的丰富度、多样性和遗传多样性均显著高于感病土壤(表 2)。基于OTU的主坐标轴分析(PCoA)结果显示(图 2c),感病土壤根际和抑病土壤的群落结构差异明显(P = 0.002,R2 = 0.544 7),能沿着轴一明显区分(35.8%),且群落差异分析(PERMANOVA)表明感病土壤和抑病土壤根际微生物群落差异极显著(图 2d)。
|
|
表 2 不同处理的alpha多样性指数 Table 2 The alpha diversity indices in different treatments |
驱动感病土微生物群落构建的生态过程主要由扩散限制过程(52%)控制,同质选择和生态漂变过程各占34%和14%;而在抑病土中,扩散限制过程占61%,同质选择与异质选择各占19%和9%。从感病土壤到抑病土壤,同质选择过程明显降低(从34%降至19%),扩散限制过程则从52% 增至62%(表 3)。进一步分析发现,βNTI值呈上升趋势,但并未达到显著(图 3a):感病土中确定性过程的贡献(34%)略高于其在抑病土中的贡献(28%)。确定性过程均由同质性选择主导,且该过程在感病土中较高;而随机性过程均以扩散限制过程占主导,且其在抑病土中较高(图 3b)。
|
|
表 3 不同处理微生物群落构建的确定性、随机性过程 Table 3 Deterministic and stochastic processes influencing community assembly under different treatment |

|
注:多组比较使用克鲁斯卡尔-沃利斯(Kruskal-Wallis)检验,成对比较使用Wilcoxon检验。“*”表示处理间差异显著,“ns”表示处理间差异不显著(P < 0.05),n=4。 Note: Kruskal-Wallis test was used for multi-group comparison and Wilcoxon test for paired comparison."*"indicates significant differences and"ns"indicates there is no significance at the 0.05 level, n=4. 图 3 不同处理的微生物群落的βNTI值(a,c)及其群落组装过程(b,d) Fig. 3 The βNTI(a, c)and its assembly processes(b, d)of bacterial communities in different treatments |
随着抑病土比例的增加,开花期时番茄病情指数下降,其病情指数分别为41. 67、29.17与16.67(图 4a)。3种处理的根际样品中均都能检测到青枯菌,且病原菌丰度随着抑病土比例的增加呈现明显降低的趋势(图 4b),与感病土样品相比,掺混土和抑病土样品根际中青枯病菌的密度分别减少了2.51倍和5.37倍。此外,3种处理中番茄的株高呈上升趋势,但并未达到显著(图 4c)。

|
注:“*”表示处理间差异显著,“ns”表示处理间差异不显著(P < 0.05),n=4。 Note: "*"indicates significant differences and"ns"indicates there is no significance at the 0.05 level, n=4. 图 4 掺混试验的番茄青枯病的病情指数(a)、根际土壤青枯病菌丰度(b)、番茄株高(c) Fig. 4 The disease index of tomato wilt(a), Ralstonia solanacearum abundance of rhizopheric soil(b), and plant height (c)in mixing experiment |
与感病土相比,变形菌门(Proteobacteria)和布克氏菌科的丰度明显成梯度降低(图 5a),而厚壁菌门、链霉菌科和芽孢杆菌科的丰度明显增加,且随着抑病土的比例增加呈明显上升(图 5b)。3种处理的微生物多样性也存在明显不同,掺混土和抑病土样品中的细菌群落的丰富度和遗传多样性较感病土增加,且遗传多样性随着抑病土的比例增加呈明显上升趋势(表 2)。基于OTU的主坐标轴分析(PCoA)结果显示,感病土壤、抑病土壤以及掺混土的根际细菌群落结构差异明显(图 5c,R2 = 0.7928,P = 0.001),能沿着轴1明显区分(45.4%),且相似性分析(PERMANOVA)表明群落结构差异极显著(图 5d)。
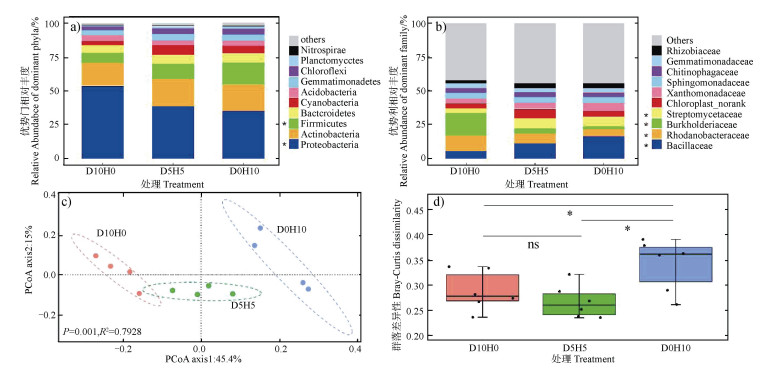
|
注:“*”表示处理间差异显著,“ns”表示处理间差异不显著(P < 0.05),n=4。 Note: "*"indicates significant differences and"ns"indicates there is no significance at the 0.05 level, n=4. 图 5 掺混试验的优势细菌类群在门水平(a)和科水平(b)上的相对丰度以及细菌群落的主坐标轴分析(PCoA,c)和群落差异性分析(d) Fig. 5 The relative abundance of dominant bacterial groups at the phylum(a), family(b)level, and Principal coordinate analysis(PCoA, c)and its community dissimilarity analysis(d)in mixing experiment |
βNTI值随着抑病土的比例增加呈显著上升梯度(图 3c,表 3):确定性过程主导感病土样品根际细菌群落(67%);掺混土样品中,确定性过程降低至34%,随机性过程增加到66%;而随机性过程主导抑病土样品根际细菌群落。随着抑病土比例的增加,确定性过程的贡献逐渐降低,随机性过程的贡献增加。进一步发现,感病土中确定性过程均由同质性选择主导,随机性则由生态漂变过程主导(图 3d)。
3 讨论 3.1 抑病土壤的根际微生物组特征根际土壤中病原菌的含量在感病土壤中明显高于抑病土壤,它的大量存在影响了植物的生长。在我们的研究中,病原菌的入侵降低了根际土壤微生物多样性,如丰富度、Chao1和遗传多样性在感病土中病株根际样本中显著下降。大量研究表明,根际土壤微生物多样性越低,越有利于病原菌入侵[26]。病原菌的入侵还能影响宿主植物的根际微生物组成。例如,与健康番茄根际相比,发病番茄根际样品中的有益的革兰氏阳性细菌群落的失衡会加重病害的发生,如放线菌门和厚壁菌门的丰度失调[27-28]。同样的,在本研究中,与感病土中的发病番茄根际相比,抑病土中富集了较高丰度的放线菌门、厚壁菌门、芽孢杆菌科、链霉菌科。而厚壁菌门和放线菌门及其类别被报道与病害抑制相关[29]。具体地,链霉菌科属于放线菌门,而放线菌常因其抗生作用被用来生产临床用的抗生素[30],故能对青枯菌的生长产生抑制[31];而芽孢杆菌科属于厚壁菌门,也因其对青枯病菌以及其他病原菌的拮抗活性(脂肽类抗生素)而闻名[28,32]。因此,病原菌的入侵破坏了宿主植物的根际微生物组,并促使宿主植物根际招募更多有益微生物以抵御病害的进一步入侵。
3.2 细菌群落重组与病害抑制研究发现,抑病土壤的抑病特性会传递至下一代,即形成“土壤遗产”,而形成的“土壤遗产”会帮助同种下茬作物抵御同种病原菌的入侵,从而有利于后代植物生长[10,33]。本研究通过抑病土壤与感病土壤等质量掺混评估了抑病土壤抑病特性的传递性。结果发现抑病土与感病土进行掺混后,番茄的病情指数下降,从D10H0的41.67%降低至D5H5的29.17%,番茄根际土壤中的青枯菌丰度也明显降低,而根际有益微生物,厚壁菌门、链霉菌科和芽孢杆菌科等的丰度也随着抑病土的比例的降低而明显降低。因此,可以合理推测,抑病土壤的“土壤遗产”的形成与抑病土中上一代植物根际募集的有益微生物有关,而募集的有益微生物能在土壤中蛰伏以对病害再次入侵时作出响应。本研究再次验证了抑病土壤的存在能够对病害产生一定的抑制作用从而促使植物与土壤保持健康,并且抑病土壤会通过形成“土壤遗产”来辅助后代作物应对病害的侵袭,如同人体的免疫反应。
3.3 植物病害与群落构建无论是抑病土壤还是感病土壤,随机性过程和确定性过程均共同驱动微生物群落结构的形成(图 3和表 3)。研究表明,确定性过程可以强化特定环境条件下的土壤微生物群落的生态功能,而随机性过程则能产生更多样化的生态功能以维持微生物群落的稳定性和持续性 [34],同时,随机性过程主导的微生物群落可以对于外界环境的变化所带来的扰动起到一定的缓冲作用,故随机性过程对于系统的稳定性有一定的正反馈调节作用[35-36]。因此,抑病土壤根际中增加的随机性过程可提供更为丰富的微生物多样性。相似地,本研究中,无论是丰富度还是系统遗传发育多样性,细菌群落alpha多样性从感病土到抑病土显著增加(表 2),而抑病土壤中的随机性过程的重要性高于感病土壤。抑病土与感病土的盆栽掺混试验进一步证明了这一观点。从D10H0到D5H5,再到D0H10,随着抑病土比例的增加,其细菌alpha多样性增加,而随机性过程的在微生物群落构建过程中的作用也随之明显增加,而由于随机性过程主导的微生物群落可以对于土传病害的入侵所带来的扰动起到一定的缓冲作用,因此番茄的发病率明显降低。本研究从微生物群落构建的层面更好地揭示了抑病土壤和感病土壤根际微生物群落和植物病害的关系,同时抑病土壤对植物病害的拮抗、保护土壤健康与植物健康方面具有不可替代的重要作用。应进一步探究抑病土壤微生物群落构建过程调控根际微生物群落结构、组成、多样性与宿主植物之间的相互关系,以便为抑病土在土传病害防控应用方面提供理论支撑。
4 结论抑病土和感病土在根际微生物群落结构多样性、组成以及群落构建过程存在差异。抑病土壤中植物根际能富集较为丰富的有益微生物,与感病土壤相比,随机性过程增加,确定性过程降低。随机性过程维持了抑病土根际微生物的多样性,使之加强了根际对病害胁迫的抵抗能力,有助于根际微生物帮助宿主植物适应病害胁迫。然而,抑病土壤中青枯病害的具体影响机制还有待进一步深入研究,以便为抑病土在土传病害防控上提供理论参考。
| [1] |
Hayward A C. Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum[J]. Annual Review of Phytopathology, 1991, 29(1): 65-87. DOI:10.1146/annurev.py.29.090191.000433
(  0) 0) |
| [2] |
Jiang G F, Wei Z, Xu J, et al. Bacterial wilt in China: History, current status, and future perspectives[J]. Frontiers in Plant Science, 2017, 8: 1549. DOI:10.3389/fpls.2017.01549
(  0) 0) |
| [3] |
Kwak M J, Kong H G, Choi K, et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature Biotechnology, 2018, 36(11): 1100-1109. DOI:10.1038/nbt.4232
(  0) 0) |
| [4] |
Wei Z, Wang J N, Jiang G F, et al. Survival-virulence trade-off of soil-borne pathogenic bacteria (In Chinese)[J]. Acta Pedologica Sinica, 2021. DOI:10.11766/trxb202008310399 [土传病原细菌的生存与致病权衡[J]. 土壤学报, 2021.]
(  0) 0) |
| [5] |
Zhang P, Wang X H, Li R, et al. Effect of bio-organic fertilizer on pathogenic and functional bacteria composition in rhizospheric soil of field vegetables (In Chinese)[J]. Acta Pedologica Sinica, 2013, 50(2): 381-387. [生物有机肥对田间蔬菜根际土壤中病原菌和功能菌组成的影响[J]. 土壤学报, 2013, 50(2): 381-387.]
(  0) 0) |
| [6] |
Wei Z, Gu Y A, Friman V P, et al. Initial soil microbiome composition and functioning predetermine future plant health[J]. Science Advances, 2019, 5(9): eaaw0759. DOI:10.1126/sciadv.aaw0759
(  0) 0) |
| [7] |
Chapelle E, Mendes R, Bakker P A, et al. Fungal invasion of the rhizosphere microbiome[J]. The ISME Journal, 2016, 10: 265-268. DOI:10.1038/ismej.2015.82
(  0) 0) |
| [8] |
Weller D M, Raaijmakers J M, Gardener B B M, et al. Microbial populations responsible for specific soil suppressiveness to plant pathogens[J]. Annual Review of Phytopathology, 2002, 40(1): 309-348. DOI:10.1146/annurev.phyto.40.030402.110010
(  0) 0) |
| [9] |
Liu X J, Zhang S T, Jiang Q P, et al. Using community analysis to explore bacterial indicators for disease suppression of tobacco bacterial wilt[J]. Scientific Reports, 2016, 6: 36773. DOI:10.1038/srep36773
(  0) 0) |
| [10] |
Raaijmakers J M, Mazzola M. Soil immune responses[J]. Science, 2016, 352(6292): 1392-1393. DOI:10.1126/science.aaf3252
(  0) 0) |
| [11] |
Henry A W. The natural microflora of the soil in relation to the foot-rot problem of wheat[J]. Canadian Journal of Research, 1931, 4(1): 69-77. DOI:10.1139/cjr31-006
(  0) 0) |
| [12] |
Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health[J]. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001
(  0) 0) |
| [13] |
Dini-Andreote F. Endophytes: The second layer of plant defense[J]. Trends in Plant Science, 2020, 25(4): 319-322. DOI:10.1016/j.tplants.2020.01.007
(  0) 0) |
| [14] |
Lozupone C A, Knight R. Global patterns in bacterial diversity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(27): 11436-11440. DOI:10.1073/pnas.0611525104
(  0) 0) |
| [15] |
Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(3): 626-631. DOI:10.1073/pnas.0507535103
(  0) 0) |
| [16] |
Chase J M, Myers J A. Disentangling the importance of ecological niches from stochastic processes across scales[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2011, 366(1576): 2351-2363. DOI:10.1098/rstb.2011.0063
(  0) 0) |
| [17] |
Liu W J, Graham E B, Dong Y, et al. Balanced stochastic versus deterministic assembly processes benefit diverse yet uneven ecosystem functions in representative agroecosystems[J]. Environmental Microbiology, 2021, 23(1): 391-404. DOI:10.1111/1462-2920.15326
(  0) 0) |
| [18] |
Feng M M, Adams J M, Fan K K, et al. Long-term fertilization influences community assembly processes of soil diazotrophs[J]. Soil Biology & Biochemistry, 2018, 126: 151-158.
(  0) 0) |
| [19] |
Shi Y, Li Y T, Xiang X J, et al. Spatial scale affects the relative role of stochasticity versus determinism in soil bacterial communities in wheat fields across the North China Plain[J]. Microbiome, 2018, 6(1): 27. DOI:10.1186/s40168-018-0409-4
(  0) 0) |
| [20] |
Huang X Q, Zhou X, Zhang J B, et al. Highly connected taxa located in the microbial network are prevalent in the rhizosphere soil of healthy plant[J]. Biology and Fertility of Soils, 2019, 55(3): 299-312. DOI:10.1007/s00374-019-01350-1
(  0) 0) |
| [21] |
Gómez Expósito R, de Bruijn I, Postma J, et al. Current insights into the role of rhizosphere bacteria in disease suppressive soils[J]. Frontiers in Microbiology, 2017, 8: 2529. DOI:10.3389/fmicb.2017.02529
(  0) 0) |
| [22] |
Lu R K. Analytical methods for soil and agrochemistry (In Chinese). Beijing: China Agricultural Science and Technology Press, 2000. [土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000.]
(  0) 0) |
| [23] |
Schönfeld J, Heuer H, van Elsas J D, et al. Specific and sensitive detection of Ralstonia solanacearum in soil on the basis of PCR amplification of fliC fragments[J]. Applied and Environmental Microbiology, 2003, 69(12): 7248-7256. DOI:10.1128/AEM.69.12.7248-7256.2003
(  0) 0) |
| [24] |
Stegen J C, Lin X J, Konopka A E, et al. Stochastic and deterministic assembly processes in subsurface microbial communities[J]. The ISME Journal, 2012, 6(9): 1653-1664. DOI:10.1038/ismej.2012.22
(  0) 0) |
| [25] |
Stegen J C, Lin X J, Fredrickson J K, et al. Quantifying community assembly processes and identifying features that impose them[J]. The ISME Journal, 2013, 7(11): 2069-2079. DOI:10.1038/ismej.2013.93
(  0) 0) |
| [26] |
Locey K J, Lennon J T. Scaling laws predict global microbial diversity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(21): 5970-5975. DOI:10.1073/pnas.1521291113
(  0) 0) |
| [27] |
Lee S M, Kong H G, Song G C, et al. Disruption of Firmicutes and Actinobacteria abundance in tomato rhizosphere causes the incidence of bacterial wilt disease[J]. The ISME Journal, 2021, 15(1): 330-347. DOI:10.1038/s41396-020-00785-x
(  0) 0) |
| [28] |
Wei Z, Hu J, Gu Y A, et al. Ralstonia solanacearum pathogen disrupts bacterial rhizosphere microbiome during an invasion[J]. Soil Biology and Biochemistry, 2018, 118: 8-17. DOI:10.1016/j.soilbio.2017.11.012
(  0) 0) |
| [29] |
Mendes R, Kruijt M, de Bruijn I, et al. Deciphering the rhizosphere microbiome for disease-suppressive bacteria[J]. Science, 2011, 332(6033): 1097-1100. DOI:10.1126/science.1203980
(  0) 0) |
| [30] |
de Lima Procópio R E, da Silva I R, Martins M K, et al. Antibiotics produced by Streptomyces[J]. The Brazilian Journal of Infectious Diseases, 2012, 16(5): 466-471. DOI:10.1016/j.bjid.2012.08.014
(  0) 0) |
| [31] |
Yuliar, Nion Y A, Toyota K. Recent trends in control methods for bacterial wilt diseases caused by Ralstonia solanacearum[J]. Microbes and Environments, 2015, 30(1): 1-11. DOI:10.1264/jsme2.ME14144
(  0) 0) |
| [32] |
Wei Z, Yang X M, Yin S X, et al. Efficacy of Bacillus-fortified organic fertiliser in controlling bacterial wilt of tomato in the field[J]. Applied Soil Ecology, 2011, 48(2): 152-159. DOI:10.1016/j.apsoil.2011.03.013
(  0) 0) |
| [33] |
Bakker P A, Pieterse C M J, de Jonge R, et al. The soil-borne legacy[J]. Cell, 2018, 172(6): 1178-1180. DOI:10.1016/j.cell.2018.02.024
(  0) 0) |
| [34] |
Knelman J, Nemergut D. Changes in community assembly may shift the relationship between biodiversity and ecosystem function[J]. Frontiers in Microbiology, 2014, 5: 424.
(  0) 0) |
| [35] |
Graham E, Stegen J C. Dispersal-based microbial community assembly decreases biogeochemical function[J]. Processes, 2017, 5.
(  0) 0) |
| [36] |
Chase J M. Stochastic community assembly causes higher biodiversity in more productive environments[J]. Science, 2010, 328(5984): 1388-1391. DOI:10.1126/science.1187820
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


