2. 中国科学院大学, 北京 100049;
3. 南京农业大学生命科学学院, 南京 210095;
4. 浙江农林大学亚热带森林培育国家重点实验室, 杭州 311300;
5. 华东师范大学地理科学学院地理信息科学教育部重点实验室, 上海 200241;
6. 南京农业大学资源与环境科学学院, 南京 210095;
7. 盐城师范学院海洋与生物工程学院, 江苏盐城 224007
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Life Sciences, Nanjing Agricultural University, Nanjing 210095, China;
4. State Key Laboratory of Subtropical Silviculture, Zhejiang Agriculture and Forestry University, Hangzhou 311300, China;
5. Key Laboratory of Geographic Information Science (Ministry of Education), School of Geographic Sciences, East China Normal University, Shanghai 200241, China;
6. College of Resources and Environmental Science, Nanjing Agricultural University, Nanjing 210095, China;
7. School of Marine and Biological Engineering, Yancheng Teachers University, Yancheng, Jiangsu 224007, China
地球上酸性土壤约占无冰陆地面积的30%(pH < 5.5)[1],而几乎所有微生物均须维持细胞内的中性稳态环境[2],极可能通过消耗更多能量克服细胞膜内外巨大的质子梯度适应酸性胁迫。尤其是考虑到土壤中微生物数量巨大,每克土壤可能栖息着数以亿计的微生物细胞,其耐酸生理生长机制不仅是重要科学前沿,也是农业生产的重要实践问题[3]。例如,全球每年施用氮肥一亿多吨,大量氮肥进入土壤后促进氨氧化微生物生长并释放质子,进一步加剧土壤酸化,可能导致微生物功能丧失,降低作物氮磷钾养分利用效率,而我国氮肥施用量占全球比例高达32%,土壤酸化及其次生障碍问题尤为突出[4]。
氮素是所有生物生长的重要营养元素,而微生物将氨氧化为亚硝酸盐则是地球氮素循环的关键限速步骤[5]。2005年科学家发现海洋古菌具有氨氧化能力,根本改变了地球氮循环的研究理念[6]。随后大量分子生态学研究表明,氨氧化古菌(Ammonia-oxidizing archaea,AOA)是酸性土壤氨氧化的主要作用者,并与海洋古菌AOA具有更近的系统发育进化关系[7-10],但其嗜酸机制报道较少。已有研究表明AOA含有脲酶,通过水解尿素获得底物氨分子,同时提高胞内pH适应酸性胁迫[10-11],而基于纯菌株Ca. Nitrosotaleales devanaterra的研究则认为AOA嗜酸机制与已知的其他嗜酸菌基本一致[12]。嗜酸菌维持胞内pH稳态机制主要包括:(1)形成具有极低质子渗透率的特殊细胞膜[13];(2)通过吸收K+等形成膜内正电位抑制H+流入[14];(3)通过谷氨酸和精氨酸脱羧作用消耗质子,然后将质子运送出细胞等[15]。值得注意的是,酸性土壤并非严格的极端环境,迄今尚未发现最适pH低于3.0、严格意义的极端嗜酸氨氧化微生物。事实上,酸性土壤中极可能存在中性和碱性微域,分离到的氨氧化古菌通常为中度嗜酸菌[8],最佳生长pH在3.0~5.0之间;而最近分离的菌株则为耐酸菌[16],最佳生长pH高于5.0,但在较低的pH下也能生长[17]。
ATP是所有生物生长的能量货币,而氨氧化古菌是严格化能无机自养菌,通过氧化氨态氮获得能量,同化无机碳自养生长。然而,值得注意的是,尽管氨单加氧酶amoA基因与核糖体16S rRNA基因均可作为氨氧化古菌和细菌的分类标靶,但二者均无法解释氨氧化古菌的生境偏好性。例如,通过稳定性同位素示踪氨氧化古菌核酸DNA,2014年我们首次发现酸性土壤中存在着尚未可知的嗜酸/耐酸AOA,这些古菌AOA在酸性土壤中分裂生殖产生新细胞被13C所标记,具有较强的氨氧化活性,但其amoA基因和16S rRNA基因却与中性环境中的Nitrososphaerales古菌具有更近的亲缘关系,与已知的嗜酸古菌Ca. Nitrosotaleales截然不同[18]。我们推测,与氨单加氧酶相比,古菌AOA的ATPase可能在适应酸性环境过程中发挥了更为重要的作用。事实上,质子进入AOA细胞与ATP酶(ATPase)密切相关,AOA通过氨氧化与电子转移系统耦合产生质子动力,并通过ATPase合成生命活动所需能量ATP[19]。因此,编码ATP合酶的基因不仅是微生物能量产生的遗传基础,也可能在古菌AOA在适应酸性胁迫环境过程中发挥了重要作用。
ATPase广泛存在于古菌、细菌和真核生物,主要包括古菌A-ATPase,细菌、线粒体和叶绿体中常见的F-ATPase,以及真核生物V-ATPase等[20]。V-ATPase是由ATP驱动的离子泵,最初是在细胞器膜中发现,其功能主要是酸化细胞器(如溶酶体、核内体、液泡等),但越来越多的研究表明,V-ATPase在多种细胞的质膜质子转运中发挥着重要作用[21-23],如在巨噬细胞和中性粒细胞中,质膜V-ATPase能够将细胞内质子转运到细胞外,使细胞内保持中碱性pH稳态环境[24]。古菌的A-ATPase在功能上类似于F-ATPase,利用质子动力催化ATP合成(反应可逆),在结构上却更类似于V-ATPase,并且一系列证据表明V-ATPase可能由A-ATPase演变而来[20]。因此,古菌A-ATPase可能具有与质膜V-ATPase类似的调节细胞内pH的功能。我们最近的研究则表明,V-ATPase可能在古菌AOA嗜酸过程中发挥了重要作用,将AOA的V-ATPase基因转入大肠杆菌异源表达,显著增加了大肠杆菌在低pH条件下的生长速率,表明V-ATPase是AOA适应酸性胁迫的关键模块[25]。同时,所有陆地生态系统中性和碱性AOA菌株均含有A-ATPase,而嗜酸/耐酸AOA菌株含有V-ATPase。这一结果表明,尽管ATPase基因不能作为古菌AOA的系统发育分类分子标靶基因,但却能较好解释其生境分布规律[25]。但这一结论是否具有一定的普适性,尚缺乏明确证据。
据此,本研究利用深度宏基因组测序技术,针对浙江省建德市不同种植年限的马尾松人工林酸性土壤,研究不同林龄马尾松人工林土壤中AOA群落组成及影响AOA生态分化的环境因素,通过对amoA基因和ATPase基因的宏基因组数据分析,探究酸性环境中AOA与ATPase之间的关系,以期为AOA在地球环境中的生境扩张及遗传适应机制提供更多证据。
1 材料与方法 1.1 供试材料土壤样品采自浙江省建德市马尾松人工林(29.48°N,119.28°E),土壤类型为凝灰岩发育的红壤。该区为亚热带海洋型季风气候,海拔约200 m,年均气温17.4℃,年均降雨量1 600 mm。选择中龄林(15 a)、近熟林(24 a)、成熟林(45 a)、过熟林Ⅰ(55 a)、过熟林Ⅱ(63 a)的马尾松人工林土壤样品采样。具体设计如下:针对每一种年限,选择3个不同小区作为重复,空间间隔约2 000 m,每个小区由5个土柱样品混匀而成,采样深度为0~20cm,样品分成两份,一份保存于–20℃用于提取土壤总DNA,另一份经风干、磨细、过2 mm筛、混匀后在4℃保存,参照《土壤农化分析》[26],测定土壤理化指标。
1.2 土壤DNA提取和深度宏基因组测序土壤微生物组总DNA使用FastDNA Spin Kit for Soil(MP Biomedicals,USA)试剂盒提取,并通过DNeasy PowerClean Pro Cleanup Kit(QIAGEN,Germany)试剂盒纯化获得高质量DNA,具体步骤参考相关操作指南。通过微量紫外分光光度计(Nanodrop ND-1000)测定DNA的浓度和纯度后,保存于–20℃。
宏基因组测序主要通过Illumina Hi Seq 2000平台完成。首先利用自动聚焦声波基因组剪切仪Covaris M220,将土壤总DNA随机打断为约300 bp的片段,然后采用TruSeqTM DNA Sample Prep Kit试剂盒构建PE(Paired-End)文库,最后通过HiSeq 3000/4000 PE Cluster Kit试剂盒进行桥式PCR反应,质量检测合格后进行双末端(Paired-End,PE)测序。5种不同种植年限的森林土壤及其3个生物学重复均开展了深度测序,每个样品约50 G bp原始数据,共计获得约750 G bp高通量数据。
高质量数据分析具体如下:利用软件SeqPrep剪切序列3´端和5´端的adaptor序列,随后删除所有长度小于50 bp的序列reads;通过软件Sickle去除平均质量值低于20以及含N碱基的序列reads,保留高质量的reads,每个样品最终得到约49 G bp高质量数据。
1.3 高通量深度测序宏基因组注释策略及丰度分析以NCycDB数据库中AOA amoA基因序列[27]和最近发表的ATP酶A亚基(ATPase subunit A)蛋白序列[25]为参考序列,通过比对提取E≤10–4的序列reads,将其定义为AOA序列,并使用SPAdes软件进行拼接组装为大片段重叠群(contigs)(k-mer设置为21~29),所有样品测序数据最后均一化成50 G bp进行分析。
根据已有的AOA物种多样性报道[25,28],构建AOA amoA基因和ATPase subunit A蛋白数据库。根据AOA的分类学文献[28]对AOA进行分类。每个土壤样品中AOA的注释及其丰度计算方法如下:(1)方法一:首先利用BLAST将contigs与amoA基因数据库比对,通过聚类软件CD-HIT以默认参数对contigs(Identity≥0.90,Coverage≥120)进行聚类,构建amoA基因系统发育树以进一步筛选并确定代表序列的物种分类地位,进而确定聚类中各contigs所属类群。随后通过BLAST将contigs与ATPase subunit A蛋白数据库比对,通过ORF Finder将核酸序列(Identity≥0.60,Coverage≥120)翻译为蛋白序列并通过聚类软件CD-HIT以默认参数对蛋白序列聚类,构建ATPase subunit A蛋白系统发育树筛选并确定代表序列的物种分类地位,进而确定聚类中各contigs所属类群。统计用于拼接contigs序列上属于目标基因片段的reads(参与拼接长度≥30 bp)作为丰度。(2)方法二:利用BLAST将contigs分别与amoA基因和ATPase subunit A蛋白数据库比对,得到候选contigs,然后分别通过构建amoA基因和ATPase subunit A蛋白系统发育树明确各候选contigs的物种分类地位,丰度计算与方法一相同。(3)方法三:利用BLAST将contigs分别与AOA amoA基因和ATPase subunit A蛋白数据库比对,根据比对结果确定amoA contigs(Identity≥0.90,Coverage≥120)和ATPase subunit A contigs(Identity≥0.60,Coverage≥120)的物种分类地位,丰度计算与方法一相同。(4)方法四:直接利用BLAST获得短片段序列reads(E≤10–4),分别与amoA基因和ATPase subunitA蛋白数据库比对,根据比对结果确定amoA reads(Identity≥0.95,Coverage≥ 30)和ATPase subunit A reads(Identity≥0.80,Coverage≥60)的物种分类地位,并将reads数均一化后作为其丰度。
1.4 氨氧化古菌分子系统发育树构建利用CD-HIT软件对amoA contigs和ATPase subunit A contigs序列进行聚类,软件设置为默认参数得到代表性序列。利用MAFFT软件根据默认参数对代表性序列进行比对,然后用IQ-TREE软件构建amoA基因和ATPase subunit A蛋白系统发育树,参数设置为“–MFP –bb 1000”。系统发育树中的经典物种采用已有的纯菌株和富集物序列作为参考。
1.5 数据处理利用Microsoft Excel 2016对数据进行前处理,IBM SPSS Statistics 20进行差异显著性分析(LSD法,α=0.05)和Spearman分析;利用R软件vegan包开展Mantel分析、ANOISM分析;利用OriginPro 8.0制作箱线图;并利用软件Adobe Illustrator CS5优化调整。
2 结果 2.1 马尾松人工林土壤理化性质人工种植导致马尾松林土壤性质发生显著变化。如表 1所示,低龄林(15 a和24 a)的土壤含水量显著大于高龄林土壤(45 a、55 a和63 a)。与初始15 a中龄林土壤相比,长期种植导致马尾松林土壤pH、有机碳、全氮、水解性氮、速效钾含量均呈下降趋势,尤其在63 a过熟林Ⅱ均达到统计显著性(P < 0.05)。有效磷则表现出相反的规律,与初始15 a中龄林土壤相比,其含量呈增加趋势,尤其在63 a过熟林Ⅱ土壤,从6.60 mg·kg–1增至17.0 mg·kg–1。此外,与45 a成熟林相比,55 a和63 a土壤含水率、pH、有机碳、全氮和水解性氮均未有显著差异,有效磷和速效钾均呈增加趋势并在63 a土壤具有统计显著性。
|
|
表 1 马尾松人工林土壤基本理化性质 Table 1 Physico-chemical properties of Pinus massoniana plantation soil |
针对5种土壤3个重复的750 G bp高质量测序数据,通过4种方法重构amoA基因和ATPase subunit A基因的系统发育进化规律。方法一、方法二和方法三均通过将高质量序列拼接为大片段contigs后,构建系统发育进化关系或直接比对进行注释;而方法四则通过直接分析高通量序列reads并注释。结果表明,方法一注释策略最佳,故选择方法一结果构建系统发育进化关系。
系统发育分析amoA基因发现其进化距离较远,在分类学上归属于两个目,分别为Ca.Nitrosotaleales(NT)和Nitrososphaerales(NS)。而NT和NS目可进一步细分为3个亚类,NT-α、NS-γ-2.3、NS-ζ亚类,这些类群占所有AOA的丰度分别为28.1%、60.3%和10.8%,合计99.2%(图 1a)。此外,土壤中也检测到少量NS-δ亚类,其丰度约为0.9%(图 1a)。
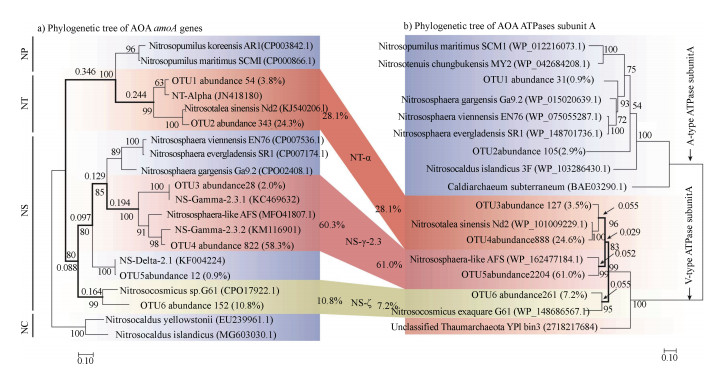
|
图 1 马尾松人工林土壤AOA amoA基因(a)和ATPase subunit A(b)系统发育进化树及趋同分析 Fig. 1 Phylogenetic tree and convergence analysis of AOA amoA gene(a)and ATPase subunit A(b)in Pinus massoniana plantation soil |
然而,ATPase subunit A基因分析则得到完全不同的结果,土壤中优势AOA具有较高的序列亲缘相似度,均可归类为V-ATPase并占所有序列96.3%。采用amoA基因的物种命名并用斜体作为区分,则发现V-ATPase可细分为3个亚类NT-α、NS-γ-2.3、NS-ζ、其丰度分别为28.1%、61.0%、7.2%(图 1b)。而仅有3.8%的序列可归类为A-ATPase,其amoA基因的分类地位为NS和NP目,亚类为NS-α和NP-γ。
这些结果表明,V-ATPase基因可能是古菌AOA适应酸性胁迫并发生趋同进化的分子基础。本研究中所有5种土壤均为酸性(pH < 5.0),而基于amoA基因的分类无法解释AOA古菌的酸性胁迫适应规律。如图 1a所示,AOA古菌NS目和NT目具有显著的系统进化分异,前者占比高达71.1%,后者占比仅为18.1%,但前者的亲缘物种多见于中碱性土壤,而后者的亲缘物种主要存在于酸性土壤。相反,ATPase subunit A系统进化分析则发现,96.3%的AOA均可归类为V-ATPase古菌AOA,表明不同类型的古菌AOA(NS和NT目)长期适应酸性胁迫过程中,可能通过基因水平转移,获得了相同/相似的V-ATPase基因适应酸性胁迫,发生了趋同进化。
2.3 不同种植年限下酸性森林土壤氨氧化古菌amoA和ATPase subunit A基因丰度变化进一步在更精细化水平,研究了不同种植年限下酸性森林土壤AOA amoA和ATPase subunit A基因分化及其丰度变化规律(图 2)。针对amoA基因,四种注释方法均发现土壤中amoA基因具有较远的进化亲缘关系,在分类学上可归类为NS和NT两个AOA目。通过拼接contigs注释的方法一(图 2a)、方法二(图 2b)和方法三(图 2c),均发现NS目含有3个亚类NS-γ-2.3、NS-ζ和NS-δ,而通过直接注释高通量测序序列的方法四也发现了这三个亚类,并检测到一个数量上极为弱势的NS-β(图 2d)。同时,所有方法均发现NT目仅有一个亚类NT-α。
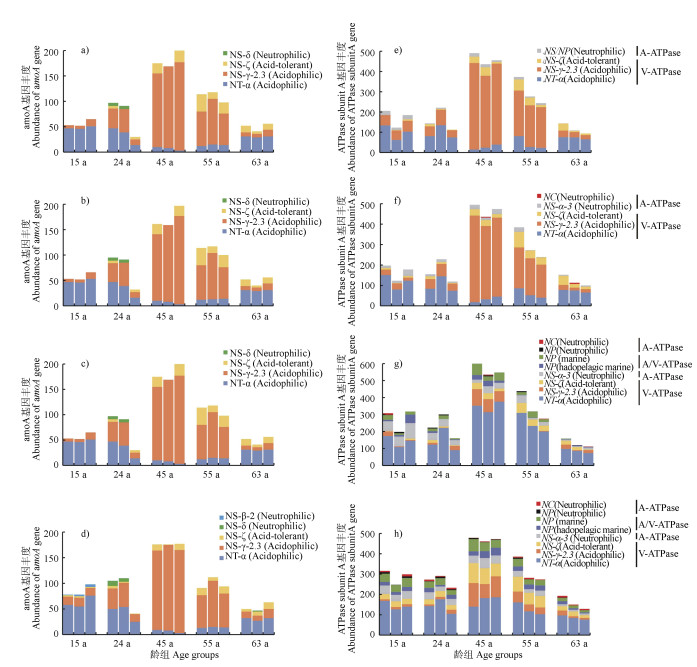
|
注:(a)、(b)、(c)、(d)分别是通过方法一、方法二、方法三、方法四得出的AOA amoA基因分类及其丰度;(e)、(f)、(g)、(h)分别是通过方法一、方法二、方法三、方法四得出的ATPase subunit A基因分类及其丰度。 Note: (a), (b), (c), (d)are the AOA amoA gene classifications and their abundances obtained by method 1, method 2, method 3, and method 4 respectively; (e), (f), (g), (h)are the classification and abundance of ATPase subunit A gene obtained by method 1, method 2, method 3, and method 4 respectively. NP, NT, NS, NC are the Nitrosopumilales, Ca. Nitrosotaleales, Nitrososphaerales, and Ca. Nitrosocaldales respectively. 图 2 不同注释方法马尾松人工林土壤AOA amoA基因和ATPase subunit A基因丰度 Fig. 2 The results of different annotation methods for AOA amoA gene and ATPase subunit A gene in Pinus massoniana plantation soil |
尽管AOA古菌ATPase subunit A基因与amoA基因的亲缘进化规律明显不同(图 1),但在精细化分类水平ATPase基因的相对丰度则与amoA基因丰度有较为一致的规律。采用amoA基因的物种命名并用斜体作为区分,ATPase subunit A占绝对优势的类群均为NT-α和NS-γ-2.3。值得注意的是,四种分类方法针对NT-α和NS-γ-2.3具有不同的分类结果,与方法一(图 2e)和方法二(图 2f)相比,方法三(图 2g)和方法四(图 2h)将部分NS-γ-2.3亚类AOA也归类至NT-α,导致后者丰度相对较高。但由于这一分类不确定性仅存在于NT-α和NS-γ-2.3之间,对其相对丰度及本文结论无影响,故在本文中重点采用方法一的数据。
amoA分析结果均表明,NT-α和NS-γ-2.3亚类是不同种植年限下马尾松林土壤中的优势AOA类群(图 2a),在不同种植年限的5种土壤中,其总丰度分别为100%(15 a中龄林)、88.5%(24 a近熟林)、92.4%(45 a成熟林)、77.6%(55 a过熟林Ⅰ)和80.5%(63 a过熟林Ⅱ)。其中,NT-α亚类占比分别为85.2%、46.0%、3.98%、12.5%和61.9%;而NS-γ-2.3亚类占比分别为14.8%、42.5%、88.4%、66.4%和18.6%(图 2a)。此外,NS-ζ丰度在中龄林土壤(15 a)中低于检测限,但随着种植年限增加而增加,其相对丰度在过熟林Ⅰ(55 a)土壤最高为21.1%。
ATPase subunit A基因分析结果也发现NT-α和NS-γ-2.3亚类是土壤AOA优势类群(图 2e),在不同种植年限的5种土壤中,其总丰度分别为87.8%(15 a中龄林)、94.1%(24 a近熟林)、91.1%(45 a成熟林)、86.0%(55 a过熟林Ⅰ)和86.4%(63 a过熟林Ⅱ)。其中,NT-α和NS-γ-2.3的占比分别为:15 a中龄林(57.0%和30.7%)、24 a近熟林(61.2%和32.9%)、45 a成熟林(5.70%和85.4%)、55 a过熟林Ⅰ(13.6%和72.4%)和63 a过熟林Ⅱ(63.6%和22.8%)(图 2e)。此外,NS-ζ丰度随着种植年限增加而增加,其相对丰度由15 a中龄林土壤1.8%增加至55 a过熟林Ⅱ土壤的12.8%。
总体而言,系统发育进化发现酸性土壤中占据绝对优势AOA的ATPase subunit A序列具有高度同源性,这些NT-α和NS-γ-2.3亚类与已知的嗜酸/耐酸AOA相似并可归类为V-ATPase。同时,这些AOA的amoA基因同源性较低,表明AOA古菌可能通过基因水平转移获得V-ATPase基因适应酸性胁迫。
2.4 土壤氨氧化古菌分异的环境驱动机制在微生物分类学不同水平,如纲(Class)、目(Order)、亚支(Subclade),将不同林龄马尾松人工林土壤AOA分类,计算其相对丰度的变异系数并与土壤理化性质进行相关分析。结果表明,AOA amoA基因和ATPase subunit A基因分类越细,其丰度的变异系数越大,即重复间变异越大(图 3a、图 3b)。同时,土壤理化因子的变异系数低于AOA类群的变异系数。Spearman统计分析发现,AOA类群丰度与其变异系数极显著负相关(r =–0.679,P < 0.001)。例如,在亚支分类水平,当AOA古菌amoA基因和ATPase subunit A基因丰度低于40个序列reads,其变异系数高达34.4%~86.7%,而当AOA丰度高于40个reads,其变异系数集中在9.07%~33.9%之间(图 3c)。
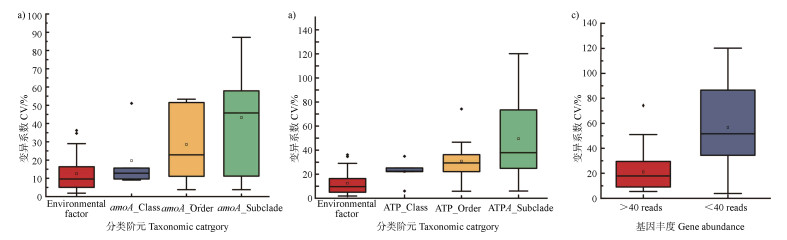
|
图 3 不同分类水平下AOA amoA基因(a)和ATPase subunit A(b)丰度变异系数及其与丰度关系(c) Fig. 3 Coefficient of variation of abundance of AOA amoA gene(a)and ATPase subunit A(b)at different classification levels and their relationship with abundance(c) |
Spearman分析不同AOA基因型与土壤理化性质发现,不同环境要素对AOA基因型的影响不同。pH、速效钾与NT-α亚支极显著正相关,但与NS-γ-2.3和NS-ζ亚支显著或极显著负相关(表 2)。利用总amoA基因型研究AOA群落多样性,通过ANOISM分析,发现不同林龄马尾松人工林土壤AOA群落存在显著差异(r=0.83,P < 0.001),进一步Mantel分析发现,速效钾是影响AOA群落结构的最重要环境因子(r=0.56,P < 0.001)。
|
|
表 2 不同马尾松种植年限下氨氧化古菌AOA类群与土壤理化性质的相关性 Table 2 Relationship of AOA groups with soil physicochemical properties |
已有研究表明,酸性土壤中存在大量尚未可知的嗜酸/耐酸氨氧化古菌,基于经典的16S rRNA基因和氨单加氧酶amoA基因系统发育进化分析均表明,这些AOA与已知的嗜酸性AOA古菌亲缘关系较远,与中碱性环境中的古菌AOA亲缘关系较近[29-31],AOA古菌在酸性环境中的生境分布规律与致。我们通过稳定同位素核酸探针技术,首次系统16S rRNA和amoA基因的系统发育进化关系并不一阐述了这一现象,并提供了这些未知AOA在酸性环境中分裂增殖的明确证据[18]。同时,几乎所有微生物细胞内部均为中性稳态环境,为了在酸性条件下生长,AOA必须克服细胞膜内外巨大的pH梯度,消耗更多能量适应酸性胁迫。我们前期的研究发现能量货币ATP合成相关的基因极可能发生水平转移,是酸性土壤中未知AOA及已知嗜酸性AOA适应酸性胁迫环境的重要机制[25],但其普适性尚需进一步证据。本研究则针对不同种植年限的马尾松人工林酸性土壤,发现V-ATPase基因水平转移可能是古菌AOA适应酸性胁迫环境的重要机制。
3.1 土壤氨氧化古菌适应酸性胁迫机制分析通过对马尾松人工林土壤总DNA深度宏基因组测序结果分析,表明V-ATPase基因协助AOA适应酸性胁迫机制可能具有普适性。目前AOA耐酸机制研究主要集中在NT类群,推测其可能通过一些典型机制适应酸性环境[12]。但是,已知嗜酸NT类群四株AOA古菌研究表明,其中有两种菌株不含有与钾离子代谢相关的基因,说明通过吸收钾离子抑制质子渗透的耐酸机制并不适用于所有嗜酸NT类群[32]。此外,酸性土壤中存在丰度更高的与NT类群具有较远亲缘关系的NS-γ-2.3类群,目前尚未有纯菌株报道,其耐酸机制尚不清楚。因此,不同AOA类群适应酸性胁迫的机制是否具有共性,仍然需要进一步研究。本研究通过深度宏基因组测序,对AOA amoA基因和ATPase subunit A基因分析发现,在马尾松人工林酸性土壤中,各酸性AOA类群的V-ATPase比例与其对应的amoA基因比例基本相同,而不同林龄土壤中两者的变化规律也基本相同。这一结果从生境适应性角度,提供了V-ATPase基因水平转移的重要证据,表明V-ATPase基因是NT类群和NS-γ-2.3类群适应酸性胁迫环境的重要机制。
3.2 马尾松人工林种植年限对土壤氨氧化古菌群落分化的影响规律人工林种植可能促进了酸性AOA的种群分化。通过深度宏基因组测序发现,马尾松人工林酸性土壤中AOA主要是NT和NS-γ-2.3类群,并且随林龄增加,NT类群丰度先减少后增加,而NS-γ-2.3类群丰度先增加后减少,这可能是不同林龄土壤理化性质差异导致的。有研究表明pH是影响AOA群落的重要因子[33-34],但是本研究通过Mantel分析发现,速效钾与AOA群落结构相关性最强。这可能是由于本研究样品皆为酸性土壤,pH在小范围变化内对AOA影响有限,而钾离子与AOA适应酸性胁迫的代谢活动密切相关,因此,速效钾显著影响酸性马尾松人工林土壤AOA群落结构。进一步通过Spearman相关分析发现,NT类群与环境因子显著正相关,而NS-γ-2.3类群则与环境因子显著负相关,导致这种差异的原因以及环境因子对AOA群落的生态分化影响仍需进一步研究。本研究同时发现,不同林龄马尾松人工林土壤AOA群落结构组间差异显著大于组内差异,当同一林龄土壤样品重复间的环境因子变异程度较小时,不同林龄马尾松人工林土壤中AOA类群丰度随着分类越细,重复间变异程度越大,且通过Spearman分析发现,AOA丰度与变异系数极显著负相关(r = –0.679,P < 0.001)。由此可以推测,当采集样品的真重复间环境因子相差较小且在较大级别进行微生物分类时,生物学重复的单个样品深度宏基因组测序结果也能反映该样品中古菌的整体情况。
3.3 宏基因组测序氨氧化古菌注释策略的差异分析针对马尾松人工林酸性土壤总DNA深度宏基因组测序,研究发现通过不同注释策略对amoA基因和ATPase subunit A基因的注释结果具有显著差异。Illumina测序技术是目前宏基因组研究中最为广泛应用的测序方法,而在对测序结果进行注释时,一般是通过reads[35-37]或contigs[38-40]与数据库比对进行物种分类。本研究通过对马尾松人工林土壤总DNA使用Illumina Hi Seq 2000测序平台进行双末端测序,对总共约750G bp原始数据质控、组装获得高质量序列后,使用不同方法对AOA注释。针对amoA基因注释,无论直接将reads或contigs与数据库比对,还是通过将这些序列构建系统发育确定其进化分类地位,其结果基本一致。然而,ATPase subunit A基因注释则必须通过构建系统发育进化树。造成这种差异的可能原因是:氨氧化古菌amoA基因和ATPase subunit A基因注释差异可能取决于数据库中不同基因的丰富度。氨单加氧酶amoA基因一直是氨氧化古菌分子生态学研究的分子标靶,数据库中数量高达33 256个amoA基因序列,聚类分析则可得到1 190个amoA基因代表序列[28]。本研究以这些代表序列为基础构建amoA基因数据库,最大程度涵括所有AOA类群信息。相反,由于古菌AOA的ATPase研究较少,本文构建的ATPase subunit A数据库只含有215个序列,仅57个序列属于AOA,其中25个序列与陆地AOA相关,同时,ATPase基因作为系统发育分类的意义有限。因此,本研究缺少足够的AOA古菌ATPase subunit A序列信息,导致不同ATPase subunit A注释方法结果差异较大。
此外,大片度基因拼接也可能产生误差,尤其是AOA的ATPase subunit A序列更长,拼接难度更大。尽管高通量测序导致微生物学、生态学、进化生物学等学科交叉发展态势迅猛[41],但短读取长度始终是二代测序最明显的缺点[42]。借助各种生物信息学工具可将短序列组装至原始长度模板中,也可能对基于序列的生物学研究准确性产生重要影响。本研究中拼接组装得到的AOA古菌V-ATPase subunit A的大片段contigs均长为528 bp,与amoA contigs均长511 bp几乎相同,而AOA的ATPase subunit A序列更长,约是amoA基因的2.8倍。由于酸性土壤的中性AOA数量较少,导致拼接出的AOA古菌A-ATPase subunit A contigs均长更短(310 bp),因此,即便通过构建系统发育进化关系,也很难准确对古菌AOA进行分类(图 1b)。因此,尽管高通量测序技术为研究环境微生物和微生物生态学研究提供了强有力的工具,并已出现了大量的算法用于海量的宏基因组数据分析,然而,目前尚没有一种计算软件可解决所有问题,针对各种生境中丰度各异、类群多样的微生物区系仍需针对具体问题构建新的算法。
4 结论基于16S rRNA基因和amoA基因的氨氧化古菌系统发育分类地位基本一致,是氨氧化古菌多样性研究的经典标靶基因。针对5个不同种植年限的马尾松人工林酸性土壤,通过深度宏基因组测序并重构古菌AOA的氨单加氧酶amoA基因系统发育,发现所有酸性森林土壤中主要包括Ca. Nitrosotaleales和Nitrososphaerales古菌,前者是已知的嗜酸氨氧化菌,而后者则与已知嗜酸的Ca. Nitrosotaleales亲缘关系较远,却与中碱性土壤中的AOA亲缘进化更近,表明amoA基因系统发育进化无法解释Nitrososphaerales在酸性森林土壤中的定殖。然而,所有这些类群均含有亲缘关系高度相近的V-ATPase subunit A基因,表明氨氧化古菌可能通过基因水平转移获得V-ATPase基因适应酸性胁迫。同时,不同种植年限下酸性人工林土壤中氨氧化古菌种群发生了明显的分化,速效钾是显著影响AOA群落结构的重要环境因子。
| [1] |
Von Uexküll H R, Mutert E. Global extent, development and economic impact of acid soils[J]. Plant and Soil, 1995, 171(1): 1-15. DOI:10.1007/BF00009558
(  0) 0) |
| [2] |
Slonczewski J L, Fujisawa M, Dopson M, et al. Cytoplasmic pH measurement and homeostasis in bacteria and Archaea[J]. Advances in Microbial Physiology, 2009, 55: 1-79.
(  0) 0) |
| [3] |
Yu C Q, Huang X, Chen H, et al. Managing nitrogen to restore water quality in China[J]. Nature, 2019, 567(7749): 516-520. DOI:10.1038/s41586-019-1001-1
(  0) 0) |
| [4] |
Guo J H, Liu X J, Zhang Y, et al. Significant acidification in major Chinese croplands[J]. Science, 2010, 327(5968): 1008-1010. DOI:10.1126/science.1182570
(  0) 0) |
| [5] |
Galloway J N, Townsend A R, Erisman J W, et al. Transformation of the nitrogen cycle: Recent trends, questions, and potential solutions[J]. Science, 2008, 320(5878): 889-892. DOI:10.1126/science.1136674
(  0) 0) |
| [6] |
Könneke M, Bernhard A E, de la Torre J R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437(7058): 543-546. DOI:10.1038/nature03911
(  0) 0) |
| [7] |
Zhang L M, Hu H W, Shen J P, et al. Ammonia-oxidizing archaea have more important role than ammoniaoxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. The ISME Journal, 2012, 6(5): 1032-1045. DOI:10.1038/ismej.2011.168
(  0) 0) |
| [8] |
Lehtovirta-Morley L E, Stoecker K, Vilcinskas A, et al. Cultivation of an obligate acidophilic ammonia oxidizer from a nitrifying acid soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(38): 15892-15897. DOI:10.1073/pnas.1107196108
(  0) 0) |
| [9] |
Nicol G W, Leininger S, Schleper C, et al. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria[J]. Environmental Microbiology, 2008, 10(11): 2966-2978. DOI:10.1111/j.1462-2920.2008.01701.x
(  0) 0) |
| [10] |
Lu L, Han W Y, Zhang J B, et al. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea[J]. The ISME Journal, 2012, 6(10): 1978-1984. DOI:10.1038/ismej.2012.45
(  0) 0) |
| [11] |
Lu L, Jia Z J. Urease gene-containing Archaea dominate autotrophic ammonia oxidation in two acid soils[J]. Environmental Microbiology, 2013, 15(6): 1795-1809. DOI:10.1111/1462-2920.12071
(  0) 0) |
| [12] |
Lehtovirta-Morley L E, Sayavedra-Soto L A, Gallois N, et al. Identifying potential mechanisms enabling acidophily in the ammonia-oxidizing archaeon "Candidatus Nitrosotalea devanaterra"[J]. Applied and Environmental Microbiology, 2016, 82(9): 2608-2619. DOI:10.1128/AEM.04031-15
(  0) 0) |
| [13] |
Konings W N, Albers S V, Koning S, et al. The cell membrane plays a crucial role in survival of bacteria and Archaea in extreme environments[J]. Antonie Van Leeuwenhoek, 2002, 81(1/2/3/4): 61-72.
(  0) 0) |
| [14] |
Baker-Austin C, Dopson M. Life in acid: pH homeostasis in acidophiles[J]. Trends in Microbiology, 2007, 15(4): 165-171. DOI:10.1016/j.tim.2007.02.005
(  0) 0) |
| [15] |
Castanie-Cornet M P, Penfound T A, Smith D, et al. Control of acid resistance in Escherichia coli[J]. Journal of Bacteriology, 1999, 181(11): 3525-3535. DOI:10.1128/JB.181.11.3525-3535.1999
(  0) 0) |
| [16] |
Lehtovirta-Morley L E, Ge C R, Ross J, et al. Characterisation of terrestrial acidophilic archaeal ammonia oxidisers and their inhibition and stimulation by organic compounds[J]. FEMS Microbiology Ecology, 2014, 89(3): 542-552. DOI:10.1111/1574-6941.12353
(  0) 0) |
| [17] |
Johnson D B, Schippers A. Editorial: Recent advances in acidophile microbiology: Fundamentals and applications[J]. Frontiers in Microbiology, 2017, 8: 428.
(  0) 0) |
| [18] |
Wang B Z, Zheng Y, Huang R, et al. Active ammonia oxidizers in an acidic soil are phylogenetically closely related to neutrophilic archaeon[J]. Applied and Environmental Microbiology, 2014, 80(5): 1684-1691. DOI:10.1128/AEM.03633-13
(  0) 0) |
| [19] |
Stahl D A, de la Torre J R. Physiology and diversity of ammonia-oxidizing archaea[J]. Annual Review of Microbiology, 2012, 66: 83-101. DOI:10.1146/annurev-micro-092611-150128
(  0) 0) |
| [20] |
Zubareva V M, Lapashina A S, Shugaeva T E, et al. Rotary ion-translocating ATPases/ATP synthases: Diversity, similarities, and differences[J]. Biochemistry: Moscow, 2020, 85(12/13): 1613-1630.
(  0) 0) |
| [21] |
Forgac M. Vacuolar ATPases: Rotary proton pumps in physiology and pathophysiology[J]. Nature Reviews Molecular Cell Biology, 2007, 8(11): 917-929. DOI:10.1038/nrm2272
(  0) 0) |
| [22] |
Wagner C A, Finberg K E, Breton S, et al. Renal vacuolar H+-ATPase[J]. Physiological Reviews, 2004, 84(4): 1263-1314. DOI:10.1152/physrev.00045.2003
(  0) 0) |
| [23] |
Beyenbach K W, Wieczorek H. The V-type H+ ATPase: Molecular structure and function, physiological roles and regulation[J]. Journal of Experimental Biology, 2006, 209(4): 577-589. DOI:10.1242/jeb.02014
(  0) 0) |
| [24] |
Nanda A, Brumell J H, Nordström T, et al. Activation of proton pumping in human neutrophils occurs by exocytosis of vesicles bearing vacuolar-type H+-ATPases[J]. Journal of Biological Chemistry, 1996, 271(27): 15963-15970. DOI:10.1074/jbc.271.27.15963
(  0) 0) |
| [25] |
Wang B Z, Qin W, Ren Y, et al. Expansion of Thaumarchaeota habitat range is correlated with horizontal transfer of ATPase operons[J]. The ISME Journal, 2019, 13(12): 3067-3079. DOI:10.1038/s41396-019-0493-x
(  0) 0) |
| [26] |
Bao S D. Soil and agricultural chemistry analysis (In Chinese). Beijing: China Agriculture Press, 2000. [土壤农化分析[M]. 3版. 北京: 中国农业出版社, 2000.]
(  0) 0) |
| [27] |
Tu Q C, Lin L, Cheng L, et al. NCycDB: a curated integrative database for fast and accurate metagenomic profiling of nitrogen cycling genes[J]. Bioinformatics, 2019, 35(6): 1040-1048. DOI:10.1093/bioinformatics/bty741
(  0) 0) |
| [28] |
Alves R J E, Minh B Q, Urich T, et al. Unifying the global phylogeny and environmental distribution of ammonia-oxidising archaea based on amoA genes[J]. Nature Communications, 2018, 9: 1517. DOI:10.1038/s41467-018-03861-1
(  0) 0) |
| [29] |
He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9): 2364-2374. DOI:10.1111/j.1462-2920.2007.01358.x
(  0) 0) |
| [30] |
Gubry-Rangin C, Hai B, Quince C, et al. Niche specialization of terrestrial archaeal ammonia oxidizers[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(52): 21206-21211. DOI:10.1073/pnas.1109000108
(  0) 0) |
| [31] |
Huang R, Wu Y C, Zhang J B, et al. Nitrification activity and putative ammonia-oxidizing archaea in acidic red soils[J]. Journal of Soils and Sediments, 2012, 12(3): 420-428. DOI:10.1007/s11368-011-0450-4
(  0) 0) |
| [32] |
Herbold C W, Lehtovirta-Morley L E, Jung M Y, et al. Ammonia-oxidising archaea living at low pH: Insights from comparative genomics[J]. Environmental Microbiology, 2017, 19(12): 4939-4952. DOI:10.1111/1462-2920.13971
(  0) 0) |
| [33] |
Xi R J, Long X E, Huang S, et al. pH rather than nitrification and urease inhibitors determines the community of ammonia oxidizers in a vegetable soil[J]. AMB Express, 2017, 7(1): 129. DOI:10.1186/s13568-017-0426-x
(  0) 0) |
| [34] |
Gubry-Rangin C, Kratsch C, Williams T A, et al. Coupling of diversification and pH adaptation during the evolution of terrestrial Thaumarchaeota[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(30): 9370-9375. DOI:10.1073/pnas.1419329112
(  0) 0) |
| [35] |
Petersen T N, Lukjancenko O, Thomsen M C F, et al. MGmapper: Reference based mapping and taxonomy annotation of metagenomics sequence reads[J]. PLoS One, 2017, 12(5): e0176469. DOI:10.1371/journal.pone.0176469
(  0) 0) |
| [36] |
Chen J R, McIlroy S E, Archana A, et al. A pollution gradient contributes to the taxonomic, functional, and resistome diversity of microbial communities in marine sediments[J]. Microbiome, 2019, 7(1): 104. DOI:10.1186/s40168-019-0714-6
(  0) 0) |
| [37] |
Müller A, Hundt C, Hildebrandt A, et al. MetaCache: context-aware classification of metagenomic reads using minhashing[J]. Bioinformatics, 2017, 33(23): 3740-3748. DOI:10.1093/bioinformatics/btx520
(  0) 0) |
| [38] |
Yu C D, Hou L J, Zheng Y L, et al. Evidence for complete nitrification in enrichment culture of tidal sediments and diversity analysis of clade a comammox Nitrospira in natural environments[J]. Applied Microbiology and Biotechnology, 2018, 102(21): 9363-9377. DOI:10.1007/s00253-018-9274-0
(  0) 0) |
| [39] |
Levy Karin E, Mirdita M, Söding J. MetaEuk-sensitive, high-throughput gene discovery, and annotation for large-scale eukaryotic metagenomics[J]. Microbiome, 2020, 8(1): 48. DOI:10.1186/s40168-020-00808-x
(  0) 0) |
| [40] |
Liao S L, Wang Y Y, Liu H, et al. Deciphering the microbial taxonomy and functionality of two diverse mangrove ecosystems and their potential abilities to produce bioactive compounds[J]. mSystems, 2020, 5(5): e00851-19.
(  0) 0) |
| [41] |
Metzker M L. Sequencing technologies-The next generation[J]. Nature Reviews Genetics, 2010, 11(1): 31-46. DOI:10.1038/nrg2626
(  0) 0) |
| [42] |
Midha M K, Wu M C, Chiu K P. Long-read sequencing in deciphering human genetics to a greater depth[J]. Human Genetics, 2019, 138(11/12): 1201-1215.
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


