2. 中国科学院大学, 北京 100049;
3. 江苏省农业科学院农业资源与环境研究所, 南京 210014;
4. 江苏大学环境与安全工程学院, 江苏镇江 212013
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Agricultural Resources and Environment, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China;
4. School of Environmental and Safety Engineering, Jiangsu University, Zhenjiang, Jiangsu 212013, China
自然环境中微生物具有极其丰富的多样性且仅有部分物种主导关键生态功能,因此,直接甄别这些执行特定生态功能的活性物种组成具有较大难度。目前,DNA稳定性同位素核酸探针(DNA-based stable isotope probing,DNA-SIP)技术在生态学和环境科学领域中被广泛应用于揭示活性功能微生物。但其也存在方法学上的不足,例如依赖于同位素标记底物的专一性[1],即当底物具有代谢广谱性时,物种间的互营养使得诸多非靶标物种也会被标记上,进而会影响结论的可靠性。此外,DNA-SIP技术无法示踪底物异化型微生物[2],且微生物基因组鸟嘌呤和胞嘧啶所占的比率过高也会影响实验结果和结论的准确性[3]。上述技术上的缺陷需要研究人员利用其他技术方法进一步研究,以更全面地了解自然环境中的活性功能微生物组成及其生态生理功能。
磁性纳米粒子介导分离技术(magnetic nanoparticle- mediated isolation,MMI)是从复杂微生物群落中鉴定、分离具有特定代谢功能活性物种的有力工具。其原理为,用生物相容性高的磁性纳米粒子(magnetic nanoparticles,MNPs)均匀包裹复杂环境中所有微生物的细胞表面,使其均具有磁性; 在底物定向培养过程中,仅有具有代谢活性的微生物会随着细胞不断分裂增殖而逐渐降低其表面MNPs,直至失去磁性。最终借助外加磁场将无磁性(或低磁性)的活性功能物种与有磁性无代谢活性的物种分离[2]。MMI无需同位素标记目标底物,可弥补DNA-SIP的诸多不足,此外,该技术相较操作复杂的DNA-SIP技术以及传统菌种筛选方法也更为简便快捷[2,4]。更重要的是,通过MMI方法分离所获得的功能微生物仍具活性,为进一步研究其生理特征及生态功能提供了技术上的便利。MMI技术已初步应用于环境微生物研究中。例如,2015年,Zhang等[2]应用MMI技术成功地从污染场地中分离和表征有活性且尚未培养的功能微生物; 2016年,Wang等[5]结合MMI和BIOLOG表型芯片(BIOLOG phenotype microarray plates)技术成功从原油污染土壤中筛选出了主导烷烃代谢的功能微生物,并鉴定了其碳源利用能力。
秸秆是农业生产系统中的一项重要生物资源。秸秆还田有助于提高土壤肥力、减少化肥使用以及促进农业废弃物资源化,是经济可行和环境友好的综合利用方式之一[6]。秸秆主要成分为纤维素(38%~41%)、半纤维素(26%~35%)、木质素(15%)和水溶性多糖(8%)[7]。纤维素是秸秆组成中最重要且占比最高的一类,以其为主要成分的胞壁包裹住了秸秆的大部分营养成分,因此,纤维素降解直接影响秸秆养分的再利用[8]。土壤微生物是秸秆降解的主要驱动者,前者通过分泌纤维素酶等方式促进后者降解。而农业措施会通过改变土壤微生物群落结构、功能而影响土壤秸秆的降解。因此,揭示土壤中活性纤维素降解菌群落及其对不同施肥处理的响应,可为农田秸秆的资源化管理提供理论依据。
基于以上认知,本研究希望通过应用MMI技术解析三种不同施肥处理间水稻土活性纤维素降解菌群落(组成和分子生态网络)的变化,以期明确施肥对其群落的影响。基于此,选取鹰潭红壤长期定位试验站三种不同施肥处理的土壤为供试对象,选取纤维素降解菌常用底物羧甲基纤维素钠(carboxymethyl cellulose sodium,CMC-Na)为碳源[9-10],开展了微域培养试验以及后期的分子生态研究。
1 材料与方法 1.1 供试土壤样品本实验所用土壤采自鹰潭红壤长期定位试验站(28°15′N,116°55′E)。经过28年不同施肥处理(有机肥OM、化肥NPK和不施肥CK),具体土壤化学性质详见表 1。
|
|
表 1 三种不同施肥土壤的化学特性 Table 1 The soil chemical properties under three fertilization strategies |
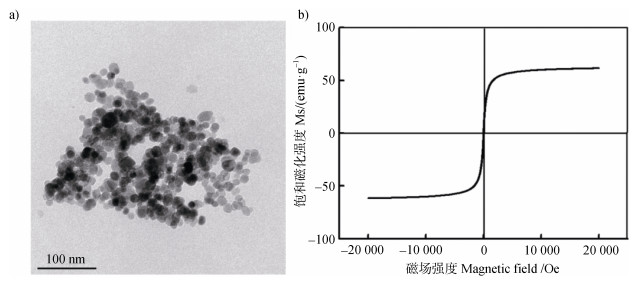
氧化铁磁性纳米粒子制备方法采用共沉淀法[11]。具体信息如下,量取300 mL超纯水倒入500 mL三口烧瓶中,在室温下边搅拌边通氮气30 min,排尽烧瓶里的空气。称量4.490 g FeCl3·6H2O和2.307 g FeSO4·7H2O于上述三口烧瓶内,机械搅拌至完全溶解、充分混匀,溶液呈橙红色。用恒流泵将30 mL浓氨水注入上述混合溶液中,立即有大量黑色沉淀生成,在60℃水浴锅继续搅拌反应2 h,整个实验过程在氮气保护下进行。边搅拌边室温冷却,用磁铁将其吸附分离。用超纯水洗涤五次,无水乙醇洗涤三次,在60℃真空干燥箱干燥20 h,即得到干燥的块状纳米Fe3O4。用研钵研磨后得粉末状纳米Fe3O4,将其加至无菌水中用40 kHz超声振荡10 min,备用。制备好的纳米Fe3O4颗粒分别用透射电镜(JEM-200EX,JEOL,日本)和振动样品磁强计(PPMS-9T,Quantum Design,美国)进行表征。透射电镜观察可见,制备的磁性Fe3O4纳米粒子为球形,粒径为10~20 nm(图 1a)。纳米材料的磁滞回线如图 1b所示,其饱和磁场强度为61.94 emu·g–1,具有超顺磁性。
|
图 1 磁性纳米粒子(MNPs)的透射电镜图(a)和磁滞回线图(b) Fig. 1 Transmission electron microscope image(a)and magnetization curve(b)of the produced magnetic nanoparticles |
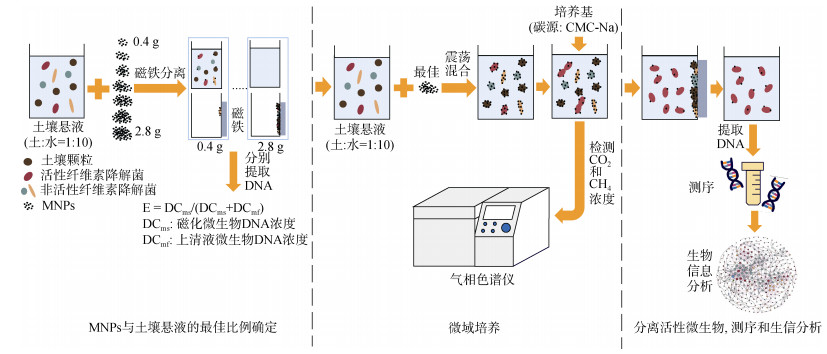
本研究MMI流程详见图 2,主要分为3个环节:(1)确定MNPs与土壤悬液的最佳比例; (2)CMC-Na对磁性纳米材料包裹后的微生物进行微域培养; (3)活性微生物的分离、测序和生物信息分析。
|
注:MNPs:磁性纳米粒子; CMC-Na:羧甲基纤维素钠; E:土壤磁化效率。下同。 Note: MNPs: magnetic nanoparticles; CMC-Na: carboxymethyl cellulose sodium; E: soil magnetic functionalization efficiency. The same below. 图 2 磁性纳米粒子介导分离技术(MMI)流程 Fig. 2 Magnetic nanoparticle-mediated isolation(MMI)process |
具体而言,首先制备三种施肥处理的土壤悬液(水土比为10:1),每种处理三个重复,各取10 mL至7个离心管中,1 000 r·min–1离心2 min去除大颗粒土壤及杂质。为了测试土壤悬液磁化率并优化以进行有效分离,分别添加0.4、0.8、1.2、1.6、2.0、2.4和2.8 g MNPs至土壤悬液混合震荡(150 r·min–1)20 min。用磁铁吸附磁化微生物10 min,分别收集磁化微生物与去除磁化微生物的剩余部分,10 000 r·min–1离心2 min获得剩余部分的菌体沉淀,使用细菌基因组DNA提取试剂盒(TIANGEN,北京),根据其说明书提取DNA。提取的基因组DNA溶于50 μL TE缓冲液(Tris-EDTA buffer solution)中,使用Qubit(Thermo Scientific,美国)荧光法测定有磁性细菌的DNA浓度(DCms)和剩余部分中无磁性细菌的DNA浓度(DCmf),计算土壤磁化效率(E = DCms/(DCms+DCmf))。在土壤磁化效率值初始到达100%即拐点时,说明土壤悬液与MNPs的比例最佳,土壤悬液中所有菌体刚好被MNPs包裹; 当土壤磁化效率高于100%时,MNPs含量过高,包裹所有菌体后仍有剩余,不利于后期新生目标菌体的分离鉴定; 反之,当土壤磁化效率低于100%时,MNPs含量过低,不足以包裹全部菌体,则会丢失部分目标微生物[5]。
1.4 实验室微域培养方法配制以CMC-Na为唯一碳源的培养基:CMC-Na 10.0 g,Na2HPO4 3.0 g,KH2PO4 3.0 g,NH4Cl 1.0 g,MgSO4·7H2O 0.5 g [12],调节pH至7.2,121℃灭菌20 min备用。按前期实验所得最佳比例混合MNPs与三种施肥处理的土壤悬液并加至100 mL血清瓶,于室温下振荡(150 r·min–1)20 min,每处理六个重复,共18个样本; 用磁铁吸附磁化微生物10 min,去除剩余部分后将分离出的磁化细菌重悬于等体积无菌水中,重复洗涤三遍除尽未被MNPs吸附的微生物,将其重悬于等体积CMC-Na培养基中,置于28℃培养箱中。分别于培养的第3、6、9和12天用气密注射器从血清瓶的顶空收集气体样品,通过气相色谱仪(Agilent 7890A,Agilent Technologies,美国)测定CO2和CH4的浓度,获得微生物代谢情况。12 d后结束培养,在超净台中用磁铁放置于血清瓶左侧,定向吸附有磁性材料包裹的无代谢活性微生物20 min。随后用无菌移液管将剩余部分转移至离心管内,10 000 r·min–1离心2 min获得剩余部分菌体沉淀后提取DNA(详见1.3)。
1.5 16S rRNA基因PCR扩增及高通量扩增子测序使用引物519F(3'-CAGCMGCCGCGGTAA TWC-5')和907R(3'-CCGTCAATTCMTTTRAGTTT- 5')扩增细菌16S rRNA基因的V4~V5可变区。为区分不同样品,于519F引物3'端加一段5 bp长度的已知碱基序列的分类标签(Barcode)。PCR扩增体系为50 μL:1 μL模板DNA,2 μL(15 μmol·L–1)正、反向引物,2 μmol·L–1 TaqDNA聚合酶(TaKaRa,日本),1.25 μmol·L–1脱氧核糖核苷三磷酸,阴性对照以无菌超纯水替代模板DNA。PCR扩增程序为:94℃ 5 min,35个循环(95℃ 30 s,56℃ 30 s和72℃ 45 s),72℃延伸10 min。PCR产物均通过1%琼脂糖凝胶条带检测特异性。使用QIAquick PCR Purification kit(QIAGEN)纯化试剂盒纯化PCR产物,用Qubit(Thermo Scientific,美国)测定PCR产物浓度,将所有PCR产物等摩尔混合后,使用Illumina MiSeq测序平台进行扩增子高通量测序。
基因序列处理使用微生物生态定量指标(Quantitative Insights Into Microbial Ecology,QIIME)平台完成。首先,去除平均质量分数低于20且长度小于200 bp的序列,并根据Barcode序列与样品进行匹配。使用Cutadapt和Trimmomatic分别去除扩增引物和接头序列。使用Usearch算法按照97%的相似性对序列进行聚类,同时进行嵌合体去除,每个可操作分类单元(operational taxonomic unit,OTU)中丰度最高的序列被选作代表序列。按照核糖体数据库项目(Ribosomal Database Project,RDP)分类方法使用Greengene(version 13.8)进行物种注释。使用PyNast算法比对代表序列,并使用FastTree构建系统发育树。
测序共得到1 245 632条高质量16S rRNA基因序列。为了进行样品间多样性、物种组成和分子生态网络的比较,将所有样品的序列统一抽平至19 233条。
1.6 数据统计分析计算物种丰富度指数(Richness index,in term of observed OTUs)、辛普森优势度指数(Simpson dominance index)和物种均一度指数(species evenness index)来表征物种在上述三个方面的alpha多样性。基于Bray-Curtis距离(OTUs水平)的非度量多维尺度分析(nonmetric multidimensional scaling,NMDS)用以展示群落结构差异,置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA)检验群落结构差异的显著性。以上分析均使用R软件“vegan”包。方差分析(ANOVA)用以多样性指数的组间差异分析,使用SPSS16.0计算。使用MENA在线平台(http://ieg4.rccc.ou.edu/mena)进行共发生网络分析。本文所有统计分析,处理间数据差异采用SPSS 16.0进行统计分析,并使用图基(Tukey)检验进行多重比较,显著和极显著水平分别为P < 0.05和P < 0.01。
2 结果与讨论 2.1 MNPs与土壤悬液的最佳比例为保证初始时不同处理下的每个微生物均被充分包裹且具备磁性,即磁化效率初始达到100%,对于相同量的土壤悬液,NPK与CK处理悬液中的菌体需要1.6 g MNPs,而OM处理悬液则需要2 g MNPs(图 2a)。如果统一为NPK或CK的最佳磁性纳米颗粒添加量,OM中部分物种可能未被磁性纳米材料包裹,而未参与后期的培养; 如果统一为OM的最佳磁性纳米颗粒添加量,NPK和CK中活性物种新分裂出的细胞又会被过量的MNPs包裹而无法通过后期的磁性吸附分离被检测出。图 2b中显示,按上述比例混合土壤悬液与MNPs,再用磁铁吸附磁性材料包裹的微生物后,剩余部分均澄清透明。颜色越清澈表明磁化效果越好,佐证了MNPs与土壤悬液的最佳比例。
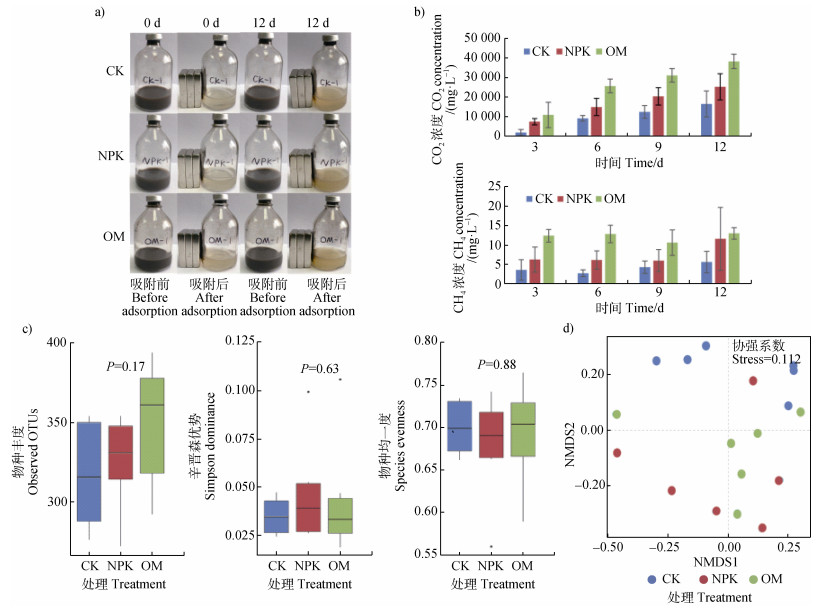
2.2 微域培养及不同施肥处理活性纤维素降解细菌的群落结构用磁铁立即吸附与CMC-Na液体培养基混合的磁性材料包裹的微生物后,培养液澄清透明(图 4a),且DCmf很低,仅在0.11~3.74 ng·μL–1之间。28℃培养12 d后,因微生物大量繁殖,培养液变得浑浊(图 4a)。此时,DCmf上升至81.29±14.96 ng·μL–1(CK)、84.60±36.64 ng·μL–1(NPK)和115.5± 29.35 ng·μL–1(OM)。12 d内血清瓶中的CO2与CH4浓度的变化也反映了培养期间的微生物增殖情况,二者均呈现由CK、NPK至OM增加的趋势(P < 0.05)(图 4b)。测气与确定最佳比例的结果共同说明同质量的土壤中,微生物数量和活性(增殖快)均呈现由OM、NPK、到CK逐渐降低的趋势,即长期施肥增加了土壤中微生物数量和活性。造成该结果的原因可能是长期施肥增加了土壤中的养分含量(表 1),且通过改善土壤理化性质使环境条件更适于微生物生存(如提高总孔隙度和土壤团聚体的稳定性,改善土壤水、气的连通性等),从而增加微生物数量与活性[13-15]。夏文建等[16]也在试验中得出了相同结论。

|
注:MMNPs为磁性纳米粒子质量。图b上方的质量代表MNPs的添加量。 Note: MMNPs is the mass of magnetic nanoparticles. The text above panel b)represents the amount of MNPs. 图 3 三种施肥处理下土壤悬液的磁化效率与悬液中MNPs含量的关系(a)及加入MNPs并去除磁化部分后的剩余土壤悬液(b) Fig. 3 Relationships between the magnetic efficiency of three fertilization strategies soil solution and the MNPs concentration(a)and the remaining of corresponding soil solution after removing magnetic part(b) |
|
注:(a)微域培养图片; (b)培养12天内的CO2和CH4浓度; (c)三种施肥处理下活性纤维素降解细菌群落的alpha多样性指数差异; (d)非度量多维尺度分析(NMDS)。图(b)中不同字母显示统计学的差异显著性。 Note: (a)Microcosm picture; (b)CO2 and CH4 concentrations for 12-day cumulative; (c)Changes in alpha diversity indexes and(d)the nonmetric multidimensional scaling of metabolically active cellulose-degrading bacterial communities under three fertilization strategies. The different letters in Figure(b)represent a significant difference. 图 4 微域培养及活性纤维素降解细菌群落多样性分析 Fig. 4 Microcosm and diversity indexes of metabolically active cellulose-degrading bacterial communities degrading bacterial communities |
微域培养后可获得不同施肥处理下的活性纤维素降解细菌群落信息,分别计算其物种丰富度指数、辛普森优势度指数和物种均一度指数值来表征物种丰富度、优势度和均一度方面的alpha多样性。结果表明,alpha多样性的三种水平均无显著性差异(P > 0.05)(图 4c)。
基于Bray-Curtis距离的NMDS(图 4d)和PERMANOVA(表 2)表明,不施肥处理CK的活性纤维素降解细菌群落结构与施肥处理OM和NPK均有显著差异(P<0.05),而OM与NPK间则无显著差异(P>0.05),表明施肥影响了活性纤维素降解细菌的群落结。
|
|
表 2 不同施肥处理下纤维素降解细菌群落结构差异 Table 2 Dissimilarity tests of metabolically active cellulose-degrading bacterial communities between fertilization strategies using Permutational multivariate analysis |
根据OTU物种注释的结果,水稻土活性纤维素降解细菌群落中超过90%来自于3个细菌门(表 3):变形菌门(Proteobacteria)(42.88%~63.99%)、拟杆菌门(Bacteroidetes)(18.37%~37.26%)和厚壁菌门(Firmicutes)(15.19%~18.74%)。不同施肥处理间进行比较,发现变形菌门在CK处理下丰度较高,而拟杆菌门和厚壁菌门在施肥(NPK和OM)处理下丰度较高。对比本课题组前期以秸秆为底物,利用DNA-SIP技术解析主导秸秆降解微生物物种的结果,不同之处为本研究还发现厚壁菌门微生物也随着施肥,特别是有机肥的施用而增加。该结果可能是因为底物不同,抑或DNA-SIP技术无法检测到底物异化型微生物所至[17]。
|
|
表 3 不同施肥处理下活性纤维素降解细菌的相对丰度 Table 3 Relative abundances of metabolically active cellulose-degrading bacterial phylotypes between fertilization strategies |
将分辨率锁定在科水平,CK处理中变形菌门的优势菌为红环菌科(Rhodocyclaceae)占据了29.03%,较NPK、OM处理的19.41%和18.79%超出10%左右; 与之相反,NPK(19.06%)和OM(15.81%)处理中拟杆菌门的优势菌是紫单胞菌科(Porphyromonadaceae),远高于CK(1.34%)处理; NPK(6.13%)和OM(6.91%)处理中厚壁菌门的优势菌为类芽孢杆菌科(Paenibacillaceae),远高于CK(2.19%)处理。此外,厚壁菌门中的毛螺菌科(Lachnospiraceae)则在CK、NPK和OM处理中占比较为均等,分别为8.41%、8.67%和8.06%。已有研究[18-19]证明在以纤维素或秸秆为底物时可从污泥中富集到紫单胞菌科; 2015年,Hahnke等[20]从沼气反应池中分离出了一株紫单胞菌科的菌株,并从其基因组中检测到了编码纤维素酶的基因。2005年,Siddique等[21]在污水样品中加入秸秆后检测到了类芽孢杆菌科的细菌; 2014年,Keita等[22]从野生大猩猩的粪便样本中分离出了一株类芽孢杆菌科的菌株,并检测到其具有纤维二糖酶活性。2010年,Wongwilaiwalin等[23]从甘蔗渣堆肥筛选出了一个包含红环菌科的高活性纤维素降解菌群落,并证实该群落可降解秸秆等含纤维素的底物且有纤维素降解酶活性。毛螺菌科微生物中含有许多与降解纤维素相关的酶,这使得它可以很好地降解纤维素,且其转运蛋白和代谢途径表明毛螺菌科微生物对复杂植物材料的降解具有较高的专一性[24-25]。上述研究表明,本研究方法鉴定出来的物种确有降解纤维素的能力,证明了该技术用于分离鉴定活性纤维素降解菌的可行性。
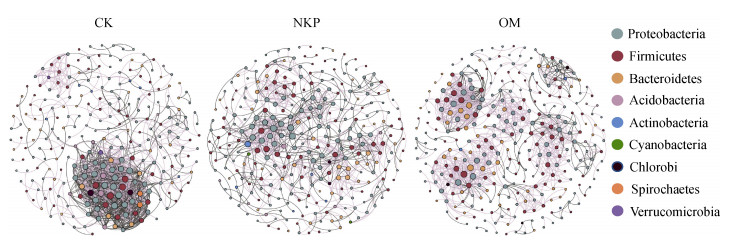
2.4 活性纤维素降解细菌群落分子生态网络分析为明确不同施肥处理对活性纤维素降解细菌种间相互作用的影响,计算了CK、NPK和OM处理下微生物分子生态网络及各拓扑结构参数(图 5和表 4)。结果显示,三种施肥处理下模块化指数均大于0.4,表明三者群落均有模块化结构,即存在生态功能模块。相比于CK,施肥处理(NPK和OM)下活性纤维素降解细菌群落的模块化指数、节点数、连接度、平均聚类系数和更高,而平均度更低,即施肥处理下有更多的微生物群落生态功能模块参与CMC-Na的降解。此外,施肥处理下微生物间的网络连接可能更复杂且网络稳定性更高; 而CK处理下所有节点间的距离均较低且连通性较高[26-29],即施肥提高了CMC-Na活性降解微生物的生态位,增加了网络的稳定性且降低了种间依赖性。
|
图 5 三种施肥处理下活性纤维素降解细菌群落分子生态网络 Fig. 5 The molecular ecological networks of metabolically active cellulose-degrading bacterial communities under three fertilization strategies |
|
|
表 4 三种施肥处理下活性纤维素降解细菌群落的分子生态网络拓扑性质 Table 4 Topological properties of the empirical molecular ecological networks(MENs)of metabolically active cellulose-degrading bacterial communities under three fertilization strategies |
分子生态网络的模块数相当于微生物潜在的生态功能[17],OM施肥处理下的模块数更多,即微生物生态功能更多。由于微生物生态功能多与碳转化相关[30-31],这可能是CMC-Na在OM处理中的降解速度最快(图 3b)的微生物机制。
3 结论本研究运用磁性纳米粒子介导分离技术(MMI)从三种施肥土壤中解析了活性羧甲基纤维素钠(CMC-Na)降解微生物,对比于本课题组前期以秸秆为底物,利用DNA稳定性同位素核酸探针(DNA-SIP)技术解析主导秸秆降解微生物物种的结果,二者均发现施用肥料,特别是有机肥增加了活性降解菌群落的生态网络稳定性和高效性,也增加了拟杆菌门微生物的数量,进而促进了秸秆的分解,且有机肥的效果优于化肥。在能获得大体相同结论的情况下,MMI相比DNA-SIP技术有无需同位素标记目标底物且实验步骤更为简便快捷的优点,具有方法学上的互补性。
| [1] |
Radajewski S, McDonald I R, Murrell J C. Stable-isotope probing of nucleic acids: A window to the function of uncultured microorganisms[J]. Current Opinion in Biotechnology, 2003, 14(3): 296-302. DOI:10.1016/S0958-1669(03)00064-8
(  0) 0) |
| [2] |
Zhang D Y, Berry J P, Zhu D, et al. Magnetic nanoparticle-mediated isolation of functional bacteria in a complex microbial community[J]. ISME Journal, 2015, 9(3): 603-614. DOI:10.1038/ismej.2014.161
(  0) 0) |
| [3] |
Hungate B A, Mau R L, Schwartz E, et al. Quantitative microbial ecology through stable isotope probing[J]. Applied and Environmental Microbiology, 2015, 81(21): 7570-7581. DOI:10.1128/AEM.02280-15
(  0) 0) |
| [4] |
Li J B, Luo C L, Zhang G, et al. Coupling magnetic-nanoparticle mediated isolation(MMI)and stable isotope probing(SIP)for identifying and isolating the active microbes involved in phenanthrene degradation in wastewater with higher resolution and accuracy[J]. Water Research, 2018, 144: 226-234. DOI:10.1016/j.watres.2018.07.036
(  0) 0) |
| [5] |
Wang X Z, Zhao X H, Li H N, et al. Separating and characterizing functional alkane degraders from crude-oil-contaminated sites via magnetic nanoparticle-mediated isolation[J]. Research In Microbiology, 2016, 167(9/10): 731-744.
(  0) 0) |
| [6] |
Pan J L, Dai W A, Shang Z H, et al. Review of research progress on the influence and mechanism of field straw residue incorporation on soil organic matter and nitrogen availability (In Chinese)[J]. Chinese Journal of Eco-Agriculture, 2013, 21(5): 526-535. [潘剑玲, 代万安, 尚占环, 等. 秸秆还田对土壤有机质和氮素有效性影响及机制研究进展[J]. 中国生态农业学报, 2013, 21(5): 526-535.]
(  0) 0) |
| [7] |
Akira W, Makoto K. Effect of rice straw application on CH4 emission from paddy fields IV. Influence of rice straw incorporated during the previous cropping period[J]. Soil Science and Plant Nutrition, 1998, 44(4): 507-512. DOI:10.1080/00380768.1998.10414474
(  0) 0) |
| [8] |
Chen X, Jiang C H, Luo A C. Study on the microbial degradation mechanism of straw (In Chinese)[J]. Journal of Anhui Agricultural Sciences, 2013, 41(23): 9728-9731, 9740. DOI:10.3969/j.issn.0517-6611.2013.23.070 [陈昕, 姜成浩, 罗安程. 秸秆微生物降解机理研究[J]. 安徽农业科学, 2013, 41(23): 9728-9731, 9740.]
(  0) 0) |
| [9] |
Wang H Y, Fan B Q. Screening of three straw-cellulose degrading microorganism (In Chinese)[J]. Acta Microbiologica Sinica, 2010, 50(7): 870-875. [王洪媛, 范丙全. 三株高效秸秆纤维素降解真菌的筛选及其降解效果[J]. 微生物学报, 2010, 50(7): 870-875.]
(  0) 0) |
| [10] |
Wang X, Hua L, Zhang H L, et al. Isolation, identification, mutagenesis of cellulose-degrading bacterium CMC-4 and its enzymatic properties (In Chinese)[J]. Soils, 2017, 49(5): 919-925. [王霞, 华琳, 张海龙, 等. 纤维素降解菌CMC-4的分离鉴定、诱变和酶学特性研究[J]. 土壤, 2017, 49(5): 919-925.]
(  0) 0) |
| [11] |
Lu A H, Salabas E L, Schueth F, et al. Magnetic Nanoparticles: Synthesis, Protection, Functionalization, and Application[J]. Angewandte Chemie International Edition, 2007, 46: 1222-1244. DOI:10.1002/anie.200602866
(  0) 0) |
| [12] |
Huang W E, Wang H. Zheng H J, et al. Chromosomally located gene fusions constructed in Acinetobacter sp ADP1 for the detection of salicylate[J]. Environmental Microbiology, 2005, 7(9): 1339-1348. DOI:10.1111/j.1462-5822.2005.00821.x
(  0) 0) |
| [13] |
Deng C, Bi L D, Qin J T, et al. Effects of long-term fertilization on soil property changes and soil microbial biomass (In Chinese)[J]. Soils, 2013, 45(5): 888-893. [邓超, 毕利东, 秦江涛, 等. 长期施肥下土壤性质变化及其对微生物生物量的影响[J]. 土壤, 2013, 45(5): 888-893.]
(  0) 0) |
| [14] |
Chen X J, Liang T, Zhao Y N, et al. Influence of long-term fertilization managements on organic C and microorganism in different aggregates in purple paddy soil (In Chinese)[J]. Scientia Agricultura Sinica, 2015, 48(23): 4669-4677. DOI:10.3864/j.issn.0578-1752.2015.23.008 [陈轩敬, 梁涛, 赵亚南, 等. 长期施肥对紫色水稻土团聚体中有机碳和微生物的影响[J]. 中国农业科学, 2015, 48(23): 4669-4677.]
(  0) 0) |
| [15] |
Zhang S T, Ren T, Zhou X Q, et al. Effects of rapeseed/wheat-rice rotation and fertilization on soil nutrients and distribution of aggregate carbon and nitrogen (In Chinese)[J]. Acta Pedologica Sinica, 2021. DOI:10.11766/trxb202004090091 [张顺涛, 任涛, 周橡棋, 等. 油/麦-稻轮作和施肥对土壤养分及团聚体碳氮分布的影响[J]. 土壤学报, 2021.]
(  0) 0) |
| [16] |
Xia W J, Liu K L, Zhang L F, et al. Effect of long-term fertilization on soil microbial biomass and enzyme activities in reddish paddy soil (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(3): 628-637. [夏文建, 柳开楼, 张丽芳, 等. 长期施肥对红壤稻田土壤微生物生物量和酶活性的影响[J]. 土壤学报, 2021, 58(3): 628-637.]
(  0) 0) |
| [17] |
Zhan Y S, Liu W J, Bao Y Y, et al. Fertilization shapes a well-organized community of bacterial decomposers for accelerated paddy straw degradation[J]. Scientific Reports, 2018, 8: 7981. DOI:10.1038/s41598-018-26375-8
(  0) 0) |
| [18] |
Sun L, Liu T, Müller B, et al. The microbial community structure in industrial biogas plants influences the degradation rate of straw and cellulose in batch tests[J]. Biotechnology for Biofuels, 2016, 9(1): 1-20. DOI:10.1186/s13068-015-0423-8
(  0) 0) |
| [19] |
Qin Y, Wang H S, Li X R, et al. Improving methane yield from organic fraction of municipal solid waste(OFMSW)with magnetic rice-straw biochar[J]. Bioresource Technology, 2017, 245: 1058-1066. DOI:10.1016/j.biortech.2017.09.047
(  0) 0) |
| [20] |
Hahnke S, Maus I, Wibberg D, et al. Complete genome sequence of the novel Porphyromonadaceae bacterium strain ING2-E5B isolated from a mesophilic lab-scale biogas reactor[J]. Journal of Biotechnology, 2015, 193: 34-36. DOI:10.1016/j.jbiotec.2014.11.010
(  0) 0) |
| [21] |
Siddique T, Okeke B C, Zhang Y Q, et al. Bacterial diversity in selenium reduction of agricultural drainage water amended with rice straw[J]. Journal of Environmental Quality, 2005, 34(1): 217-226.
(  0) 0) |
| [22] |
Keita M B, Padhmanabhan R, Caputo A, et al. Non-contiguous finished genome sequence and description of Gorillibacterium massiliense gen. nov, sp. nov., a new member of the family Paenibacillaceae[J]. Standards In Genomic Sciences, 2014, 9(3): 807-820. DOI:10.4056/sigs.5199182
(  0) 0) |
| [23] |
Wongwilaiwalin S, Rattanachomsri U, Laothanachareon T, et al. Analysis of a thermophilic lignocellulose degrading microbial consortium and multi-species lignocellulolytic enzyme system[J]. Enzyme and Microbial Technology, 2010, 47(6): 283-290. DOI:10.1016/j.enzmictec.2010.07.013
(  0) 0) |
| [24] |
Biddle A, Stewart L, Blanchard J, et al. Untangling the genetic basis of fibrolytic specialization by Lachnospiraceae and Ruminococcaceae in diverse gut communities[J]. Diversity, 2013, 5(3): 627-640. DOI:10.3390/d5030627
(  0) 0) |
| [25] |
Ozbayram E G, Kleinsteuber S, Nikolausz M, et al. Effect of bioaugmentation by cellulolytic bacteria enriched from sheep rumen on methane production from wheat straw[J]. Anaerobe, 2017, 46: 122-130. DOI:10.1016/j.anaerobe.2017.03.013
(  0) 0) |
| [26] |
Latora V, Marchiori M. Efficient behavior of small-world networks[J]. Physical Review Letters, 2001, 87(19): 198701. DOI:10.1103/PhysRevLett.87.198701
(  0) 0) |
| [27] |
Bai R, Wang J T, Deng Y, et al. Microbial community and functional structure significantly varied among distinct types of paddy soils but responded differently along gradients of soil depth layers[J]. Frontiers in Microbiology, 2017, 8: 945. DOI:10.3389/fmicb.2017.00945
(  0) 0) |
| [28] |
Deng Y, Jiang Y H, Yang Y F, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13: 113. DOI:10.1186/1471-2105-13-113
(  0) 0) |
| [29] |
Wood S A, Gilbert J A, Leff J W, et al. Consequences of tropical forest conversion to oil palm on soil bacterial community and network structure[J]. Soil Biology & Biochemistry, 2017, 112: 258-268.
(  0) 0) |
| [30] |
Zheng S X, Hu J L, Chen K, et al. Soil microbial activity measured by microcalorimetry in response to long-term fertilization regimes and available phosphorous on heat evolution[J]. Soil Biology & Biochemistry, 2009, 41(10): 2094-2099.
(  0) 0) |
| [31] |
Jing Z W, Chen R R, Wei S P, et al. Response and feedback of C mineralization to P availability driven by soil microorganisms[J]. Soil Biology & Biochemistry, 2017, 105: 111-120.
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


