2. 华中科技大学生命科学与技术学院, 武汉 430074;
3. 合肥工业大学土木与水利工程学院, 合肥 230009;
4. 北京大学地球与空间科学学院, 北京 100871;
5. 瑞士联邦水科学与技术研究所, 杜本多夫 8600
2. College of Life Science and Technology, Huazhong University of Science and Technology, Wuhan 430074, China;
3. College of Civil Engineering, Hefei University of Technology, Hefei 230009;
4. School of Earth and Space Sciences, Peking University, Beijing 100871, China;
5. Swiss Federal Institute of Aquatic Science and Technology, Dübendorf 8600, Switzerland
病毒在土壤元素生物地球化学循环中发挥重要作用,土壤生态系统中每克土含有的病毒颗粒(Virus-like particles,VLPs)丰度可达109 VLPs[1-3]。病毒基因结构组成非常复杂,大量未知的遗传基因被认为是基因库中的“暗物质”,对这些未知基因的研究,有助于揭开生命的起源、演替和更迭规律,帮助人们理解和确定病毒在生态系统中的地位和作用[4]。
自然界大量的病毒是构成生态系统的重要基础,而分离技术的缺乏是目前限制病毒研究发展的主要原因之一,近年来发展起来的基因组学技术为探索病毒的奥秘提供了有效途径,尤其是高通量测序法已经成为研究环境中微生物群落结构和功能分析的重要工具。高通量测序法具有信息量大、对样本所含信息无偏倚、操作较简单等优点,避免了传统的微生物分离培养步骤,可以直接从环境样品中提取总DNA,是有效提升微生物生态学研究的重要手段[5]。由于土壤具有复杂的空间结构(比表面积达1.1×106~8×106 cm2·g–1),并与土壤各要素(水分、养分、空气和温度等)共同形成了特有的微观异质性,为土壤病毒和其他微生物的生命活动创造了多重生态位,这些因素给土壤微生物学相关研究带来巨大的挑战[6-7]。目前仅有少数宏病毒组学研究揭示了土壤中病毒群落结构[8-11]。Yu等[12]收集了新疆独山子区火山土壤的病毒,并通过宏基因组数据分析病毒群落结构和功能,鉴定出了8个完整的新病毒基因组。Jin等[13]利用病毒宏基因组测序研究了从中国南方不同红树林生境采集的6个红树林土壤样本中的病毒群落,并从中鉴定出了丰富的辅助代谢基因。
黑土是一种结构好、肥力高的土壤,尤其适合种植玉米、小麦、大豆等农作物,被誉为耕地中的“大熊猫”。在我国,黑土主要分布于黑龙江、吉林两省,是我国重要的粮食生产基地。解析黑土微生物群落结构和功能,尤其是病毒的研究对我国的作物生产和安全有重要意义。当前,关于黑土地土壤病毒的研究还十分匮乏,Wang等[14]利用分子生物学技术,以T4型噬菌体主要衣壳蛋白基因(g23)作为目标基因,揭示了东北黑土中不同施肥处理条件下T4型噬菌体的多样性。但目前对于黑土病毒多样性特征及其生态功能的认识仍十分有限。
基于目前发展的宏病毒组测序技术以及生物信息学的数据挖掘能力,本研究拟以一个东北农田黑土样品中的病毒作为研究对象,通过宏基因组测序技术获得土壤宏病毒基因序列,利用生物信息学分析揭示土壤病毒多样性及其生态功能。
1 材料与方法 1.1 土壤样品采集于2019年11月从位于吉林省梨树县大房身乡高家村的中国科学院保护性耕作实验基地(43°19' N,124°14' E)采集耕层土壤样品(0~20 cm)。该处土壤以玉米连作为主,经历多年传统耕作,质地为壤质黏土。利用直径为2 cm的土钻采用网格抽样法随机选取每个样地内采样点,剔除杂物和2 mm以上的沙砾,以四分法将5份样品等量混匀得到一份土样;采样过程使用无菌手套操作,并提前去除样品外围可能污染的部分;采集好的土壤样品放置无菌袋中并放置于冰盒中暂时保存,当日运回实验室后4℃保存备用。
1.2 病毒的收集利用切向流膜过滤系统切向流过滤法收集病毒[15]。具体步骤:称取500 g土壤样品,加入800 mL浸提液(1%柠檬酸钾、250 mmol·L–1甘氨酸)中。将土壤悬液置于180 r·min–1摇床震荡30 min后,转移至离心机3 500×g离心20 min收集上层溶液备用。离心管底的土壤样品加入1.2 L浸提液重悬并搅匀,180 r·min–1摇床震荡30 min后,再次将土壤悬液转移至离心机3500 g离心并收集上层溶液备用。该浸提液洗脱过程,重复3次。所收集的上层溶液用10层纱布过滤去除漂浮物后转移溶液至切向流膜过滤系统(AKTA Flux S)中,分别用0.45 μm、0.22 μm和30 kDa滤管进行过滤和浓缩,得到200 mL病毒浓缩液。将病毒浓缩液转移至50 mL 30 kDa超滤管,3500×g离心8 min,移除滤液。每管超滤管加入1 mL PBS缓冲液充分重悬滤膜中富集的病毒颗粒,置入–80℃冰箱保存。
1.3 基因测序为确保收集的病毒不被环境中游离的DNA干扰,使用DNase I降解土壤溶液中游离的DNA,并使用16S rRNA引物E9F GAGTTTGATCCTGGCT CAG / U1510r GGTTACCTTGTTACGACTT[16]通过PCR扩增检测样品无细菌DNA污染。病毒DNA提取采用改良CTAB法提取[17],消化体系为:200 μL病毒溶液、140 μL 5×TE、50 μL蛋白酶K(2 mg·mL–1)、40 μL 20% SDS。所提取的病毒DNA用全基因扩增试剂盒(illustraTM Ready-To-GoTM GenomiPhiTM V3 DNA Amplification Kit)进行扩增,扩增后浓度为284.63 ng·μL–1,并利用超声破碎仪进行随机打断得到0~23 kb的短片段DNA,用于测序文库构建,质检合格的文库将采用Illumina NovaSeq 6000(Illumina,San Diego,CA,USA)平台进行测序。将测序所得宏病毒组原始序列提交至NCBI Sequence Read Archive(SRA)数据库,获得数据收录号。
1.4 序列数据预处理使用Trimmomatic[18],对原始数据进行质量控制,去除低质量读段得到高质量的过滤后读段条数(Clean reads)。随后使用BWA v0.7.17[19]将Clean reads与NT数据库分离的病毒参考序列进行比对,初步获得样本中的病毒分类信息。最后使用Megahit v1.1.2[20]高质量Clean reads进行组装获得不短于300 bp的Contigs。
1.5 群落结构分析使用两种方法对病毒Contigs进行注释以提高注释的广度和精度。方法一,从NT和NR数据库中分离出相应的病毒序列,使用BLAST将组装得到的Contigs与构建的病毒数据库进行比对,基于参考序列进行物种注释。得到可信度较高(Identity > = 80%,length > =500bp,E-value < =1e–5)的确认序列。方法二,使用BLAST将Contigs分别与从NT和NR分离得到的病毒数据库进行比对,同时将MetaGeneMark v3.38[21]预测得到的基因使用Hmmsearch[22]在病毒的HMM数据库(VPFs[23]和vFam[24])进行搜索比对得到候选的病毒Contigs。使用Blastn将候选Contigs与NT数据库进行比对,随后将未比对上的Contigs使用Diamond v0.9.10[25]与NR数据库进行比对。将比对结果使用NCBI Taxonomy数据库进行注释,若比对前50个结果中注释结果超过80%为病毒,则认为该Contig为病毒序列。方法二得到的新病毒序列和方法一得到的高可信度序列则认为是病毒Contigs,进行后续分析。
使用BWA v0.7.17[19]将Clean reads分别与病毒Contigs比对,过滤比对长度低于80% Reads总长的比对结果,统计病毒Reads比例。随后计算每条病毒Contig的RPKM值和样本Alpha多样性的Shannon指数。
1.6 基因注释以及功能分析采用Prokka v1.13[26]来预测病毒Contigs上长度大于200 bp的基因序列。将预测得到的基因蛋白序列与UniProtKB/Swiss-Prot数据库的病毒序列(ViralZone[27],reviewed proteins,https://viralzone.expasy.org/)进行比对,过滤E-value > 1e–3的比对结果获得病毒基因功能注释信息[28]。
1.7 宿主预测利用CRISPR-Cas系统,研究噬菌体与细菌的关系,推测噬菌体可能的宿主。使用CRISPR Recognition Tool[28](CRT,http://www.room220.com/crt/)从RefSeq数据库的细菌基因组中构建CRISPR-Cas spacer数据库,然后使用Blastn-short v2.9.0与前述鉴定的病毒Contigs比对,获取噬菌体可能的宿主信息。由于目前已知的Spacer序列有限,因此只有一部分病毒Contigs能预测出可能的宿主。
1.8 特征基因组分析使用Prodigal v2.6.3[29]对两条最长的Contigs进行基因预测,使用Blastp将预测得到的基因蛋白序列与UniProtKB/Swiss-Prot数据库的病毒序列(ViralZone[27],reviewed proteins,https://viralzone.expasy.org/)进行比对,过滤E-value > 1e–3的比对结果获得病毒基因功能注释信息。病毒基因组使用SnapGene进行可视化。
2 结果与讨论 2.1 病毒丰度及群落结构通过高通量测序,从东北农田黑土样品中获得35 611 102条原始下机序列(Raw reads),测序数据量为12.2 Gbp,原始数据在NCBI数据库的收录号为SRR14209300。经过质控后Clean reads数占比为78.85%(28 078 029条)。对Clean reads使用Megahit进行拼接,获得229 988条Contigs(> 300 bp)。
通过两种方法对病毒Contigs进行物种注释,注释得到9 851条可能的病毒Contigs(> 300 bp)。由于土壤环境的复杂性导致病毒收集过程中其他微生物碎片所携带基因组的干扰,以及目前土壤病毒基因组库还尚未完善,大量未知的基因无法被注释,因此仅有4%组装的Contigs被注释为病毒。此外,虽然采用全基因组扩增后测定序列有偏好性,偏向于单链病毒,而本文得到最多的是双链病毒,可能由于样品中dsDNA病毒占比较大,因此能够被注释的Contigs也多属于dsDNA病毒。病毒群落主要归属于有尾噬菌体目(Caudovirales)和疱疹病毒目(Herpesvirales)2个病毒目,以及29个病毒科和一些未分类的病毒(图 1)。其中长尾噬菌体科(Siphoviridae)、微小噬菌体科(Microviridae)、肌病毒科(Myoviridae)相对丰度最高,分别为44.48%、20.53%和8.38%。噬菌体占55.42%,而其他的病毒占44.58%。基于病毒序列的RPKM值计算该东北农田土壤病毒的香农指数为8.93。研究发现,噬菌体是该农田土壤的主要类群,其中长尾噬菌体科(Siphoviridae)是一类dsDNA病毒,可以感染细菌和古菌,不仅在深海沉积物[30-31]、沙漠土壤[32]、农田土壤[33]以及森林土壤[11]广泛分布,在生活污水[34]、人体肠道[35]等与人类息息相关的环境中也均被报道,是一类重要的环境病毒。微小噬菌体科则是一类ssDNA病毒,有研究报道在部分农田土壤和森林土壤中微小噬菌体科病毒数量占比最大[36]。其他以微生物为宿主的病毒科肌病毒科(Myoviridae)、短尾噬菌体科(Podoviridae)、代列尔噬菌体科(Herelleviridae)、Ackermannviridae科、三层病毒科(Tristromaviridae,侵染古菌)、丝杆病毒科(Inoviridae)、Lavidaviridae科。除噬菌体外,该样品中还有以原生动物为宿主的拟菌病毒科(Mimiviridae)、阔口罐病毒科(Pithoviridae)、马赛病毒科(Marseilleviridae),它们属于核质巨型DNA病毒,拥有较大的衣壳直径和庞大的基因组。与植物相关的病毒则有藻类DNA病毒科(Phycodnaviridae)、Genomoviridae科、双生病毒科(Geminiviridae)、矮化病毒科(Nanoviridae)、Alphasatellitidae科。此外,还发现了多种与动物致病相关的病毒,包含杆状病毒科(Baculoviridae)、虹彩病毒科(Iridoviridae)、Polydnaviridae科、Nudiviridae科、细小病毒科(Parvoviridae)、疱疹病毒科(Herpesviridae)、圆环病毒科(Circoviridae)、痘病毒科(Poxviridae)、异疱疹病毒科(Alloherpesviridae)、腺病毒科(Adenoviridae)、乳头瘤病毒科(Papillomaviridae)。而其中人类健康息息相关的病毒,有疱疹病毒科(Herpesviridae)、痘病毒科(Poxviridae)、异疱疹病毒科(Alloherpesviridae)、腺病毒科(Adenoviridae)、乳头瘤病毒科(Papillomaviridae)和圆环病毒科(Circoviridae)等,这也暗示土壤成为了人类病毒的储存库,对人类的健康有着潜在的影响。
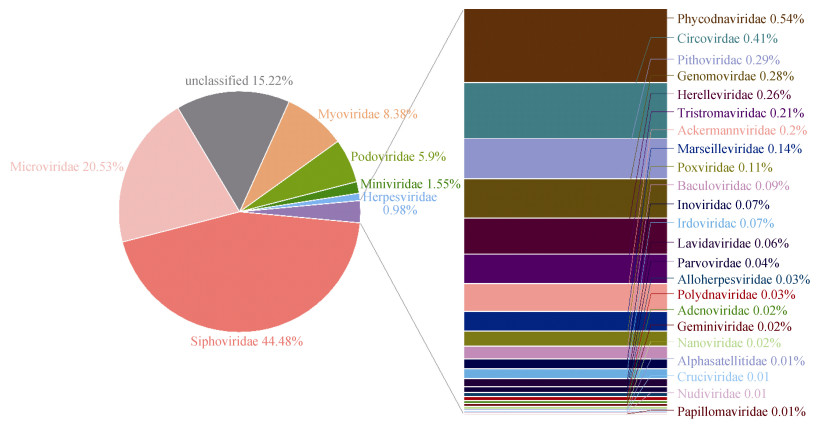
|
图 1 病毒物种注释结果(科水平) Fig. 1 Taxonomic composition of viromes(family level) |
病毒在生态系统中发挥着关键作用。病毒可以通过辅助代谢基因(Auxiliary metabolic genes,AMGs)重新编码宿主基因而影响其新陈代谢[37-38]。同时,病毒还可以通过介导水平基因转移驱动宿主进化[39-40]。本研究中,使用Prokka v1.13对病毒Contigs进行基因预测,一共预测到808个tRNA和12 058个CDS(Coding sequence)。在UniprotKB/ Swiss-Prot数据库中有效注释病毒序列1 867个基因。
注释结果中,分子功能相关基因共有179个,其中占比较高的是催化活性基因(Catalytic activity),共有146个,包含氧化还原酶活性、转移酶活性、水解酶活性、裂解酶活性、肽聚糖的遗传活性、催化蛋白质活性、催化DNA活性、催化RNA活性。这些微生物催化酶活性方面的相关基因在微生物细胞生长和遗传发育中起着不可替代的调控作用。维持病毒繁殖和生理结构的相关基因共有124个,包含病毒衣壳蛋白、病毒膜蛋白、尾部和末端酶等。生物过程代谢相关基因共有105个,包含氮化合物代谢过程、分解代谢过程、生物合成过程、甲基化、多生物代谢过程、细胞代谢过程、初级代谢过程、含碱基小分子代谢过程以及有机物代谢过程,相关基因参与了微生物介导的诸多物质循环过程,如代谢过程。这些过程在不同程度下均影响了微生物参与的地球元素化学循环。
2.3 噬菌体宿主预测目前土壤病毒资源的获取在微生物学中仍然是个巨大的挑战,病毒宿主的预测可以有效地指导单病毒的分离工作。本研究从Refseq数据库中的细菌基因组挖掘CRISPR-Cas序列构建CRISPR-Cas spacer数据库,对病毒Contigs比对搜索获取可能的宿主信息。但由于已知的Spacer序列有限,因此样品中仅有66条病毒Contigs能预测出可能的宿主,具体结果详见表 1。其中有30条(45.45%)Contigs被预测宿主属变形菌门,占能被预测宿主最多比例,宿主分属于无色杆菌属(Achromobacter)、不动杆菌属(Acinetobacter)、气单胞菌属(Aeromonas)、弓形杆菌属(Arcobacter)、伯克氏菌属(Burkholderia)、Candidimonas属、柠檬酸杆菌属(Citrobacter)、克洛诺菌属(Cronobacter)、克雷伯氏菌属(Klebsiella)、军团菌属(Legionella)、Methylocaldum属、甲基单胞菌属(Methylomonas)、新鞘氨醇杆菌属(Novosphingobium)、寡养杆菌属(Oligella)、草酸杆菌属(Oxalobacter)、潘隆尼亚碱湖杆菌(Pannonibacter)、假单胞菌属(Pseudomonas)、假螺菌属(Pseudospirillum)、假黄色单胞菌属(Pseudoxanthomonas)、红假单胞菌属(Rhodopseudomonas)、盐弧菌属(Salinivibrio)、沙门氏菌属(Salmonella)、Sutterella属、弧菌属(Vibrio)、耶尔森氏菌属(Yersinia)15个菌属中。有20条(30.30%)Contigs被预测宿主属放线菌门,宿主分属于放线菌属(Actinomyces)、双歧杆菌属(Bifidobacterium)、纤维菌属(Cellulomonas)、小单胞菌属(Micromonospora)、诺卡式菌属(Nocardia)、Plantactinospora属、四折叠球菌属(Quadrisphaera)、盐胞菌属(Salinispora)、链霉菌属(Streptomyces)、Thermobifida属10个菌属中。有9条(13.63%)Contigs被预测宿主属厚壁菌门,宿主分属于芽孢杆菌属(Bacillus)、Clostridioides属、Cohnella属、Desulfitobacterium属、乳酸菌属(Lactobacillus)、巨球菌属(Megasphaera)6个菌属中。有5条(7.58%)Contigs被预测宿主属于拟杆菌门,宿主分属于Niastella属、类土地杆菌属(Parapedobacter)、地杆菌属(Pedobacter)3个菌属中。有2条(3.03%)Contigs被预测宿主属于绿菌门,宿主属于突柄绿菌属(Prosthecochloris)。该农田土壤病毒可预测的宿主分属于放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿菌门(Chlorobi)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)5个菌门中的35个菌属,其中以变形菌门和放线菌门的病毒预测宿主居多,分别占45.45%和30.30%。变形菌门和放线菌门作为土壤中高丰度的菌门被广泛报道,目前已得到分离的噬菌体也大多来自于这两个菌门当中。
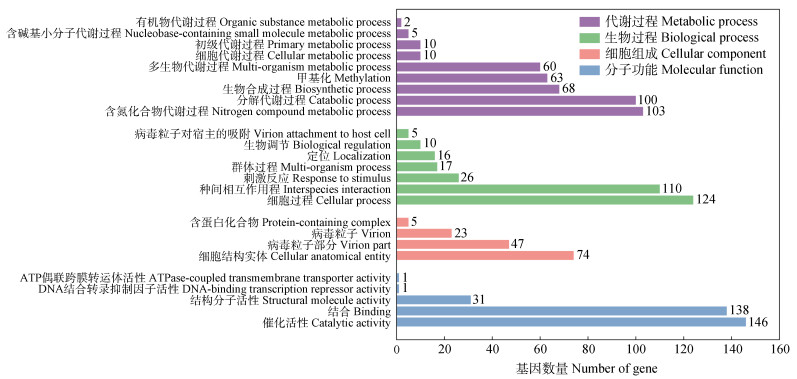
|
图 2 病毒基因组在UniProt数据库中的注释结果 Fig. 2 Annotation of viral genes against UniProt database |
|
|
表 1 土壤噬菌体宿主预测结果 Table 1 Prediction results of soil phage host |
在土壤的宏病毒组中组装注释得到2 284条病毒Contigs(> 1 kb)。筛选出其中最长的两条Contigs分别为Contig 1(54 552 bp)和Contig 2(55 789 bp),分析其功能基因组成(图 3)。使用BLAST基于NCBI的Complete bacteriophages数据库进行序列比对并进行物种注释,比对发现Contig 1与Streptomyces phage VWB相似性最高,相似度为76.85%[41]。Contig 2与Pseudomonas phage PS-1相似性最高,相似度为81.13%[42]。Contig 1预测得到115个编码序列,Contig 2预测得到75个编码序列。使用Blastp基于Uniprot/Swiss-Prot数据库进行基因注释,Contig 1有7个基因能被注释,Contig 2有17个基因能被注释。由于土壤病毒研究较少,相关数据库并不完善,因此只有少部分基因能够进行功能注释。虽然能够进行功能注释的基因不多,但是大部分都与病毒的组装、转录翻译及裂解等重要的生理过程相关。

|
注:箭头方向为转录方向,紫色箭头为无法进行功能注释的假象蛋白,蓝色箭头为能够进行功能注释的蛋白。 Note: The arrow direction is the transcription direction, and the purple arrow is the hypothetical protein that cannot be annotated functionally, and the blue arrow is the protein that can be annotated functionally. 图 3 典型contig基因注释图(> 1 kb) Fig. 3 Genomic features of representative viral contigs(> 1 kb) |
本研究首次通过宏病毒组学方法报道了一个东北农田黑土样品中病毒的群落组成和功能特征。通过数据库比对分析,发现病毒主要归属于有尾噬菌体目(Caudovirales,59.38%)和疱疹病毒目(Herpesvirales,2.56%)2个病毒目以及29个病毒科,其中长尾噬菌体科(Siphoviridae)、微小噬菌体科(Microviridae)相对丰度最高,分别为44.48%和20.53%。功能基因注释分析表明了土壤病毒在微生物酶催化和生物代谢等过程中发挥着潜在的作用。对病毒基因组进行了宿主预测,可预测的宿主归属于放线菌门、拟杆菌门、绿菌门、厚壁菌门和变形菌门等5个菌门的35个菌属中。
| [1] |
Breitbart M, Rohwer F. Here a virus, there a virus, everywhere the same virus?[J]. Trends in Microbiology, 2005, 13(6): 278-284. DOI:10.1016/j.tim.2005.04.003
(  0) 0) |
| [2] |
Reavy B, Swanson M M, Taliansky M. Viruses in soil[M]//Dighton J, Krumins J A. Interactions in soil: promoting plant growth. Netherlands: Springer, 2014: 163—180.
(  0) 0) |
| [3] |
Chen L, Xun W B, Sun L, et al. Effect of different long-term fertilization regimes on the viral community in an agricultural soil of Southern China[J]. European Journal of Soil Biology, 2014, 62: 121-126. DOI:10.1016/j.ejsobi.2014.03.006
(  0) 0) |
| [4] |
Wang G H, Liu J J, Zhu D, et al. A review of researches on viruses in soil–advancement and challenges (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(6): 1319-1332. [王光华, 刘俊杰, 朱冬, 等. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319-1332.]
(  0) 0) |
| [5] |
Weinbauer M G. Ecology of prokaryotic viruses[J]. FEMS Microbiology Reviews, 2004, 28(2): 127-181. DOI:10.1016/j.femsre.2003.08.001
(  0) 0) |
| [6] |
Wilpiszeski R L, Aufrecht J A, Retterer S T, et al. Soil aggregate microbial communities: Towards understanding microbiome interactions at biologically relevant scales[J]. Applied and Environmental Microbiology, 2019, 85(14). DOI:10.1128/aem.00324—19
(  0) 0) |
| [7] |
Fierer N, Breitbart M, Nulton J, et al. Metagenomic and small-subunit rRNA analyses reveal the genetic diversity of bacteria, Archaea, fungi, and viruses in soil[J]. Applied and Environmental Microbiology, 2007, 73(21): 7059-7066. DOI:10.1128/AEM.00358-07
(  0) 0) |
| [8] |
Diemer G S, Stedman K M. A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses[J]. Biology Direct, 2012, 7: 13. DOI:10.1186/1745-6150-7-13
(  0) 0) |
| [9] |
Emerson J B, Thomas B C, Andrade K, et al. Dynamic viral populations in hypersaline systems as revealed by metagenomic assembly[J]. Applied and Environmental Microbiology, 2012, 78(17): 6309-6320. DOI:10.1128/AEM.01212-12
(  0) 0) |
| [10] |
Yoshida M, Takaki Y, Eitoku M, et al. Metagenomic analysis of viral communities in(hado)pelagic sediments[J]. PLoS One, 2013, 8(2): e57271. DOI:10.1371/journal.pone.0057271
(  0) 0) |
| [11] |
Kuzyakov Y, Blagodatskaya E. Microbial hotspots and hot moments in soil: Concept & review[J]. Soil Biology & Biochemistry, 2015, 83: 184-199.
(  0) 0) |
| [12] |
Yu D T, He J Z, Zhang L M, et al. Viral metagenomics analysis and eight novel viral genomes identified from the Dushanzi mud volcanic soil in Xinjiang, China[J]. Journal of Soils and Sediments, 2019, 19(1): 81-90. DOI:10.1007/s11368-018-2045-9
(  0) 0) |
| [13] |
Jin M, Guo X, Zhang R, et al. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7(1): 58. DOI:10.1186/s40168-019-0675-9
(  0) 0) |
| [14] |
Wang G H, Yu Z H, Liu J J, et al. Molecular analysis of the major capsid genes(g23)of T4-type bacteriophages in an upland black soil in Northeast China[J]. Biology and Fertility of Soils, 2011, 47(3): 273-282. DOI:10.1007/s00374-010-0533-1
(  0) 0) |
| [15] |
Thurber R V, Haynes M, Breitbart M, et al. Laboratory procedures to generate viral metagenomes[J]. Nature Protocols, 2009, 4(4): 470-483. DOI:10.1038/nprot.2009.10
(  0) 0) |
| [16] |
Ramond J B, Welz P J, Tuffin M I, et al. Assessment of temporal and spatial evolution of bacterial communities in a biological sand filter mesocosm treating winery wastewater[J]. Journal of Applied Microbiology, 2013, 115(1): 91-101. DOI:10.1111/jam.12203
(  0) 0) |
| [17] |
Wang H N, Guo Z Q, Feng H L, et al. Novel sulfolobus virus with an exceptional capsid architecture[J]. Journal of Virology, 2018, 92(5): e01727-e01717.
(  0) 0) |
| [18] |
Bolger A M, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics: Oxford, England, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170
(  0) 0) |
| [19] |
Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760. DOI:10.1093/bioinformatics/btp324
(  0) 0) |
| [20] |
Li D H, Luo R B, Liu C M, et al. MEGAHIT v1.0: A fast and scalable metagenome assembler driven by advanced methodologies and community practices[J]. Methods, 2016, 102: 3-11. DOI:10.1016/j.ymeth.2016.02.020
(  0) 0) |
| [21] |
Zhu W H, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences[J]. Nucleic Acids Research, 2010, 38(12): e132. DOI:10.1093/nar/gkq275
(  0) 0) |
| [22] |
Eddy S R. A new generation of homology search tools based on probabilistic inference[J]. Genome Informatics International Conference on Genome Informatics, 2009, 23(1): 205-211.
(  0) 0) |
| [23] |
Paez-Espino D, Eloe-Fadrosh E A, Pavlopoulos G A, et al. Uncovering Earth's virome[J]. Nature, 2016, 536(7617): 425-430. DOI:10.1038/nature19094
(  0) 0) |
| [24] |
Skewes-Cox P, Sharpton T J, Pollard K S, et al. Profile hidden Markov models for the detection of viruses within metagenomic sequence data[J]. PLoS One, 2014, 9(8): e105067. DOI:10.1371/journal.pone.0105067
(  0) 0) |
| [25] |
Buchfink B, Xie C, Huson D H. Fast and sensitive protein alignment using DIAMOND[J]. Nature Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176
(  0) 0) |
| [26] |
Seemann T. Prokka: rapid prokaryotic genome annotation[J]. Bioinformatics, 2014, 30(14): 2068-2069. DOI:10.1093/bioinformatics/btu153
(  0) 0) |
| [27] |
Hulo C, de Castro E, Masson P, et al. ViralZone: A knowledge resource to understand virus diversity[J]. Nucleic Acids Research, 2011, 39(suppl_1): D576-D582. DOI:10.1093/nar/gkq901
(  0) 0) |
| [28] |
Bland C, Ramsey T L, Sabree F, et al. CRISPR recognition tool(CRT): A tool for automatic detection of clustered regularly interspaced palindromic repeats[J]. BMC Bioinformatics, 2007, 8: 209. DOI:10.1186/1471-2105-8-209
(  0) 0) |
| [29] |
Hyatt D, Chen G L, Locascio P F, et al. Prodigal: Prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11: 119. DOI:10.1186/1471-2105-11-119
(  0) 0) |
| [30] |
Zheng X W, Liu W, Dai X, et al. Extraordinary diversity of viruses in deep-sea sediments as revealed by metagenomics without prior virion separation[J]. Environmental Microbiology, 2021, 23(2): 728-743. DOI:10.1111/1462-2920.15154
(  0) 0) |
| [31] |
Cai L L, Jørgensen B B, Suttle C A, et al. Active and diverse viruses persist in the deep sub-seafloor sediments over thousands of years[J]. The ISME Journal, 2019, 13(7): 1857-1864. DOI:10.1038/s41396-019-0397-9
(  0) 0) |
| [32] |
Adriaenssens E M, Van Zyl L, De Maayer P, et al. Metagenomic analysis of the viral community in Namib Desert hypoliths[J]. Environmental Microbiology, 2015, 17(2): 480-495. DOI:10.1111/1462-2920.12528
(  0) 0) |
| [33] |
Bi L, Yu D T, Du S, et al. Diversity and potential biogeochemical impacts of viruses in bulk and rhizosphere soils[J]. Environmental Microbiology, 2021, 23(2): 588-599. DOI:10.1111/1462-2920.15010
(  0) 0) |
| [34] |
Guerrero L D, Pérez M V, Orellana E, et al. Long-run bacteria-phage coexistence dynamics under natural habitat conditions in an environmental biotechnology system[J]. The ISME Journal, 2021, 15(3): 636-648. DOI:10.1038/s41396-020-00802-z
(  0) 0) |
| [35] |
Mirzaei M K, Maurice C F. Ménage à trois in the human gut: Interactions between host, bacteria and phages[J]. Nature Reviews Microbiology, 2017, 15(7): 397-408. DOI:10.1038/nrmicro.2017.30
(  0) 0) |
| [36] |
Bi L, Du S, Yu D T, et al. Compositional and functional characteristics of viruses in soil under two types of land-use in Xinjiang, China (In Chinese)[J]. Acta Ecologica Sinica, 2021, 41(7): 2728-2737. [毕丽, 杜帅, 于丹婷, 等. 新疆两种土地利用方式下土壤病毒的群落组成与功能特征[J]. 生态学报, 2021, 41(7): 2728-2737.]
(  0) 0) |
| [37] |
Roux S, Brum J R, Dutilh B E, et al. Ecogenomics and potential biogeochemical impacts of globally abundant ocean viruses[J]. Nature, 2016, 537(7622): 689-693. DOI:10.1038/nature19366
(  0) 0) |
| [38] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880. DOI:10.1038/s41564-018-0190-y
(  0) 0) |
| [39] |
Mann N H, Cook A, Millard A, et al. Bacterial photosynthesis genes in a virus[J]. Nature, 2003, 424(6950): 741.
(  0) 0) |
| [40] |
Suttle C A. Marine viruses—major players in the global ecosystem[J]. Nature Reviews Microbiology, 2007, 5(10): 801-812. DOI:10.1038/nrmicro1750
(  0) 0) |
| [41] |
Anné J, Van Mellaert L, Decock B, et al. Further biological and molecular characterization of actinophage VWB[J]. Journal of General Microbiology, 1990, 136(7): 1365-1372. DOI:10.1099/00221287-136-7-1365
(  0) 0) |
| [42] |
Yoshida M, Yoshida-Takashima Y, Nunoura T, et al. Identification and genomic analysis of temperate Pseudomonas bacteriophage PstS-1 from the Japan trench at a depth of 7000 M[J]. Research in Microbiology, 2015, 166(9): 668-676. DOI:10.1016/j.resmic.2015.05.001
(  0) 0) |
 2022, Vol. 59
2022, Vol. 59


