2. 瑞士联邦水科学与技术研究所, 杜本多夫 8600;
3. 合肥工业大学土木与水利工程学院, 合肥 230009;
4. 中国农业大学国家黑土地现代农业研究院, 北京 100193
2. Swiss Federal Institute of Aquatic Science and Technology, Dübendorf 8600, Switzerland;
3. College of Civil Engineering, Hefei University of Technology, Hefei 230009, China;
4. National Black Soil and Agriculture Research, China Agricultural University, Beijing 100193, China
氮素是作物及微生物生长必需元素之一,其在土壤中的周转对于土壤供氮能力、作物生长、粮食安全及生态系统服务等具有重要意义[1]。因此,深入了解土壤氮循环过程及机理对农业可持续发展及生态环境安全至关重要。然而,土壤氮循环过程极为复杂,同时涵盖固氮、硝化、反硝化、氨化、厌氧氨氧化、硝酸盐异化还原成铵等不同形态氮素转化过程[2-3]。在复杂的氮素循环过程中,土壤微生物(如固氮菌、硝化细菌和反硝化细菌等)发挥着不可或缺的作用[3]。而氮循环相关功能微生物主要通过功能基因的表达来编码各过程中关键酶,进而调控土壤氮循环过程[4-5]。因此,土壤氮循环相关功能基因可应用于评估微生物参与下的氮循环过程。
近年来,基因芯片、高通量测序、宏基因组学等技术迅速兴起,使得土壤样品中氮循环功能基因的检测成为可能[6-9]。研究表明,nifH、amoA、nirS、nirK和nosZ等氮循环相关功能基因丰度被广泛应用于指示土壤氮循环相关功能微生物丰度及种群变化[4,10-12]。Petersen等[13]也发现与氮循环相关的微生物功能基因丰度可以作为相关氮循环过程潜在周转速率的预测指标。通过定量检测微生物氮循环相关功能基因,进而探索土壤氮循环与相关功能基因多样性之间的联系,可综合分析不同生境中土壤氮循环的整体水平及影响因素,从而为土壤氮循环机制提供基因水平上的证据。
目前关于土壤氮循环功能基因方面的研究报道较多,然而该研究领域现状、热点及历史发展趋势等尚有待进行系统性梳理。科学知识图谱是一种通过统计分析和计算机技术,对大量文献信息(如关键词、被引频次、参考文献等)按照信息单元的相似性、测度来构建矩阵,通过可视化分析(如网络和概念结构图谱等)展示各信息单元间的联系和结构的文献计量学方法[14],近年来广泛应用在农学[15-17]和环境[18-21]、植物[22]、地学[23]等学科领域,但具体到土壤氮循环功能基因研究领域的文献计量分析研究尚未见报道。
当前用于科学知识图谱分析的软件较多,如CiteSpace、VOSviewer和R语言中bibliometrix软件包等。其中,R语言作为一种开源的可应用于计算、统计及可视化分析的语言[24],bibliometrix包是基于R语言的科学知识图谱绘制软件包,能通过对文献信息进行统计分析,有效检索出对一个学科(或研究领域/方向)发展产生重要影响的关键文献,并展示其在一定时间尺度上的历史发展趋势和研究热点及动态[14]。
本研究基于Web of Science核心合集数据库和R语言bibliometrix软件包(ver. 3.0.4),从发文量趋势、高被引论文、高频关键词及历史直接引文四个方面,对近20年(2001—2020年)发表的有关土壤氮循环功能基因方面文献信息进行科学知识图谱分析,进而对该研究领域最新研究进展、热点及历史发展脉络进行综述并提出一定展望,以期为相关研究者提供一定参考。
1 材料与方法 1.1 数据来源2021年1月27日,以“土壤氮循环功能基因”为主题,以“TS =(soil AND nitrogen AND(functional AND gene))”为检索式在Web of Science核心合集数据库进行文献检索。检索时设置文献类型为文章(article)、语种为英语(English)、文献时间跨度为最近20年(2001—2020年),共获取1 219篇相关文献,下载全部文献信息(其中涵盖文章作者、标题、关键词、发表年份、摘要、参考文献等)。
1.2 研究方法运用R语言bibliometrix软件包(ver. 3.0.4)对获取到的文献信息进行科学知识图谱分析,主要包括发文量趋势分析、高被引论文分析、高频关键词分析以及历史直接引文分析,以探讨土壤氮循环功能基因研究领域的最新研究进展、现状和热点及历史发展脉络和趋势,具体方法参考之前研究结果[14,25-26]。发文量趋势分析是指对2001—2020年发表的土壤氮循环功能基因研究领域逐年发文量进行可视化分析,进而判断该领域发展状况。高被引论文分析是指分别基于全球/本地数据集对文献被引篇次进行统计,从而确定高被引论文。其中全球数据集是指Web of Science核心合集数据库,本地数据集是指2001—2020年间土壤氮循环功能基因研究领域相关文献数据集。总被引频次(Total citations,TC)和本地被引频次(Local citations,LC)分别指该篇文章在全球/本地数据集中被引用的次数,TC(或LC)愈高说明该文章与该研究领域愈相关[26]。高频关键词分析主要是指对文献中的最高频的关键词绘制词云图、共现网络图谱、聚类图谱及概念结构图,从而进行可视化分析。词云图中字体越大,表示该关键词出现频次越高;关键词共现网络图谱中圆圈愈大,则说明该关键词度中心性越大[27],而度中心性作为网络分析中度量节点中心性的最直接表征指标,其越高则说明该节点关键词在网络中愈发重要[14]。关键词聚类图谱及概念结构图是在关键词共现网络图谱基础上,利用多重对应分析(Multiple Correspondence Analysis,MCA)方法,以网络映射和聚类的方式利用文献中关键词共现网络绘制框架概念结构图,不同关键词间距离越小越相似,相关性较强,联系越紧密[28]。历史直接引文分析是通过快速识别出研究领域历史发展过程中最直接相关、直接引用的关键文献,再按照时间序列绘制历史直接引文图谱,从而逐年溯源分析出该研究领域的历史发展脉络[29]。
2 结果 2.1 发文量趋势2001—2020年土壤氮循环功能基因研究领域发文量可划分为2个阶段:平稳发展阶段(2001—2006年)和迅速增长(2006—2020年)阶段。2001—2006年,每年发文量不超过10篇,该时期土壤氮循环相关功能基因研究发展缓慢;2006—2020年,发文量极速增长,尤其是近3年发展更为迅速(图 1)。这说明土壤氮循环相关功能基因研究领域正处于高速发展阶段,同时也证实该研究领域目前为国际土壤学研究热点,推测可能与近年来分子生物学技术的快速发展有关[30]。

|
图 1 2001—2020年土壤氮循环功能基因研究领域全球发文量趋势 Fig. 1 Global trends in the number of published articles in the field of functional gene related to soil nitrogen cycling from 2001 to 2020 |
通过对2001—2020年土壤氮循环功能基因研究领域全球/本地Top 10高被引论文(表 1和表 2)进行汇总、排序,去重后共计16篇高被引论文,研究发现这些论文主要聚焦于以下3个方面:
|
|
表 1 2001—2020年土壤氮循环功能基因研究领域全球数据库中Top 10高被引论文 Table 1 Top 10 most-cited papers on functional genes of soil nitrogen cycling in the global database from 2001 to 2020 |
|
|
表 2 2001—2020年土壤氮循环功能基因研究领域本地数据库中Top 10高被引论文 Table 2 Top 10 most-cited papers on functional genes of soil nitrogen cycling in the local database from 2001 to 2020 |
1)通过测定土壤氮循环过程(如固氮[31-32]、硝化[33-36]、反硝化[34,37-38]等)相关功能基因丰度,来探究土壤氮循环及潜在微生物学机制[13]。如Gubry-Rangin等[33]通过测定氨氧化过程相关的amoA功能基因丰度,发现土壤氨氧化古菌(而非氨氧化细菌)控制着农业酸性土壤中的硝化作用。Poly等[31]和Tourna等[35]分别对固氮作用(nifH基因)和氨氧化作用(amoA)相关的功能基因进行分析,进而研究了土壤固氮菌和氨氧化古菌和细菌活性及结构变化。值得注意的是,高被引论文中排名第一的是Daims等2015年发表于Nature上的“Complete nitrification by Nitrospira bacteria”,研究发现了单步硝化菌(Nitrospira)及相关功能基因,并指出单步硝化菌是土壤氮循环微生物群落中的重要组成部分,从根本上改变了学术界对硝化作用的认识[36]。
2)通过基因组测序或宏基因组学等分子生物学技术,筛选、鉴定及识别功能基因[32,39]。如Young等[32]通过对豆科根瘤菌(Rhizobium leguminosarum biovar viciae strain 3841)进行基因组测序,确定该基因组中可识别的核心和附属组成部分,并识别出了固氮作用相关的功能基因。Jones等[38]通过对公共数据库中的N2O还原酶的编码功能基因nosZ进行系统发育分析,发现在不同生境类型和环境条件下,nosZ谱系广泛多样且相对丰度不同这一特征。
3)通过测定土壤氮循环相关功能基因丰度这一指标,以研究土壤氮素转化过程的主要影响因素,如土地利用类型[31]、养分添加[40-41]、生物炭施用[34]、温度(气候变暖)[35,42-43]、CO2浓度升高[42-43]等,进而评估这些影响因子对土壤氮循环过程及相关微生物活性、丰度、功能基因、群落结构的影响。如Harter等[34]通过qPCR技术对土壤微生物固氮(nifH)、硝化(amoA)和反硝化(nirK、nirS和nosZ)过程相关功能基因的丰度和活性进行定量分析,发现施加生物炭增加了编码N2O还原酶的nosZ基因相对丰度,且该结果与观测到的N2O排放量减少联系紧密,从而揭示了生物炭对土壤N2O排放的微生物学影响机制。Leff等[41]通过对功能基因进行高通量测序,在全球尺度上阐明了土壤真菌、古菌以及细菌群落对高养分投入量的响应特征,结果发现,氮、磷养分输入量的增加引发了代表性功能基因相对丰度的变化,即土壤微生物群落的分类和功能性状发生变化,包括快速生长、富营养细菌类群的相对丰度增加等,这些变化可能会影响全球地下生态系统。Fierer等[40]通过运用16S rRNA测序及宏基因组学技术,对不同氮素水平下的土壤微生物群落结构及功能进行对比分析,发现施氮可以直接或间接地诱导主要微生物生活史策略发生改变,有利于形成更活跃的富营养微生物群落。Yergeau等[42]通过16S rRNA基因测序和qPCR基因芯片分析,揭示了气候变暖对功能微生物群落具有显著的增温影响,尤其是对于氮循环相关功能微生物。Zhou等[43]也利用16S rRNA高通量宏基因组测序数据来识别微生物群落系统发育分子生态网络,确定了CO2浓度升高对土壤微生物群落系统发育的影响,且发现网络结构的变化与土壤碳氮含量显著相关。Mackelprang等[44]通过宏基因组学测序技术,确定多年冻土融化对微生物系统发育及功能基因的影响以及甲烷(CH4)和氮素在冻土融化过程中快速循环的重要性。从冷冻状态到融化状态转变过程中,大量微生物、系统发育及功能基因丰度和路径发生快速改变。且在融化过程中,参与碳氮循环的多个基因会迅速转移。综上可得,人为管理或自然环境条件的变化均可能导致土壤氮素转化过程及相应功能微生物群落及生态功能发生转变,功能基因在表征功能微生物丰度及群落变化方面具有重要应用价值,通过确定土壤氮循环功能基因丰度及其主要影响因子有助于解释土壤氮循环的微生物学机制。
2.3 高频关键词1 219篇土壤氮循环功能基因文献中共含3 447个关键词,对高频关键词进行分析(图 2—图 4;表 3)。关键词共现网络图谱结果(图 2、图 3;表 3)发现,2001—2020年土壤氮循环功能基因研究领域Top 10高频关键词依次为:多样性(diversity)、氮(nitrogen)、土壤(soil)、反硝化作用(denitrification)、细菌(bacteria)、碳(carbon)、基因(genes)、丰度(abundance)、硝化作用(nitrification)、微生物群落结构(microbial communities)。这说明,土壤氮循环功能基因研究主要涉及到不同氮素转化过程(如反硝化作用和硝化作用)、微生物(细菌)多样性及群落结构、功能基因丰度等内容[34,45-46]。此外,图 2和图 3结果也表明土壤氮循环功能基因研究领域所涉及的学科交叉现象极为常见,涉及到农学、微生物学、植物学、环境学、生态学及大气科学等学科,未来可能会有更多学科相互交叉,产生一些新的研究方向、热点,如结合地理学、工学(计算机模拟方面)等学科。

|
图 2 2001—2020年土壤氮循环功能基因关键词云图 Fig. 2 Keywords cloud map about functional genes related to soil nitrogen cycling from 2001 to 2020 |
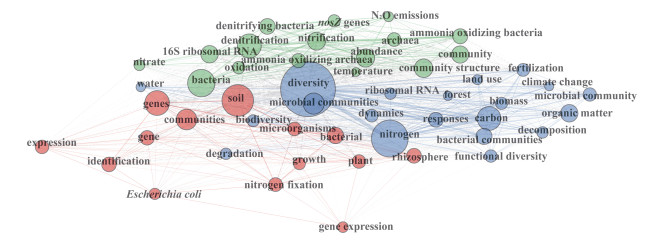
|
图 3 2001—2020年土壤氮循环功能基因关键词共现网络图谱 Fig. 3 Keywords co-occurrence network about functional genes related to soil nitrogen cycling from 2001 to 2020 |

|
图 4 2001—2020年土壤氮循环功能基因关键词聚类图谱 Fig. 4 Keywords clustering map of functional genes related to soil nitrogen cycling from 2001 to 2020 |
|
|
表 3 2001—2020年土壤氮循环功能基因关键词共现频次及度中心性 Table 3 Keywords co-occurrence network analysis about soil nitrogen cycling functional genes from 2001 to 2020 |
关键词聚类图及概念结构图(图 4)结果表明,关键词主要划分为3类,其中Dim 1(轴1)和Dim 2(轴2)分别解释了总方差的41.35%和16.38%,累积方差解释率达57.73%。通过对3类中关键词进行细化分析,发现图 4与图 3结果较为一致并相互验证,图 4中聚类1、聚类2和聚类3分别对应图 3中红色、绿色及蓝色部分关键词。聚类1部分研究热点主要是围绕土壤/根际微生物中关于有机质分解及固氮作用功能基因表达、识别及其对植物生长影响方面开展,其中将大肠杆菌作为模式菌株进行研究[31-32,47-48],主要关键词有:植物(plant)、生长(growth)、根际(rhizosphere)、群落(communities)、基因(gene)、基因表达(gene expression)、序列(sequence)、识别(identification)、固氮作用(nitrogen fixation)、大肠杆菌(Escherichia coli)等;聚类2部分研究热点主要包含土壤氮循环过程(如硝化和反硝化作用等)相关功能基因丰度、功能微生物数量与群落结构间关系等方面[13,34,45-46],主要关键词有土壤(soil)、N2O排放(nitrous oxide emissions)、反硝化作用(denitrification)、功能基因(nirK/nosZ genes)、硝化作用(nitrification)、氨氧化古菌/细菌(ammonia oxidizing archaea/bacteria)等;聚类3部分研究热点主要集中于土壤微生物群落、功能多样性、功能基因、酶活性等微生物指标的影响因素(或环境变化因子)方面[37,40,42-43],主要关键词有:土壤类型(森林(forest)和草地(grassland))、气候变化(climate change)、土地利用(land use)、氮肥施用(nitrogen fertilization)、施肥(fertilization)、管理措施(management)、微生物/细菌群落(microbial/bacterial community)、功能基因(functional genes)、酶活性(enzyme activities)、生物多样性(biodiversity)、功能多样性(functional diversity)等。
2.4 历史直接引文通过历史直接引文分析,共得到21篇2001—2020年土壤氮循环功能基因研究领域历史发展过程中的关键文献(图 5和表 4),且发现历史直接引文与表 1和表 2中高被引论文重叠率较高。通过溯源分析发现,其中有6篇文献在土壤氮循环功能基因研究领域发展过程中具有里程碑意义,按照时间序列主要集中于以下几方面:

|
图 5 2001—2020年土壤氮循环功能基因历史直接引文图谱 Fig. 5 A direct citation map of the history of soil nitrogen cycling functional genes from 2001 to 2020 |
|
|
表 4 2001—2020年土壤氮循环功能基因历史直接引文 Table 4 Historical direct citations about soil nitrogen cycling functional gene from 2001 to 2020 |
1)土壤固氮过程研究。2001年,Poly等[31]对固氮作用相关的功能基因nifH分析方法进行了改进,发现不同类型(农田和牧场)土壤中nifH基因多样性显著不同,且土壤固氮菌群落结构受到土壤性质的影响。Patra等[49]在其基础上探索了管理方式与植物种类对草地土壤固氮菌群落、酶活性及功能基因结构的影响。Wakelin等[50]通过测定nifH(固氮)功能基因丰度,从而确定了农艺管理措施(留茬和氮肥施用)对土壤氮循环相关功能微生物群落的影响。Hsu和Buckley[51]在Patra等[49]和Wakelin等[50]的研究基础上探讨了土壤固氮菌群落结构功能的重要性,发现固氮量与土壤性质、固氮菌群落结构紧密相关,而土壤性质主要受耕作管理的影响,固氮量和固氮菌群落结构受生物量管理、耕作及生物量管理交互作用的影响。
2)土壤氮循环多过程研究。2005年,Wallenstein和Vilgalys[52]开发出qPCR技术,通过对5个重要的氮循环相关功能基因(nifH、amoA、nirS、nirK和nosZ)进行定量化分析,来研究土壤氮循环功能微生物群落组成与氮循环过程间的关系。Yoshida等[53]和Stone等[45]在此基础上,分别评估了细菌群落结构和氮周转多过程相关功能基因随时间[53]和土层深度[45]的变化,从而提供土壤微生物在时间和垂直梯度上调控氮循环的新见解。
3)土壤氮循环过程影响因素研究。2007年,Yergeau等[47]利用功能基因微列阵技术,研究了南极纬度梯度上气候变暖对土壤微生物群落的影响,发现反硝化功能基因与土壤温度升高紧密相关,且升温显著提高了土壤真菌和细菌丰度。并验证了功能基因在检测环境样本中功能微生物群落变化方面的实用性,并提供了基因层面上微生物控制养分循环过程的证据。Yoshida、Morales及Yergeau等也在此基础上继续开展了一些相关研究,以深入了解控制土壤养分循环的主要影响因子如时间[53]、土地利用方式及管理措施[54]、气候变暖[47]等,且发现土壤细菌功能基因丰度可以作为温室气体排放量的表征[47]。Fierer等[40]通过运用16S rRNA测序及宏基因组学技术,对不同氮素水平下的土壤微生物群落结构及功能进行对比分析,发现施氮可以直接或间接地诱导形成更活跃的富营养微生物类群。Su等[46]在Fierer等[40]的基础上也探讨了不同施肥处理水稻土中微生物群落组成和功能基因结构的变化,并发现长期施肥不仅显著改变了微生物的整体功能结构,且显著增加了碳氮磷硫循环中大部分基因的多样性和丰度。功能基因结构和丰度、相关土壤酶活性与水稻产量之间存在显著相关关系,表明施肥引起的微生物群落变化可能加速土壤养分周转,进而影响水稻生长。
4)土壤硝化过程研究。2008年,Tourna等[35]通过对氨氧化作用相关的功能基因amoA进行分析,证实了温度对土壤微环境中氨氧化古菌和细菌生长、活性的影响,发现随着温度升高,氨氧化古菌和细菌群落变化极为显著。Gubry-Rangin、Rasche和Daims等也随后进行了相关研究,Gubry-Rangin等[33]通过测定氨氧化过程相关的amoA功能基因丰度,发现土壤氨氧化古菌(而非氨氧化细菌)控制着农业酸性土壤中的硝化作用。Rasche等[55]通过测定nirS和nosZ基因以及氨氧化细菌和古菌amoA基因丰度来评估硝化和反硝化微生物群落变化。
值得注意的是,历史直接引文中LCS排名第一的是Petersen等[13]在2012年发表于Environmental Microbiology期刊上的“Abundance of microbial genes associated with nitrogen cycling as indices of biogeochemical process rates across a vegetation gradient in Alaska”一文(表 4),该研究发现与氮循环相关的微生物功能基因丰度是预测土壤氮循环(如硝化和反硝化)速率的最重要指标,其中硝化速率的最佳预测变量为细菌amoA基因丰度,其次是铵态氮含量;反硝化速率的最佳预测变量为nosZ基因丰度,其次是nirK/nirS基因丰度。Jones等[38]和Philippot等[37]随后开展了反硝化作用相关功能基因方面的研究;而Harter等[34]和Graham等[56]对土壤氮循环多个过程进行了同步研究。Graham等[56]通过测定氮循环过程速率和相关功能基因丰度,利用逐步线性回归来检验土壤性质和功能基因相对丰度对氮循环过程的解释度,从而确定最主要解释变量。
综上所述,2001—2020年土壤氮循环相关功能基因研究领域历史直接引文主要聚焦于:①土壤氮循环过程(如固氮[31,52]、硝化[33-34,36,52]和反硝化[34,38,52]等)相关的功能基因丰度及活性变化及其与土壤性质(环境条件)、微生物群落结构变化间的联系;②土壤氮循环功能基因的影响因素,如土地利用类型[31]、管理方式[50]、植物种类[49]、耕作[51]、留茬[50]、养分投入[41]、施用生物炭[34]、温度(气候变暖)[35,42,47]、pH[33]等;③土壤氮循环过程相关功能基因的筛选、鉴定与识别,如nosZ基因[38]、nifH基因[31]等。
且该研究领域历史发展脉络:早期阶段主要是针对土壤氮循环过程进行相关功能基因筛选、鉴定、识别、设计相应引物及确定分析方法等;随后聚焦于土壤氮循环功能基因的影响因素(或环境条件)方面,尤其是在当前全球气候变暖大背景下,气候变暖/升温(或大气CO2浓度升高)是当前研究热点;此外,关于土壤氮循环功能基因丰度与功能微生物种群、群落结构的关系,也是目前研究前沿热点,尤其是利用如今迅速更新发展的分子生物学技术如宏基因组学和高通量基因芯片技术等,从基因层面上探讨土壤氮循环相关的功能微生物群落结构及驱动机制。
3 结论与展望微生物不同的氮代谢途径和过程显著影响土壤氮循环过程(图 6)[4-5,10,57]。如今分子生物学技术及生物信息学迅猛发展,解析参与不同氮循环过程的关键酶与编码功能基因,并对新功能基因进行筛选、鉴定,完善相应引物设计,进而从微生物功能基因多样性角度深入探讨土壤氮循环过程及驱动机制。与此同时,运用土壤氮循环相关功能基因多样性、丰度来指示土壤功能微生物的生态功能(如提供土壤生态系统服务等)也具有重要实践意义[4,58-59]。

|
注:不同颜色箭头表示土壤中不同氮循环过程,箭头上基因是该周转过程中关键酶的标记功能基因。固氮作用:固氮酶基因nifH;硝化作用:氨单加氧酶基因amoA/amoB,羟胺氧化还原酶基因hao,亚硝酸盐氧化还原酶基因nxrA/nor;反硝化作用:硝酸盐还原酶基因narG,亚硝酸盐还原酶基因nirK/nirS,一氧化氮还原酶基因norB,氧化亚氮还原酶基因nosZ;厌氧氨氧化作用:N2H4合成酶基因hzsA,N2H4氧化还原酶hzo;同化氮还原作用:硝酸盐同化还原酶基因nasA/narB,亚硝酸盐同化还原酶基因nirA/nirB;异化氮还原作用:硝酸盐异化还原酶基因napA,亚硝酸盐异化还原酶基因nrfA;氨化作用:尿素酶基因ureC。 Note:The arrows indicate soil N turnover processes,and the genes on the arrows are the functional genes of coding key enzymes in the N turnover process. Nitrogen fixation:nitrogenase gene nifH. Nitrification:ammonia monooxygenase gene amoA/amoB,hydroxylamine oxidoreductase gene hao,nitrite oxidoreductase gene nxrA/nor. Denitrification:nitrate reductase gene narG,nitrite reductase gene nirK/nirS,nitric oxide reductase gene norB,nitrous oxide reductase gene nosZ. Anaerobic ammonia oxidation:N2H4 synthase gene hzsA,N2H4 oxidoreductase hzo. Assimilatory nitrogen reduction:nitrate assimilation reductase gene nasA/narB,nitrite assimilation reductase gene nirA/nirB;dissimilatory nitrogen reduction:nitrate dissimilation reductase gene napA,nitrite dissimilation reductase gene nrfA. Ammoniation:urease gene ureC. 图 6 土壤氮循环路径及相关功能基因[5,9,30] Fig. 6 The nitrogen cycling pathways and key functional genes[5, 9, 30] |
本研究结果表明,近20年来土壤氮循环功能基因研究主要集中于3个方面:①利用宏基因组学等分子生物学技术对土壤氮循环相关的功能基因进行筛选、识别、注释,从而发现新的微生物功能基因序列、更新引物数据库等;②环境因子及管理措施对土壤氮循环相关微生物指标的影响;③利用功能基因丰度表征土壤氮循环过程相关功能微生物数量、群落结构变化,分析功能基因与土壤性质、微生物群落结构间的关系,以期揭示土壤氮循环过程的分子生物学机制。
以土壤氮循环功能基因为靶基因,是研究土壤样品中氮素转化功能微生物行之有效的手段之一,但受限于土壤氮循环过程及相关功能基因自身的复杂性。随着未来对土壤氮循环的进一步研究及分子生物学技术推陈出新,随着新微生物功能基因序列、引物数据库及氮循环路径的发现与信息更新,对于目前不能扩增的序列或许可以设计出新引物,从而获取更全面精准的功能微生物群落组成、丰度及活性等方面信息,进而推动土壤氮循环相关功能基因领域的研究与发展[60]。
土壤生态系统中涵盖土壤真菌、细菌、古菌、病毒、动物及植物等,其联系极为紧密,而目前发现大量研究聚焦于土壤细菌、真菌多样性,对土壤病毒、动物等的关注及对地上与地下、微生物与植物、动物间整体效应(相互作用)的研究较少。土壤微生物(细菌、古菌、真菌及病毒)互作机制及土壤-微生物-植物系统中三者是否存在协同进化的交互作用及机制亟待进一步研究,从而解析土壤微生物群落与生态功能间的关联机制。
目前大量研究主要关注细菌反硝化、固氮、氨氧化、硝化作用这几个主要氮循环过程,且研究单过程较多,关于厌氧氨氧化、硝酸盐异化还原成铵及真菌反硝化等方面的研究鲜有报道。土壤氮循环需要考虑土壤生态系统的整体效应,因此未来的研究有必要同步关注固氮、硝化、反硝化及硝酸盐异化还原成铵等氮循环过程,从而更精准评估土壤氮循环模式。此外,经典的酶驱动‑地球生态系统模型普遍认为胞外酶,而非特定微生物功能基因的丰度,是微生物介导土壤养分循环的主导因素[61]。另一方面,研究结果表明,微生物功能基因丰度的变化往往与相关的酶活性紧密关联,因此,微生物功能基因变化与酶调节的土壤碳氮动态之间可能存在直接联系[61-63]。Guo等[64]通过模型论证了酶活性与土壤微生物功能基因之间的相关性,发现在经典的微生物模型中,微生物功能基因丰度可以被视为微生物介导碳氮循环的有效替代工具。值得注意的是,功能基因并不能系统量化氮循环周转过程,土壤氮循环不仅仅由微生物功能基因决定,还与相关的酶活性(或功能基因的表达量)以及其他土壤生物和非生物因素紧密关联。因此,通过功能基因丰度直接量化土壤氮转化速率尚存在一定的不确定性,需充分考虑土壤本身的基本性质、环境要素以及具体转化过程[65]。全面考量微生物功能基因和酶活性可能是未来土壤氮循环研究领域行之有效的新途径之一[10]。
环境变化因子能够通过影响土壤氮循环关键酶活性来调控土壤氮循环过程,因此将环境因子与氮循环功能微生物相结合具有重要意义。当前关于土壤氮循环过程相关功能基因及微生物群落受到不同环境变化因子影响的研究报道较多,但全面、系统地研究环境因子如何对参与各氮素转化过程的微生物种群产生影响以及效应大小这方面的报道较少。此外,与土壤氮循环相关的环境因素较多,目前研究大多针对于单一环境因子(如温度、水分、pH等),多环境影响因素间是否存在交互作用及其对土壤氮循环关键过程的影响值得深入探讨、研究[9,66],且量化并确定影响土壤氮循环的主要控制因子具有重要意义。此外,长期观测试验报道较少,有必要增加观测试验周期,明晰环境条件变化对土壤氮循环关键过程的长期影响。很多关于土壤氮循环功能基因研究大多关注功能基因及功能微生物数量的变化,结合微生物群落活性、组成和核心菌群(或关键微生物)等方面综合分析功能微生物对影响因子的响应较少,因此,功能微生物对土壤氮循环的调控机制目前仍不清晰。未来有必要注重分析土壤氮循环功能微生物群落组成、活性、关键类群、群落组装过程等对不同环境因子的综合响应,将氮循环功能微生物群落特征与氮循环过程进行深层次结合研究,深入探究不同环境因子影响土壤氮循环的微生物学机制。
为更好地利用土壤氮转化功能基因来解释土壤氮循环机理,进而服务于中国现代绿色农业发展,未来可以加强以下几个方面的研究:
1)利用稳定性同位素示踪、新一代高通量测序及宏基因组学技术,结合单细胞尺度研究方法(如传统纯菌培养法等),在基因组和转录组水平上筛选、识别土壤氮循环新功能微生物及相应功能基因,明确土壤微生物群落组成、功能信息及丰度较低但发挥关键功能作用的微生物类群,了解关键微生物类群、优势类群和稀有类群在土壤氮循环过程中的作用[60,67-69]。
2)影响土壤氮转化过程功能基因丰度的因素很多,有必要结合室内控制性培养试验,设置不同环境因子梯度,明确各环境因子的阈值范围,这有助于揭示外界环境干扰/胁迫对氮转化过程功能微生物的作用机制及发展氮素有效性及氮通量的定量化模型,改进农田生态系统中氮素管理策略[4,10,70]。
3)土壤氮循环是一个整体过程,应对一系列氮循环功能基因开展系统性研究,而不仅仅是针对单个功能基因(酶活性),这样可以避免造成氮循环功能基因认识的盲区,同时可以反映不同氮循环(如硝化作用、反硝化作用、硝酸盐异化还原成铵、氨化作用等)的效率及贡献。此外,将氮循环功能基因与土壤性质、养分数据相结合,进而预测土壤生态系统功能,有利于更深刻地揭示土壤氮素周转过程的微生物作用机理以及环境因子变化对其产生的影响,也可以深入地阐释土壤氮素周转内在机制[71]。
4)从细胞尺度、田块尺度(或实验室控制/培养试验)拓展延伸到区域/全球尺度,利用土壤微生物生物地理学理论和方法,解析不同时空尺度下土壤微生物分布规律、格局及群落组装机制,进而对土壤氮循环机制进行解译[72-74]。
5)在当前全球气候变化大背景下,N2O作为强效温室气体,关注土壤氮循环相关微生物对气候变化的响应过程,以及明确气候变化对土壤微生物的影响过程及机制,同时加强实用技术研发、推广,如保护性耕作(包括秸秆还田、免耕、有机肥施用等)、硝化抑制剂、增效控释肥等管理措施及技术方法,进而提出高效可用的温室气体减排措施,实现中国农业可持续发展、碳达峰及碳中和[75-76]。
| [1] |
Robertson G P, Vitousek P M. Nitrogen in agriculture: Balancing the cost of an essential resource[J]. Annual Review of Environment and Resources, 2009, 34(1): 97-125. DOI:10.1146/annurev.environ.032108.105046
(  0) 0) |
| [2] |
Gruber N, Galloway J N. An Earth-system perspective of the global nitrogen cycle[J]. Nature, 2008, 451(7176): 293-296. DOI:10.1038/nature06592
(  0) 0) |
| [3] |
Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9
(  0) 0) |
| [4] |
Ouyang Y, Evans S E, Friesen M L, et al. Effect of nitrogen fertilization on the abundance of nitrogen cycling genes in agricultural soils: A meta-analysis of field studies[J]. Soil Biology & Biochemistry, 2018, 127: 71-78.
(  0) 0) |
| [5] |
Levy-Booth D J, Prescott C E, Grayston S J. Microbial functional genes involved in nitrogen fixation, nitrification and denitrification in forest ecosystems[J]. Soil Biology & Biochemistry, 2014, 75: 11-25.
(  0) 0) |
| [6] |
He Z L, Gentry T J, Schadt C W, et al. GeoChip: a comprehensive microarray for investigating biogeochemical, ecological and environmental processes[J]. The ISME Journal, 2007, 1(1): 67-77. DOI:10.1038/ismej.2007.2
(  0) 0) |
| [7] |
Li P, Jiang Z, Wang Y H, et al. Analysis of the functional gene structure and metabolic potential of microbial community in high arsenic groundwater[J]. Water Research, 2017, 123: 268-276. DOI:10.1016/j.watres.2017.06.053
(  0) 0) |
| [8] |
Zhou J Z, He Z L, Yang Y F, et al. High-throughput metagenomic technologies for complex microbial community analysis: Open and closed formats[J]. mBio, 2015, 6(1): e02288-14.
(  0) 0) |
| [9] |
He Z L, Xu M Y, Deng Y, et al. Metagenomic analysis reveals a marked divergence in the structure of belowground microbial communities at elevated CO2[J]. Ecology Letters, 2010, 13(5): 564-575. DOI:10.1111/j.1461-0248.2010.01453.x
(  0) 0) |
| [10] |
Ouyang Y, Reeve J R, Norton J M. Soil enzyme activities and abundance of microbial functional genes involved in nitrogen transformations in an organic farming system[J]. Biology and Fertility of Soils, 2018, 54(4): 437-450. DOI:10.1007/s00374-018-1272-y
(  0) 0) |
| [11] |
Li X M, Qiao J T, Li S, et al. Bacterial communities and functional genes stimulated during anaerobic arsenite oxidation and nitrate reduction in a paddy soil[J]. Environmental Science & Technology, 2020, 54(4): 2172-2181.
(  0) 0) |
| [12] |
Hou H J, Qin H L, Chen C L, et al. Research progress of the molecular ecology on microbiological processes in soil nitrogen cycling (In Chinese)[J]. Research of Agricultural Modernization, 2014, 35(5): 588-594. DOI:10.13872/j.1000-0275.2014.0032 [侯海军, 秦红灵, 陈春兰, 等. 土壤氮循环微生物过程的分子生态学研究进展[J]. 农业现代化研究, 2014, 35(5): 588-594.]
(  0) 0) |
| [13] |
Petersen D G, Blazewicz S J, Firestone M, et al. Abundance of microbial genes associated with nitrogen cycling as indices of biogeochemical process rates across a vegetation gradient in Alaska[J]. Environmental Microbiology, 2012, 14(4): 993-1008. DOI:10.1111/j.1462-2920.2011.02679.x
(  0) 0) |
| [14] |
Aria M, Cuccurullo C. Bibliometrix: An R-tool for comprehensive science mapping analysis[J]. Journal of Informetrics, 2017, 11(4): 959-975. DOI:10.1016/j.joi.2017.08.007
(  0) 0) |
| [15] |
Shi C, Qu L Q, Zhang Q W, et al. A systematic review on comprehensive sloping farmland utilization based on a perspective of scientometrics analysis[J]. Agricultural Water Management, 2021, 244: 106564. DOI:10.1016/j.agwat.2020.106564
(  0) 0) |
| [16] |
Padilla F M, Gallardo M, Manzano-Agugliaro F. Global trends in nitrate leaching research in the 1960-2017 period[J]. Science of the Total Environment, 2018, 643: 400-413. DOI:10.1016/j.scitotenv.2018.06.215
(  0) 0) |
| [17] |
Liu Y N, Wu K N, Zhao R. Bibliometric analysis of research on soil health from 1999 to 2018[J]. Journal of Soils and Sediments, 2020, 20(3): 1513-1525. DOI:10.1007/s11368-019-02519-9
(  0) 0) |
| [18] |
Zhang Y, Pu S Y, Lv X, et al. Global trends and prospects in microplastics research: A bibliometric analysis[J]. Journal of Hazardous Materials, 2020, 400: 123110. DOI:10.1016/j.jhazmat.2020.123110
(  0) 0) |
| [19] |
Mao G Z, Hu H Q, Liu X, et al. A bibliometric analysis of industrial wastewater treatments from 1998 to 2019[J]. Environmental Pollution, 2021, 275: 115785. DOI:10.1016/j.envpol.2020.115785
(  0) 0) |
| [20] |
Mao G Z, Huang N, Chen L, et al. Research on biomass energy and environment from the past to the future: A bibliometric analysis[J]. Science of the Total Environment, 2018, 635: 1081-1090. DOI:10.1016/j.scitotenv.2018.04.173
(  0) 0) |
| [21] |
Liu X M, Zhao J, Xu J M. Research on immobilization of heavy metals in contaminated agricultural soils-Bibliometric analysis based on Web of Science database (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(2): 445-455. [刘杏梅, 赵健, 徐建明. 污染农田土壤的重金属钝化技术研究——基于Web of Science数据库的计量分析[J]. 土壤学报, 2021, 58(2): 445-455.]
(  0) 0) |
| [22] |
Chen R R, Zhu Y, Cao W X, et al. A bibliometric analysis of research on plant critical dilution curve conducted between 1985 and 2019[J]. European Journal of Agronomy, 2021, 123: 126199. DOI:10.1016/j.eja.2020.126199
(  0) 0) |
| [23] |
Li T, Cui L Z, Xu Z H, et al. Quantitative analysis of the research trends and areas in grassland remote sensing: A scientometrics analysis of Web of Science from 1980 to 2020[J]. Remote Sensing, 2021, 13(7): 1279. DOI:10.3390/rs13071279
(  0) 0) |
| [24] |
The R Development Core Team. R: A language and environment for statistical computing[EB/OL]. https://www.R-project.org.
(  0) 0) |
| [25] |
Wu H Q, Zhang B G, Li Q, et al. Bibliometric analysis for factors of influencing agricultural soil N2O emission based on Web of Science (In Chinese)[J]. Chinese Journal of Soil Science, 2021, 52(1): 221-232. [吴汉卿, 张宝贵, 李强, 等. 基于Web of Science对农业土壤N2O排放影响因素研究的文献计量分析[J]. 土壤通报, 2021, 52(1): 221-232.]
(  0) 0) |
| [26] |
Wu H Q, Zhang B G, Wang X X, et al. The method of literature retrieval via biliometric analysis: Taking soil ammonia volatilization as an example (In Chinese)[J]. Chinese Agricultural Science Bulletin, 2021, 37(1): 147-157. [吴汉卿, 张宝贵, 王学霞, 等. 文献计量分析在快速检索文献中的应用——以土壤氨挥发为例[J]. 中国农学通报, 2021, 37(1): 147-157.]
(  0) 0) |
| [27] |
Opsahl T, Agneessens F, Skvoretz J. Node centrality in weighted networks: Generalizing degree and shortest paths[J]. Social Networks, 2010, 32(3): 245-251. DOI:10.1016/j.socnet.2010.03.006
(  0) 0) |
| [28] |
Cuccurullo C, Aria M, Sarto F. Foundations and trends in performance management. A twenty-five years bibliometric analysis in business and public administration domains[J]. Scientometrics, 2016, 108(2): 595-611. DOI:10.1007/s11192-016-1948-8
(  0) 0) |
| [29] |
Garfield E. Historiographic mapping of knowledge domains literature[J]. Journal of Information Science, 2004, 30(2): 119-145. DOI:10.1177/0165551504042802
(  0) 0) |
| [30] |
Wang Z J, Wang S, Liu Y Y, et al. The applications of metagenomics in the detection of environmental microbes involving in nitrogen cycle (In Chinese)[J]. Biotechnology Bulletin, 2018, 34(1): 1-14. DOI:10.13560/j.cnki.biotech.bull.1985.2018-0024 [王朱珺, 王尚, 刘洋荧, 等. 宏基因组技术在氮循环功能微生物分子检测研究中的应用[J]. 生物技术通报, 2018, 34(1): 1-14.]
(  0) 0) |
| [31] |
Poly F, Monrozier L J, Bally R. Improvement in the RFLP procedure for studying the diversity of nifH genes in communities of nitrogen fixers in soil[J]. Research in Microbiology, 2001, 152(1): 95-103. DOI:10.1016/S0923-2508(00)01172-4
(  0) 0) |
| [32] |
Young J P W, Crossman L C, Johnston A W B, et al. The genome of Rhizobium leguminosarum has recognizable core and accessory components[J]. Genome Biology, 2006, 7(4): R34. DOI:10.1186/gb-2006-7-4-r34
(  0) 0) |
| [33] |
Gubry-Rangin C, Nicol G W, Prosser J I. Archaea rather than bacteria control nitrification in two agricultural acidic soils[J]. FEMS Microbiology Ecology, 2010, 74(3): 566-574. DOI:10.1111/j.1574-6941.2010.00971.x
(  0) 0) |
| [34] |
Harter J, Krause H M, Schuettler S, et al. Linking N2O emissions from biochar-amended soil to the structure and function of the N-cycling microbial community[J]. The ISME Journal, 2014, 8(3): 660-674. DOI:10.1038/ismej.2013.160
(  0) 0) |
| [35] |
Tourna M, Freitag T E, Nicol G W, et al. Growth, activity and temperature responses of ammonia-oxidizing Archaea and bacteria in soil microcosms[J]. Environmental Microbiology, 2008, 10(5): 1357-1364. DOI:10.1111/j.1462-2920.2007.01563.x
(  0) 0) |
| [36] |
Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461
(  0) 0) |
| [37] |
Philippot L, Spor A, Hénault C, et al. Loss in microbial diversity affects nitrogen cycling in soil[J]. The ISME Journal, 2013, 7(8): 1609-1619. DOI:10.1038/ismej.2013.34
(  0) 0) |
| [38] |
Jones C M, Graf D R H, Bru D, et al. The unaccounted yet abundant nitrous oxide-reducing microbial community: A potential nitrous oxide sink[J]. The ISME Journal, 2013, 7(2): 417-426. DOI:10.1038/ismej.2012.125
(  0) 0) |
| [39] |
Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616. DOI:10.1038/nature13377
(  0) 0) |
| [40] |
Fierer N, Lauber C L, Ramirez K S, et al. Comparative metagenomic, phylogenetic and physiological analyses of soil microbial communities across nitrogen gradients[J]. The ISME Journal, 2012, 6(5): 1007-1017. DOI:10.1038/ismej.2011.159
(  0) 0) |
| [41] |
Leff J W, Jones S E, Prober S M, et al. Consistent responses of soil microbial communities to elevated nutrient inputs in grasslands across the globe[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(35): 10967-10972. DOI:10.1073/pnas.1508382112
(  0) 0) |
| [42] |
Yergeau E, Bokhorst S, Kang S, et al. Shifts in soil microorganisms in response to warming are consistent across a range of Antarctic environments[J]. The ISME Journal, 2012, 6(3): 692-702. DOI:10.1038/ismej.2011.124
(  0) 0) |
| [43] |
Zhou J Z, Deng Y, Luo F, et al. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2[J]. mBio, 2011, 2(4): e00122-11.
(  0) 0) |
| [44] |
Mackelprang R, Waldrop M P, DeAngelis K M, et al. Metagenomic analysis of a permafrost microbial community reveals a rapid response to thaw[J]. Nature, 2011, 480(7377): 368-371. DOI:10.1038/nature10576
(  0) 0) |
| [45] |
Stone M M, Kan J J, Plante A F. Parent material and vegetation influence bacterial community structure and nitrogen functional genes along deep tropical soil profiles at the Luquillo Critical Zone Observatory[J]. Soil Biology & Biochemistry, 2015, 80: 273-282.
(  0) 0) |
| [46] |
Su J Q, Ding L J, Xue K, et al. Long-term balanced fertilization increases the soil microbial functional diversity in a phosphorus-limited paddy soil[J]. Molecular Ecology, 2015, 24(1): 136-150. DOI:10.1111/mec.13010
(  0) 0) |
| [47] |
Yergeau E, Kang S, He Z L, et al. Functional microarray analysis of nitrogen and carbon cycling genes across an Antarctic latitudinal transect[J]. The ISME Journal, 2007, 1(2): 163-179. DOI:10.1038/ismej.2007.24
(  0) 0) |
| [48] |
Liu F T, Kou D, Chen Y L, et al. Altered microbial structure and function after thermokarst formation[J]. Global Change Biology, 2021, 27(4): 823-835. DOI:10.1111/gcb.15438
(  0) 0) |
| [49] |
Patra A K, Abbadie L, Clays-Josserand A, et al. Effects of management regime and plant species on the enzyme activity and genetic structure of N-fixing, denitrifying and nitrifying bacterial communities in grassland soils[J]. Environmental Microbiology, 2006, 8(6): 1005-1016. DOI:10.1111/j.1462-2920.2006.00992.x
(  0) 0) |
| [50] |
Wakelin S A, Colloff M J, Harvey P R, et al. The effects of stubble retention and nitrogen application on soil microbial community structure and functional gene abundance under irrigated maize[J]. FEMS Microbiology Ecology, 2007, 59(3): 661-670. DOI:10.1111/j.1574-6941.2006.00235.x
(  0) 0) |
| [51] |
Hsu S F, Buckley D H. Evidence for the functional significance of diazotroph community structure in soil[J]. The ISME Journal, 2009, 3(1): 124-136. DOI:10.1038/ismej.2008.82
(  0) 0) |
| [52] |
Wallenstein M D, Vilgalys R J. Quantitative analyses of nitrogen cycling genes in soils[J]. Pedobiologia, 2005, 49(6): 665-672. DOI:10.1016/j.pedobi.2005.05.005
(  0) 0) |
| [53] |
Yoshida M, Ishii S, Otsuka S, et al. Temporal shifts in diversity and quantity of nirS and nirK in a rice paddy field soil[J]. Soil Biology & Biochemistry, 2009, 41(10): 2044-2051.
(  0) 0) |
| [54] |
Morales S E, Cosart T, Holben W E. Bacterial gene abundances as indicators of greenhouse gas emission in soils[J]. The ISME Journal, 2010, 4(6): 799-808. DOI:10.1038/ismej.2010.8
(  0) 0) |
| [55] |
Rasche F, Knapp D, Kaiser C, et al. Seasonality and resource availability control bacterial and archaeal communities in soils of a temperate beech forest[J]. The ISME Journal, 2011, 5(3): 389-402. DOI:10.1038/ismej.2010.138
(  0) 0) |
| [56] |
Graham E B, Wieder W R, Leff J W, et al. Do we need to understand microbial communities to predict ecosystem function? A comparison of statistical models of nitrogen cycling processes[J]. Soil Biology & Biochemistry, 2014, 68: 279-282.
(  0) 0) |
| [57] |
Liu Y Y, Wang S, Li S Z, et al. Advances in molecular ecology on microbial functional genes of carbon cycle (In Chinese)[J]. Microbiology China, 2017, 44(7): 1676-1689. DOI:10.13344/j.microbiol.china.160941 [刘洋荧, 王尚, 厉舒祯, 等. 基于功能基因的微生物碳循环分子生态学研究进展[J]. 微生物学通报, 2017, 44(7): 1676-1689.]
(  0) 0) |
| [58] |
Zhang J, Lin X G, Yin R. Advances in functional gene diversity of microorganism in relation to soil nitrogen cycling (In Chinese)[J]. Chinese Journal of Eco-Agriculture, 2009, 17(5): 1029-1034. [张晶, 林先贵, 尹睿. 参与土壤氮素循环的微生物功能基因多样性研究进展[J]. 中国生态农业学报, 2009, 17(5): 1029-1034.]
(  0) 0) |
| [59] |
Zhang J, Zhang H W, Li X Y, et al. Soil microbial ecological process and microbial functional gene diversity (In Chinese)[J]. Chinese Journal of Applied Ecology, 2006, 17(6): 1129-1132. DOI:10.3321/j.issn:1001-9332.2006.06.034 [张晶, 张惠文, 李新宇, 等. 土壤微生物生态过程与微生物功能基因多样性[J]. 应用生态学报, 2006, 17(6): 1129-1132.]
(  0) 0) |
| [60] |
Ji H F, Wang Y. Advances in molecular approach applications in microbial ecology studies (In Chinese)[J]. Acta Ecologica Sinica, 2016, 36(24): 8234-8243. [姬洪飞, 王颖. 分子生物学方法在环境微生物生态学中的应用研究进展[J]. 生态学报, 2016, 36(24): 8234-8243.]
(  0) 0) |
| [61] |
Chen J, Sinsabaugh R L. Linking microbial functional gene abundance and soil extracellular enzyme activity: Implications for soil carbon dynamics[J]. Global Change Biology, 2021, 27(7): 1322-1325. DOI:10.1111/gcb.15506
(  0) 0) |
| [62] |
Chen J, Luo Y Q, van Groenigen K J, et al. A keystone microbial enzyme for nitrogen control of soil carbon storage[J]. Science Advances, 2018, 4(8): eaaq1689. DOI:10.1126/sciadv.aaq1689
(  0) 0) |
| [63] |
Moore J A M, Anthony M A, Pec G J, et al. Fungal community structure and function shifts with atmospheric nitrogen deposition[J]. Global Change Biology, 2021, 27(7): 1349-1364. DOI:10.1111/gcb.15444
(  0) 0) |
| [64] |
Guo X, Gao Q, Yuan M, et al. Gene-informed decomposition model predicts lower soil carbon loss due to persistent microbial adaptation to warming[J]. Nature Communications, 2020, 11: 4897. DOI:10.1038/s41467-020-18706-z
(  0) 0) |
| [65] |
Li W J, Cai Y J, Zhu T B, et al. Release of nitrous oxide from soil aggregates and its microbial mechanism (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(5): 1132-1144. [李文娟, 蔡延江, 朱同彬, 等. 土壤团聚体氧化亚氮排放及其微生物学机制研究进展[J]. 土壤学报, 2021, 58(5): 1132-1144.]
(  0) 0) |
| [66] |
Rillig M C, Ryo M, Lehmann A, et al. The role of multiple global change factors in driving soil functions and microbial biodiversity[J]. Science, 2019, 366(6467): 886-890. DOI:10.1126/science.aay2832
(  0) 0) |
| [67] |
Chen Q L, Ding J, Zhu D, et al. Rare microbial taxa as the major drivers of ecosystem multifunctionality in long-term fertilized soils[J]. Soil Biology & Biochemistry, 2020, 141: 107686.
(  0) 0) |
| [68] |
Xiong C, He J Z, Singh B K, et al. Rare taxa maintain the stability of crop mycobiomes and ecosystem functions[J]. Environmental Microbiology, 2021, 23(4): 1907-1924. DOI:10.1111/1462-2920.15262
(  0) 0) |
| [69] |
Jiao S, Xu Y Q, Zhang J, et al. Core microbiota in agricultural soils and their potential associations with nutrient cycling[J]. mSystems, 2019, 4(2): e00313-18.
(  0) 0) |
| [70] |
Li Y Q, Ma J W, Yu Y, et al. Effects of multiple global change factors on soil microbial richness, diversity and functional gene abundances: A meta-analysis[J]. Science of the Total Environment, 2022, 815: 152737. DOI:10.1016/j.scitotenv.2021.152737
(  0) 0) |
| [71] |
Qin H L, Chen A L, Sheng R, et al. A review on the microbial regulation mechanism of N2O production and emission of rice paddy ecosystems (In Chinese)[J]. Research of Agricultural Modernization, 2018, 39(6): 922-929. [秦红灵, 陈安磊, 盛荣, 等. 稻田生态系统氧化亚氮(N2O)排放微生物调控机制研究进展及展望[J]. 农业现代化研究, 2018, 39(6): 922-929.]
(  0) 0) |
| [72] |
Jiao S, Lu Y H. Abundant fungi adapt to broader environmental gradients than rare fungi in agricultural fields[J]. Global Change Biology, 2020, 26(8): 4506-4520. DOI:10.1111/gcb.15130
(  0) 0) |
| [73] |
Deng Y L, Ruan Y J, Ma B, et al. Multi-omics analysis reveals niche and fitness differences in typical denitrification microbial aggregations[J]. Environment International, 2019, 132: 105085. DOI:10.1016/j.envint.2019.105085
(  0) 0) |
| [74] |
Chu H Y, Gao G F, Ma Y Y, et al. Soil microbial biogeography in a changing world: Recent advances and future perspectives[J]. mSystems, 2020, 5(2): e00803-19.
(  0) 0) |
| [75] |
Liu Z, Deng Z, He G, et al. Challenges and opportunities for carbon neutrality in China[J]. Nature Reviews Earth & Environment, 2022, 3(2): 141-155.
(  0) 0) |
| [76] |
Wang F, Harindintwali J D, Yuan Z Z, et al. Technologies and perspectives for achieving carbon neutrality[J]. The Innovation, 2021, 2(4): 100180. DOI:10.1016/j.xinn.2021.100180
(  0) 0) |
 2023, Vol. 60
2023, Vol. 60


