2. 中国科学院地理科学与资源研究所, 生态网络观测与模拟重点实验室, 北京 100101
2. Key Laboratory of Ecosystem Network Observation and Modeling /Institute of Geographic Sciences and Natural Resources Research, Chinese Academy of Sciences, Beijing 100101, China
土壤微生物多样性或群落结构在不同距离尺度或环境梯度下的分布格局和机制是近年来生态学及生物地理学中的关键议题之一[1]。海拔高度的变化能够在相对小的空间距离内产生气候、植被、土壤等一系列环境因子的综合变化[2],并显著影响土壤微生物的分布格局[3-4]。因此,研究微生物海拔分布格局有助于促进微生物生态学理论的发展及全球气候变化影响预测[5]。真菌是土壤生态系统的重要组成部分,是凋落物降解、养分周转循环等多种生态系统过程或功能的核心介导及驱动者[6-7]。目前关于真菌多样性沿海拔梯度分布特征的研究结果不一,真菌丰富度或多样性随海拔的升高而表现出降低[8-10]、单峰或U型变化模式[11-12]以及无明显的海拔分布特征[13-15]。总体而言,自然生态系统中土壤真菌具有十分复杂的生物地理分布格局,这通常归因于真菌与植物密切的关系(共生/寄生),植物群落的组成和多样性对土壤真菌的分布具有显著的影响[16-17]。此外,气候、土壤等非生物因素在全球尺度上也被证明是决定真菌生物地理分布格局的关键因子。
明确土壤真菌多样性的形成和维持(即群落构建)的机理是理解真菌生物地理分布格局形成的关键。目前关于群落构建的机制理论主要有两种不同的观点:一种认为物种多样性的形成和维持是基于扩散和随机作用的生态过程,即中性理论,假设群落结构独立于物种特征,受随机过程(如出生、死亡、物种形成、灭绝)控制;另一种观点则强调共存物种间的生态位分化等确定性因素,即生态位理论,认为环境条件、物种间相互作用(如竞争、共生)等决定性因素控制着群落组成[18]。物种的生态位特征,包括生态位宽度、生态位重叠等,是影响环境选择、物种竞争等对作用于群落组成特征的关键性状[19]。并且,物种的生境或者环境生态位宽度可以作为其应对气候变化敏感性的重要指标[20]。根据生态位理论,生态位较窄的种群(分布范围有限)在环境变化压力下面临着更高的灭绝风险,而生态位较宽(分布广泛)的种群则能表现出对环境变化的更高适应性。目前已有研究揭示了海拔梯度对物种生态位宽度的影响,但是主要集中在植物、动物、昆虫等生物类群。例如,Schellenberger Costa等[21]在乞力马扎罗山西坡的研究发现植物生态位宽度随海拔高度、降雨量以及干扰程度的上升而增加,而Ahmad等[22]在喜马拉雅山脉西侧的研究则发现非本土植物的生态位宽度随海拔上升而降低。对传粉昆虫的研究发现,由于高海拔地区可选择的开花植物种类有限,传粉昆虫的生态位宽度相比于低海拔地区显著增加[23]。尽管生态位宽度的信息对于预测生物应对环境变化的响应十分必要,关于微生物生态位宽度的研究却非常有限,微生物生态位宽度随海拔梯度的变化特征尚未明确。
青藏高原高寒生态系统对外界环境变化和人为扰动十分敏感,对我国乃至全球的气候变化和调控均至关重要。因此,研究青藏高原土壤微生物多样性及群落组成的海拔分布模式,有助于深入理解典型高寒生态系统下土壤微生物群落构建机制及全球变化影响预测。色季拉山是青藏高原东南地区极具代表性的高山生态系统,其植被具有典型的垂直分布特征[24],随海拔升高由温带森林过渡至高寒荒漠。目前对色季拉山土壤微生物生物量、多样性及群落组成的海拔分布格局已有一些报道。例如,Xu等[24]使用磷脂脂肪酸(Phospholipid fatty acid,PLFA)谱图和生态微平板法(Biolog Ecoplate)分析发现色季拉山主要土壤微生物类群的生物量没有表现出随海拔变化的分布特征,但是碳源利用活性随海拔升高而显著降低。利用高通量测序分析,Wang等[12,25]报道了色季拉山森林生态系统中土壤细菌和真菌的多样性具有不同的海拔分布模式。但是,色季拉山土壤真菌的群落组成的影响机制目前尚未明确。相较于表层土壤(0~20 cm),底层土壤贮藏了更多的有机碳,是全球碳循环的重要组分[26],然而目前对土壤微生物多样性、群落组成及功能的研究多集中在表层土壤[27]。最近研究发现,色季拉山暗针叶林表层和底层土壤微生物群落特征具有显著差异,并且物种间相互作用关系随土壤深度变化发生改变[28],这暗示深层土壤中微生物群落多样性的分布格局和物种共存机制与表层土壤可能存在显著差异。因此,本研究在色季拉山不同海拔梯度采集表层(0~20 cm)和底层(40~60 cm)土壤样品,基于ITS rDNA高通量测序和分子生态学方法,分析真菌群落组成及其影响因素和生态位特征,揭示色季拉山土壤真菌群落组成的海拔分布模式及其影响机制,以及其在表层和底层土壤的异同,有助于深入了解真菌多样性在高寒生态系统土壤中的形成和维持机制。
1 材料与方法 1.1 研究区概况色季拉山位于西藏林芝市八一区境内(29°5′—29°57′ N,94°25′—94°45′ E),主峰高度5 200 m,处于藏东南湿润气候区与半湿润气候区的过渡地带,年平均气温为–0.73℃,年平均降水量为1 134 mm。西坡自最低海拔至最高处具有5个植被带,分别为(1)山地温带针阔混交林带(3 000~3 500 m),主要为高山松(Pinus densata)、西藏红杉(Larix grifithiana)、川滇高山栎(Quercus aquifolioides)等;(2)亚高山寒温带-湿暗针叶林带(3 500~4 200 m),优势种为林芝云杉(Picea likiangensis var. linzhensis)和急尖长苞冷杉(Abies georgei var. smithii);(3)高山亚寒带-疏林、灌丛、草甸带(4 200~4 500 m),优势灌木为方枝柏(Sabina saltuaila)、雪层杜鹃(Rhododendron hiale)和林芝杜鹃(Rhododendron nyingchiense)等;(4)高山寒带草甸带(4 500~4 700 m),优势物种为散鳞杜鹃(Rhododendron bulu)、苔草(Carex sp.)、蓼(Polygonum spp.)等;(5)高山荒漠带(海拔4 700 m以上),零星分布有岩须(Cassiope selaginodes)、塔黄(Rheum nobile)和红景天(Rhodiola spp.)。
1.2 土壤样品采集与处理本研究于2018年在色季拉山西坡海拔3 000~4 600 m范围内选取了6个不同的采样海拔(表 1),根据各海拔的植被类型可分为低海拔区间(3 356~3 689 m,植被类型为森林)和高海拔区间(4 284~4 590 m,植被类型为高寒灌丛草甸)。在每个采样海拔,沿水平方向随机选取4个采样点,每个采样点间隔不小于100 m。在每个采样点设置3个1 m× 1 m样方,先去除土壤表面的凋落物,再用五点法(样方四角和中心点)用土钻采集表层(0~20 cm)和底层(40~60 cm)土壤样品,每个采样点采集的土样混合成一个样品(重复),共获得24个表层土样和24个底层土样。采集的土壤样品装入密封口袋中,保存于冰盒里运输回实验室进行后续分析。土壤样品挑出可见的石块、土壤动物和植物残体后过筛(2 mm),均匀分取一部分样品用于DNA提取(–20℃下保存),其余土壤样品风干后研磨用于测定土壤基础理化指标。
|
|
表 1 不同土壤深度下土壤理化性质的海拔变化特征 Table 1 Variations in soil physical and chemical properties with elevation at two soil depths |
土壤pH用1 mol·L–1 KCl浸提(土水比为1:2.5)并用电位法测定。土壤有机碳含量使用重铬酸钾容量法-外加热法测定,土壤全氮含量使用元素分析仪测定(EA1108,CarloErba,Turin,Italy)。土壤湿度用质量含水量表征,即通过测定新鲜土壤样品在105℃下烘干前后的质量差获得。颗粒组成采用激光粒度分析仪(Longbench Mastersizer 2000)进行测定。
1.4 高通量测序和分析土壤DNA的提取使用PowerSoil DNA提取试剂盒(MoBio Laboratories Inc.,Carlsbad,CA,USA),并依照试剂盒说明书上的详细步骤进行提取。提取的DNA使用NanoDrop ND-1000分光光度计(ThermoScientific,Wilmington,DE,USA)测定质量,之后于–80℃保存。使用1737F(5′-GGAAGT AAAAGTCGTAACAAGG-3′)和2043R(5′-GCTGCG TTCTTCATCGATGC-3′)扩增真菌ITS基因,基因扩增产物送至上海美吉生物医药有限公司进行高通量测序(Illumina MiSeq platform)。测序获得的原始序列保存为FASTQ格式,使用Quantitative Insights Into Microbial Ecology(QIIME)平台对序列进行优化和数据质控,之后使用Usearch软件平台(version7.0,http://drive5.com/uparse/),依照97%的相似度划分OTU(Operational taxonomic units)。为了得到每个OTU对应的物种分类信息,采用RDP classifier(version 2.2,http://sourceforge.net/projects/rdp-classifier/)贝叶斯算法对OTU代表序列进行分类学分析,对比数据库为UNITE真菌ITS数据库(Release 6.0,http://unite.ut.ee/index.php)。根据每个样本测序量的总体概况,使用最小测序量(10 466)进行抽平后再进行多样性指数分析。真菌的α-多样性指数用丰富度(Chao1 Index)和香农多样性(Shannon Index)表征,使用Mothur软件(version v.1.30.1,http://www.mothur.org/wiki/Schloss_SOP# Alpha_diversity)计算得到。真菌功能群使用FUNGuild功能预测分析对真菌ITS基因进行功能注释[29]。
1.5 数据分析图表中数据表征为平均值±标准误差。真菌群落组成沿不同海拔梯度的差异(β多样性)基于Bray-Curtis距离进行计算,不同海拔梯度或土壤深度下真菌群落组成的差异用相似性分析(Analysis of similarity,ANOSIM)进行检验。真菌β多样性与海拔距离及环境因子梯度(基于Euclidean距离计算)的Pearson相关性用Mantel检验进行分析。使用基于距离矩阵的冗余分析(Distance-based redundancy analysis,db-RDA)分析环境因子对不同土层真菌群落组成沿海拔梯度变异的影响,db-RDA之前先用膨胀因子分析对环境因子进行筛选,剔除共线性高的因子。上述分析均使用R软件vegan包进行。
真菌种群的生态位特征,包括Levins生态位宽度(Niche breadth,B)指数[30]和Pianka生态位重叠值(Oik)[31]根据下式进行计算:
| $B_j=1 / \sum _{i=1}^N P_{i j}^2 $ | (1) |
| $ {O_{ik}} = \frac{{\sum\nolimits_{j = 1}^N {\left( {{P_{ij}} \times {P_{kj}}} \right)} }}{{\sqrt {\sum\nolimits_{j = 1}^N {P_{ij}^2 \times \sum\nolimits_{j = 1}^N {P_{kj}^2} } } }} $ | (2) |
式中,Bj为真菌群落中OTUj的生境生态位宽度,N为样点的总数;Pij为OTUj在样点i的群落中占据的比例。如果某个OTU在某一生境中(例如某海拔)具有较高的B值,则表明该种的生境生态位宽度较宽。在每个海拔,每个采集的土壤样品的真菌群落中出现的所有OTU的B值的平均值即为该海拔下群落水平的生境生态位宽度(Bcom)。生态位重叠是指不同种生物对同一生态位的共享或对同一资源的共同利用的现象,生态位重叠值反映了物种间对生境适应和资源利用的相似程度[30]。根据高斯竞争排斥原理,生态位重叠值越大,种群间的竞争越激烈;相反重叠值越小,种群间对资源或生境的分享更加协调和平衡。生态位宽度和生态位重叠值均使用R软件spaa包进行计算。
2 结果 2.1 土壤基础理化性质海拔梯度及土壤深度显著影响色季拉山西坡土壤基础理化性质(表 1)。随着海拔升高,表层(0~20 cm)与底层(40~60 cm)土壤中的有机碳、总氮含量及土壤含水量均显著增加,并且在表层土壤显著高于底层土壤。相反的,土壤pH随海拔升高显著下降,并且在底层土壤显著高于表层土壤。土壤碳氮比在表层土壤随海拔升高而显著降低,在底层土壤则呈现先增高后降低的特征。随着海拔升高,土壤砂粒含量在表层和底层土壤均显著降低,而土壤粉粒和黏粒含量均显著增加。土壤深度对土壤颗粒组成没有显著影响。
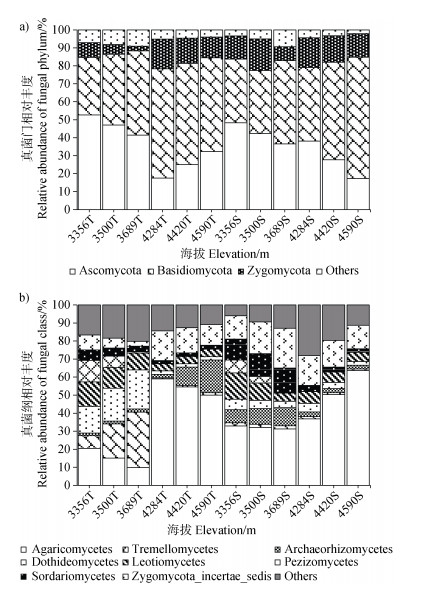
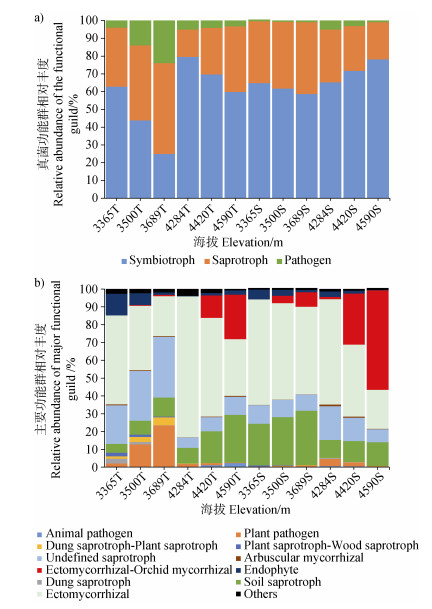
2.2 真菌群落组成与多样性本研究采集的土壤样品经高通量测序共检测到真菌序列622 797条,按照97%序列相似性水平共鉴定出2 879个OTU。经过分类学分析,共鉴定出子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)、球囊菌门(Glomeromycota)、壶菌门(Chytridiomycota)5个真菌门,包括23个纲、80个目、169个科、316个属和501个种。子囊菌门、担子菌门和接合菌门是优势种群,三者相对丰度之和在不同采样海拔和土壤深度均达到90%以上(图 1)。真菌优势门和纲的相对丰度显著受到海拔梯度和土壤深度的影响,且不同真菌类群存在明显异同。无论是表层还是底层土壤,子囊菌门的相对丰度在低海拔区间(3 356~3 689 m)均显著高于高海拔区间(4 284~4 590 m)。子囊菌门下的优势纲中,座囊菌纲(Dothideomycetes)、锤舌菌纲(Leotiomycetes)、盘菌纲(Pezizomycetes)及粪壳菌纲(Sordariomycetes)均表现出在低海拔富集的特征,而古根菌纲(Archaeorhizomycetes)的海拔变化则因土层而异。与子囊菌门相反,担子菌门的相对丰度随海拔升高而增加;其优势纲中,伞菌纲(Agaricomycetes)在高海拔区间的表层及底层土壤中显著增加,而银耳纲(Tremellomycetes)则在低海拔表层土壤中相对富集。接合菌门的相对丰度仅在表层土壤随海拔升高而显著增加。土壤深度对真菌优势种群的影响主要在低海拔梯度:相比于表层土壤,伞菌纲、古根菌纲、粪壳菌纲和接合菌某纲的相对丰度在底层土壤显著增加,而银耳纲和座囊菌纲则显著降低。基于FUNGild功能预测分析,共生真菌是色季拉山土壤真菌最主要的功能群(平均相对丰度61.8%),其中最丰富的类型为外生菌根真菌(46.6%)和外生菌根-兰科菌根真菌(11.4%)(图 2)。在表层土壤,外生菌根真菌的相对丰度在海拔4 284 m处最高,其次是4 590 m和3 356 m;而在底层土壤,外生菌根真菌则随海拔升高而显著降低。表层及底层土壤中的外生-兰科菌根真菌的相对丰富均在海拔4 420 m和4 590 m处显著增加。腐生真菌的相对丰度仅次于共生真菌(32.8%),主要有土壤腐生真菌和未知腐生菌。土壤腐生真菌在表层土壤随海拔升高而增加,但在底层土壤则正相反。病原真菌仅在低海拔区间的表层土壤中较为富集(14%),主要是植物病原菌,在其他生境的相对丰度均小于4%。
|
注:Ascomycota:子囊菌门;Basidiomycota:担子菌门;Zygomycota:接合菌门;Others:其他真菌;Agaricomycetes:伞菌纲;Tremellomycetes:银耳纲;Archaeorhizomycetes:古根菌纲;Dothideomycetes:座囊菌纲;Leotiomycetes:锤舌菌纲;Pezizomycetes:盘菌纲;Sordariomycetes:粪壳菌纲;Zygomycota_incertae_sedis:接合菌某纲。3356T、3500T、3689T、4284T、4420T、4590T表示各海拔表层土壤,3356S、3500S、3689S、4284S、4420S、4590S表示各海拔底层土壤。下同。 Note: 3356T, 3500T, 3689T, 4284T, 4420T and 4590T indicate the topsoil at each elevation, while 3356S, 3500S, 3689S, 4284S, 4420S and 4590S indicate the subsoil at each elevation. The same below. 图 1 不同土壤深度下真菌门a)及优势纲b)相对丰度的海拔变化特征 Fig. 1 Variation in the relative abundance of fungal phylum a)and dominant class b)with elevation at two soil depths |
|
注:Pathogen:病原真菌;Saprotroph:腐生真菌;Symbiotroph:共生真菌;Animal pathogen:动物病原菌;Plant pathogen:植物病原菌;Dung saprotroph:粪腐菌;Dung saprotroph-Plant saprotroph:粪腐菌-植物腐生菌;Plant saprotroph-Wood saprotroph:植物腐生菌-木腐菌;Soil saprotroph:土壤腐生菌;Undefined saprotroph:未知腐生菌;Arbuscular Mycorrhizal:丛枝菌根真菌;Ectomycorrhizal:外生菌根真菌;Ectomycorrhizal-Orchid Mycorrhizal:外生菌根-兰科菌根真菌;Endophyte:内生真菌;Others:其他真菌。 图 2 不同土壤深度下真菌功能群相对丰度的海拔变化特征 Fig. 2 Variation in the relative abundance of the fungal functional guild with elevation at two soil profile depths |
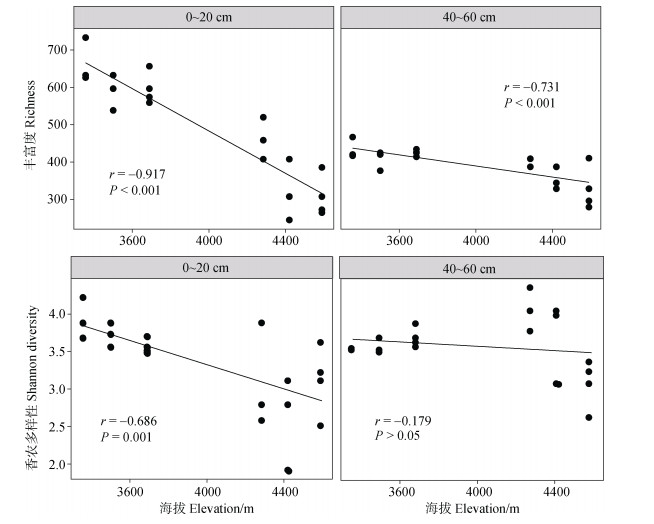
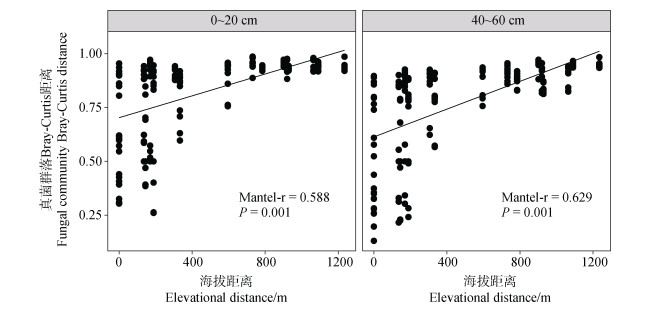
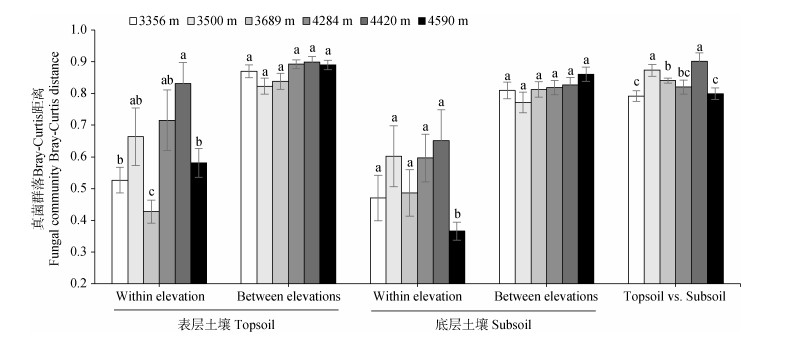
土壤真菌α多样性受到海拔梯度的显著影响(图 3)。相关性分析结果表明,真菌丰富度在土壤表层和底层均与海拔高度呈极显著的负相关关系;真菌香农多样性指数也随海拔升高而降低,但是仅在表层土壤与海拔高度呈显著负相关。Mantel检验分析表明,真菌群落的β多样性与海拔距离在表层和底层土壤均呈显著的正相关关系(图 4),即土壤真菌群落的相异性随着海拔距离的增加而显著升高。进一步分析显示,对于每个采样海拔(除了4 420 m的表层土壤),同海拔不同采样点间真菌群落的Bray-Curtis距离显著小于不同海拔间真菌群落的Bray-Curtis距离;同海拔的不同采样点间(海拔距离为0)的真菌群落组成差异在4 420 m处(表层及底层Bray-Curtis距离分别为0.83和0.65)最大(图 5)。ANOSIM分析表明,海拔梯度在表层(P=0.001)及底层(P=0.001)土壤均对真菌群落有显著影响。土壤深度也显著影响真菌群落组成(P=0.001),不过其影响在低海拔区域(ANOSIM R=0.638,P=0.001)明显强于高海拔区域(ANOSIM R=0.239,P=0.016)。
|
图 3 表层(0~20 cm)和底层(40~60 cm)土壤真菌α多样性指数随海拔高度的变化特征 Fig. 3 Variations in fungal α-diversity indices with elevation in the topsoil(0~20 cm)and subsoil(40~60 cm) |
|
图 4 表层(0~20 cm)和底层(40~60 cm)土壤深度下真菌群落组成差异性(β多样性)与海拔距离的关系 Fig. 4 Relationships between the dissimilarities in fungal community composition(β-diversity)and elevational distance in the topsoil(0~20 cm)and subsoil(40~60 cm) |
|
注:Within elevation:同海拔不同采样点之间比较;Between elevations:不同海拔之间比较;Topsoil vs. Subsoil:同海拔表层和底层之间比较。不同小写字母表示每个分组中不同海拔之间存在显著差异。 Note: Different lowercase letters indicate significant differences among different sampling elevations in each group. 图 5 同海拔内、不同海拔间以及不同土层间真菌群落组成的相异性 Fig. 5 Pairwise Bray-Curtis dissimilarities of fungal community composition in sampling sites within the same elevation, between different elevations and between two soil depths |
Mantel检验分析表明,表层及底层土壤真菌群落组成沿海拔梯度的变化与气候因子(年均温、生长季降水量)、pH、土壤颗粒组成(砂粒、粉粒、黏粒)及土壤含水量均有显著正相关关系(表 2),即土壤真菌群落组成的相异性随这些环境因子变化梯度的增加而升高。土壤有机碳和全氮含量的梯度变化也与真菌群落组成变化有正相关关系,但是仅在表层土壤达到显著。db-RDA分析进一步揭示,影响土壤真菌群落沿海拔梯度变化的关键因子在表层和底层土壤有明显差异(表 3):在表层土壤,环境因子能够解释54.9%的真菌群落变化(F=3.41,P < 0.001),其中土壤pH的解释度最大(29.8%);在底层土壤,环境因子共能够解释60.12%的真菌群落变化(F=4.22,P < 0.001),其中土壤含水量的解释度最大(17.3%)。
|
|
表 2 土壤真菌α及β多样性与环境因子的Pearson相关性系数(r) Table 2 Pearson correlation coefficients(r)of soil fungal α and β diversity with environmental variables |
|
|
表 3 不同土壤深度下影响真菌群落组成沿海拔梯度变异的环境因子及其贡献 Table 3 Driving factors and their contributions to the variation in fungal community composition along the elevation gradient |
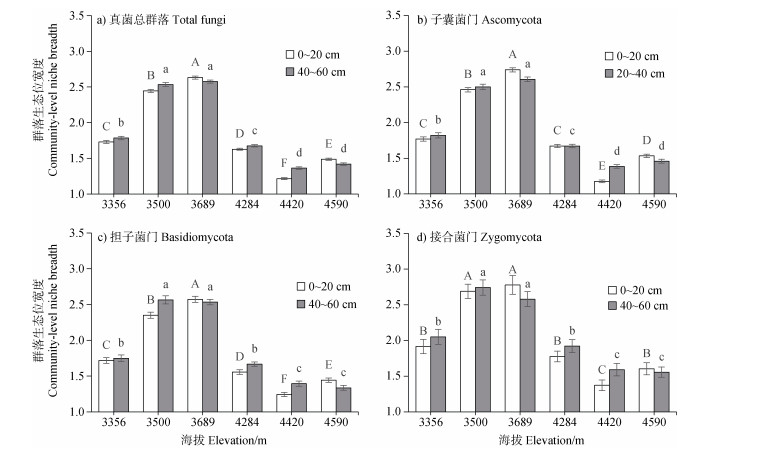
基于Levins法估算得到的真菌总群落及各门的群落水平生态位宽度如图 6所示。海拔梯度显著影响土壤真菌群落的生态位宽度:无论是在土壤表层还是底层,真菌总群落的生态位宽度表现出先升高后降低的特征,在海拔3 500 m和3 689 m处生态位最宽,而在4 420 m和4 590 m处生态位最窄。三个主要的真菌门的群落生态位宽度的海拔变化特征与真菌总群落基本一致。土壤深度对真菌群落生态位宽度的影响显著弱于海拔梯度,且真菌群落生态位宽度在表层和底层土壤的差异在不同海拔处有明显异同:3 500 m、4 284 m和4 420 m处底层土壤真菌的生态位更宽,而3 689 m和4 590 m处表层土壤真菌的生态位更宽。
|
注:不同大写和小写字母分别表示表层和底层土壤中真菌种群生态位宽度在不同海拔间有显著差异。 Note: Different upper-and lowercase letters indicate significant differences among sampling elevations in the topsoil and subsoil, respectively. 图 6 不同土壤深度下真菌总群落及主要门的群落水平生境生态位宽度(Bcom)的海拔变化特征 Fig. 6 Variation in community-level habitat niche breadth(Bcom)of the total community and major fungal phylum with elevation at two soil depths |
土壤真菌种群的生态位重叠值(Oik)分布在0.42~0.99之间(表 4),说明色季拉山各真菌种群之间对资源利用或者生态适应性的相似度有一定的差异性。在土壤表层,生态位高度重叠(Oik > 0.8)的在低海拔区间和高海拔区间分别共有21对和15对,占总对数的80.8%和57.7%;中度重叠(0.6 < Oik ≤0.8)的分别有3和9对,占总对数的11.5%、34.6%;低度重叠(Oik ≤ 0.6)的均为有2对,占总对数的7.7%。在土壤底层,海拔区间对真菌中区间的生态位重叠没有明显影响:低海拔区间和高海拔区间具有相同的生态位高度重叠(共14对)和中度重叠(共4对)对数。总体而言,生态位高度重叠的对数在高海拔地区低于低海拔地区,且在土壤表层高于底层,而生态位中低度重叠占总对数的比例在高海拔和土壤底层更高。生态位高度重叠的种对中,以子囊菌、接合菌与其他种群形成的种对为主:子囊菌、接合菌与其他种群的生态位重叠值大于0.8的均能占到总对数的70%。而生态位低度重叠的种对均是以壶菌、球囊菌与其他种群形成的对为主。
|
|
表 4 不同海拔梯度及土壤深度下真菌群落的生态位重叠值(Oik) Table 4 Niche overlap values(Oik)of fungal communities at different elevations and soil depths |
海拔分布特征是微生物地理学的基本模式之一,然而目前相关研究多关注表层土壤。本研究发现,色季拉山土壤真菌α和β多样性的海拔分布特征在表层和底层土壤存在一定的相异性(图 3和图 4),并且驱动土壤真菌群落海拔变异的关键环境因子在土壤表层和底层不同(表 3)。在土壤表层,土壤pH与真菌α和β多样性有极强的正相关性,db-RDA分析进一步揭示土壤pH是表层土壤真菌群落海拔变异的关键驱动因子,这与以往研究结果一致[9-10,13-15]。而在土壤底层,土壤湿度的变化则是真菌群落组成沿海拔梯度变异的重要驱动。土壤湿度是影响微生物活性及细菌和真菌生长的重要因子之一[32]。近期研究表明,土壤湿度对真菌多样性的生物地理分布模式至关重要[7]。并且,与土壤水分有效性密切相关的一些气候变量,例如年降雨量和潜在蒸散量,也与真菌物种多样性或群落组成显著相关[6]。
土壤剖面深度的变化能够显著影响土壤性质,进而改变微生物群落组成[33]。本研究发现,色季拉山真菌群落的组成在表层和底层土壤有显著差异,且真菌群落组成在不同土层之间的相异性与群落在不同海拔之间的相异性没有显著差异(图 5)。这表明在色季拉山,真菌群落组成的差异性在土壤剖面上(厘米距离范围)的变化程度与其在海拔梯度上(千米距离范围)的变化程度几乎相近。Chu等[33]在西藏阿里的研究也揭示了细菌群落组成在单一采样点的表层(0~15 cm)和底层(15~30 cm)土壤之间的差异与其在不同采样点之间(相隔数公里)的差异大致相同,而这归因于不同土层间显著变化的土壤因子例如土壤有机碳、土壤碳氮比等。在色季拉山,随着土壤剖面加深,土壤pH显著增加,而土壤有机碳、土壤总氮以及土壤湿度则显著降低(表 1),这些剧烈变化的土壤性质显著改变了土壤剖面不同深度下真菌群落的组成和多样性。
3.2 色季拉山土壤真菌群落分布格局的影响机制db-RDA模型中环境变量对色季拉山表层及底层土壤真菌群落组成变异的解释度分别为54.9%和60.1%,说明生境过滤作用是影响色季拉山土壤真菌多样性海拔分布格局的关键机制。赵鹏宇等[34]在庞泉沟自然保护区的研究和罗正明等[9]在五台山的研究也报道了生境过滤作用对土壤真菌群落海拔分布格局的主导作用。除了环境因子,植物群落组成沿海拔梯度的变化也是可能影响色季拉山土壤真菌群落分布格局。FUNGuild分析发现,色季拉山土壤真菌群落中有44%~72%属于共生真菌,其中大部分为外生菌根真菌及外生-兰科菌根真菌(图 2),其宿主植物的多样性和群落组成的空间变异对菌根真菌群落构建有主导作用[35]。此外,随机性过程(例如扩散限制)也是调控土壤真菌群落组成的重要机制。Sheng等[8]在神农架的研究发现了土壤真菌群落组成在同一海拔(同一植被类型)的不同采样点间表现出了较大的差异,本研究也发现了类似的结果(图 4)。扩散过程是影响同一植被类型下土壤真菌群落组成变异的重要机制,这主要是由于生境异质性和环境波动程度在同一植被类型下通常弱于不同植被类型间[36]。另外,色季拉山降水充沛,土壤孔隙充满水后能够相互连通,有助于孢子/菌丝在土壤中的扩散、漂移过程,从而增加真菌群落组成在同一植被类型下的随机性并导致群落组成的明显差异。
3.3 色季拉山土壤真菌群落的生态位特征物种的生态位决定了其在生态系统中发挥的功能,而生态位宽度则界定了物种资源利用能力和生境分布的范围[37]。在色季拉山,不同海拔高度下真菌总群落生态位宽度的变化范围为1.21~2.63(图 6)。这表明通过高通量测序检测到的真菌物种(OTU)中有很大部分属于Habitat specialists(生态位宽度B值小于1.5[38]),分布范围有限。真菌群落生态位宽度在低海拔(3 500 m和3 689 m)显著增加,这可能是由于低海拔地区变化频繁的水热环境降低了微生物群落对于环境变化的敏感性[39],从而导致真菌种群具有较高的环境适应性和更为灵活的群落代谢方式[19]。在高海拔地区(4 420 m和4 590 m),真菌群落的生态位宽度显著降低。在南极地区的研究也发现,由于随海拔升高而降低的温度减缓了细菌生长速率,土壤细菌的生态位宽度随着海拔的升高而变窄[40]。温度变化能够显著影响到代谢等生物学过程的速率,因而物种对温度的适应性是影响生物多样性海拔分布特征的关键因素之一[3]。因此,本研究的结果表明色季拉山高海拔地区的土壤真菌对环境变化的敏感性更高,在未来全球变暖情景下可能面临着更高的灭绝风险[20]。
生态位重叠值的高低能够反映物种间对资源利用或生境分布(环境适应)的相似程度。本研究的结果表明,色季拉山土壤真菌种群间对资源利用或生境分布存在竞争关系,并且其竞争的激烈程度随海拔的升高及土壤剖面的加深而降低。主要真菌种群中,子囊菌门与担子菌门种群之间的生态位重叠度较高(表 4),表明这两类种群之间以及与其他种群的竞争十分激烈,而这主要是由于这两类真菌在凋落物分解和养分循环过程中起着核心作用[41]。接合菌与子囊菌、担子菌及球囊菌之间也具有较高的生态位重叠度,这是由于接合菌门中的物种具有多种功能类型,既有腐生菌,也有病原菌,有些接合菌还可以形成专性或者兼性寄生关系,从而导致接合菌与其他真菌种群的生态位重叠较高。生态位低度重叠的种对中以壶菌、球囊菌与其他种群形成的比例最高,说明这两类真菌与其他种群在资源利用或生境分布上有一定差异。例如,壶菌门的真菌多生活在淡水或者潮湿的土壤中,且很多种属能够寄生在浮游植物、藻类(绿藻、硅藻、蓝藻等)上[42];而球囊菌门的真菌属于丛枝菌根真菌(专性共生),其生长和分布更多地依赖于寄主植物的群落多样性和组成[43],受环境因子的影响弱于其他主要真菌种群。正是这种与植物寄生或者共生的关系使壶菌和球囊菌在资源利用和生境分布上与子囊菌、担子菌等相似程度低,种群间可以协调地分享资源,使群落处于相对稳定的平衡状态。
4 结论总体上随着海拔的升高,色季拉山真菌的丰富度在土壤表层及底层均显著下降,但是香农多样性仅在表层显著下降,而在底层表现出先增高后降低的单峰特征。真菌的群落组成(β多样性)在表层和底层土壤均表现出距离衰减模式,土壤pH是表层土壤真菌群落组成沿海拔梯度变化的关键驱动因子,而土壤湿度则对底层土壤真菌群落变异的解释度最高。环境因子改变引起的生境过滤是影响色季拉山土壤真菌群落组成沿海拔梯度变异的关键过程。真菌总群落及优势种群的生态位宽度表现出先升高后降低的特征,高海拔地区的真菌种群生态位宽度最窄,可能对未来全球变暖的响应更加敏感。真菌种群间的生态位重叠度随海拔的升高和土壤剖面的加深而减弱。子囊菌、接合菌与其他种群生态位重叠度较高,其种群间的竞争较激烈;而球囊菌、壶菌由于其与植物共生或寄生的生理特性,能与其他种群协调地分享资源,生态位重叠度较低。本研究结果为深入了解青藏高原高寒生态系统中土壤真菌的群落构建和共存机制提供了科学依据。
| [1] |
Nemergut D R, Schmidt S K, Fukami T, et al. Patterns and processes of microbial community assembly[J]. Microbiology and Molecular Biology Reviews, 2013, 77(3): 342-356. DOI:10.1128/MMBR.00051-12
(  0) 0) |
| [2] |
Li D W, Wang Z Q, Tian H X, et al. Carbon, nitrogen and phosphorus contents in soils on Taibai Mountain and their ecological stoichiometry relative to elevation (In Chinese)[J]. Acta Pedologica Sinica, 2017, 54(1): 160-170. [李丹维, 王紫泉, 田海霞, 等. 太白山不同海拔土壤碳、氮、磷含量及生态化学计量特征[J]. 土壤学报, 2017, 54(1): 160-170.]
(  0) 0) |
| [3] |
Sundqvist M K, Sanders N J, Wardle D A. Community and ecosystem responses to elevational gradients: Processes, mechanisms, and insights for global change[J]. Annual Review of Ecology, Evolution, and Systematics, 2013, 44: 261-280. DOI:10.1146/annurev-ecolsys-110512-135750
(  0) 0) |
| [4] |
Li G X, Ma K M. Progress in the study of elevational patterns of soil microbial diversity (In Chinese)[J]. Acta Ecologica Sinica, 2018, 38(5): 1521-1529. [厉桂香, 马克明. 土壤微生物多样性海拔格局研究进展[J]. 生态学报, 2018, 38(5): 1521-1529.]
(  0) 0) |
| [5] |
Bryant J A, Lamanna C, Morlon H, et al. Microbes on mountainsides: Contrasting elevational patterns of bacterial and plant diversity[J]. PNAS, 2008, 105(1): 11505-11511.
(  0) 0) |
| [6] |
Tedersoo L, Bahram M, Põlme S, et al. Global diversity and geography of soil fungi[J]. Science, 2014, 346(6213): 1256688. DOI:10.1126/science.1256688
(  0) 0) |
| [7] |
Peay K G, Kennedy P G, Talbot J M. Dimensions of biodiversity in the earth mycobiome[J]. Nature Reviews Microbiology, 2016, 14(7): 434-447. DOI:10.1038/nrmicro.2016.59
(  0) 0) |
| [8] |
Sheng Y Y, Cong W, Yang L S, et al. Forest soil fungal community elevational distribution pattern and their ecological assembly processes[J]. Frontiers in Microbiology, 2019, 10: 2226. DOI:10.3389/fmicb.2019.02226
(  0) 0) |
| [9] |
Luo Z M, Liu J X, Bao J B, et al. Elevational distribution patterns and assembly mechanisms of soil fungal community in Mount Wutai, Shanxi, China (In Chinese)[J]. Acta Ecologica Sinica, 2020, 40(19): 7009-7017. [罗正明, 刘晋仙, 暴家兵, 等. 五台山亚高山土壤真菌海拔分布格局与构建机制[J]. 生态学报, 2020, 40(19): 7009-7017.]
(  0) 0) |
| [10] |
Zhou Y J, Jia X, Han L, et al. Fungal community diversity in soils along an elevation gradient in a Quercus aliena var. acuteserrata forest in Qinling Mountains, China[J]. Applied Soil Ecology, 2021, 167: 104104. DOI:10.1016/j.apsoil.2021.104104
(  0) 0) |
| [11] |
Miyamoto Y, Nakano T, Hattori M, et al. The mid-domain effect in ectomycorrhizal fungi: Range overlap along an elevation gradient on Mount Fuji, Japan[J]. The ISME Journal, 2014, 8(8): 1739-1746. DOI:10.1038/ismej.2014.34
(  0) 0) |
| [12] |
Wang J T, Zheng Y M, Hu H W, et al. Soil pH determines the alpha diversity but not beta diversity of soil fungal community along altitude in a typical Tibetan forest ecosystem[J]. Journal of Soils and Sediments, 2015, 15(5): 1224-1232. DOI:10.1007/s11368-015-1070-1
(  0) 0) |
| [13] |
Shen C C, Liang W J, Shi Y, et al. Contrasting elevational diversity patterns between eukaryotic soil microbes and plants[J]. Ecology, 2014, 95(11): 3190-3202. DOI:10.1890/14-0310.1
(  0) 0) |
| [14] |
Siles J A, Margesin R. Abundance and diversity of bacterial, archaeal, and fungal communities along an altitudinal gradient in alpine forest soils: What are the driving factors?[J]. Microbial Ecology, 2016, 72(1): 207-220. DOI:10.1007/s00248-016-0748-2
(  0) 0) |
| [15] |
Liu D, Liu G H, Chen L, et al. Soil pH determines fungal diversity along an elevation gradient in Southwestern China[J]. Science China Life Sciences, 2018, 61(6): 718-726. DOI:10.1007/s11427-017-9200-1
(  0) 0) |
| [16] |
Tedersoo L, Bahram M, Cajthaml T, et al. Tree diversity and species identity effects on soil fungi, protists and animals are context dependent[J]. The ISME Journal, 2016, 10(2): 346-362. DOI:10.1038/ismej.2015.116
(  0) 0) |
| [17] |
Chen X B, Zhu D Q, Zhao C C, et al. Community composition and diversity of fungi in soils under different types of Pinus koraiensis forests (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(5): 1221-1234. [陈秀波, 朱德全, 赵晨晨, 等. 不同林型红松林土壤真菌群落组成和多样性[J]. 土壤学报, 2019, 56(5): 1221-1234.]
(  0) 0) |
| [18] |
Niu K C, Liu Y N, Shen Z H, et al. Community assembly: The relative importance of neutral theory and niche theory (In Chinese)[J]. Biodiversity Science, 2009, 17(6): 579-593. [牛克昌, 刘怿宁, 沈泽昊, 等. 群落构建的中性理论和生态位理论[J]. 生物多样性, 2009, 17(6): 579-593.]
(  0) 0) |
| [19] |
Pandit S N, Kolasa J, Cottenie K. Contrasts between habitat generalists and specialists: An empirical extension to the basic metacommunity framework[J]. Ecology, 2009, 90(8): 2253-2262. DOI:10.1890/08-0851.1
(  0) 0) |
| [20] |
Thuiller W, Lavorel S, Araújo M B. Niche properties and geographical extent as predictors of species sensitivity to climate change[J]. Global Ecology and Biogeography, 2005, 14(4): 347-357. DOI:10.1111/j.1466-822X.2005.00162.x
(  0) 0) |
| [21] |
Schellenberger Costa D, Gerschlauer F, Kiese R, et al. Plant niche breadths along environmental gradients and their relationship to plant functional traits[J]. Diversity and Distributions, 2018, 24(12): 1869-1882. DOI:10.1111/ddi.12815
(  0) 0) |
| [22] |
Ahmad M, Sharma P, Rathee S, et al. Niche width analyses facilitate identification of high-risk endemic species at high altitudes in western Himalayas[J]. Ecological Indicators, 2021, 126: 107653. DOI:10.1016/j.ecolind.2021.107653
(  0) 0) |
| [23] |
Lara-Romero C, Seguí J, Pérez-Delgado A, et al. Beta diversity and specialization in plant-pollinator networks along an elevational gradient[J]. Journal of Biogeography, 2019, 46(7): 1598-1610.
(  0) 0) |
| [24] |
Xu M, Li X L, Cai X B, et al. Soil microbial community structure and activity along a montane elevational gradient on the Tibetan Plateau[J]. European Journal of Soil Biology, 2014, 64: 6-14. DOI:10.1016/j.ejsobi.2014.06.002
(  0) 0) |
| [25] |
Wang J T, Cao P, Hu H W, et al. Altitudinal distribution patterns of soil bacterial and archaeal communities along Mt. Shegyla on the Tibetan Plateau[J]. Microbial Ecology, 2015, 69(1): 135-145. DOI:10.1007/s00248-014-0465-7
(  0) 0) |
| [26] |
Jackson R B, Lajtha K, Crow S E, et al. The ecology of soil carbon: Pools, vulnerabilities, and biotic and abiotic controls[J]. Annual Review of Ecology, Evolution, and Systematics, 2017, 48: 419-445. DOI:10.1146/annurev-ecolsys-112414-054234
(  0) 0) |
| [27] |
Yost J L, Hartemink A E. How deep is the soil studied–an analysis of four soil science journals[J]. Plant and Soil, 2020, 452(1/2): 5-18.
(  0) 0) |
| [28] |
Jiao K, Zhang X B, Xu M, et al. Depth- related characteristics of soil microbial community along the soil profile of typical dark coniferous forest in southeast Tibet (In Chinese)[J]. Acta Ecologica Sinica, 2021, 41(12): 4864-4875. [焦克, 张旭博, 徐梦, 等. 藏东南典型暗针叶林不同土壤剖面深度微生物群落特征研究[J]. 生态学报, 2021, 41(12): 4864-4875.]
(  0) 0) |
| [29] |
Nguyen N H, Song Z W, Bates S T, et al. FUNGuild: An open annotation tool for parsing fungal community datasets by ecological guild[J]. Fungal Ecology, 2016, 20: 241-248. DOI:10.1016/j.funeco.2015.06.006
(  0) 0) |
| [30] |
Levins R. Evolution in changing environments . Princeton, New Jersey: Princeton University Press, 1968.
(  0) 0) |
| [31] |
Pianka E R. The structure of lizard communities[J]. Annual Review of Ecology, Evolution, and Systematics, 1973, 4: 53-74. DOI:10.1146/annurev.es.04.110173.000413
(  0) 0) |
| [32] |
Odriozola I, Navrátilová D, Tláskalová P, et al. Predictors of soil fungal biomass and community composition in temperate mountainous forests in Central Europe[J]. Soil Biology and Biochemistry, 2021, 161: 108366. DOI:10.1016/j.soilbio.2021.108366
(  0) 0) |
| [33] |
Chu H Y, Sun H B, Tripathi B M, et al. Bacterial community dissimilarity between the surface and subsurface soils equals horizontal differences over several kilometers in the western Tibetan Plateau[J]. Environmental Microbiology, 2016, 18(5): 1523-1533. DOI:10.1111/1462-2920.13236
(  0) 0) |
| [34] |
Zhao P Y, Li C, Chai B F. Environmental filters drive the assembly of the soil fungal community in the Larix principis-rupprechtii forests of the Guandi Mountains (In Chinese)[J]. Environmental Science, 2018, 39(8): 3876-3884. [赵鹏宇, 李毳, 柴宝峰. 生境过滤驱动庞泉沟华北落叶松林土壤真菌群落的构建过程[J]. 环境科学, 2018, 39(8): 3876-3884.]
(  0) 0) |
| [35] |
Boeraeve M, Honnay O, Jacquemyn H. Effects of host species, environmental filtering and forest age on community assembly of ectomycorrhizal fungi in fragmented forests[J]. Fungal Ecology, 2018, 36: 89-98. DOI:10.1016/j.funeco.2018.08.003
(  0) 0) |
| [36] |
Bahram M, Kohout P, Anslan S, et al. Stochastic distribution of small soil eukaryotes resulting from high dispersal and drift in a local environment[J]. The ISME Journal, 2016, 10(4): 885-896. DOI:10.1038/ismej.2015.164
(  0) 0) |
| [37] |
Pielou E C. Niche width and niche overlap: A method for measuring them[J]. Ecology, 1972, 53(4): 687-692.
(  0) 0) |
| [38] |
Logares R, Lindström E S, Langenheder S, et al. Biogeography of bacterial communities exposed to progressive long-term environmental change[J]. The ISME Journal, 2013, 7(5): 937-948.
(  0) 0) |
| [39] |
Griffiths B S, Philippot L. Insights into the resistance and resilience of the soil microbial community[J]. FEMS Microbiology Reviews, 2013, 37(2): 112-129. DOI:10.1111/j.1574-6976.2012.00343.x
(  0) 0) |
| [40] |
Okie J G, van Horn D J, Storch D, et al. Niche and metabolic principles explain patterns of diversity and distribution: Theory and a case study with soil bacterial communities[J]. Proceedings of the Royal Society B: Biological Sciences, 2015, 282(1809): 20142630.
(  0) 0) |
| [41] |
Schneider T, Keiblinger K M, Schmid E, et al. Who is who in litter decomposition? Metaproteomics reveals major microbial players and their biogeochemical functions[J]. The ISME Journal, 2012, 6(9): 1749-1762.
(  0) 0) |
| [42] |
Gleason F H, Jephcott T G, Küpper F C, et al. Potential roles for recently discovered chytrid parasites in the dynamics of harmful algal blooms[J]. Fungal Biology Reviews, 2015, 29(1): 20-33.
(  0) 0) |
| [43] |
Gai J P, Tian H, Yang F Y, et al. Arbuscular mycorrhizal fungal diversity along a Tibetan elevation gradient[J]. Pedobiologia, 2012, 55(3): 145-151.
(  0) 0) |
 2023, Vol. 60
2023, Vol. 60


