2. 河南省土壤肥料站, 郑州 450002;
3. 郑州大学政治与公共管理学院, 郑州 450001
2. Henan Provincial Station of Soil and Fertilizer, Zhengzhou 450002, China;
3. School of Public Administration, Zhengzhou University, Zhengzhou 450001, China
随着世界人口的持续增长与全球性资源环境问题的逐渐凸显,作为人类生存与发展最重要物质基础之一的土壤资源面临严峻挑战,有关土壤功能、土壤质量、土壤安全(Soil security)的风险日益引发关注,土壤资源在保障粮食安全、供给生态服务、实现联合国可持续发展目标(the United Nations Sustainable Development Goals,SDGs)中的核心作用成为公众关注的焦点[1-4]。在高强度土壤资源利用和全球变化宏观背景下,高质量土壤空间基准信息是监测土壤性能、土壤条件动态变化的前提,是警示土壤退化过程发生、优化未来土壤利用选择的重要依据[5-7]。另一方面,多尺度、高分辨率、高精度的土壤信息是现代农业发展的基石,是在环境友好、资源可持续利用基础上实现高产稳产、满足世界人口对粮食生产与生态服务日益增长需求的重要支撑。此外,精确、详细的土壤信息可广泛应用于其他相关领域,如区域生态环境建模、流域水文水资源评价等[8-10]。快速、高效、精准获取并表征土壤信息近年来已成土壤相关学科的研究热点之一[11-12]。21世纪初,数字土壤制图(Digital soil mapping,DSM)逐步取代常规土壤调查制图成为土壤信息获取与表达的新范式,引领这一领域的研究与应用走进全新时代[11,13]。数字土壤制图又称土壤预测制图(Predictive soil mapping,PSM),定义为“利用野外观测和实验室分析手段,结合空间和非空间土壤推绎系统,组织和创建空间土壤信息系统的技术途径”[14]。它的核心技术框架是McBratney等[15]基于道库恰耶夫土壤发生学理论、Jenny土壤状态因子方程提出的SCORPAN模型(其中,S:其他土壤类型或属性,C:气候因子,O:生物因子,R:地形因子,P:母质因子,A:时间因子,N:地理坐标)。DSM通过定量拟合土壤空间依赖结构、模拟土壤与环境协变量之间复杂的线性及非线性关系,实现对目标土壤属性或土壤类型的空间预测与数字化制图。经过20年的快速发展,当前数字土壤制图已经从研究层面逐步走向应用环节[16-17]。DSM包括三类数学模型:地理空间模型(Geographic-space-based models)、属性空间模型(Feature-space-based models)以及混合模型(Hybrid models)[13,18]。其中,地理空间模型又称随机模型(Stochastic models),它只拟合土壤属性观测值的空间依赖结构,不考虑土壤属性与环境协变量之间相互关系导致的确定性趋势,土壤属性预测结果及精度完全取决于观测土壤样点的密度与质量。属性空间模型又称确定性模型(Deterministic models),此类模型通过定量拟合目标土壤变量与各协变量之间的复杂关系,揭示土壤变量空间分布的确定性趋势。作为一类最为重要的属性空间预测模型,机器学习(Machine learning,ML)算法能有效整合、处理和利用不同类型、不同分辨率的多源数据,且在进行模型拟合前无需对数据做任何统计性假设,非常适合用于拟合和揭示目标土壤类型和属性与其他土壤因子、环境因子等各种协变量之间复杂的非线性关系。最近二十年,得益于土壤数据、环境信息快速获取技术和共享计算能力的持续提高,ML算法在数字土壤制图中的应用日益广泛[19-21]。从简单的多元线性回归(Multivariate linear regression,MLR)[8,22]、分类与回归树(Classification and regression trees,CART)[18,23],到相对复杂的人工神经网络(Artificial neural network,ANN)[24-25]、Cubist(Cu)[26]和随机森林(Random forests,RF)[25,27-28],在土壤空间预测与数字化制图实践中均有相当多的应用案例。在上述ML模型中,RF模型因其预测精度高、过度拟合少、容噪能力强等优势,成为广受好评的佼佼者。根据Lamichhane等[25]的统计,在2013至2019年前两个月报道的834个SOC空间预测与数字化制图案例研究中,RF模型的应用频率在所有DSM模型中排名第二;在对17种模型性能的对比研究中,RF模型在其中13个案例中的表现优于比较对象。
土壤空间预测与数字化制图输出结果的精度与可靠性,不仅取决于预测模型,同时也取决于目标土壤变量的空间变异特征、观测土壤样点密度(即特定研究区内的样点规模)、环境协变量信息可用性、准确性、空间分辨率以及与选用预测模型的匹配程度等多种因素[29-30]。理论上,没有适合于一切应用的最优模型,只有适用于特定场景的最佳模型。众多案例研究结果显示,样点规模、采样方法对土壤空间预测精度的影响超过预测模型[31-32]。某种程度上,土壤空间预测模型选择就是在预测精度、样点规模、模型复杂程度之间的相互折中。有关土壤样点密度与采样策略对土壤空间预测精度及ML模型性能表现影响的诸多案例研究已经见诸报道[33-38],但绝大部分案例研究只涉及样点密度或采样策略对一种ML模型预测精度的影响,在多种样点密度、多种采样方法下多种常用ML模型的预测精度、性能表现的多维度比较研究,迄今鲜有报道。
以位于中国中部地区的河南省为案例研究区,基于新近更新的河南省耕地质量评价数据库和土壤属性数据库,采用不同的采样方法抽取不同的土壤样点密度,分别应用MLR、CART、Cu、ANN和RF等五种常用的ML算法对耕地表层土壤pH实施空间预测与数字化制图:(1)揭示样点密度、采样方法对五种选用ML模型性能表现与预测精度的影响;(2)确定预测精度最高的样点密度、采样方法与ML模型组合,输出研究区耕地表层土壤pH预测与不确定性评价结果。
1 材料与方法 1.1 研究区域河南省位于我国中东部、黄河中下游(31°23′— 36°22′N,110°21′—116°39′E)(图 1)。全省国土面积1.67×105 km2,其中耕地面积8.1×106 hm2,约占全国耕地总面积的6.0%。河南省是我国农业大省,至2020年,河南省粮食产量已连续多年稳定在650×108 kg以上,全国占比超过10%,在国家粮食生产和粮食安全战略中发挥着至关重要的作用。河南省地处暖温带与北亚热带过渡带,属大陆性季风气候,年均气温12.7~16.2℃,年均降水量478~1 167 mm,年无霜期208~272 d。省内地势西高东低,山地和丘陵占陆地总面积的44.3%,平原和盆地占总面积的55.7%。河南省属于北方典型的两熟制地区,北部以冬小麦—夏玉米轮作为主,南部则以水稻—冬小麦轮作为主。本研究区为河南省耕地土壤分布区,按照《中国土壤系统分类》土壤类型划分标准,河南省境内耕作土壤主要包括新成土、雏形土、淋溶土和人为土[39]。主要受氮肥长期高强度施用影响,近年来全省耕地表土pH呈明显下降趋势。根据我们最近研究结果,2008—2018年河南省超过总面积94%的耕地表层土壤pH出现不同程度的下降,平均下降幅度高达0.36个pH单位[40]。
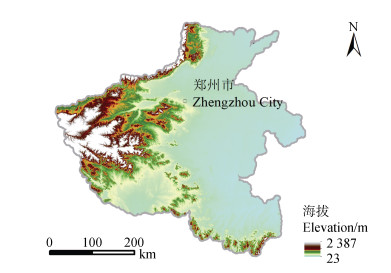
|
图 1 研究区地理位置与地貌示意图 Fig. 1 Geographical location and general landform of the study region |
2017—2019年,河南省启动测土配方施肥项目补充采样和耕地质量调查监测与评价土壤采样,先后共采集耕地表层土壤样品2万余份。采样按《NY/T1634-2008耕地地力调查与质量评价技术规程》和《GB/T 36197-2018土壤质量:土壤采样技术指南》进行。土壤样点以土地利用形式、土壤类型和地貌单元为基本数据层通过分层随机策略生成,野外通过手持GPS确定坐标位置,定位精度在5 m以内。表层土壤采样深度0~20 cm,采集20 m×20 m正方形四角和中心位置混合样。样品处理与实验室测试分析按照《NYT 1121 2-2006土壤检测第2部分:土壤pH的测定》进行,土壤样品风干、研磨并过2 mm筛,使用标准pH计在1:2.5的土壤-水悬浮液中测定pH。
1.3 协变量集基于DSM-SCORPAN技术框架[15],从研究区土壤发生学背景、主要成土因素与成土过程、土壤农业利用历史、现代改良与干预活动等方面出发,收集整理了5类31个具备耕地表层pH空间变异性解释潜力的因素构成协变量集。其中,气候类因子包括年均气温、年均降水量、年均蒸发量和干旱指数,观测数据可在国家气象科学数据中心网站(http://data.cma.cn/data/cdcdetail/dataCode)下载,最终数值通过对分布在河南省的115座气象站的地面观测数据进行Anusplin插值生成。地形因子包括高程、坡度、坡向、平面曲率、剖面曲率、地形湿度指数(TWI)和地形位置指数(TPI)。30 m分辨率DEM从ASTER全球数字高程模型(http://www.tuxingis.com/resource/aster_v3.html)获取,并使用ArcGIS10.4 Spatial Analyst Tools获得其他衍生地形因子。生物因素包括逐月归一化植被指数(NDVI)和净初级生产力指数(NPP),由中国资源环境科学与数据中心(http://www.resdc.cn/Default提供的SPOT/VEGETATION和MODIS卫星图像生成.aspx)。土壤因子主要来自新近更新的1:500,000河南耕地质量评价数据库和河南省土壤图,包括土壤类型(土壤发生分类)、表层土壤质地、土壤容重、表层厚度、剖面构型、土壤温度状况和土壤水分状况。
除上述4类自然因子外,本研究还收集整理了一组表征耕地人为干预活动的因子,作为干预类或管理类协变量,包括耕地类型(根据第二次全国土地调查土地利用分类)、灌溉保证率、排水能力、化肥施用总量、氮肥施用量和有机肥施用量,相关数据均来自河南省耕地质量评价数据库。其中耕地类型、灌溉保证率和排水能力为图斑数据层,施肥数据为样点土壤背景调查中获得的肥料施用量经普通克里格插值而成的栅格数据。由于上述协变量分辨率不统一,需在ArcGIS环境中对其重采样为100 m分辨率。
1.4 样点规模2017—2019年采样周期内,河南省耕地表层土壤样点总规模超过2万个。团队前期研究表明,当训练样点规模超过8 000个时,省尺度表层土壤pH空间预测输出结果的精度不再随样点规模增大而提高。本研究样点足够丰富,故采用独立验证(hold out validation)将数据划分为验证集和测试集。首先从2万多个有效样点中采集(抽取)2 000样点作为独立验证集。从互斥的样点中再抽取8 000个样点组成规模最大的样点集,然后再以倍减梯度构建样点规模分别为6 000、4 000、2 000、1 600、1 200、800、400和200的样点集作为不同采样方法的训练集,分别参与构建用于表层pH空间预测的ML模型,用以对比分析不同样点规模对预测精度及各选用ML模型性能表现的影响。每个样点规模重复抽样10次,以避免随机性的影响和单个随机样本可能造成的不稳定性。
1.5 采样方法为对比分析不同采样方法对ML模型性能表现与表层土壤pH预测精度的影响,本研究分别采用简单随机采样、分层随机采样、网格法采样、k-均值(k-means)法采样和条件拉丁超立方采样(Conditioned latin hypercube sampling)从研究区全部2万余个土壤样点中抽取样点规模从8000至200的9个样点集构建预测模型。五种采样方法下400个样点的空间分布图如图 2。

|
图 2 五种采样方法下研究区土壤样点空间分布图(以样点规模400个为例) Fig. 2 Spatial distribution of the soil sites sampled with five strategies(taking the sample size of 400 as an example) |
(1) 简单随机采样。无替换简单随机采样是最简单的抽样方法之一,它以等概率的途径从总样点中独立地选择采样个体,即个体样点被选中进入特定子集的概率完全相同,且采样过程无需任何关于目标土壤类型和属性在研究区空间变异的先验知识[27]。本研究使用R语言基础包中的样本函数完成简单随机采样[41]。
(2) 分层随机采样。将研究区主要环境协变量进行分层,在协变量层相互重叠区域随机选择样点,以达到以较小样点规模最大程度覆盖协变量—目标土壤变量相互关系的变异空间[42-43]。分层随机采样的设计需要土壤学专业知识以及对研究区土壤发生学背景、成土环境与主要成土过程以及土壤利用与改良历史有较为深入的了解。本研究以耕地利用方式、地貌类型、土壤类型作为分层协变量,分层随机采样使用R语言基础包中的样本函数实施[41]。
(3) 网格法采样。按照参与ML预测模型构建的样点集规模不同,将研究区多次格网化,依次划分出8 000,6 000、4 000、2 000、1 600、1 200、800、400和200个正方形网格,每个网格里选中靠近格网中心一个样点参与构建不同规模的样点子集,网格中无样点者,则选择邻近格网中空间距离最小的样点替代。本研究使用R软件包中的函数spsample()实施土壤样点的网格法采样[44]。
(4) k-均值(k-means)聚类采样。k-均值聚类是最著名一种迭代求解的分类算法。简单而言,如果指定将研究区土壤样点归入k个聚类中,k-均值算法会根据距离函数反复计算并依据距离最小原则将样点分入k个不同聚类中。本研究中k-均值聚类使用欧氏距离(Euclid Distance),土壤样点之间、土壤样点与聚类中心之间欧式距离就是其协变量多维空间上的距离。聚类完成后,每个土壤样点只能归属一个类别,本研究中,聚类k的数量即为拟选样点的数量,距离k个聚类欧式距离最小的样点即为被选定的样点。本研究中k-均值聚类采样在MATLAB中用Statistics and Machine Learning Toolbox中完成。
(5) 条件拉丁超立方采样。条件拉丁超立方采样(Conditioned Latin hypercube sampling cLHS)理论上也是一种分层随机抽样程序,它首先将样本空间划分为与预测协变量数量相同的维度,从而形成“超立方体”。将每个变量独立地分层为连续的非重叠区间,使区间数量等于样本量,从每层随机抽取一个样本。然后以随机方式或基于特定规则的方式从每个协变量层中确定样本点,以便样本覆盖所有变量区间。cLHS使用退火模拟的方法,通过最小化样点数量与环境特征相关矩阵的目标函数来实现[45]:
| $\begin{array}{llll} \mathrm{O}=\mathrm{w} 1 & \mathrm{O}_1+\mathrm{w}_2 & \mathrm{O}_2+\mathrm{w}_3 & \mathrm{O}_3 \end{array} $ | (1) |
式中,O1为连续变量的子函数,O2为分类变量的子函数,O3为确保采样协变量最大限度代表原始数据而另外增加的一个目标。w为目标函数各分量的权重,本研究所有的w均设置为1。本研究条件拉丁超立方采样在R语言中的clhs包完成[46]。
1.6 预测模型(1) 线性多元回归(MLR)。MLR是最简单的机器学习方法之一,是普通最小二乘(OLS)回归的扩展。它涉及一个以上变量。模型构建基于因变量和解释变量之间存在线性关系、且各解释变量之间无相关性的统计学假设[47]。由于相对简单,MLR在土壤空间预测与数字化制图的早期发展阶段应用广泛,常见与国内外相关研究报道[29,48]。在本研究中,MLR模型经基于赤池信息准则(Akaike information criterion,AIC)的逐步变量选择算法简化,在R中caret包中运行[49-52]。
(2) 分类与回归树(CART)。CART是一种重要且应用广泛的机器学习算法,既可用于分类,也可用于回归。目标变量是离散型变量则用分类树,目标变量是连续型变量则用回归树。决策树生成包括特征选择、树的生成以及剪枝组成三个环节,其生成准则为平方差最小化,即预测误差最小化。CART采用二分递归分割技术将训练集D分成两个子集D1和D2,并且使子集中各自的平方差最小。然后再分别在D1和D2中找类似的分界点,继续循环,直至满足终止条件。因此,CART算法生成的决策树是结构简洁的二叉树[18,53-54]。本研究使用R中rpart包构建CART模型[55]。
(3) Cubist(Cu)。Cubist是一种基于规则和面向预测的回归模型,它是M5模型树的扩展[56-57]。在执行预测时,Cubist首先创建一个树结构,然后回归树简化为由“如果”和“否则”引导的简洁、易理解的规则,每个规则均与一个多元线性模型关联[47]。因此,与CART不同,Cu预测是基于线性回归模型而不是离散值[51]。由于每个规则均基于特定条件,所以不同的多元线性模型能够捕获预测变量空间中的局部特征,因此通常较CART预测具有更好的精度[50-51]。本研究中,模型使用R环境中Cubist包的默认选项训练[58]。
(4) 随机森林(RF)。RF是由Therneau[55]在21世纪初作为CART模型的扩展而开发的一种树结构的集成学习(Ensemble learning)算法。它通过从训练数据中抽取的引导样本训练独立决策树并以它们的中值作为最终预测值,以此克服CART预测准确性方面的不足。RF随机选择特定数量、而非全部预测变量来拟合单个树,从而集成效率、减少过度拟合[38,53,56]。RF的突出优点包括:能够处理具有高维特征的输入样本而无需做降维处理;能够评估预测变量的相对重要性;能够获取到内部生成误差的无偏估计(OOB);能很好处理缺省值问题等。构建RF模型需要设置两个用户定义的参数:回归树的数量(ntree)和每次分裂时选择预测变量的数量(mtry)。本研究RF模型在R环境中的randomForest包中拟合[59]。
(5) 人工神经网络(ANN)。ANN由大量通过节点相互连接的、被称为处理单元的人工神经元组成。代表自变量的输入神经元根据内部加权系统分配权重后连接到单层或多层隐藏神经元,后者连接着代表目标土壤变量的输出神经元[21]。信息通过这个多层网络系统从一个神经元传输至另一个神经元。ANN使用一组称为反向传播的学习规则来修改输出结果,在此过程中ANN会在必要时返回各层以调整网络方程,并通过比较输入和输出计算残差。这种反向传播过程会重复进行直至误差最小[47,60]。本研究使用R环境中的nnet包拟合ANN模型[49]。
1.7 预测精度评价与不确定性分析(1) 预测精度评价。采用独立验证(hold out validation)评价选用ML模型基于不同样点规模和采样策略的获得的研究区表层土壤pH空间预测精度。本研究使用的精度评价指标包括均方根误差(RMSE)和决定系数(R2)。
(2) 不确定性分析。DSM技术突出的优势之一,就是可以对土壤类型和土壤属性空间预测与数字化制图输出结果进行定量的空间分析。土壤空间预测的不确定性包括三种类型:输入不确定性、结构不确定性和参数不确定性[60],三者共同导致了模型拟合目标土壤变量与协变量复杂定量关系的不准确和不完整性[61]。在本研究中,ML模型预测表层土壤pH的不确定性评估通过分析GlobalSoilMap技术规范中定义的90%预测区间(Prediction Interval,PI)的方式进行。所谓90%PI,即pH实测值10中有9次落入其中的预测值分布范畴[62]。本研究利用重抽样方法(bootstrap)计算pH预测结果的平均值和标准偏差μ±σ。假若pH预测结果为正态分布,利用μ±1.645σ计算90%PI。
2 结果与讨论 2.1 研究区耕地表层土壤pH描述性统计研究区表层土壤pH在4.1~9.4之间,均值为7.21,中值为7.5。变异系数(CV)为14%,指示中等变异性。偏度和峰度的值显示出左(负)偏斜分布,范围相对较短。K-S检验显示表层土壤pH实测值为非正态分布(P < 0.05)。
比较不同采样方法,不同样点规模训练集与总体2万样点之间表层土壤pH实测值的分布差异,采用Kruskal-Wallis H检验。结果表明只有分层随机采样下表层土壤pH实测值分布不完全相同,差异具有统计学意义(H=25.946,P < 0.05),其他各组表层与总体样点的土壤pH实测值分布相同。采用Bonferroni法校正显著性水平的事后两两比较发现,分层随机采样表层土壤pH分布只有在200密度下与2万密度的差距有统计学意义(调整后P < 0.05)。
2.2 耕地表层土壤pH预测精度与选用ML模型性能表现相关结果如图 3、图 4、图 5所示,从中可以发现几个重要现象:(1)使用任何采样方法和任何ML模型,研究区耕地表层土壤pH预测精度均表现出随土壤样点规模增大而提高的总体趋势;在样本规模小于2 000个时,各选用ML预测结果的R2快速提升,但当样本规模达到和超过2 000个时,R2提升速率突降并最终趋于稳定。相同的结果也被国内外其他学者在相关案例研究中获得[32,63-64]。这表明,在其他因素如ML模型性能以及协变量完整性、可靠性、准确性、分辨率等未得到实质性改善的前提下,无法单纯依靠增加样点规模、提高样点密度提高土壤属性空间预测精度。在现有技术条件和数据基础上,2 000个土壤样点可以认为是用于研究区耕地土壤表层pH空间预测的样点规模阈值。
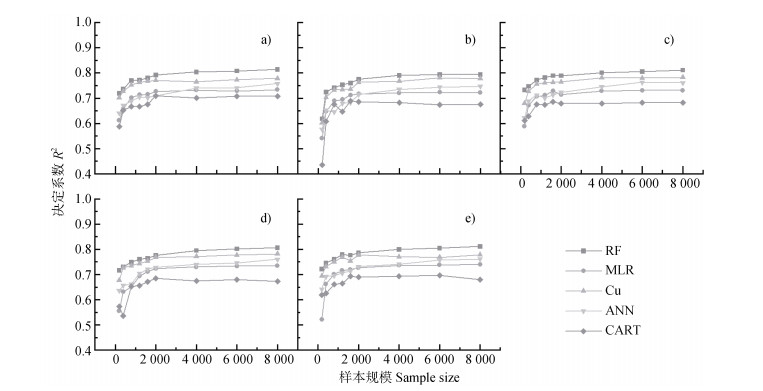
|
注:a)简单随机采样;b)分层随机采样;c)网格法采样;d)k-均值聚类采样;e)条件拉丁超立方采样。 Note: a)Simple random sampling; b)Stratified random sampling; c)Grid sampling; d)k-means cluster sampling; e)Conditioned latin hypercube sampling. 图 3 使用不同采样方法与不同样点规模时ML模型预测性能(R2) Fig. 3 R2 of topsoil pH prediction from the applied ML models with different sample sizes and sampling methods |
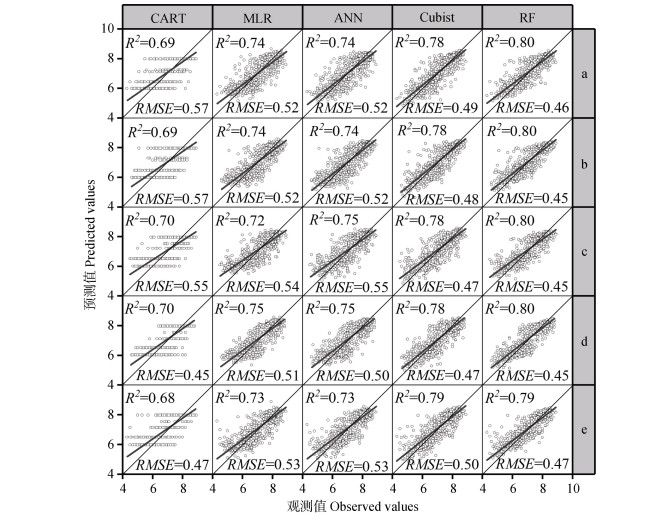
|
注:a)简单随机采样;b)分层随机采样;c)网格法采样;d)k-均值聚类采样;e)条件拉丁超立方采样。 Note: a)Simple random sampling; b)Stratified random sampling; c)Grid sampling; d)k-means cluster sampling; e)Conditioned latin hypercube sampling. 图 4 使用不同采样方法ML模型的预测性能(R2)(以土壤样点规模4 000个为例) Fig. 4 Prediction performance(R2)of ML models with different sampling methods(taking 4 000 soil samples as an example) |

|
注:a)简单随机采样;b)分层随机采样;c)网格法采样;d)k-均值聚类采样;e)条件拉丁超立方采样 Note: a)Simple random sampling; b)Stratified random sampling; c)Grid sampling; d)k-means cluster sampling; e)Conditioned latin hypercube sampling 图 5 使用不同采样方法与不同样点规模时ML模型预测精度(RMSE) Fig. 5 Prediction accuracy(RMSE)of topsoil pH prediction from the applied ML models with different sample sizes and sampling methods |
(2) 尽管所有ML模型随样点规模变化表现出一致的性能变化趋势,但不同的ML模型之间预测表现差别明显。在五种选用的ML模型,随机森林的性能表现最好,Cubist模型次之,二者在任何情况下预测性能均强于使用相同样点规模和采样方法的其他三类ML模型。在土壤样点规模大于2 000个的情况下,无论使用哪种采样方法,预测结果的R2均可稳定在0.75~0.80之间(图 3,图 4),即可以解释研究区75%~80%耕地表层土壤pH空间变异。上述两种ML模型,尤其是随机森林的性能与预测精度得到了之前国内外相关研究的普遍认可[18,28,37,49]。相对而言,多元线性回归与人工神经网络表现一般,二者在不同样点规模与采样方法时的预测精度互有优劣,R2维持在0.70~0.75之间。在所有选用的ML模型中,分类与回归模型表现最差,几乎在所有情况下R2均最低。究其原因,可能是由于分类与回归模型依赖单棵分类树难以有效拟合目标土壤变量与大批协变量之间复杂的非线性关系。
(3) 当土壤样点规模较小时,采样方法对模型表现和土壤目标变量空间预测精度的影响逐渐凸显。样点规模小于2 000个时,条件拉丁超立方采样方法下ML模型在使用同等样点规模时具有较好的性能表现;样点规模为1 000个时,随机森林和Cubist预测结果的R2仍能维持在0.78左右;当样点规模最小(200个)时,除MLR外,其他四种ML模型的R2均保持在0.60以上。比较而言,分层随机采样在样点规模较小时效果最差。当样点规模低于2 000个时,ML预测性能快速下降且表现极不稳定;当样点规模小至200个,使用分层随机采样的ML模型预测精度最低,R2低至0.44左右。以环境协变量高程(DEM)为例,如图 6所示,在密度较高时,各抽样方法下DEM均值较为接近,在低密度时,各采样方法下DEM均值波动明显。Kruskal-Wallis H检验结果表明,在条件拉丁方抽样方法下,各样点规模之间DEM分布相同,差距没有统计学意义(H=15.095,P=0.088);而在分层采样方法下,各样点规模之间的DEM分布不相同,差异具有统计学意义(H=58.816,P < 0.05),采用Bonferroni法校正显著性水平的事后两两比较发现,400样点规模开始与2万样点DEM分布有差异显著(调整后P < 0.05)。在样点规模较大时,各抽样方法均能与2万样点的环境协变量分布相同,当样点规模较少时,合理的采样方法会使环境协变量与总体样本分布相同。所以当采样预算不足、样点规模受限时,合理的采样策略可以在一定程度上提升土壤空间预测与数字化制图的精度[35,65-66]。
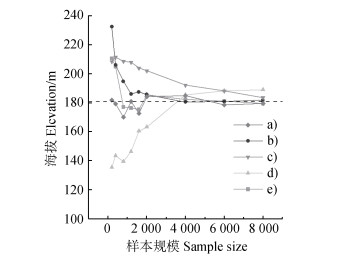
|
注:a)简单随机采样;b)分层随机采样;c)网格法采样;d)k-均值聚类采样;e)条件拉丁超立方采样。 Note: a)Simple random sampling; b)Stratified random sampling; c)Grid sampling; d)k-means cluster sampling; e)Conditioned latin hypercube sampling. 虚线表示2万样本土壤pH均值。The dotted line indicates the mean soil pH of the 20 thousand sample. 图 6 不同采样方法与不同样点规模时DEM均值 Fig. 6 Elevation of topsoil pH with different sample sizes and sampling methods |
(4) RMSE通常用于表征预测精度,它描述的主要是目标变量预测值与观测值之间的差异。而R2不同,它主要用来指示目标变量空间变异可以被协变量解释的程度以及模型的预测能力。以往的大量案例研究表明,预测土壤特性时具有较高R2的模型不一定必然输出更准确的预测结果[47]。但在本研究中,ML性能表现与表层土壤pH表现出几乎完全一致的随样点规模、采样方法不同而变化的趋势预测,R2越高,RMSE越小,反之亦然(图 3,图 5)。
2.3 耕地表层土壤pH预测制图以五个ML模型中性能表现最好的随机森林为例,输出基于不同样点规模和采样方法的研究区耕地表层土壤pH预测制图结果(图 7)。首先可以看到,使用任何样点规模和采样方法,随机森林模型输出的预测结果总体上均表现出非常相似的空间分布格局,Kruskal-Wallis H检验结果表明,分层随机抽样在200、400样本规模下的预测图与其他土壤pH预测图的差异较大(P < 0.05),而其他各土壤pH预测图的土壤表层pH分布相同,充分体现了随机森林模型数据挖掘和有效捕捉土壤目标变量与协变量之间复杂非线性关系的能力。其次,随着样点规模的增加,耕地表层土壤pH预测结果空间分布的细节特征逐渐清晰,主要体现在预测高值区与低值区的范围变大、且高值更高、低值更低。以2万样点与本研究中的最优预测方法(RF)的表层土壤pH预测图视作该地区“最准确”的土壤图,表 1展示不同样本规模、抽样方法的RF预测图与“最准确”的土壤图的相关系数。随着样点规模增加,各抽样方法的相关系数均增加,当样点规模超过2 000时,相关系数达到0.98以上。
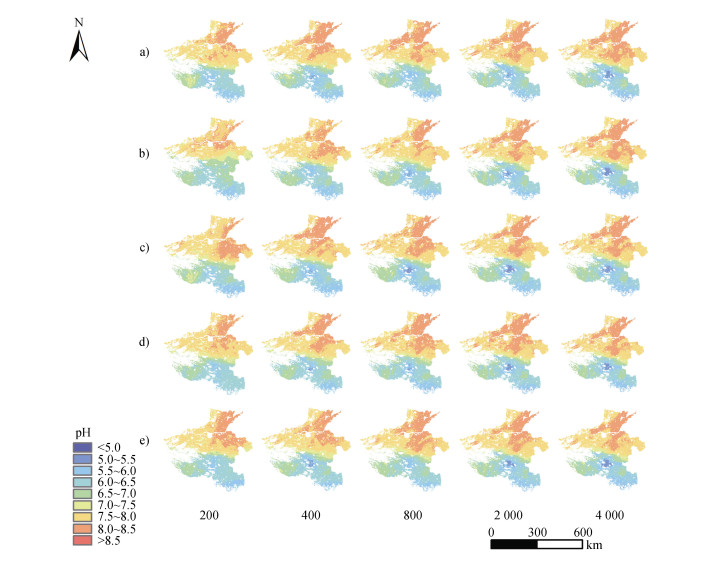
|
注:a)简单随机采样;b)分层随机采样;c)网格法采样;d)k-均值聚类采样;e)条件拉丁超立方采样。 Note: a)Simple random sampling; b)Stratified random sampling; c)Grid sampling; d)k-means cluster sampling; e)Conditioned latin hypercube sampling. 图 7 随机森林模型输出的研究区耕地表层土壤pH预测制图 Fig. 7 Predictive map of cropland topsoil pH in the study region generated from RF model |
|
|
表 1 不同样点规模、采样方法预测土壤表层pH与总体样点预测土壤表层pH相关系数 Table 1 Correlation coefficient between soil surface pH predicted by different site scales and sampling methods and soil surface pH predicted by overall sampling sites |
最后,以随机森林模型性能表现最好(R2= 0.80)、预测精度最高(RMSE=0.45)时的样点规模(4 000个)与采样方法(条件拉丁超立方抽样)组合为例,对研究区耕地表层土壤pH预测制图结果进行简要述评。根据预测,研究区耕地表层土壤pH变幅介于4.94~8.52之间,平均值为7.15,标准差为0.9,变异系数为13%。与样点描述性统计结果相比,pH变幅收窄,均值基本一致,变异系数小幅降低。从空间分布上看,研究区耕地表层土壤pH以沙颍河为界南北分异明显,北部以pH≥7.0的中性至碱性土壤为主,南部以pH < 7.0的酸性土壤为主。表层pH最高的土壤主要分布于黄河古道、沿黄背河洼地、研究区西北石灰性母岩区域,表土pH最低的土壤分布于沙颍河上中游南岸地区,这一区域也是过去研究区内10年耕地表层土壤pH下降最快的区域[40]。
2.4 不确定性图 8为随机森林模型基于不同样点规模输出研究区耕地表层土壤pH预测结果均值和第5百分位、第95百分位预测结果的空间可视化表达。常用的ML模型输出结果一般是预测结果的均值,而5百分位、95百分位预测结果可分别视为低估和高估的界限。
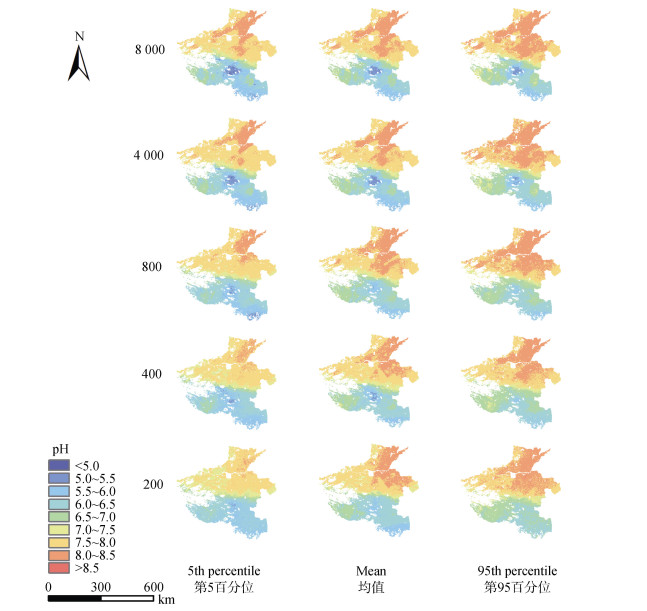
|
图 8 基于不同样点规模(条件拉丁超立方采样)随机森林输出的表层土壤pH预测结果及不确定性制图 Fig. 8 Uncertainty maps of cropland topsoil pH prediction from ML with different sample sizes(sampled by cLHS algorithm) |
按照Globalsoilmap技术规程,本研究将验证样点表层土壤pH观测值落入90%预测区域的百分比作为评价模型可靠性的依据,观测值落入90%预测区间的百分比越高,真实的预测区间范围越大,预测的不确定性就越小。有关计算结果见表 2,从中可以看到,使用不同样点规模时,平均73.9%的验证样点pH观测值落入90%预测区间,表明随机森林模型的稳定性被轻微高估,但模型可靠性在可接受范围内。分析表 2中数据发现,当样点规模低至200个和400个时,验证样点pH实测值落入90%预测区间的百分比并不比样点规模很大(如2 000个和4 000个)时低,表明样点规模小于特定阈值可对模型预测精度产生显著影响,但却不影响模型的稳定性与可靠性。
|
|
表 2 验证样点表层土壤pH实测值落入90%预测区间百分比 Table 2 Percentage of the topsoil pH observations that fell inside the 90% PI/% |
(1) 无论采用哪种采样方法,用于研究区耕地表层土壤pH空间预测的五种ML模型的性能表现和输出结果精度均随土壤样点规模增大而提高,当样点规模达到特定阈值(2 000个)时,大多数模型性能表现趋于稳定,预测精度也不再有明显提升。这表明,进一步提高土壤目标变量空间预测与数字化制图精度无法单纯依靠扩大样点规模、加大样点密度实现,需要同时提升模型预测性能和协变量数据质量。(2)具有树结构的机器学习模型随机森林和Cubist在本案例中表现突出,尤其是随机森林。作为一种具有树结构的集成学习算法,随机森林的性能表现与预测精度优于使用同样样点规模和采样方法的其他四种ML模型,表明其在定量模拟目标土壤变量与环境协变量之间复杂非线性关系方面具有优势。(3)当样点规模足够大时,采样方法对ML模型性能表现和表层土壤pH预测精度的影响可以忽略不计,这是因为密集分布的样点已经包含足够多的环境协变量信息,无论采用哪种采样方法均可以体现总样点的协变量分布;当样点规模小于特定阈值时,采样方法对模型表现与预测结果影响显著。因此,当采样预算不足时,需要制订一个更加科学、合理的采样策略。(4)不确定性分析结果表明,随机森林输出耕地表层土壤pH空间预测结果的可靠性在可接受范围内;样点规模对随机森林模型的稳定性与预测结果的可靠性没有显著影响。
| [1] |
FAO and ITPS. 2015. Status of the World's Soil Resources(SWSR)– Main Report. Food and Agriculture Organization of the United Nations and Intergovernmental Technical Panel on Soils, Rome, Italy, ISBN 978-92-5-109004-6.
(  0) 0) |
| [2] |
FAO. 2017. Soil Organic Carbon: the hidden potential. Food and Agriculture Organization of the United Nations, Rome, Italy. ISBN 978-92-5-109681-9.
(  0) 0) |
| [3] |
Bouma J, Montanarella L, Evanylo G. The challenge for the soil science community to contribute to the implementation of the UN Sustainable Development Goals[J]. Soil Use and Management, 2019, 35(4): 538-546. DOI:10.1111/sum.12518
(  0) 0) |
| [4] |
Berhe A A. Drivers of soil change[M]//Developments in Soil Science. Elsevier, 2019, 36: 27—42.
(  0) 0) |
| [5] |
McBratney A B, Field D, Morgan C L S, et al. On soil capability, capacity, and condition[J]. Sustainability, 2019, 11(12): 3350. DOI:10.3390/su11123350
(  0) 0) |
| [6] |
Kuzyakov Y, Zamanian K. Reviews and syntheses: Agropedogenesis–humankind as the sixth soil-forming factor and attractors of agricultural soil degradation[J]. Biogeosciences, 2019, 16(24): 4783-4803. DOI:10.5194/bg-16-4783-2019
(  0) 0) |
| [7] |
Román Dobarco M, McBratney A, Minasny B, et al. A framework to assess changes in soil condition and capability over large areas[J]. Soil Security, 2021, 4: 100011. DOI:10.1016/j.soisec.2021.100011
(  0) 0) |
| [8] |
Forkuor G, Hounkpatin O K L, Welp G, et al. High resolution mapping of soil properties using remote sensing variables in south-western Burkina Faso: A comparison of machine learning and multiple linear regression models[J]. PLoS One, 2017, 12(1): e0170478. DOI:10.1371/journal.pone.0170478
(  0) 0) |
| [9] |
Hartemink A E, McBratney A. A soil science renaissance[J]. Geoderma, 2008, 148(2): 123-129. DOI:10.1016/j.geoderma.2008.10.006
(  0) 0) |
| [10] |
Rodrigo-Comino J, Senciales J M, Cerdà A, et al. The multidisciplinary origin of soil geography: A review[J]. Earth-Science Reviews, 2018, 177: 114-123. DOI:10.1016/j.earscirev.2017.11.008
(  0) 0) |
| [11] |
Arrouays D, McBratney A, Bouma J, et al. Impressions of digital soil maps: The good, the not so good, and making them ever better[J]. Geoderma Regional, 2020, 20: e00255. DOI:10.1016/j.geodrs.2020.e00255
(  0) 0) |
| [12] |
Thompson J A, Kienast-Brown S, D'Avello T, et al. Soils2026 and digital soil mapping - A foundation for the future of soils information in the United States[J]. Geoderma Regional, 2020, 22: e00294. DOI:10.1016/j.geodrs.2020.e00294
(  0) 0) |
| [13] |
Minasny B, McBratney A B. Digital soil mapping: A brief history and some lessons[J]. Geoderma, 2016, 264: 301-311. DOI:10.1016/j.geoderma.2015.07.017
(  0) 0) |
| [14] |
Lagacherie P, McBratney A B. Chapter 1 spatial soil information systems and spatial soil inference systems: Perspectives for digital soil mapping[J]. Developments in Soil Science, 2006, 31: 3-22.
(  0) 0) |
| [15] |
McBratney A B, Mendonça Santos M L, Minasny B. On digital soil mapping[J]. Geoderma, 2003, 117(1/2): 3-52.
(  0) 0) |
| [16] |
Arrouays D, Lagacherie P, Hartemink A E. Digital soil mapping across the globe[J]. Geoderma Regional, 2017, 9: 1-4. DOI:10.1016/j.geodrs.2017.03.002
(  0) 0) |
| [17] |
Dharumarajan S, Kalaiselvi B, Suputhra A, et al. Digital soil mapping of key Global Soil Map properties in Northern Karnataka Plateau[J]. Geoderma Regional, 2020, 20: e00250. DOI:10.1016/j.geodrs.2019.e00250
(  0) 0) |
| [18] |
Keskin H, Grunwald S, Harris W G. Digital mapping of soil carbon fractions with machine learning[J]. Geoderma, 2019, 339: 40-58. DOI:10.1016/j.geoderma.2018.12.037
(  0) 0) |
| [19] |
Hengl T, Mendes de Jesus J, Heuvelink G B M, et al. Soil Grids 250m: Global gridded soil information based on machine learning[J]. PLoS One, 2017, 12(2): e0169748. DOI:10.1371/journal.pone.0169748
(  0) 0) |
| [20] |
Wadoux A M J C, Minasny B, McBratney A B. Machine learning for digital soil mapping: Applications, challenges and suggested solutions[J]. Earth-Science Reviews, 2020, 210: 103359. DOI:10.1016/j.earscirev.2020.103359
(  0) 0) |
| [21] |
Heung B, Ho H C, Zhang J, et al. An overview and comparison of machine-learning techniques for classification purposes in digital soil mapping[J]. Geoderma, 2016, 265: 62-77. DOI:10.1016/j.geoderma.2015.11.014
(  0) 0) |
| [22] |
Mondejar J P, Tongco A F. Estimating topsoil texture fractions by digital soil mapping - A response to the long outdated soil map in the Philippines[J]. Sustainable Environment Research, 2019, 29: 31. DOI:10.1186/s42834-019-0032-5
(  0) 0) |
| [23] |
Gutiérrez Á G, Schnabel S, Lavado Contador J F. Using and comparing two nonparametric methods(CART and MARS)to model the potential distribution of gullies[J]. Ecological Modelling, 2009, 220(24): 3630-3637. DOI:10.1016/j.ecolmodel.2009.06.020
(  0) 0) |
| [24] |
Coelho F F, Giasson E, Campos A R, et al. Geographic object-based image analysis and artificial neural networks for digital soil mapping[J]. Catena, 2021, 206: 105568. DOI:10.1016/j.catena.2021.105568
(  0) 0) |
| [25] |
Lamichhane S, Kumar L, Wilson B. Digital soil mapping algorithms and covariates for soil organic carbon mapping and their implications: A review[J]. Geoderma, 2019, 352: 395-413. DOI:10.1016/j.geoderma.2019.05.031
(  0) 0) |
| [26] |
Pouladi N, Møller A B, Tabatabai S, et al. Mapping soil organic matter contents at field level with Cubist, Random Forest and kriging[J]. Geoderma, 2019, 342: 85-92. DOI:10.1016/j.geoderma.2019.02.019
(  0) 0) |
| [27] |
Zhou Y, Zhao X M, Guo X. Prediction of total nitrogen distribution in surface soil based on multi-source auxiliary variables and random forest approach (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(2): 451-460. [周洋, 赵小敏, 郭熙. 基于多源辅助变量和随机森林模型的表层土壤全氮分布预测[J]. 土壤学报, 2022, 59(2): 451-460.]
(  0) 0) |
| [28] |
Hengl T, Nussbaum M, Wright M N, et al. Random forest as a generic framework for predictive modeling of spatial and spatio-temporal variables[J]. PeerJ, 2018, 6: e5518. DOI:10.7717/peerj.5518
(  0) 0) |
| [29] |
Veronesi F, Schillaci C. Comparison between geostatistical and machine learning models as predictors of topsoil organic carbon with a focus on local uncertainty estimation[J]. Ecological Indicators, 2019, 101: 1032-1044. DOI:10.1016/j.ecolind.2019.02.026
(  0) 0) |
| [30] |
Rial M, Cortizas A M, Rodríguez-Lado L. Understanding the spatial distribution of factors controlling topsoil organic carbon content in European soils[J]. Science of the Total Environment, 2017, 609: 1411-1422. DOI:10.1016/j.scitotenv.2017.08.012
(  0) 0) |
| [31] |
Loiseau T, Arrouays D, Richer-de-Forges A C, et al. Density of soil observations in digital soil mapping: A study in the Mayenne region, France[J]. Geoderma Regional, 2021, 24: e00358. DOI:10.1016/j.geodrs.2021.e00358
(  0) 0) |
| [32] |
Somarathna P D S N, Minasny B, Malone B P. More data or a better model? figuring out what matters most for the spatial prediction of soil carbon[J]. Soil Science Society of America Journal, 2017, 81(6): 1413-1426. DOI:10.2136/sssaj2016.11.0376
(  0) 0) |
| [33] |
Long J, Liu Y L, Xing S H, et al. Effects of sampling density on interpolation accuracy for farmland soil organic matter concentration in a large region of complex topography[J]. Ecological Indicators, 2018, 93: 562-571. DOI:10.1016/j.ecolind.2018.05.044
(  0) 0) |
| [34] |
Lai Y Q, Wang H L, Sun X L. A comparison of importance of modelling method and sample size for mapping soil organic matter in Guangdong, China[J]. Ecological Indicators, 2021, 126: 107618. DOI:10.1016/j.ecolind.2021.107618
(  0) 0) |
| [35] |
Ma T W, Brus D J, Zhu A X, et al. Comparison of conditioned Latin hypercube and feature space coverage sampling for predicting soil classes using simulation from soil maps[J]. Geoderma, 2020, 370: 114366. DOI:10.1016/j.geoderma.2020.114366
(  0) 0) |
| [36] |
Sena N C, Veloso G V, Lopes A O, et al. Soil sampling strategy in areas of difficult acess using the cLHS method[J]. Geoderma Regional, 2021, 24: e00354. DOI:10.1016/j.geodrs.2020.e00354
(  0) 0) |
| [37] |
Wadoux A M J C, Brus D J, Heuvelink G B M. Sampling design optimization for soil mapping with random forest[J]. Geoderma, 2019, 355: 113913. DOI:10.1016/j.geoderma.2019.113913
(  0) 0) |
| [38] |
Walvoort D J J, Brus D J, de Gruijter J J. An R package for spatial coverage sampling and random sampling from compact geographical strata by k-means[J]. Computers & Geosciences, 2010, 36(10): 1261-1267.
(  0) 0) |
| [39] |
Wu K N, Li L, Ju B, et al. Soil Series of China·Henan volume (In Chinese). Beijing: Science Press, 2019: 476. [吴克宁, 李玲, 鞠兵, 等. 中国土系志·河南卷[M]. 北京: 科学出版社, 2019: 476.]
(  0) 0) |
| [40] |
Wu Z F, Sun X M, Sun Y Q, et al. Soil acidification and factors controlling topsoil pH shift of cropland in central China from 2008 to 2018[J]. Geoderma, 2022, 408: 115586. DOI:10.1016/j.geoderma.2021.115586
(  0) 0) |
| [41] |
Team R C. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria[J/OL]. http://www.R-project.org/, 2020.
(  0) 0) |
| [42] |
McKenzie N J, Ryan P J. Spatial prediction of soil properties using environmental correlation[J]. Geoderma, 1999, 89(1/2): 67-94.
(  0) 0) |
| [43] |
Huang S H, Pu L J, Xie X F, et al. Review and outlook of designing of soil sampling for digital soil mapping (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(2): 259-272. [黄思华, 濮励杰, 解雪峰, 等. 面向数字土壤制图的土壤采样设计研究进展与展望[J]. 土壤学报, 2020, 57(2): 259-272.]
(  0) 0) |
| [44] |
Pebesma E J, Bivand R S. Classes and methods for spatial data in R. R News, 2005, 5(2): 9–13.
(  0) 0) |
| [45] |
Minasny B, McBratney A B. A conditioned Latin hypercube method for sampling in the presence of ancillary information[J]. Computers & Geosciences, 2006, 32(9): 1378-1388.
(  0) 0) |
| [46] |
Roudier P. Package "clhs". R package version 0.7-0. https://CRAN.R-project.org/package=clhs. 2018.
(  0) 0) |
| [47] |
Khaledian Y, Miller B A. Selecting appropriate machine learning methods for digital soil mapping[J]. Applied Mathematical Modelling, 2020, 81: 401-418. DOI:10.1016/j.apm.2019.12.016
(  0) 0) |
| [48] |
Pahlavan-Rad M R, Dahmardeh K, Hadizadeh M, et al. Prediction of soil water infiltration using multiple linear regression and random forest in a dry flood plain, eastern Iran[J]. Catena, 2020, 194: 104715. DOI:10.1016/j.catena.2020.104715
(  0) 0) |
| [49] |
Venables W N, Ripley B D. Modern applied statistics with S-plus[M]. New York: Springer, 2002.
(  0) 0) |
| [50] |
Hounkpatin O K L, Op de Hipt F, Bossa A Y, et al. Soil organic carbon stocks and their determining factors in the Dano catchment(Southwest Burkina Faso)[J]. Catena, 2018, 166: 298-309. DOI:10.1016/j.catena.2018.04.013
(  0) 0) |
| [51] |
Odhiambo B O, Kenduiywo B K, Were K. Spatial prediction and mapping of soil pH across a tropical Afro-montane landscape[J]. Applied Geography, 2020, 114: 102129. DOI:10.1016/j.apgeog.2019.102129
(  0) 0) |
| [52] |
Kuhn M, Wing J, Weston S et al. The caret package: Classification and regression training. R package version 6.0-47. 2015.
(  0) 0) |
| [53] |
Lamsal S, Grunwald S, Bruland G L, et al. Regional hybrid geospatial modeling of soil nitrate-nitrogen in the Santa Fe River Watershed[J]. Geoderma, 2006, 135: 233-247. DOI:10.1016/j.geoderma.2005.12.009
(  0) 0) |
| [54] |
Krzywinski M, Altman N. Classification and regression trees[J]. Nature Methods, 2017, 14(8): 757-758. DOI:10.1038/nmeth.4370
(  0) 0) |
| [55] |
Therneau T, Atkinson B. rpart: Recursive partitioning and regression trees. R package version 4.1-15. 2019.
(  0) 0) |
| [56] |
Quinlan R. Learning with continuous classes. Proceedings of the 5th Australian Joint Conference on Artificial Intelligence, 1992: 343—348.
(  0) 0) |
| [57] |
Quinlan R. C4.5: Programs for machine learning. Morgan Kaufmann Publishers. 1993.
(  0) 0) |
| [58] |
Kuhn M, Quinlan R. Cubist: Rule-and instance-based regression modeling. R package version 0.2.3[J]. 2020.
(  0) 0) |
| [59] |
Liaw A, Wiener M. Classification and regression by random Forest. R News, 2007, 2(3): 18—22.
(  0) 0) |
| [60] |
Were K, Bui D T, Dick Ø B, et al. A comparative assessment of support vector regression, artificial neural networks, and random forests for predicting and mapping soil organic carbon stocks across an Afromontane landscape[J]. Ecological Indicators, 2015, 52: 394-403. DOI:10.1016/j.ecolind.2014.12.028
(  0) 0) |
| [61] |
McBratney A B, Minasny B, Cattle S R, et al. From pedotransfer functions to soil inference systems[J]. Geoderma, 2002, 109(1/2): 41-73.
(  0) 0) |
| [62] |
Arrouays D, Grundy M G, Hartemink A E, et al. Global Soil Map: Toward a fine-resolution global grid of soil properties. Advances in Agronomy 2014, 125: 93—134.
(  0) 0) |
| [63] |
Lagacherie P, Arrouays D, Bourennane H, et al. Analysing the impact of soil spatial sampling on the performances of Digital Soil Mapping models and their evaluation: A numerical experiment on Quantile Random Forest using clay contents obtained from Vis-NIR-SWIR hyperspectral imagery[J]. Geoderma, 2020, 375: 114503. DOI:10.1016/j.geoderma.2020.114503
(  0) 0) |
| [64] |
Li N, Arshad M, Zhao D X, et al. Determining optimal digital soil mapping components for exchangeable calcium and magnesium across a sugarcane field[J]. Catena, 2019, 181: 104054. DOI:10.1016/j.catena.2019.04.034
(  0) 0) |
| [65] |
Rudiyanto, Minasny B, Setiawan B I, et al. Open digital mapping as a cost-effective method for mapping peat thickness and assessing the carbon stock of tropical peatlands[J]. Geoderma, 2018, 313: 25-40. DOI:10.1016/j.geoderma.2017.10.018
(  0) 0) |
| [66] |
Zhang L, Yang L, Cai Y Y, et al. A multiple soil properties oriented representative sampling strategy for digital soil mapping[J]. Geoderma, 2022, 406: 115531. DOI:10.1016/j.geoderma.2021.115531
(  0) 0) |
 2023, Vol. 60
2023, Vol. 60


