几丁质是自然界中含量仅次于纤维素的第二大生物聚合物,是无脊椎动物外骨骼和真菌细胞壁的主要成分,广泛存在于土壤中[1]。由于几丁质是土壤碳氮主要来源之一,土壤中几丁质的生物降解对陆地生态系统,尤其是农业土壤的养分循环有重要贡献[2]。同时几丁质也被称作控释氮肥,可通过释放铵态氮或其他形式氮素来促进植物生长[3]。此外,几丁质调控也是一种先进的农业管理措施,可加强植物对病虫害的防御,提高作物产量[4]。几丁质降解微生物能够分解线虫、昆虫和致病性真菌的几丁质结构,从而成为一种生物防治剂[5]。几丁质降解过程中首先被内切几丁质酶分解成水溶性低聚体,再被外切几丁质酶(脱乙酰几丁质酶和β-N-乙酰葡糖氨糖苷酶)分解成二聚体和单体[6-7]。根据氨基酸序列,几丁质酶可分为不同的糖苷水解酶(GH)家族,如GH18、GH19和GH20[8]。GH18内切几丁质酶存在于细菌、真菌、动物和部分植物内,GH19主要在植物和细菌中[9-10]。GH18几丁质酶形成三个深度系统发育链,分别由chiA、chiВ和chiC三个基因控制。其中chiA最大,所以通常使用chiA基因作为几丁质降解微生物多样性的标记[11]。
土壤环境中存在多种几丁质降解菌,国内外对几丁质降解菌的分离、纯化和鉴定进行了大量研究,其菌群主要以细菌为主,常见的包括放线菌、厚壁菌和变形菌等,在农业生防和培肥中作为生物防治剂、土壤改良剂等得到广泛应用[12]。放线菌是重要的土壤腐生菌,能生产抗生素、次生代谢物以及合成几丁质酶,是几丁质降解菌群中最重要的种群之一,其数量超过已检测陆地生态系统几丁质降解菌基因组公共数据库的50%[13]。厚壁菌群中的很多芽孢杆菌是生产几丁质酶的菌株,可筛选出作为微生物肥料,施用后能促进玉米生长[14]。灰化棕壤土表层分离出的几丁质降解菌中发现单胞菌和色杆菌能抑制马铃薯包囊线虫卵的孵化[15]。假单胞菌能分离得到GH18几丁质酶,具有体外抗真菌几丁质溶解活性,有效增加鹰嘴豆的结瘤数[16]。土壤几丁质降解菌群与土壤含水量、可溶性有机碳氮、有效磷和铵态氮等养分含量显著相关[17]。这些结果显示,土壤中几丁质降解菌存在极高生物多样性,农业管理的诸多措施中以不同耕作方式对土壤扰动最大,其对几丁质降解微生物和酶活性的影响值得深入研究。
不同耕作方式通过改变耕层土壤结构和作物残茬在土壤中的空间分布,影响土壤微生物群落和酶活性[18-19]。已有报道指出,免耕5~20 cm土层较0~5 cm表层细菌多样性显著降低,放线菌相对丰度显著提高,而旋耕垄作两土层间差异均不显著,同时底层细菌多样性明显高于免耕[20]。全球141个试验的Meta分析发现免耕后显著降低放线菌相对丰度,变形菌相对丰度差异不显著,施氮量高于200 kg·hm–2会较翻耕降低厚壁菌相对丰度[21]。19年免耕后进行2年深松处理,0~20 cm土层chiA基因丰度较连续免耕差异不显著[22]。免耕表层土壤几丁质酶活性显著高于翻耕处理[23]。以上研究多通过测定16S rRNA分析不同耕作方式下细菌群落结构,很少测定chiA功能基因直接反应几丁质降解菌群特征,也未明晰菌群与几丁质酶活性的互作关系。此外,研究大多集中于0~20 cm土层,很少对深层土壤进一步探索。基于以上研究背景和现状,本研究依托黑土区耕法长期定位平台,采用荧光定量和高通量测序技术分析长期耕作对农田黑土0~40 cm土层几丁质降解菌群及酶活性的影响,研究结果将为东北黑土保育和适宜耕作措施的选择与应用提供理论依据。
1 材料与方法 1.1 研究区概况定位试验位于吉林省农业科学院公主岭院内试验田(43°52.23′N,124°81.18′E),该地区是中温带大陆性季风气候,年均气温4.5℃,有效积温2 800℃,太阳有效辐射4 800 MJ·m–2,年均降雨量567 mm。土壤类型为中层黑土、壤质黏土。
1.2 试验设计耕法长期定位试验始于1983年,设灭茬起垄(CT)、免耕(NT)、间隔深松(ST)和翻耕(MP)4种耕作措施。具体田间机械操作如下:灭茬起垄为秋季将秸秆移出农田后旋耕灭茬起垄,春季播种机等行距65 cm播种;免耕为秋季收获后留茬40 cm,春季免耕播种机等行距65 cm播种,苗带行间交替休闲,生育期内不进行任何田间机械作业;间隔深松为秋季收获后留茬40 cm,春季免耕播种机宽窄行播种,宽行90 cm,窄行40 cm,宽窄行交替休闲,并在玉米拔节期采用刀踵式深松铲进行宽行深松35 cm;翻耕为秋季秸秆移除后深翻35 cm埋茬后耙平镇压,春季播种机等行距65 cm机械播种。各小区面积1 950 m2,3次重复,20行区,150 m行长,供试玉米品种为翔玉998。每年5月1日左右播种,10月1日左右收获。种植密度为60 000株·hm–2。施纯N 225 kg·hm–2、P2O5 90 kg·hm–2和K2O 80 kg·hm–2,全部的磷、钾肥和1/3氮肥作基肥,2/3氮肥在玉米拔节前追施(6月中下旬)。耕法定位试验除耕整地和播种为机械作业外,打药、除草、追肥、收获及秸秆移除均为人工操作,避免其他环节机械压实对耕层产生影响。
1.3 样品采集及测定方法土壤样品于2020年10月中旬玉米收获后秋整地前采集,每个处理3次重复,各小区使用S型采样法分别采集5个苗带和5个行间0~20 cm和20~40 cm土层混合样品2 kg、原状土2 kg和环刀样品。混合样品分成两份,一份存储在–80℃下用于荧光定量PCR测定和DNA提取。一份自然风干,过筛研磨后用于土壤理化性质和几丁质酶活性测定。原状土带回实验室后将大土块按自然裂痕剥离为1 cm3左右小土块,待自然风干后再挑除粗根系和小石块。
将风干原状土样分别过孔径为5 mm和2 mm的筛子,土样被分为 > 5 mm、5~2 mm、< 2 mm三个级别。然后计算三个级别土样在原状土中所占比例,取混合土样50 g,慢慢放入振荡式土壤团粒筛分仪(DM200-Ⅳ型,中国),筛网没入水面的套筛(孔径依次分别为2,0.25及0.053 mm)顶部,浸泡10 min,再以3 cm振幅、20次·min–1的运动频率振荡2 min后,从上到下依次取下套筛,收集筛上土样于60℃烘干称重保存。土壤基本理化性质按照《土壤农化分析》的方法测定[24]:土壤酸碱度采用土水比1︰1电极法;容重采用烘干法;有机碳采用高温外加热重铬酸钾氧化容量法;全氮用凯氏法;全磷用HClO4-H2SO4消煮—钼锑抗比色法;有效氮采用碱解扩散法;有效磷采用Olsen法;几丁质酶采用酶标法测定。测定结果见表 1。
使用FastDNA® SPIN kit试剂盒提取土壤总DNA,称取–80℃下保存的新鲜土壤样品0.5 g,按照说明书步骤,使用NanoDropND-1000光谱仪测定260 nm的OD值,检测提取DNA的浓度和纯度。使用ABI7500实时PCR系统对chiA基因的拷贝数进行测定。chiA基因的引物为chif2(5′-GACGGCAT CGACATCGATTGG-3′)和chir(5′-CSGTCCAGCCG CGSCCRTA-3′)[25]。扩增程序为98℃预变性5 min;95℃变性60 s,35个循环,60℃退火30 s;72℃延伸60 s;每个样品3次重复。qPCR反应体系包括10 μL(5×SYBR® Premix Ex-TaqTM)PCR缓冲液,正反引物各0.8 μL,1 μL DNA模板,超纯水(ddH2O)补充至20 μL。每个反应后绘制溶解曲线分析,用质量分数为1.5%的琼脂糖凝胶电泳确保PCR扩增的特异性。样品的基因丰度基于干土重进行计算,单位为copies·g–1。
|
|
表 1 不同耕作措施0~40 cm土层理化性质 Table 1 Soil physicochemical properties of 0~40 cm soil layer under different tillage practices |
对上述引物进一步进行扩增子测序测定chiA几丁质降解菌群落多样性和物种组成。利用QIIME2软件DADA2方法进行去引物、质量过滤、去噪、拼接和去嵌合体等步骤。共获得3 080 842条有效DNA序列,每个样本序列数在77 970~247 454之间,按照97%的序列相似性对所有序列进行聚类,共获得27 131个可操作分类单元(Operational taxonomic unit,OTU),各样品文库的覆盖率均达到99%以上。选择每个OTU中最具代表性序列进行系统发育分类。利用本地化的nt或nr(2019.8下载,ftp://ftp.ncbi.nih.gov/blast/db/)数据库进行注释。利用QIIME2软件分析各样本多样性和物种分类。用Chao 1丰富度指数和Shannon多样性指数表征chiA群落Alpha多样性,用PCoA排序表征chiA群落Beta多样性。
1.4 数据处理水稳性团聚体稳定性指标使用平均重量直径(MWD)[26]表示:
| $ \text{MWD}=\sum\limits_{i = 1}^n {{x_i}{w_i}} $ |
式中,xi为任一级别范围内团聚体的平均直径(mm),wi为对应于xi的团聚体百分含量。
在SPSS 25.0软件中利用单因素方差分析chiA基因丰度和α多样性在不同耕作措施中的差异,采用Duncan法进行多重比较。用PCoA排序的第一轴(PCoA1)表征微生物的β多样性。用Pearson相关分析解析环境因子与chiA基因丰度和多样性的关系。采用Canoco 4.5软件中冗余分析(Redundancy anlysis,RDA)解析环境因子与微生物群落结构的相关性。采用Amos 24.0构建结构方程[27]揭示土壤环境因子和几丁质降解菌群对几丁质酶活性的调控机制,并利用比较拟合指数(Comparative fitting index,CFI))、拟合优度(Goodness-of-fit,GFI)和近似误差均方根(Root mean square error of approximation,RMSEA)评价结构方程模型拟合度。采用Origin 9.0进行制图。
2 结果 2.1 不同耕作措施下chiA基因丰度和微生物群落多样性由图 1可知,chiA基因丰度在4种耕作措施中差异显著,chiA基因拷贝数在两土层中变化幅度为6 867~20 888 copies·g–1。在0~20 cm土层中,NT处理的chiA基因丰度显著高于其他3种耕作措施,提高幅度为35.8%~80.9%;在20~40 cm土层中,ST和MP处理的chiA基因丰度较CT和NT处理更高。
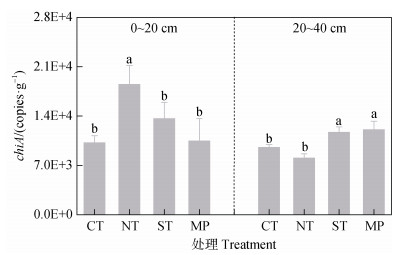
|
注:CT、NT、ST、MP分别代表灭茬起垄、免耕、间隔深松、翻耕。下同。 Note: CT、NT、ST、MP means conventional tillage, no-tillage, sub-soiling tillage, and moldboard plowing tillage, respectively. The same below. 图 1 不同耕作措施下chiA基因丰度 Fig. 1 Gene abundance of chiA in different tillage practices |
不同耕作措施几丁质降解菌群的Chao 1丰富度和Shannon多样性指数见图 2。在0~20 cm土层,ST处理Chao 1丰富度显著高于CT和MP,而MP较NT和ST处理显著提高Shannon多样性,此外NT处理Shannon多样性显著低于CT处理。在20~40 cm土层,深耕(ST和MP)对比NT显著提高Chao 1丰富度,同时,NT会显著降低Shannon多样性指数。主坐标分析(PCoA)表明(图 3),PCoA1轴和PCoA2轴分别解释了土壤几丁质降解菌群落结构差异的35.5%和21.6%,不同耕作措施和土壤深度下几丁质降解菌群β多样性具有显著差异,不同处理及深度间chiA微生物群落聚集为8组,表明不同耕作措施和采样深度对几丁质降解菌群落结构有显著影响。

|
图 2 不同耕作措施下chiA微生物的alpha多样性 Fig. 2 Alpha diversity of chiA microbial in different tillage practices |

|
注:20和40分别代表 0~20 cm土层和20~40 cm土层。下同。 Note: 20 and 40 means 0~20 cm soil layer and 20~40 cm soil layer, respectively. The same below. 图 3 几丁质降解菌的主坐标分析 Fig. 3 Principal coordinates analysis(PCoA)ordinations of microbial community for chitin-degrading bacteria |
相关分析表明(表 2):chiA基因丰度与土壤平均重量直径、有机碳、全氮、全磷、有效氮和有效磷含量呈显著正相关,与土壤pH显著负相关(P < 0.05);几丁质降解菌群β多样性与土壤容重和pH显著正相关,与土壤有机碳、全氮、全磷、有效氮和有效磷含量呈极显著负相关(P < 0.01)。几丁质降解菌群α多样性与已测定的土壤理化指标相关性不显著。
|
|
表 2 土壤理化性质与chiA微生物多样性指标的相关性 Table 2 Pearson correlation of soil physicochemical properties and chiA microbial diversity index |
不同耕作措施和采样深度下土壤几丁质降解菌群中总共检测出优势菌群(相对丰度 > 1%)为2个菌门,2个菌纲,6个菌目,5个菌科和3个菌属(图 4a)。在0~20 cm土层,NT较MP处理显著提高变形菌门相对丰度。在20~40 cm土层,NT较其他耕作处理显著提高放线菌门相对丰度,降低变形菌门的相对丰度,增幅和降幅度分别为8.3%~9.9%和49.2%~60.1%;NT和ST处理γ变形菌纲相对丰度显著低于CT;此外,NT处理奈瑟菌目、黄单孢菌目、色杆菌科和拟无枝酸菌属相对丰度同样低于CT处理;而MP较其他处理显著提高β变形菌纲和伯克霍尔德氏菌目相对丰度。在0~40 cm土层,NT较其处理显著提高小单孢菌目相对丰度,降低链霉菌目相对丰度,增幅和降幅分别为28.4%~84.7%和51.9%~65.1%;此外,NT还较CT处理显著降低假诺卡氏菌目相对丰度。
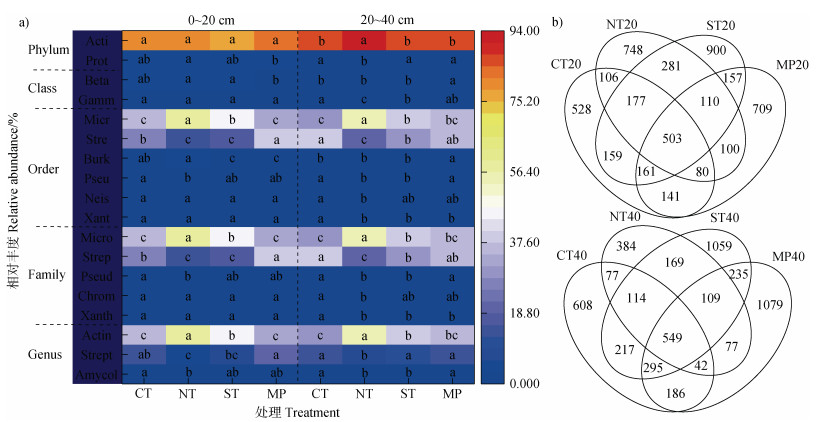
|
注:Phylum,Class,Order,Family,Genus分别代表门水平、纲水平、目水平、科水平、属水平;Acti放线菌门,Prot变形菌门,Beta β变形菌纲,Gamm γ变形菌纲,Micr小单孢菌目,Stre链霉菌目,Burk伯克霍尔德氏菌目,Pseu假诺卡氏菌目,Neis奈瑟菌目,Xant黄单孢菌目,Micro小单孢菌科,Strep链霉菌科,Pseud假诺卡氏菌科,Chrom色杆菌科,Xanth黄单孢菌科,Actin游动放线菌属,Strept链霉菌属,Amycol拟无枝酸菌属。下同。 Note: Acti, Actinobacteria; Prot, Proteobacteria; Beta, Betaproteobacteria; Gamm, Gammaproteobacteria; Micr, Micromonosporales; Stre, Streptomycetales; Burk, Burkholderiales; Pseu, Pseudonocardiales; Neis, Neisseriales; Xant, Xanthomonadales; Micro, Micromonosporaceae; Strep, Streptomycetaceae; Pseud, Pseudonocardiaceae; Chrom, Chromobacteriaceae; Xanth, Xanthomonadaceae; Actin, Actinoplanes; Strept, Streptomyces; Amycol Amycolatopsis. The same below. 图 4 不同耕作措施下土壤几丁质降解菌群落组成(a)和ASV/OTU韦恩图(b) Fig. 4 Microbial community(a)and venn diagram(b)of chitin-degrading bacteria under different tillage practices |
ASV/OTU韦恩图(图 4b)反映不同耕作措施间的共有物种和独有物种。在0~20 cm土层,NT、ST和MP均较CT处理显著提高ASV/OTU个数,增幅为34.3%~70.5%,其中以ST增幅最高。在20~40 cm土层,深耕(ST和MP)较浅免耕(CT和NT)处理显著增加ASV/OTU个数,提高幅度为1.7倍~2.8倍,其较NT处理增幅最大。
采用冗余分析进一步明确土壤理化性质对chiA几丁质降解菌群落结构的影响(图 5)。结果表明,冗余分析两坐标轴共解释了chiA几丁质降解菌群落结构73.6%的变异,且已测定的土壤理化指标均对chiA微生物群落结构变异有较大贡献。变形菌门、β变形菌纲和伯克霍尔德氏菌目均与土壤有机碳、全氮、全磷、有效氮和有效磷含量显著正相关,与土壤容重和pH显著负相关。与之相反,放线菌门、黄单胞菌目和黄单胞菌科均与土壤有机碳、全氮、全磷、有效氮和有效磷含量显著负相关,与土壤容重和pH显著正相关。小单孢菌目、小单孢菌科和游动放线菌属与平均重量直径显著正相关,而γ变形菌纲、链霉菌目、假诺卡氏菌目、链霉菌科、假诺卡氏菌科、链霉菌属和拟无枝酸菌属均与平均重量直径显著负相关。此外,奈瑟菌目和色杆菌科均与全氮和有效氮含量显著正相关,与pH显著负相关。
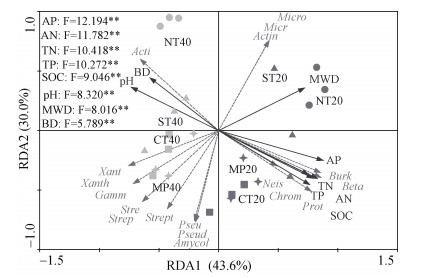
|
图 5 几丁质降解菌群与土壤理化性质的冗余分析(RDA) Fig. 5 Redundancy analysis(RDA)of chitin-degrading bacteria community with soil physicochemical properties |
图 6为不同耕作措施下土壤几丁质酶活性,对比可发现,在0~20 cm土层中,NT和MP处理的几丁质酶活性显著高于其他2种耕作措施,同时ST较CT处理显著提高几丁质酶活性。在20~40 cm土层中,深耕处理(ST和MP)对比浅免耕处理(CT和NT)几丁质酶活性更高。

|
图 6 不同耕作措施下几丁质酶活性 Fig. 6 Chitinase activity in different tillage practices |
如图 7所示,本研究所选择结构方程模型可以解释几丁质酶活性变化的97%,主要解释因子包含耕作方式、土壤深度、土壤有机碳、全氮、全磷、平均重量直径、chiA基因丰度、放线菌门和变形菌门相对丰度,该模型整体拟合效果可接受(χ2=0.164,df=4,P=0.997,RMSEA=0.000,CFI=1.000,GFI=0.999,)。模型结果表明,耕作方式、土壤深度、土壤有机碳、全氮、全磷、平均重量直径和chiA基因丰度对几丁质酶活性具有显著的直接和间接影响,而放线菌门相对丰度对几丁质酶活性具有显著的直接影响。综合直接与间接影响发现,增加耕作强度、提升土壤平均重量直径、有机碳和全磷含量,促进chiA基因丰度和放线菌门相对丰度均显著提高几丁质酶活性,而随着土壤深度增加、全氮含量提高则显著降低几丁质酶活性。
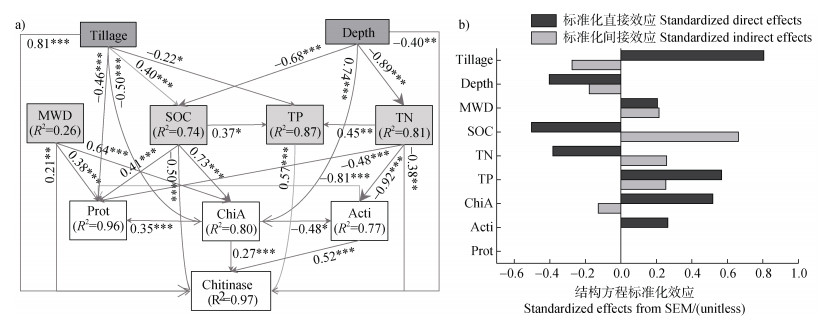
|
注:Tillage耕作方式,Depth土壤深度,ChiA chiA基因丰度,Chitinase几丁质酶活性。 Note: Tillage, tillage practice; Depth, soil depth; ChiA, chiA gene abundance. 图 7 几丁质降解菌群与几丁质酶活性的结构方程模型分析(a)及标准化效应(b) Fig. 7 Chitin-degrading bacteria community and chitinase using structural equation modeling(a)and standardized effects(b) |
长期耕作会对农田耕层和亚耕层土壤养分循环和土壤肥力维持造成不同的影响,该过程首先是调控土壤微生物活性,再进一步作用于土壤生物化学循环[28]。本研究中,与免耕相比,长期耕作后0~20 cm土层chiA基因丰度显著降低,表明耕作后显著降低该类功能微生物数量,进而降低土壤几丁质降解潜力(图 1)。该结论与Chaudhary等[23]的研究结果相反,可能是其进行19年的免耕后连续进行2年深松并没有显著影响耕层土壤养分情况,而本研究中连续37年不同耕作措施定位,土壤基础理化性质差异较大,耕作显著降低耕层有机碳、全磷和速效磷的表聚,使土壤微生物数量受到养分限制。与耕层情况相反,在20~40 cm土层,深耕处理(间隔深松和翻耕)显著提高chiA基因丰度。相关分析结果表明,chiA基因丰度与土壤结构稳定性和养分含量均显著正相关(表 2)。深耕显著降低亚耕层土壤容重,增加其通透性,利于深层根系发育,而根茬作为新鲜碳源对chiA微生物组成中大部分的异养微生物的生长繁殖起到重要作用[29]。此外,Turner等[30]通过磷添加试验证明,土壤磷养分限制将降低chiA几丁质降解微生物活性。本研究中免耕和灭茬起垄亚耕层土壤全磷和有效磷含量均低于深耕处理,这很可能是几丁质降解细菌数量降低的原因之一。
3.2 长期耕作对农田黑土几丁质降解微生物多样性与群落组成的影响本研究中,对比传统耕作,长期免耕后显著降低几丁质降解菌群Shannon多样性(图 2)。免耕处理每年进行秸秆留高茬还田,显著降低风蚀和提高耕层土壤结构稳定性,促进有机碳固定,但还田秸秆与土壤接触面积小,不利于土壤中微生物利用秸秆碳源,同时紧实的土壤环境和难获得的有机底物,促进其优势微生物活性的提高,降低群落多样性。该结果与邓超超等[31]相反,主要是因为其连续15年免耕秸秆全量覆盖还田,为土壤微生物提供有机能源,改善生态环境,进而提高细菌多样性指数。本研究中放线菌和变形菌是优势菌群,并以放线菌为主导,其相对丰度高达78%~94%(图 4a)。放线菌是农田土壤中分布最广的细菌之一,在有机底物分解中发挥重要作用,同时其也是相对寡营养菌,能在低水平碳环境中生活[20]。本研究中免耕20~40 cm土层中的放线菌相对丰度最高,这些放线菌主要包括小单孢菌目、小单孢菌科和游动放线菌属(图 4a)。长期免耕增加土壤养分表聚,促进养分分层,同时免耕玉米根系主要集中在0~20 cm土层,降低亚耕层根系发育[32],造成亚耕层土壤养分匮乏,新鲜有机物料投入较少,只能促进放线菌等寡营养菌群活性的提高,增加土壤有机碳分解与利用。此外,非优势菌种数量的减少和缺失,也使得免耕亚耕层独有物种数目远低于耕作处理(图 4b)。
本研究中,放线菌门相对丰度与土壤容重和pH显著正相关,与土壤养分含量显著负相关(图 5)。土壤容重能反映土壤结构、透气性、透水性和保水能力,当土壤容重较低时,很高比例的大孔隙将充满空气,当土壤容重超过1.3 g·cm–3时,大量微孔隙则会充满水,均在一定程度上影响土壤微生物群落组成[33]。土壤pH与细菌群落组成显著相关,与Li等[20]结果相同,很可能因为以下两点原因。一是土壤pH通过影响土壤养分有效性和金属阳离子溶解性等调控微生物群落组成。二是当土壤pH超过或低于一定范围后将改变微生物间相互作用,或者影响那些无法单独生存的微生物类群的生长[34]。洪艳华[35]研究同样发现,长期耕作后黑土pH低于未耕作土壤,同时耕作土壤pH季节性差异较大,而免耕降低土壤扰动,土壤pH季节性变化较小。张淼等[36]亦发现,土壤pH是黑土区氮循环微生物空间分布的主要影响因子。免耕亚耕层较低的根茬投入量和较高土壤容重促进厌氧细菌繁殖,放线菌群同属r型和K型微生物,参与复杂及难分解底物的分解过程,当土壤中活性养分含量较低时,将增加该类微生物数量来活化土壤养分,促进微生物利用[37]。
3.3 长期耕作对农田黑土几丁质酶活性的影响土壤几丁质酶水解几丁质,参与几丁质转化氨基糖的过程,氨基糖是可矿化氮的主要来源,占土壤有机氮的5%~10%,因此几丁质酶分解几丁质是控制氮循环速率的关键步骤[38]。土壤中不仅几丁质降解菌能合成几丁质酶,还有其他生物体,包括动物、植物和病毒等也能合成几丁质酶[9]。不同耕作措施能改变土壤有机碳含量,从而调控微生物底物供应,进而影响微生物数量和几丁质酶分泌[38]。本研究中,免耕和翻耕均显著提高0~20 cm土层几丁质酶活性,且两者差异不显著(图 6),与Chaudhary等[23]研究发现免耕对比翻耕显著提高表层土壤几丁质酶活性的结果有差异,是因为本研究中长期翻耕后耕层有机碳含量和chiA基因丰度虽然低于免耕,但可能通过促进作物根系分泌等其他来源增加几丁质酶的合成。间隔深松和翻耕显著提高20~40 cm土层几丁质酶活性,可能是因为深耕降低亚耕层土壤容重,增加通气性,提高土壤养分含量和微生物数量,从而促进几丁质酶的分泌。
土壤碳氮的可用性取决于土壤聚合物(例如木质素、几丁质等)的分解情况,这些聚合物受微生物所分泌的分解酶活性调控,以优化土壤碳氮供需平衡,当土壤碳利用率高,而氮利用率低时,则增加几丁质等富氮聚合物的分解,这是微生物氮开采(Microbial N mining)理论[39]。本研究结构方程模型结果表明,增加耕作强度、提高土壤结构稳定性、有机碳和全磷含量,提升chiA基因丰度和放线菌门相对丰度均显著提高几丁质酶活性,而增加土壤深度和全氮含量均显著降低几丁质酶活性(图 7)。深耕促进几丁质酶活性提高主要表现在亚耕层,其中翻耕直接将作物根茬翻入该土层,提高有机底物供应,而间隔深松则打破犁底层,降低亚耕层土壤容重,促进深层根系发育[40],从而提高几丁质降解菌群数量和几丁质酶活性。土壤平均重量直径对几丁质酶活性有显著的直接和间接正向效应(图 7b),表现在少免耕降低土壤扰动,提高耕层土壤结构稳定性,促进chiA基因丰度的提高和几丁质酶的合成。虽然土壤有机碳对几丁质酶总效应表现为促进作用,但直接效应为负向(图 7b)。Kumeta等[41]进行90 d培养实验发现,当土壤中添加几丁质后,为满足微生物碳需求,会促进土壤中有机碳的降解,提高铵态氮含量,而全氮含量则变化较小。表明当土壤中有机底物供应不足时,几丁质降解过程需要分解土壤碳来满足微生物增殖的碳需求。土壤全磷对几丁质酶活性具有显著的直接和间接正向效应,表明土壤全磷含量较高时,磷养分不受限制,将提高几丁质降解微生物活性,增加几丁质酶分泌[30]。Wild等[42]土壤培养试验发现,将富含碳的木质素、几丁质和蛋白质添加到同一土壤中,再添加氮素,几丁质解聚反应显著降低,而蛋白质和木质素解聚反应不受氮添加影响,表明几丁质在土壤低氮条件下会被降解为微生物氮源,并被优先分解。本研究与其结论相同,当土壤全氮含量较低时,会促进参与几丁质降解的放线菌数量增加,进而提高几丁质酶的活性,达到增加土壤有机氮含量的目的,满足微生物的氮需求。
4 结论东北黑土区不同耕作措施的长期应用使农田土壤几丁质降解菌群和酶活性发生显著分异。免耕促进效果仅表现在耕层土壤,显著降低亚耕层几丁质降解菌群数量及多样性,而间隔深松和翻耕则增加亚耕层几丁质降解菌数量,并维持其群落多样性,提高几丁质酶活性。几丁质降解菌群和土壤理化性质密切相关。耕作方式和土壤深度直接影响几丁质酶活性,也通过调控土壤结构稳定性、全量养分含量和几丁质降解菌群间接影响几丁质酶活性。
| [1] |
LeCleir G R, Buchan A, Maurer J, et al. Comparison of chitinolytic enzymes from an alkaline, hypersaline lake and an estuary[J]. Environmental Microbiology, 2007, 9(1): 197-205. DOI:10.1111/j.1462-2920.2006.01128.x
(  0) 0) |
| [2] |
Wieczorek A S, Hetz S A, Kolb S. Microbial responses to chitin and chitosan in oxic and anoxic agricultural soil slurries[J]. Biogeosciences, 2014, 11(12): 3339-3352. DOI:10.5194/bg-11-3339-2014
(  0) 0) |
| [3] |
Sharp R. A review of the applications of chitin and its derivatives in agriculture to modify plant-microbial interactions and improve crop yields[J]. Agronomy, 2013, 3(4): 757-793. DOI:10.3390/agronomy3040757
(  0) 0) |
| [4] |
Kielak A M, Cretoiu M S, Semenov A V, et al. Bacterial chitinolytic communities respond to chitin and pH alteration in soil[J]. Applied and Environmental Microbiology, 2013, 79(1): 263-272. DOI:10.1128/AEM.02546-12
(  0) 0) |
| [5] |
Cretoiu M S, Kielak A M, Schluter A, et al. Bacterial communities in chitin-amended soil as revealed by 16S rRNA gene based pyrosequencing[J]. Soil Biology & Biochemistry, 2014, 76: 5-11.
(  0) 0) |
| [6] |
Brzezinska M S, Jankiewicz U, Walczak M. Biodegradation of chitinous substances and chitinase production by the soil actinomycete Streptomyces rimosus[J]. International Biodeterioration & Biodegradation, 2013, 84: 104-110.
(  0) 0) |
| [7] |
Patil R S, Ghormade V, Deshpande M V. Chitinolytic enzymes: An exploration[J]. Enzyme and Microbial Technology, 2000, 26(7): 473-483. DOI:10.1016/S0141-0229(00)00134-4
(  0) 0) |
| [8] |
Adrangi S, Faramarzi M A. From bacteria to human: A journey into the world of chitinases[J]. Biotechnology Advances, 2013, 31(8): 1786-1795. DOI:10.1016/j.biotechadv.2013.09.012
(  0) 0) |
| [9] |
Horn S J, Sørbotten A, Synstad B, et al. Endo/exo mechanism and processivity of family 18 chitinases produced by Serratia marcescens[J]. The FEBS Journal, 2006, 273(3): 491-503. DOI:10.1111/j.1742-4658.2005.05079.x
(  0) 0) |
| [10] |
Kawase T, Saito A, Sato T, et al. Distribution and phylogenetic analysis of family 19 chitinases in Actinobacteria[J]. Applied and Environmental Microbiology, 2004, 70(2): 1135-1144. DOI:10.1128/AEM.70.2.1135-1144.2004
(  0) 0) |
| [11] |
Cretoiu M S, Kielak A M, Abu Al-Soud W, et al. Mining of unexplored habitats for novel chitinases—chiA as a helper gene proxy in metagenomics[J]. Applied Microbiology and Biotechnology, 2012, 94(5): 1347-1358. DOI:10.1007/s00253-012-4057-5
(  0) 0) |
| [12] |
Veliz E A, Martínez-Hidalgo P, Hirsch A M. Chitinase- producing bacteria and their role in biocontrol[J]. AIMS Microbiology, 2017, 3: 689-705. DOI:10.3934/microbiol.2017.3.689
(  0) 0) |
| [13] |
Bai Y, Eijsink V G H, Kielak A M, et al. Genomic comparison of chitinolytic enzyme systems from terrestrial and aquatic bacteria[J]. Environmental Microbiology, 2016, 18(1): 38-49. DOI:10.1111/1462-2920.12545
(  0) 0) |
| [14] |
Yan H Y, Jin R D, Piao G Y, et al. Effect of decomposition chitin microbial metabolites on maize growth-promoting under different fertility soils (In Chinese)[J]. Journal of Maize Sciences, 2015, 23(3): 119-123. [闫海洋, 金荣德, 朴光一, 等. 不同肥力土壤中分解几丁质微生物代谢产物对玉米的促生效果研究[J]. 玉米科学, 2015, 23(3): 119-123.]
(  0) 0) |
| [15] |
Cronin D, Moënne-Loccoz Y, Dunne C, et al. Inhibition of egg hatch of the potato cyst nematode Globodera rostochiensis by chitinase-producing bacteria[J]. European Journal of Plant Pathology, 1997, 103: 433-440. DOI:10.1023/A:1008662729757
(  0) 0) |
| [16] |
Sindhu S S, Dadarwal K R. Chitinolytic and cellulolytic Pseudomonas sp. antagonistic to fungal pathogens enhances nodulation by Mesorhizobium sp. Cicer in chickpea[J]. Microbiological Research, 2001, 156(4): 353-358. DOI:10.1078/0944-5013-00120
(  0) 0) |
| [17] |
Zhang L, Wang J, Wang X T, et al. Effect of restoration types on the community structure of microbes harboring nifH and chiA genes in alpine meadow (In Chinese)[J]. Chinese Journal of Applied Ecology, 2021, 32(12): 4349-4358. [张路, 王杰, 王向涛, 等. 不同恢复方式对退化高寒草甸土壤nifH和chiA微生物群落结构的影响[J]. 应用生态学报, 2021, 32(12): 4349-4358.]
(  0) 0) |
| [18] |
Anderson C, Beare M, Buckley H L, et al. Bacterial and fungal communities respond differently to varying tillage depth in agricultural soils[J]. PeerJ, 2017, 5: e3930. DOI:10.7717/peerj.3930
(  0) 0) |
| [19] |
Song X J, Wu H J, Wu X P, et al. Long-term conservation tillage improves surface soil carbon and nitrogen content and rhizosphere soil enzyme activities (In Chinese)[J]. Journal of Plant Nutrition and Fertilizers, 2018, 24(6): 1588-1597. [宋霄君, 吴会军, 武雪萍, 等. 长期保护性耕作可提高表层土壤碳氮含量和根际土壤酶活性[J]. 植物营养与肥料学报, 2018, 24(6): 1588-1597.]
(  0) 0) |
| [20] |
Li M, He P, Guo X L, et al. Fifteen-year no tillage of a Mollisol with residue retention indirectly affects topsoil bacterial community by altering soil properties[J]. Soil & Tillage Research, 2021, 205: 104804.
(  0) 0) |
| [21] |
Li Y, Song D P, Liang S H, et al. Effect of no-tillage on soil bacterial and fungal community diversity: A meta- analysis[J]. Soil and Tillage Research, 2020, 204: 104721. DOI:10.1016/j.still.2020.104721
(  0) 0) |
| [22] |
Liu H W, Carvalhais L C, Crawford M, et al. Strategic tillage increased the relative abundance of Acidobacteria but did not impact on overall soil microbial properties of a 19-year no-till Solonetz[J]. Biology and Fertility of Soils, 2016, 52(7): 1021-1035. DOI:10.1007/s00374-016-1138-0
(  0) 0) |
| [23] |
Chaudhary M, Naresh R K, Vek V, et al. Soil organic carbon fractions, soil microbial biomass carbon, and enzyme activities impacted by crop rotational diversity and conservation tillage in north west IGP: A review[J]. International Journal of Current Microbiology and Applied Sciences, 2018, 7(11): 3573-3600. DOI:10.20546/ijcmas.2018.711.410
(  0) 0) |
| [24] |
Bao S D. Soil and agricultural chemistry analysis (In Chinese). 3rd ed[M]. Beijing: China Agriculture Press, 2000: 42-108. [鲍士旦. 土壤农化分析[M]. 3版. 北京: 中国农业出版社, 2000: 42-108.]
(  0) 0) |
| [25] |
Xiao X, Yin X B, Lin J, et al. Chitinase genes in lake sediments of Ardley Island, Antarctica[J]. Applied and Environmental Microbiology, 2005, 71(12): 7904-7909. DOI:10.1128/AEM.71.12.7904-7909.2005
(  0) 0) |
| [26] |
Zheng H B, Liu W R, Zheng J Y, et al. Effect of long-term tillage on soil aggregates and aggregate- associated carbon in black soil of Northeast China[J]. PLoS One, 2018, 13(6): e0199523. DOI:10.1371/journal.pone.0199523
(  0) 0) |
| [27] |
Sui P X, Tian P, Lian H L, et al. Straw incorporation management affects maize grain yield through regulating nitrogen uptake, water use efficiency, and root distribution[J]. Agronomy, 2020, 10(3): 324. DOI:10.3390/agronomy10030324
(  0) 0) |
| [28] |
Morugán-Coronado A, Pérez-Rodríguez P, Insolia E, et al. The impact of crop diversification, tillage and fertilization type on soil total microbial, fungal and bacterial abundance: A worldwide meta-analysis of agricultural sites[J]. Agriculture, Ecosystems & Environment, 2022, 329: 107867.
(  0) 0) |
| [29] |
Mu X Y, Zhao Y L, Liu K, et al. Responses of soil properties, root growth and crop yield to tillage and crop residue management in a wheat-maize cropping system on the North China Plain[J]. European Journal of Agronomy, 2016, 78: 32-43. DOI:10.1016/j.eja.2016.04.010
(  0) 0) |
| [30] |
Turner S, Mikutta R, Guggenberger G, et al. Distinct pattern of nitrogen functional gene abundances in top- and subsoils along a 120, 000-year ecosystem development gradient[J]. Soil Biology & Biochemistry, 2019, 132: 111-119.
(  0) 0) |
| [31] |
Deng C C, Li L L, Xie J H, et al. Effects of tillage on soil bacterial community in the dryland farming area of central Gansu (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(1): 207-216. [邓超超, 李玲玲, 谢军红, 等. 耕作措施对陇中旱农区土壤细菌群落的影响[J]. 土壤学报, 2019, 56(1): 207-216.]
(  0) 0) |
| [32] |
Wang X B, Zhou B Y, Sun X F, et al. Soil tillage management affects maize grain yield by regulating spatial distribution coordination of roots, soil moisture and nitrogen status[J]. PLoS One, 2015, 10(6): e0129231. DOI:10.1371/journal.pone.0129231
(  0) 0) |
| [33] |
Li C H, Ma B L, Zhang T Q. Soil bulk density effects on soil microbial populations and enzyme activities during the growth of maize(Zea mays L.)planted in large pots under field exposure[J]. Canadian Journal of Soil Science, 2002, 82(2): 147-154. DOI:10.4141/S01-026
(  0) 0) |
| [34] |
Lauber C L, Hamady M, Knight R, et al. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J]. Applied and Environmental Microbiology, 2009, 75(15): 5111-5120. DOI:10.1128/AEM.00335-09
(  0) 0) |
| [35] |
Hong Y H. Effects of long-term tillage on physicochemical properties and microbial community structure in black soil[D]. Daqing, Heilongjiang: Heilongjiang Bayi Agricultural University, 2021.[洪艳华. 长期耕作对黑土理化性质及微生物群落结构的影响[D]. 黑龙江大庆: 黑龙江八一农垦大学, 2021.]
(  0) 0) |
| [36] |
Zhang M, Liu J J, Liu Z X, et al. Distribution characteristics of microbial gene abundance in key processes of soil nitrogen cycling in black soil zone (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(5): 1258-1269. [张淼, 刘俊杰, 刘株秀, 等. 黑土区农田土壤氮循环关键过程微生物基因丰度的分布特征[J]. 土壤学报, 2022, 59(5): 1258-1269.]
(  0) 0) |
| [37] |
Li H, Zhang Y Y, Yang S, et al. Variations in soil bacterial taxonomic profiles and putative functions in response to straw incorporation combined with N fertilization during the maize growing season[J]. Agriculture, Ecosystems & Environment, 2019, 283: 106578.
(  0) 0) |
| [38] |
Zhang W, Zhang M, Bai Z, et al. Research advances in soil chitinase (In Chinese)[J]. Chinese Journal of Soil Science, 2007, 38(3): 569-575. [张威, 张明, 白震, 等. 土壤中几丁质酶的研究进展[J]. 土壤通报, 2007, 38(3): 569-575.]
(  0) 0) |
| [39] |
Chen R R, Senbayram M, Blagodatsky S, et al. Soil C and N availability determine the priming effect: Microbial N mining and stoichiometric decomposition theories[J]. Global Change Biology, 2014, 20(7): 2356-2367. DOI:10.1111/gcb.12475
(  0) 0) |
| [40] |
Bai W, Sun Z X, Zhang L Z, et al. Effects of plough layer construction on soil three phase rate and root morphology of spring maize in northeast China (In Chinese)[J]. Acta Agronomica Sinica, 2020, 46(5): 759-771. [白伟, 孙占祥, 张立祯, 等. 耕层构造对土壤三相比和春玉米根系形态的影响[J]. 作物学报, 2020, 46(5): 759-771.]
(  0) 0) |
| [41] |
Kumeta Y, Inami K, Ishimaru K, et al. Thermogravimetric evaluation of chitin degradation in soil: Implication for the enhancement of ammonification of native organic nitrogen by chitin addition[J]. Soil Science and Plant Nutrition, 2018, 64(4): 512-519. DOI:10.1080/00380768.2018.1457408
(  0) 0) |
| [42] |
Wild B, Li J, Pihlblad J, et al. Decoupling of priming and microbial N mining during a short-term soil incubation[J]. Soil Biology & Biochemistry, 2019, 129: 71-79.
(  0) 0) |
 2023, Vol. 60
2023, Vol. 60


