2. 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所), 南京 210008
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
团聚体是土壤结构的基本组成单位和土壤生态系统的功能单元,为微生物提供了空间异质的栖息场所。不同粒级团聚体上营养水平、湿度、氧气等微域环境的差异,必然会引起土壤微生物群落的空间分异,进而影响土壤养分转化和物质循环,重塑土壤团聚体的生物学性质。已有较多研究证明不同农田管理措施下,团聚体的粒级组成[1]以及细菌和真菌群落多样性发生显著变化[2-4]。但整体微生物群落多样性难以与特定生态过程建立直接联系,亟待加强关键功能微生物类群在团聚体粒级间的分配及其对生态功能影响的理论研究。
硝化作用是土壤氮素循环的关键过程,不仅降低农田土壤氮肥利用率,而且增加硝酸盐淋溶和温室气体N2O排放风险。硝化作用的两个阶段分别由氨氧化菌和亚硝酸盐氧化细菌(NOB)催化完成。氨氧化菌将铵根离子(NH4+)氧化为亚硝酸盐(NO2–),此反应亦为硝化作用的限速步骤,已发现的氨氧化菌包括氨氧化古菌(AOA)、氨氧化细菌(AOB)和新发现的全程氨氧化细菌(Comammox);继而,NOB将NO2–氧化为硝酸盐(NO3–)。国内外研究表明AOA和AOB丰度和群落结构受土壤pH、NH4+含量、有机碳、土壤质地等因素影响[5]。AOA偏好于低营养水平和酸性土壤,且具有较高的氧胁迫耐受性,AOB则相反;Comammox对NH4+亲和力更强,因此低营养状态下有更强竞争力[6]。而NOB与氨氧化菌存在共生关系,氨氧化菌为NOB供给底物NO2–,NOB为氨氧化菌清除NO2–毒害,还可以利用尿素产生氨供给AOB[7]。因此,解析硝化菌群在氮循环中的生理生态多样性一直被视为土壤生态学研究的热点和难点。
近年来,土壤团聚体和硝化菌群之间的研究取得一定进展。Zhang等[8]发现AOA和AOB对施肥和团聚体粒级的响应规律具有较大差异,但两者特征amoA基因丰度均在最小粒级2~0.1 μm中最低。Jiang等[9]研究表明AOA和AOB amoA基因在最小粒级 < 250 μm中富集,在粒级 > 250 μm中较少。相反,Han等[10]发现不同施肥措施下AOA和AOB amoA基因以及NOB nxrB基因的分布规律较为一致,且均倾向于富集在较大粒级(> 250 μm)。Hou等[11]研究表明Nitrosomonas属AOB在 < 250 μm团聚体中较为丰富,而Nitrospira属NOB在较大团聚体(> 250 μm)中更多。可见,不同类群硝化微生物随土壤团聚体粒级变化而呈现的分布模式并不一致。团聚体粒级的理化性质差异,可能是塑造硝化微生物群落分布特征的关键。深入开展不同粒级团聚体中硝化微生物功能群研究,能够进一步揭示土壤结构-微生物-土壤营养元素循环的相互影响机制[4]。
本研究以旱地黄棕壤为研究对象,通过定量PCR和高通量测序等分子生物学手段揭示了硝化微生物群落在不同粒级团聚体上的分异特征,并比较两种不同种植方式(玉米连作和玉米/花生轮作)对土壤团聚体性质和硝化微生物群落的影响。研究结果有助于理解土壤团聚体形成过程中微生物的环境适应机制,为土壤肥力评价和农田管理提供理论依据。
1 材料与方法 1.1 试验地概况试验区位于安徽省巢湖市栏杆集镇新桥村(31°58'N,117°50'E),地处江淮丘陵南部,属北亚热带湿润季风气候区,光照充分,热量条件较好,无霜期长,季风气候显著,冬寒夏热,四季分明。年平均气温15~16℃,年平均降水量约1 100 mm,主要集中在5—8月,占年总降水量的55%。典型土壤类型为下蜀黄土母质发育的黄棕壤。
1.2 试验设计和土壤采集田间定位试验于2018—2019年进行。根据试验区内典型农业生产模式,在试验地设置2种处理,分别为玉米连作(M-M)和玉米/花生轮作(M-P)。试验开始前,选取一块地面平整的、土壤性质均匀的传统旱作农田,平均划分为2个小区。试验小区第1年均种植玉米,第2年分别种植玉米和花生。作物每年4月播种,当年9月下旬收获,秋冬季抛荒,在试验第2年作物收获后采集土壤进行分析。玉米和花生种植过程中,基肥施用量相同,为复混肥料(N:P2O5:K2O = 26:10:15)900 kg·hm–2,玉米在大喇叭口期追施尿素150 kg·hm–2(N含量为46.4%),花生不追肥。其他管理措施如培土、除草等均按常规模式进行。
采集试验小区多个采样点0~20 cm土壤。取样时尽量避免挤压,以保持原状土壤结构。将采集的多点土壤样品混合组成代表土样。将稍大土块沿土壤自然结构脆弱面剥离成直径约5 mm小块,仔细挑除土样中的石块、植物根系。取其中一部分土壤用于团聚体粒径分级。M-M全土理化性质为:pH 6.16,全碳(TC)7.63 g∙kg–1,全氮(TN)0.82 g∙kg–1,NH4+-N 1.20 mg∙kg–1,NO3–-N 8.47 mg∙kg–1。M-P全土理化性质为:pH 6.65,TC 8.15 g∙kg–1,TN 0.91g∙kg–1,NH4+-N 4.69 mg∙kg–1,NO3–-N 9.53 mg∙kg–1。
1.3 土壤团聚体筛分采用干筛法[2,12]对土壤团聚体进行筛分,分离出 > 2、2~1、1~0.25、< 0.25 mm四个粒级团聚体。步骤如下:取风干土样2.0 kg,使其通过一套直径20 cm筛组(孔径从上到下顺序为2 mm,1 mm和0.25 mm),筛组上方带盖,下方有底,收集通过土筛的样品。每次过筛的土样不宜过多,约为200 g。筛组先振荡5 min,收集最上面筛子上的团聚体(> 2 mm),再取下最上面1个筛子,继续振荡5 min,收集此时最上面筛子上的团聚体(2~1 mm),依次筛分出1~0.25 mm和 < 0.25 mm团聚体。当筛分完成后,称量并计算各粒级团聚体占土样总质量的百分比(表 1)。
|
|
表 1 两种种植方式下土壤团聚体的理化性质 Table 1 Soil aggregate properties under different cropping systems |
土壤pH采用电位计法测定,首先对pH计进行校准,按水土比2.5:1向土壤加入无CO2去离子水,之后将玻璃电极和饱和甘汞电极插入土壤悬液中进行读数。土壤无机氮采用氯化钾浸提,按水土比5:1向土壤加入2 mol·L–1 KCl溶液,以200 r·min–1振荡1 h,将振荡后的溶液静置后过滤,滤液采用连续流动化学分析仪(Skalar ++,Breda,Netherlands)对铵态氮(NH4+-N)和硝态氮(NO3–-N)含量进行测定。土壤TC和TN含量的测定,将土壤冷冻干燥并过100目筛,利用碳氮元素分析仪(Vario MAX CN,Germany)进行测定。以上指标测定时,均进行3次重复。
1.5 土壤硝化潜势测定土壤硝化潜势(Nitrification Potential)通过悬浮液培养法测定[13],并略有改进。具体步骤为:分别配置0.2 mol·L–1磷酸二氢钾(KH2PO4)溶液和0.2 mol·L–1磷酸氢二钾(K2HPO4)溶液,以及0.05 mol·L–1硫酸铵((NH4)2SO4)溶液适量。取1.5 mL KH2PO4溶液,7.5 mL K2HPO4溶液和15 mL(NH4)2SO4溶液,分别溶于去离子水,定容至1.0 L,并将pH调至7.2,制成土壤缓冲液。该土壤缓冲液含1.5 mmol·L–1 NH4+和1.0 mmol·L–1 PO43–[14]。称取各土样5.0 g于50 mL离心管,加入40 mL土壤缓冲液,倾斜置于摇床上(200 r·min–1)震荡培养24 h。分别在第2、10、20、24 h采集6 mL摇匀的土壤悬浮液,4 000 r·min–1离心10 min,取上清液通过定量滤纸,滤液保存于–20℃冰箱。三次重复。通过连续流动化学分析仪(Skalar ++,Breda,Netherlands)测定滤液中NOx–-N浓度。硝化潜势计算公式如下:
| $ {{\text{N}}_{\text{P}}} = R \times \frac{{{V_1} + {V_2}}}{m} \times 24 $ |
式中,NP代表硝化潜势(mg∙ kg–1∙d–1),R为NOx--N增长速率(mg∙L–1∙h–1),V1代表所加缓冲液体积(L),V2代表土壤样品中水分体积(L),m为烘干土质量(kg)。
1.6 土壤硝化微生物功能基因丰度测定采用Fast DNA® Spin Kit for Soil(MP Biomedicals)试剂盒,按照试剂盒内说明书上的步骤提取土壤微生物DNA。最终将提取到的土壤DNA溶解于100 μL DES(无核酸酶超纯水)。利用微量紫外分光光度计(Nano Drop® ND-2000 UV-Vis)在260 nm处测定DNA浓度和纯度,确保OD260/ OD280在1.8~2.0范围内,避免腐殖质对后续PCR扩增的影响。最后,用DES将土壤DNA进行10倍稀释,保存于–20℃冰箱。各粒级团聚体DNA提取均进行三次重复。
通过实时定量PCR技术对土壤不同类型硝化微生物功能基因进行定量分析。使用引物amoA-1F/ amoA-2R[15]、arch-amoAF/Arch-amoAR[16]和comamoAF/ comamoAR[17]分别对氨氧化微生物AOB、AOA和Comammox功能基因amoA进行定量扩增,使用引物nxrB169F/nxrB638R[10]对亚硝酸盐氧化细菌NOB功能基因nxrB进行定量扩增。其中,定量PCR反应体系总体积为20 μL,包括0.2 μL前、后置引物(20 μmol∙L–1)、10 μL SYBR® Premix Ex Taq、1 μL DNA(标样或待测样),剩余体积由灭菌水补充。标准样品的配制:分别将含有待测目的基因的质粒,以10倍梯度进行稀释,制成7~8个浓度梯度的标准品,与待测样品一起,按上述步骤配制定量体系。每个DNA样品均进行3次重复扩增分析,且每次均采用灭菌双蒸水作为阴性对照。实时荧光扩增定量反应在定量PCR仪(Biorad CFX96 Optical Real- Time PCR System)上进行。
1.7 高通量测序及生物信息分析为探究土壤硝化微生物群落组成变化,开展16S rRNA基因高通量测序分析。由上海美吉生物医药科技有限公司(Shanghai Majorbio Bio-pharm Technology Co.,Ltd)对四个土壤团聚体粒级DNA样品中微生物16S rRNA基因V4区进行扩增(引物515F/907R)和建库,最后在Illumina MiSeq测序系统上进行双端测序,继而在美吉生信云平台(www.majorbio.com)上进行生物信息分析。大致分析流程如下:下机数据先进行拼接和质控,根据barcode区分样本,将所测序列在97%相似度水平划分OTU(操作分类单元),并以silva138数据库为参考进行物种注释。在属分类水平,检索已知的AOB[18]、NOB[19]和AOA[20]各属并统计丰度,最终进行硝化微生物群落组成分析,并通过Spearman相关分析探究硝化微生物群落组成变化与团聚体性质和硝化潜势的关联。
1.8 统计分析通过SPSS 19.0统计软件,采用单因素方差(One-way ANOVA)分析Tukey法对不同粒径和不同种植方式下理化因素、进行统计显著性检验;采用双因素方差(Two-way ANOVA)分析检验种植方式和团聚体粒径之间是否存在交互效应;并采用Pearson双尾t检验对硝化微生物基因丰度与理化因子间的相关性和统计显著性进行评估。P < 0.05为具有统计显著性。采用Origin 8.0进行绘图。
2 结果 2.1 土壤团聚体的理化性质土壤团聚体的理化性质见表 1。pH方面,M-M中,除了 > 2 mm团聚体外,其他粒级团聚体pH随着团聚体粒径的减小而增加。相反,M-P土壤团聚体pH总体随着团聚体粒径的减小而降低。M-M土壤NH4+-N含量随着团聚体粒径的减小而增加。而M-P土壤NH4+-N含量随团聚体粒径呈现两头低中间高的分布特点,其中,> 2 mm团聚体NH4+-N含量最低。M-M土壤NO3–-N含量大致随着团聚体粒径的减小而降低。M-P土壤NO3–-N含量随团聚体粒径呈现两头低中间高的分布趋势。土壤TC含量,在M-M土壤中随着团聚体粒径的减小而增加,在M-P土壤中,除2~1 mm外的其他三个粒级之间差异均不显著。M-M土壤TN含量总体随着团聚体粒级的减小而增加(0.75~0.88 g∙kg–1),而M-P土壤TN在 < 0.25 mm粒级最低为8.01 g∙kg–1,在其他三个粒级之间差异均不显著。双因素方差分析结果表明,种植方式能显著提高团聚体NH4+、pH和TC含量,土壤团聚体粒径对NH4+、NO3–、pH和TC均有显著影响,单一因素(种植方式或团聚体粒径)均未对TN产生显著影响,但是两因素的交互效应在以上五种理化性质的分异中均达到显著水平(P < 0.05)。
2.2 土壤团聚体的硝化活性四个粒级间的土壤团聚体硝化潜势(NP)如图 1。轮作M-P土壤团聚体的硝化潜势普遍高于连作M-M土壤。连作M-M土壤团聚体硝化潜势为5.70~15.08 mg∙kg–1∙d–1,且在较小粒级(1~0.25 mm和 < 0.25 mm)中显著高于较大粒级(2~1 mm和 > 2 mm)。轮作M-P土壤团聚体硝化潜势为19.46~28.43 mg∙kg–1∙d–1,在2~1 mm粒级中最高。双因素方差分析(图 2)表明种植方式对土壤硝化潜势有显著影响,团聚体粒径对硝化潜势影响不显著,且二者的交互效应亦不显著。
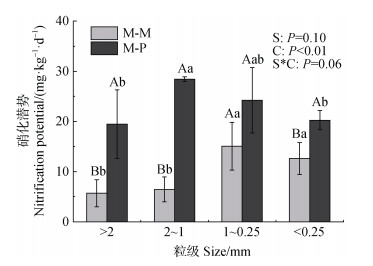
|
注:C,种植方式的影响;S,团聚体粒径的影响;C×S,种植方式和团聚体粒径的交互效应。显著性效应,P < 0.05。下同。 Note: C, the effect of cropping system; S, the effect of aggregate size; S×C, the effect of both cropping system and aggregate size. Significant effects, P < 0.05. The same below. 图 1 不同种植方式下土壤团聚体的硝化潜势差异 Fig. 1 Nitrification potentials within soil aggregates under different cropping systems |

|
图 2 不同粒径土壤团聚体上硝化微生物功能基因丰度的差异 Fig. 2 Abundance changes of nitrifying functional genes in different soil aggregate sizes |
团聚体中AOB amoA基因丰度(图 2a)在M-P土壤明显高于M-M土壤(1~0.25 mm除外),且在 < 0.25 mm粒级中数量最高,表明AOB更能适应较小团聚体的微域环境。AOA amoA基因(图 2b)在M-M土壤显著高于M-P土壤,且在团聚体上的分布趋势与AOB amoA基因大致相反,在 > 2 mm粒级中基因数量最高,表明AOA更能适应较大团聚体的微域环境。Comammox amoA基因(图 2c)在两种土壤的分布模式有较大差异,在M-M土壤 > 2 mm粒级中最高,而在M-P中随团聚体粒径呈现两头高中间低的分布趋势。NOB nxrB基因(图 2d)亦在M-M中高于M-P,且随团聚体粒径呈现两头高中间低的分布趋势,在 < 0.25 mm粒级中数量最高。双因素方差分析(图 3)表明土壤团聚体粒径和种植方式均能较大程度影响以上硝化微生物在土壤中的分布。

|
图 3 不同粒径土壤团聚体上氨氧化微生物amoA基因比值的变化 Fig. 3 amoA gene ratios of different ammonia-oxidizing groups in different soil aggregate sizes |
以amoA基因为分子标靶,连作M-M土壤中AOA/AOB比值随着团聚体粒级的减小而减小(1.56~3.92)(图 3a),Comammox/AOB比值的变化趋势与上述一致(0.15~0.46)(图 3b),表明AOA和Comammox在较大粒级团聚体上更具竞争优势,而AOB则相反。M-P土壤中上述比值的变化趋势与M-M土壤相似,但随团聚体粒级减小,上述比值的变化幅度均小于M-M土壤。M-P中AOA/AOB和Comammox/AOB比值的变化范围分别为0.87~1.58和0.18~0.24。Comammox/AOA比值在M-M土壤团聚体之间无显著变化(0.10~0.12),在M-P土壤中呈现中间高两头低(0.13~0.21)(图 3c),且普遍高于M-M(2~1 mm粒级除外)。综上所述,不同氨氧化微生物在团聚体上的分布规律具有明显差异。
2.4 土壤团聚体性质与硝化微生物功能基因丰度及硝化活性的关联硝化微生物amoA基因丰度在团聚体间的分布,受微域环境强烈影响(表 2)。在不同粒级团聚体间,就氨氧化微生物而言,AOB amoA基因丰度与pH、TC和NH4+含量显著正相关,与NO3–含量显著负相关。AOA与AOB则完全相反,其amoA基因丰度与pH、TC和NH4+含量均呈现显著负相关。而Comammox amoA基因丰度仅与TC显著负相关。对于NOB,团聚体pH、NH4+和NO3–含量的增加,均能显著降低nxrB基因丰度。并且,进一步分析土壤理化性质及硝化微生物与团聚体硝化潜势间的相关性发现,pH和NH4+含量是影响团聚体硝化潜势NP的主要因素(P < 0.01,r分别为0.800和0.889),TN亦对NP存在显著性影响(r = 0.485)。而硝化微生物中AOB amoA基因丰度与NP显著正相关(r = 0.588),AOA amoA基因丰度与NP显著负相关(r = –0.686),Comammox amoA基因与NP不存在显著性关联。
|
|
表 2 土壤团聚体理化性质、硝化微生物功能基因丰度以及硝化潜势之间的相关分析 Table 2 Correlation analysis between soil properties, nitrifying functional gene abundances and nitrification potential |
对24个团聚体DNA样品进行高通量测序,共获得16S rRNA基因序列约111.8万条,平均每个样品约4.66万条,硝化微生物的平均相对丰度约为1.02%(473条/样),其中,AOA、AOB和NOB的平均相对丰度分别为0.51%(239条/样)、0.08%(36条/样)和0.43%(199条/样)。
通过16S rRNA基因高通量测序分析,揭示了不同种植方式下土壤团聚体中硝化微生物群落组成(图 4a)。在试验土壤中,AOA主要检测到三个属,分别为Candidatus Nitrosocosmicus,Candidatus Nitrososphaera和norank_f_Nitrososphaeraceae,其中norank_f_Nitrososphaeraceae最多;AOB主要包括Nitrosospira和Nitrosomonas两属,且Nitrosospira远高于Nitrosomonas;NOB主要包括Nitrospira和Nitrolancea两属,且Nitrospira占据主导。总体而言,各类硝化菌在整体硝化微生物群落中的占比大小顺序为:AOA > NOB > AOB。双因素方差分析(表 3)表明,种植方式能够对Nitrospira属和Nitrolancea属NOB,以及Candidatus Nitrosocosmicus属AOA在硝化群落中的占比产生显著影响,而团聚体粒径仅对Nitrosospira属AOB的占比产生显著影响,但两者交互效应在各类型硝化微生物中均不显著。
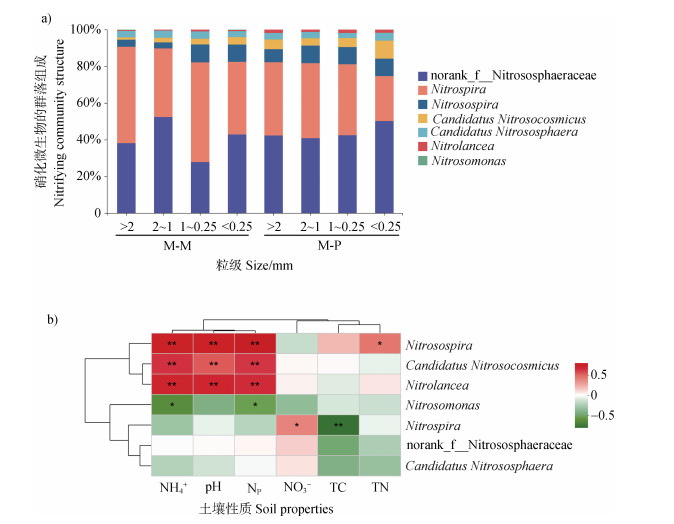
|
*,P < 0.05;**,P < 0.01。 图 4 基于16S rRNA基因的硝化微生物群落组成(a)及与土壤性质和硝化潜势的相关系数热图分析(b) Fig. 4 Nitrifying community structures based on 16S rRNA genes(a)and their correlation with soil properties and nitrification potential by heatmap analysis(b) |
|
|
表 3 双因素方差分析种植方式和团聚体粒径对氨氧化古菌、氨氧化细菌和亚硝酸盐氧化细菌组成的影响(P值) Table 3 Results (P values) of a two-way ANOVA analysis for the effects of cropping system and soil aggregate size on dominant phylotypes of AOA, AOB and NOB |
NH4+含量和pH是影响土壤团聚体硝化微生物群落组成变化的最主要因子(图 4b)。团聚体NH4+含量和pH提高,对Nitrosospira属AOB,Candidatus Nitrosocosmicus属AOA和Nitrospira属NOB均有显著促进作用。相反地,NH4+含量增加对Nitrosomonas属AOB却存在显著抑制效应。此外,TC与Nitrospira属NOB显著负相关,TN与Nitrosospira属AOB显著正相关。对硝化活性的影响方面,Nitrosospira属AOB,Candidatus Nitrosocosmicus属AOA和Nitrospira属NOB均与团聚体NP存在显著正相关关系。
3 讨论合理轮作能够协调养分供应,均衡利用土壤养分,防止连作障碍,具有很高的生态和经济效益[21]。与连作相比,轮作可改善连作土壤结构,提高土壤酶活性,促进土壤的代谢作用,促使土壤养分形态发生变化,从而提高土壤肥力[12]。花生作为一种豆科植物,被广泛用于间、套、轮作,能够增加作物产量、培肥土壤、改善农田生态条件[22-23]。与前人研究一致,本研究结果显示与玉米连作(M-M)相比,玉米/花生轮作(M-P)能够提高土壤全土及团聚体pH、NH4+和TC含量。
同时,轮作能够通过根系向土壤输送更加多样的营养物质,提高土壤微生物群落多样性[24]。对于硝化微生物而言,轮作M-P提高了团聚体中AOB amoA基因丰度,而降低了AOA amoA基因丰度,导致四个粒级团聚体上AOA/AOB和Comammox/ AOB比值的差异减小,表明轮作促使土壤硝化微生物在粒级间的分布更加均匀。曹莉等[25]研究也显示轮作方式能够提高硝化细菌的数量。进一步在分类属水平对土壤团聚体中硝化菌群落组成进行分析,结果显示轮作提高了Nitrolancea属NOB和Candidatus Nitrosocosmicus属AOA的比例,降低了Nitrospira属NOB的比例,对AOB各属无显著影响。此外,M-M土壤团聚体的硝化潜势普遍低于M-P土壤,表明M-P土壤硝化活性提高。可见,种植方式对土壤团聚体养分状况、生物学特性以及生态过程均产生了较大影响,为禾豆轮作下土壤硝化微生物的生态适应理论提供了支持。
地理大尺度数据整合分析显示,陆地生态系统中AOB和AOA amoA基因丰度和群落组成受到多种环境因素影响[5,18,26],其中,pH和NH4+尤为关键[5]。本研究中同样发现在微域尺度上团聚体pH与NH4+含量显著正相关(r = 0.62,P < 0.01),均对AOA和AOB丰度和组成均产生了显著影响。两者与团聚体AOB amoA基因丰度正相关,与AOA amoA基因丰度负相关,表明AOA和AOB具有不同的营养需求和底物利用能力。较多研究已经证实低铵或酸性条件下AOA快速生长;而在高铵或中性-碱性土壤中,AOB更具生长活性[5,27]。通过生态位竞争,AOB和AOA在微域尺度上形成了各自独特分布模式,AOB偏好栖息在NH4+含量较高、比面积较大的微团聚体(< 0.25 mm)上,而AOA则在NH4+含量较低的大团聚体(> 2 mm)环境更具竞争优势。此外,TC也与AOA和AOB amoA基因丰度显著关联(P < 0.05),一方面可能由于TC矿化能够增加土壤可利用NH4+作为硝化底物,从而刺激氨氧化菌的生长,另一方面TC还能够满足异养或混合营养生长的氨氧化菌群对碳源需求[28]。团聚体中Comammox能够与AOB和AOA共存,且与AOA分布模式更为相似,表明两者的营养需求更为接近[29]。
Zhang等[8]曾对黄棕壤(pH 6.3)团聚体进行研究,发现TN和C/N能够影响氨氧化微生物丰度,但未对pH和NH4+进行检测。Han等[10]发现在酸性(pH 5.7)旱地农田土壤团聚体中NH4+与AOB和AOA amoA基因丰度正相关,但TN和TC对amoA基因丰度变化的贡献度高于NH4+,未对团聚体pH进行检测。Jiang等[9]对长期施肥管理下酸性(pH 4.62~6.79)农田土壤进行研究,发现TC和pH分别是影响团聚体上AOA和AOB丰度和组成主要因素。Li等[30]发现在碱性土壤(pH > 8)中,不同粒级团聚体AOA和AOB amoA基因丰度变化由不同环境因子所驱动,TN、SOC和NO3 –与AOA群落组成显著关联,而pH和SOC是影响AOB群落的主要因素。由此可见,硝化微生物在团聚体粒级间的分布模式受到土壤类型、耕作方式、施肥管理等多因素共同作用,与团聚体形成和性质密不可分。由于目前土壤团聚体的研究方法缺乏统一标准,粒径划分、筛分方法以及检测指标等不一致,极大增加了对已有数据的整合难度。硝化作用各功能菌群在团聚体微域环境的分异机制有待深入挖掘。
全球范围内土壤硝化速率与土壤pH、SOC、TN、NH4+、AP等显著正相关,与C/N显著负相关[31]。在微域尺度(团聚体水平)上,环境因子如何影响土壤团聚体硝化速率尚不明确。由于土壤团聚体的原位硝化速率较难获取,本研究试图通过传统的悬浮培养法测定团聚体硝化潜势,以比较不同粒级团聚体的硝化活性。结果表明pH、NH4+和TN能显著影响团聚体硝化潜势,与全球尺度下的研究结果较为一致。土壤NP与AOB amoA基因丰度显著正相关,与AOA amoA基因丰度显著负相关,表明AOB是旱地农田原位状态下硝化作用的驱动者,但与高氮投入下活性硝化微生物类群也保持高度一致[32]。由此可见,虽然微域尺度上各因素变化幅度十分微小,但仍能显著影响硝化微生物的分布和活性。
目前团聚体水平硝化活性与硝化微生物的关联报道十分有限,硝化潜势与不同类型硝化微生物(如AOB/AOA)的相关性并不一致[8-9,11],也可能与硝化潜势测定方法的局限性有关。最新研究指出土壤硝化潜势的测定方法存在不足,理论“最佳”条件下测定的土壤硝化潜势不一定代表原位土壤硝化活性[33]。然而,当前未能解决检测土壤团聚体真实硝化活性的技术难点。原位稳定同位素标记结合Nano SIMS亚细胞成像技术有望成为阐明土壤中目标微生物活跃类群空间分布规律的有力突破点[34]。通过稳定同位素(13C或15N底物)在原位条件标记土壤硝化微生物生物质,继而在实验室进行团聚体筛分,再利用Nano SIMS亚细胞成像技术进行硝化微生物细胞活性分析,或许可以提供更为准确的、能够反映自然条件下微生物活性及空间分布的信息。
值得注意的是,本研究中禾豆轮作只进行了一轮,相关土壤结构和微生物变化是在不同作物茬口下得到的,虽然能够最大程度体现种植不同作物产生的直接影响,但无法评估这些土壤结构及微生物变化的可持续性和生态效益。因此,未来可实施多年轮作,并在相同茬口和不同茬口下进行土壤和作物的多维分析,以便获取连续、详实而全面的轮作“茬口”效应研究结果。
4 结论玉米连作和玉米/花生轮作方式下,种植方式和团聚体粒径对土壤团聚体的多种理化性质均产生较大影响,且种植方式能够强烈改变土壤硝化潜势。土壤团聚体粒径和种植方式还能较大程度影响硝化微生物在土壤团聚体中的分布,不同氨氧化微生物在团聚体上的分异规律具有明显差异。AOB amoA基因丰度在轮作土壤显著高于连作土壤,且AOB更能适应较小团聚体环境;而AOA和Comammox amoA基因丰度变化与AOB则大致相反,倾向在较大团聚体中更占优势。此外,团聚体微域环境对不同类型硝化微生物的群落组成还存在不同程度影响。
| [1] |
Wang H, Han S, Tang S, et al. Continuous multi-year application of Chinese milk vetch in paddy soil and its effect on soil aggregates distribution and their carbon and nitrogen content (In Chinese)[J]. Acta Pedologica Sinica, 2022. DOI:10.11766/trxb202106090303 [王慧, 韩上, 唐杉, 等. 紫云英翻压还田对稻田土壤团聚体组成及其碳氮的影响[J]. 土壤学报, 2022.]
(  0) 0) |
| [2] |
Tong L H, Zhu L, Lv Y Z, et al. Response of organic carbon fractions and microbial community composition of soil aggregates to long-term fertilizations in an intensive greenhouse system[J]. Journal of Soils and Sediments, 2020, 20(2): 641-652. DOI:10.1007/s11368-019-02436-x
(  0) 0) |
| [3] |
Jiang X Y, Zhang Q Q, Shen H J, et al. Effects of biochar on soil aggregate stability and microbial community in paddy field (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(6): 1564-1573. [蒋雪洋, 张前前, 沈浩杰, 等. 生物质炭对稻田土壤团聚体稳定性和微生物群落的影响[J]. 土壤学报, 2021, 58(6): 1564-1573.]
(  0) 0) |
| [4] |
Li N, Han X Z, You M Y, et al. Research review on soil aggregates and microbes (In Chinese)[J]. Ecology and Environmental Sciences, 2013, 22(9): 1625-1632. DOI:10.3969/j.issn.1674-5906.2013.09.027 [李娜, 韩晓增, 尤孟阳, 等. 土壤团聚体与微生物相互作用研究[J]. 生态环境学报, 2013, 22(9): 1625-1632.]
(  0) 0) |
| [5] |
Prosser J I, Hink L, Gubry-Rangin C, et al. Nitrous oxide production by ammonia oxidizers: Physiological diversity, niche differentiation and potential mitigation strategies[J]. Global Change Biology, 2020, 26(1): 103-118. DOI:10.1111/gcb.14877
(  0) 0) |
| [6] |
Koch H, van Kessel M A H J, Lücker S. Complete nitrification: Insights into the ecophysiology of comammox Nitrospira[J]. Applied Microbiology and Biotechnology, 2019, 103(1): 177-189. DOI:10.1007/s00253-018-9486-3
(  0) 0) |
| [7] |
Koch H, Lücker S, Albertsen M, et al. Expanded metabolic versatility of ubiquitous nitrite-oxidizing bacteria from the genus Nitrospira[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(36): 11371-11376.
(  0) 0) |
| [8] |
Zhang Q, Liang G Q, Myrold D D, et al. Variable responses of ammonia oxidizers across soil particle-size fractions affect nitrification in a long-term fertilizer experiment[J]. Soil Biology & Biochemistry, 2017, 105: 25-36.
(  0) 0) |
| [9] |
Jiang Y J, Jin C, Sun B. Soil aggregate stratification of nematodes and ammonia oxidizers affects nitrification in an acid soil[J]. Environmental Microbiology, 2014, 16(10): 3083-3094. DOI:10.1111/1462-2920.12339
(  0) 0) |
| [10] |
Han S, Luo X S, Tan S, et al. Soil aggregates impact nitrifying microorganisms in a vertisol under diverse fertilization regimes[J]. European Journal of Soil Science, 2020, 71(3): 536-547. DOI:10.1111/ejss.12881
(  0) 0) |
| [11] |
Hou Q, Zuo T, Wang J, et al. Responses of nitrification and bacterial community in three size aggregates of paddy soil to both of initial fertility and biochar addition[J]. Applied Soil Ecology, 2021, 166: 104004. DOI:10.1016/j.apsoil.2021.104004
(  0) 0) |
| [12] |
Drury C F, Yang X M, Reynolds W D, et al. Influence of crop rotation and aggregate size on carbon dioxide production and denitrification[J]. Soil and Tillage Research, 2004, 79(1): 87-100. DOI:10.1016/j.still.2004.03.020
(  0) 0) |
| [13] |
Hart S C, Stark J M, Davidson E A, et al. Nitrogen mineralization, immobilization, and nitrification[M]// Weaver R W, Angle S, Bottomley P, et al. Methods of soil analysis: Part 2 Microbiological and biochemical properties. SSSA Book Series. Madison, WI, USA: Soil Science Society of America, 1994: 985—1018.
(  0) 0) |
| [14] |
Yao H Y, Gao Y M, Nicol G W, et al. Links between ammonia oxidizer community structure, abundance, and nitrification potential in acidic soils[J]. Applied and Environmental Microbiology, 2011, 77(13): 4618-4625. DOI:10.1128/AEM.00136-11
(  0) 0) |
| [15] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: Molecular fine-scale analysis of natural ammonia- oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/aem.63.12.4704-4712.1997
(  0) 0) |
| [16] |
Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688.
(  0) 0) |
| [17] |
Zhao Z R, Huang G H, He S S, et al. Abundance and community composition of comammox bacteria in different ecosystems by a universal primer set[J]. Science of the Total Environment, 2019, 691: 146-155. DOI:10.1016/j.scitotenv.2019.07.131
(  0) 0) |
| [18] |
Aigle A, Prosser J I, Gubry-Rangin C. The application of high-throughput sequencing technology to analysis of amoA phylogeny and environmental niche specialisation of terrestrial bacterial ammonia-oxidisers[J]. Environmental Microbiome, 2019, 14(1): 3. DOI:10.1186/s40793-019-0342-6
(  0) 0) |
| [19] |
Pester M, Maixner F, Berry D, et al. NxrB encoding the beta subunit of nitrite oxidoreductase as functional and phylogenetic marker for nitrite-oxidizing Nitrospira[J]. Environmental Microbiology, 2014, 16(10): 3055-3071. DOI:10.1111/1462-2920.12300
(  0) 0) |
| [20] |
Alves R J E, Minh B Q, Urich T, et al. Unifying the global phylogeny and environmental distribution of ammonia-oxidising archaea based on amoA genes[J]. Nature Communications, 2018, 9: 1517. DOI:10.1038/s41467-018-03861-1
(  0) 0) |
| [21] |
Yao X D, Li X G, Ding C F, et al. Microzone distribution characteristics of soil microbial community with peanut cropping system, monocropping or rotation (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(4): 975-985. [姚小东, 李孝刚, 丁昌峰, 等. 连作和轮作模式下花生土壤微生物群落不同微域分布特征[J]. 土壤学报, 2019, 56(4): 975-985.]
(  0) 0) |
| [22] |
Wu Y L, Zhao X S, Song W F, et al. Effects of peanut and corn planting patterns on soil nutrient and biological characteristics in horqin sandy land (In Chinese)[J]. Journal of Anhui Agricultural Sciences, 2022, 50(5): 48—54, 60. [吴宇伦, 赵雪淞, 宋王芳, 等. 花生和玉米种植模式对科尔沁沙地土壤养分和生物特性的影响[J]. 安徽农业科学, 2022, 50(5): 48—54, 60.]
(  0) 0) |
| [23] |
Yan C B, Zhao Y, Hu F C, et al. Community structure and diversity of soil bacteria in intercropping Arachis pintoi under Areca Catechu forest (In Chinese)[J]. Soil and Fertilizer Sciences in China, 2022(2): 42-53. [颜彩缤, 赵亚, 胡福初, 等. 槟榔林下间作平托花生土壤细菌群落结构及多样性研究[J]. 中国土壤与肥料, 2022(2): 42-53.]
(  0) 0) |
| [24] |
Li W J, Yang D L, Zhao J N, et al. Progress on effects of long-term continuous cropping and rotation systems on farmland soil biological characteristics (In Chinese)[J]. Chinese Agricultural Science Bulletin, 2015, 31(3): 173-178. [李文娇, 杨殿林, 赵建宁, 等. 长期连作和轮作对农田土壤生物学特性的影响研究进展[J]. 中国农学通报, 2015, 31(3): 173-178.]
(  0) 0) |
| [25] |
Cao L, Qin S H, Zhang J L, et al. Effect of leguminous forage rotations on soil microbe consortiums and enzyme activity in continuously cropped potato fields (In Chinese)[J]. Acta Prataculturae Sinica, 2013, 22(3): 139-145. [曹莉, 秦舒浩, 张俊莲, 等. 轮作豆科牧草对连作马铃薯田土壤微生物菌群及酶活性的影响[J]. 草业学报, 2013, 22(3): 139-145.]
(  0) 0) |
| [26] |
Hu H W, Zhang L M, Dai Y, et al. pH-dependent distribution of soil ammonia oxidizers across a large geographical scale as revealed by high-throughput pyrosequencing[J]. Journal of Soils and Sediments, 2013, 13(8): 1439-1449.
(  0) 0) |
| [27] |
Prosser J I, Nicol G W. Archaeal and bacterial ammonia- oxidisers in soil: The quest for niche specialisation and differentiation[J]. Trends in Microbiology, 2012, 20(11): 523-531.
(  0) 0) |
| [28] |
Walker C B, de la Torre J R, Klotz M G, et al. Nitrosopumilus maritimus genome reveals unique mechanisms for nitrification and autotrophy in globally distributed marine crenarchaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(19): 8818-8823.
(  0) 0) |
| [29] |
Lin Y X, Ye G P, Ding W X, et al. Niche differentiation of comammox Nitrospira and canonical ammonia oxidizers in soil aggregate fractions following 27-year fertilizations[J]. Agriculture, Ecosystems & Environment, 2020, 304: 107147.
(  0) 0) |
| [30] |
Li P P, Han Y L, He J Z, et al. Soil aggregate size and long-term fertilization effects on the function and community of ammonia oxidizers[J]. Geoderma, 2019, 338: 107-117.
(  0) 0) |
| [31] |
Li Z L, Zeng Z Q, Tian D S, et al. Global patterns and controlling factors of soil nitrification rate[J]. Global Change Biology, 2020, 26(7): 4147-4157.
(  0) 0) |
| [32] |
Shen X Y, Xia W W, Cirenlamu, et al. Effect of allylthiourea on soil nitrification and the underlying microbial mechanism in short-term laboratory microcosms (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(6): 1552-1563. [沈晓忆, 夏围围, 次仁拉姆, 等. 短期培养下抑制剂烯丙基硫脲对土壤硝化作用及微生物的影响[J]. 土壤学报, 2021, 58(6): 1552-1563.]
(  0) 0) |
| [33] |
Hazard C, Prosser J I, Nicol G W. Use and abuse of potential rates in soil microbiology[J]. Soil Biology & Biochemistry, 2021, 157: 108242.
(  0) 0) |
| [34] |
Hu H W, Zhang L M, He J Z. Application of nano-scale secondary ion mass spectrometry to microbial ecology study (In Chinese)[J]. Acta Ecologica Sinica, 2013, 33(2): 348-357. [胡行伟, 张丽梅, 贺纪正. 纳米二次离子质谱技术(Nano SIMS)在微生物生态学研究中的应用[J]. 生态学报, 2013, 33(2): 348-357.]
(  0) 0) |
 2023, Vol. 60
2023, Vol. 60

