土壤盐渍化是一种土壤理化和生物学性质恶化的过程,不仅严重影响耕地质量和粮食生产,同时也威胁着地区的生态环境安全[1-2]。我国现代盐渍土分布范围广泛,面积约为3.69×107 hm2,集中分布于黄河中上游的河套平原、西北内陆、东北地区和黄淮海地区[2]。河套地区是我国盐渍化土壤集中分布区之一,具有硫酸盐型盐渍土、碱化盐渍土等多种盐渍化土壤类型并存和轻度、中度、重度盐渍化土壤交叉分布的特点[3]。土壤盐渍化对土壤养分和有机质含量具有显著影响[4]。盐渍化程度的增加会降低土壤养分的含量,从而影响植物的生长[5]。作为重要的后备耕地资源,盐渍化土壤的改良和利用对于促进土壤养分周转,提高作物生产力具有重要意义。其中,适当的农垦措施是改善盐渍化土壤结构,提高盐碱土壤质量的有效方式[6]。开垦后农田表层土壤中可溶性盐份含量显著降低[7],且耕作后的盐渍化土壤中氮、磷、钾等无机营养和有机质等含量明显提高[8]。同时,盐渍化农田中作物叶片的叶绿素含量和质膜透性也得到提升[9],从而促进光合作用,提高作物产量。然而,河套地区不同土壤盐渍化程度与土壤养分和作物产量的关系仍然缺少定量的表征。
作为土壤环境的重要组成部分,土壤微生物群落维持着土壤中的元素循环,同时对植物的生长和健康起着重要的调节作用[10-11]。土壤微生物包括细菌、古菌、真菌和原生生物等。其中,以细菌和古菌为代表的土壤原核生物参与了土壤元素循环和养分周转的大部分过程,促进了植物对养分的吸收,并且对环境扰动高度敏感[12-15]。例如,氨氧化细菌和古菌是土壤中主要的硝化微生物类群,对于农田土壤氮素周转具有重要作用[16]。土壤中的原核生物也可以通过产生生长素等激素、产生铁载体、抑制病原微生物等途径促进植物的生长和抗性,是植物健康的指示性微生物类群[17-19]。大量基于培养和免培养的研究发现盐渍化显著影响土壤细菌群落组成,并富集了拟杆菌和γ变形菌等细菌类群[20-21]。与细菌相比,古菌通常与极端环境有关[22],然而不同盐渍化程度对土壤古菌群落的驱动效应仍然有待研究。此外,土壤微生物群落的改变也会影响土壤潜在功能,对生态系统的元素循环产生重要影响。因此,系统评估不同土地利用类型和盐渍化程度下土壤微生物群落特征和潜在功能及其驱动因素,对于提升盐渍土农业系统管理具有参考意义。
本研究通过土壤理化性质测定和高通量测序等方法对内蒙古河套地区不同土地利用类型和盐渍化程度下土壤原核生物群落分布及潜在功能进行分析,以揭示原核生物群落组成和潜在功能对不同盐渍化程度和农田耕作措施的响应,并在此基础上,对原核生物群落的环境驱动因子进行分析,进一步探究影响原核生物群落组成的关键环境要素,从而揭示土地利用类型和盐碱梯度变化对盐渍土生态系统的潜在影响。
1 材料与方法 1.1 试验地概况试验所用不同土地利用类型盐渍化土壤采集自内蒙古自治区巴彦淖尔市新公中镇盐渍土分布区(41°4'20"—41°5'00" N,108°0'50"—108°1'20" E)(图 1)。该地区位于河套平原,气候类型属温带大陆性气候,日照充足,昼夜温差大,年降水量约130~170 mm,年蒸发量大于2 000 mm,无霜期160 d左右,年均日照时长约3 200 h。该区域不同程度盐碱地呈插花状交叉分布,根据土壤质地分类标准,该区域土壤质地主要属于粉砂质黏壤土(黏粒20.73%、粉粒54.17%、砂粒25.10%)[23]。盐渍化农田均经过暗管排盐改良处理,种植作物为食用向日葵,耕作种植制度为一年一熟单作制,在盐渍化农田附近,存在有零星盐草和芦苇分布的盐碱荒地。
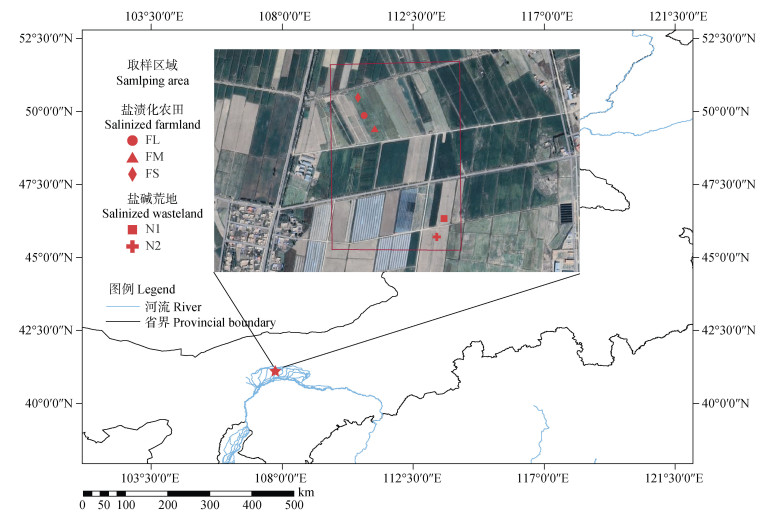
|
图 1 土壤样品采集区域 Fig. 1 Soil samples collection area |
土壤样品于2021年9月底在作物收获期后进行采集,在向日葵种植农田区域中选取盐渍化分级具有明显差异的3处集中取样点,同时根据原位土壤电导率的测定值,将其记为轻度盐渍化农田(FL)、中度盐渍化农田(FM)和重度盐渍化农田(FS)。盐碱荒地土壤样品取自盐渍化农田附近(盐碱荒地取样区域与盐渍化农田取样区域相距小于5 km,面积约1.6 hm2),记为盐碱荒地1(N1)和盐碱荒地2(N2),其中盐碱荒地1零星有盐草和芦苇分布,盐碱荒地2的地表返盐严重且无植被覆盖,分别代表了该地区两种不同程度和景观类型的盐碱荒地。随机取表层0~20 cm土壤的3个点混匀作为一个重复,每个盐碱区域取3个重复。将混匀后的土壤样品分为2份,一份(200 g)储存于4 ℃冰箱中用于测定土壤理化性质,另一份(20 g)放入干冰中运输回实验室,用于土壤总DNA提取。
土壤pH和电导率(EC)通过1︰5土水比提取浸提液并利用电极法测定(Five Easy Plus Conductivity/pH)。土壤容重通过环刀法测定,鲜土在105 ℃条件下烘干48 h后测定土壤含水率。土壤铵态氮和硝态氮经过2 mol·L–1 KCl溶液浸提,分别采用靛酚蓝比色法和紫外分光双波长法测定。土壤有机质采用重铬酸钾外加热法测定。土壤水溶性
称取0.5 g土壤样品,利用FastDNA® Spin Kit(MP Biomedicals,USA)并按照制造商的说明提取土壤总DNA。使用Nanodrop2000(Thermo Scientifc,Wilmington,DE)测定DNA的浓度和纯度。高通量测序使用原核生物特异性引物515FmodF(5’-GTGYCAGCMGCCGCGGTAA-3’)和806RmodR(5’-GGACTACNVGGGTWTCTAAT-3’)对16S rRNA基因V4区进行特异性扩增[26]。将每个样品进行三次PCR扩增并混合,并对混合后的PCR产物进行建库后,利用Illumina NovaSeq平台进行双端测序(上海美吉生物医药科技有限公司)。
原始下机序列使用QIIME2进行质控和分析。首先使用fastp软件(v0.19.6)对原始序列后端质量值低于20的碱基进行裁剪[27],之后用FLASH软件(v1.2.7)将裁剪后的序列进行拼接,允许最大错配的比率为0.2[28]。基于默认参数,利用DADA2插件对拼接后的序列进行降噪并去除嵌合体[29],并使用Naive bayes方法在0.7置信度下对ASVs(扩增子序列变体,amplicon sequence variant)代表序列进行比对(SILVA_v138数据库)。最后将每个样品的序列数抽平至96 229条,并利用抽平后的ASV表进行后续分析。土壤原核生物的原始测序数据已上传到国家微生物科学数据中心(NMDC),编号为NMDC10018162(NMDC20051850-NMDC20051864)。
1.4 数据处理采用单因素方差分析(One-way ANOVA)对不同盐渍化土壤理化性质和原核生物α多样性进行显著性检验。利用Spearman相关分析对土壤理化性质间及其与土壤原核生物α多样性间进行相关性检验。基于R语言平台,在ASV水平上利用非度量多维尺度分析(NMDS)对原核生物β多样性进行降维,并用ANOSIM方法进行显著性检验[30]。韦恩图用来展示不同盐渍化土壤共有和特有的原核生物ASVs。使用FARPROTAX方法对原核生物群落的潜在功能进行预测[31]。不同土地利用类型原核生物群落和潜在功能组成的差异采用Wilcoxon秩和检验。基于vegan包进行原核生物群落和土壤理化性质间的典范对应分析(CCA),并将膨胀因子(VIF)大于20的理化指标去除以降低共线性[30]。同时,对原核生物群落和土壤理化性质间的相关性进行Mantel检验,并进行999次置换检验(Permutation test)。
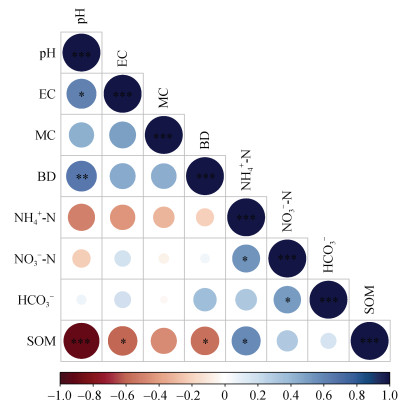
2 结果 2.1 土壤基本理化性质通过对土壤理化指标进行测定,结果表明,盐渍化农田中土壤pH和电导率(EC)显著低于盐渍化荒地(P < 0.05),平均降低了0.93个单位和92%;而有机质(SOM)含量显著高于盐渍化荒地(P < 0.05),平均含量由1.13 g·kg–1提至13.15 g·kg–1(表 1)。作物收获后,土壤无机氮主要以硝态氮为主,两种土地利用类型中盐渍化程度较高(FS和N2)的土壤中硝态氮含量显著高于较低盐渍化程度的处理(P < 0.05)。与轻度盐渍化农田相比,中度和重度盐渍化农田作物产量显著降低了15.2%和49.7%(P < 0.05)。Spearman相关性分析结果表明,pH与EC和土壤容重(BD)显著正相关,而SOM与土壤pH、EC和BD显著负相关(P < 0.05,图 2)。
|
|
表 1 不同土地利用类型下的盐渍化土壤基本理化性质 Table 1 Basic physiochemical properties in saline soils with different land use types |
|
注:1)EC:电导率;MC:水分含量;BD:容重;SOM:土壤有机质。2)不同颜色代表相关性的大小,星号表示显著性(* P < 0.05,** P < 0.01,*** P < 0.001)。下同。 Note: 1)EC: Electrical conductivity; MC: Moisture content; BD: Bulk density; SOM: Soil organic matter. 2)Different colors represent the correlation coefficient, whereas the asterisks represent a level of significance(* P < 0.05, ** P < 0.01, *** P < 0.001). The same below. 图 2 土壤理化性质间的Spearman相关性 Fig. 2 The Spearman correlations between soil physiochemical properties |
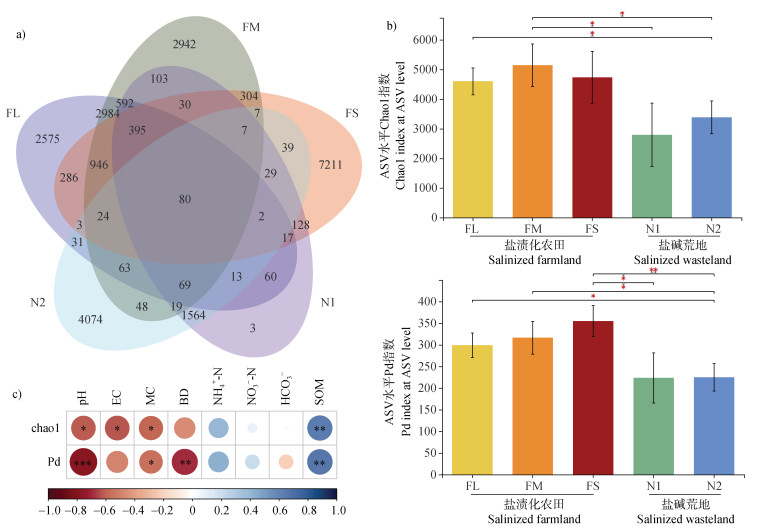
原始数据通过质控后,所有样品中共检测到27 724个原核生物ASV。维恩(Venn)图结果表明不同土地利用类型和盐渍化程度土壤共有的ASV数为80,而大多数ASV为不同盐渍化程度土壤所特有(图 3a)。FL、FM、FS、N1和N2土壤中特有的ASV数分别为2 575、2 942、7 211、3 079和4 074,表明在两种土地利用类型中相对盐度较高的土壤(FS和N2)中具有更多特有的ASV。此外,不同程度盐渍化农田(FL、FM、FS)3个处理中共有946个ASV,而不同地表景观盐渍化荒地(N1和N2)2个处理中共有1 564个ASV。
|
注:韦恩图代表不同盐渍化土壤共有和特有的原核生物ASVs(a)。不同盐渍化土壤原核生物α多样性指数及其差异显著性(b)。土壤原核生物α多样性与土壤理化性质间的Spearman相关(c)。 Note: The Venn diagram represents the number of shared and specific ASVs across different saline soils(a). The significance of α-diversity indexes in difference saline soils(b). The Spearman correlations between soil prokaryotic α-diversity and soil physiochemical properties(c). 图 3 不同土地利用类型下盐渍化土壤原核生物α多样性及其与土壤理化性质的关系 Fig. 3 The soil prokaryotic α-diversity in saline soils with different land use types and their relationship with soil physicochemical properties |
进一步在ASV水平上计算原核生物α多样性(丰富度Chao1指数和系统发育多样性Pd指数)。结果表明,盐渍化农田土壤原核生物α多样性(Chao1指数和Pd指数)显著高于盐碱荒地(P < 0.05),Chao1指数和Pd指数较盐碱荒地分别增加了56.19%和44.2%;而不同程度盐渍化农田(FL、FM、FS)和盐渍化荒地(N1和N2)两个分组内部的细菌群落α多样性均无显著差异(图 3b)。FL和FM样本的Chao1指数和Pd指数显著高于N2样本(P < 0.05),FS样品的Pd指数显著高于N1和N2样品(P < 0.05)。原核生物α多样性与pH、EC、含水率(MC)和BD呈显著负相关关系,而与SOM显著正相关(P < 0.05,图 3c)。
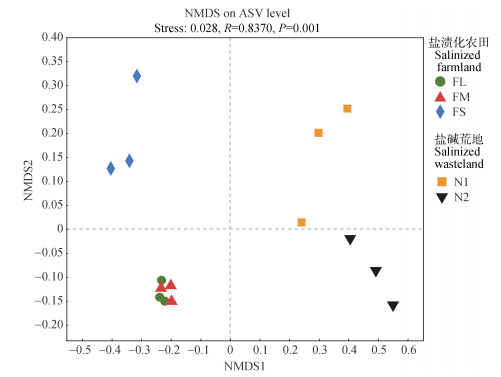
2.3 原核生物群落组成及优势类群差异基于Bray_Curtis距离在ASV水平上进行NMDS分析和ANOSIM组间差异检验,结果表明,土壤原核生物群落在不同土地利用类型和盐碱程度下存在显著差异(R = 0.837,P = 0.001)。两种土地利用类型的样品在第1轴存在明显分异,表明土地利用类型是影响原核生物群落的主要因素。进一步发现FL和FM两个处理间原核生物群落β多样性组间差异较小,其物种群落结构较相似,而与FS之间β多样性具有明显分异(图 4)。
|
图 4 不同土地利用类型下盐渍化土壤原核生物β多样性NMDS分析 Fig. 4 Non-metric multidimensional scaling(NMDS)analysis of prokaryotic β-diversity in saline soils with different land use types |
在门水平上分析不同土地利用类型和盐渍化程度中土壤优势原核生物类群的组成和差异(图 5a,图 5b)。土壤中原核生物类群主要包括变形菌门(Proteobacteria)、放线菌门(Actinobacteriota)、酸杆菌门(Acidobacteriota)、绿弯菌门(Chloroflexi)、盐杆菌门(Halobacterota)、芽单胞菌门(Gemmatimonadota)和浮霉菌门(Planctomycetota)等,分别占总序列的19.97%、14.07%、10.70%、10.70%、9.91%、5.65%和5.34%(图 5a)。由于盐碱农田和荒地土壤微生物存在显著差异,在门水平上比较了具有差异的微生物类群(图 5b)。结果表明,酸杆菌门(Acidobacteriota)、绿弯菌门(Chloroflexi)、浮霉菌门(Planctomycetota)、泉古菌门(Crenarchaeota)和黏菌门(Myxococcota)等类群在盐渍化农田中的相对丰度显著高于盐渍化荒地;而盐杆菌门(Halobacterota)和拟杆菌门(Bacteroidota)呈现相反的趋势。有趣的是,盐杆菌门(Halobacterota)仅在盐碱荒地处理中被检测出,其在中度盐碱荒地(N1)和较为重度盐碱荒地(N2)中的相对丰度分别为10.13%和39.41%,表明其具有较强的生境偏好性(图 5b)。

|
注:不同土地利用类型下盐渍化土壤原核生物在门水平上的组成(a)。两种土地利用类型盐渍化土壤原核生物在门水平(b)和科水平(c)上的差异。 Note: The stacked bar chart displays the composition of soil prokaryotic communities across diverse saline soils at the phylum level(a). The differences between soil prokaryotic communities in salinized farmland and wasteland at phylum(b)and family(c)levels. 图 5 不同土地利用类型下盐渍化土壤原核生物优势类群及差异 Fig. 5 The composition and differences of soil prokaryotic communities in saline soils with different land use types |
进一步在科水平上分析两种土地利用类型盐渍化土壤原核生物的差异类群(图 5c)。属于盐杆菌门的Haloferacaceae、Halomicrobiaceae和属于变形菌门的Halomonadaceae是盐碱荒地中富集的主要微生物类群(P < 0.01)。同时,与硝化相关的亚硝化球菌(Nitrososphaeraceae)、亚硝化单胞菌(Nitrososmonadaceae)和硝化螺菌(Nitrospiraceae)在盐碱化农田中富集。此外,紫荆菌酶科(Vicinamibacteraceae)、芽单胞菌科(Gemmatimonadaceae)、吡喃单胞菌科(Pyrinomonadaceae)、诺卡氏菌科(Nocardioidaceae)和鞘氨醇单胞菌(Sphingomonadaceae)等微生物类群在盐渍化农田中的相对丰度显著高于其在盐渍化荒地中的相对丰度(P < 0.01)。
2.4 土壤原核生物群落与环境因子的关联为检验盐渍化土壤原核生物群落和土壤环境因子之间的关系,在ASV水平上进行典范对应分析(CCA)和Mantel检验(图 6)。CCA分析结果表明,对盐渍化土壤原核生物分布具有显著影响的环境因子分别为EC(R2 = 0.94,P = 0.001)、pH(R2 = 0.89,P = 0.001)、BD(R2 = 0.71,P < 0.01)和硝态氮含量(R2 = 0.49,P < 0.05)。类似地,Mantel检验发现EC(r = 0.78,P < 0.001)、pH(r = 0.76,P < 0.001)、SOM(r = 0.72,P < 0.001)和BD(r = 0.47,P < 0.001)与土壤原核生物群落组成显著相关。
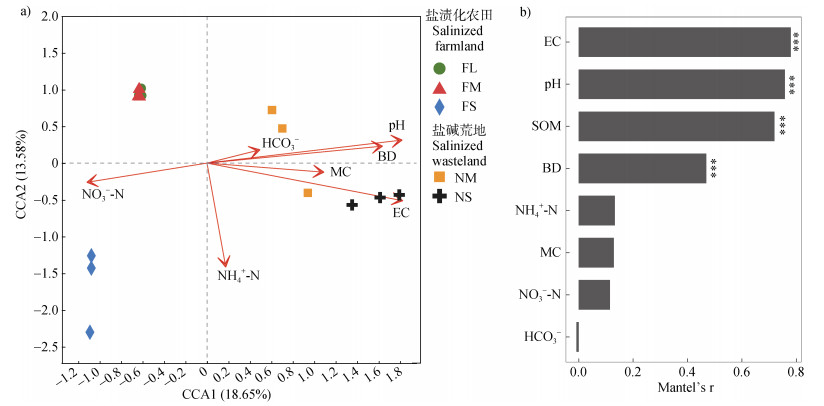
|
图 6 盐渍化土壤原核生物群落和土壤环境因子间的典范对应分析(a)和Mantel检验(b) Fig. 6 Canonical correspondence analysis(a)and Mantel test(b)between soil prokaryotic community and physiochemical properties |
基于FAPROTAX方法进行原核生物群落的功能预测分析,并对盐渍化农田和盐碱荒地两种土地利用类型下微生物功能丰度进行差异分析(图 7)。硝化(nitrification)和好氧氨氧化(aerobic ammonia oxidation)功能的相对丰度在盐渍化农田(13.51%和13.18%)中显著高于盐碱荒地(3.81%和3.42%)(P < 0.01),而碳氢化合物降解(hydrocarbon degradation)功能的相对丰度在盐碱荒地(14.55%)显著高于盐渍化农田(0.06%,P < 0.01)。此外,芳香族化合物降解(aromatic compound degradation)、光合营养(phototrophy)、光合异养(photoheterotrophy)和硝酸盐还原(nitrate reduction)等在两种土地利用类型下的盐渍化土壤中的功能丰度也存在显著差异(P < 0.05),但其相对丰度较低。
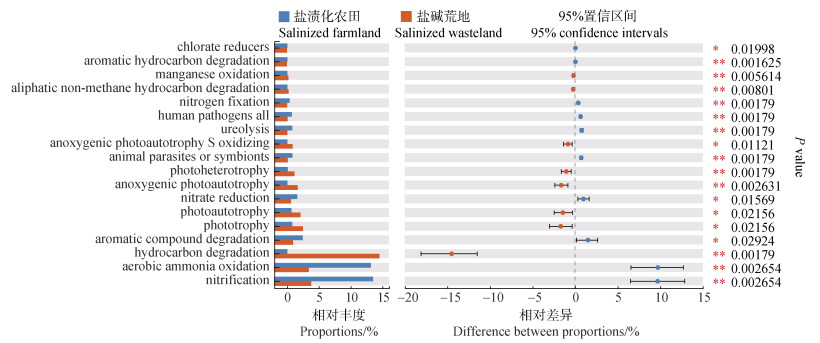
|
图 7 基于FAPROTAX预测的两种土地利用类型盐渍化土壤原核生物潜在功能差异 Fig. 7 The differences in potential soil prokaryotic functions between salinized farmland and wasteland based on the FAPROTAX method |
揭示不同盐渍化程度和土地利用方式对土壤微生物的影响对于提高植物-微生物相互作用、促进土壤养分周转具有重要意义。本研究结果表明,通过暗管排盐措施将盐碱荒地改造成农田,可以提高土壤原核生物的多样性。许多研究表明,微生物多样性对于土壤生态系统功能的维持至关重要,而较高的环境胁迫可能会降低微生物多样性[10]。同时,本研究发现从盐碱荒地到农田的转变影响了微生物的β多样性,这表明盐碱改良过程中土地利用方式的变化塑造了土壤微生物群落。类似地,土壤盐碱程度的增加降低了微生物多样性[21],而在盐碱地改良后土壤中累计呼吸速率和微生物生物量均得到显著提高[32]。进一步通过CCA和Mantel分析结果表明,原核生物群落β多样性主要受到土壤EC、pH、有机质和容重的驱动,结合盐碱地改良后较低的土壤pH、EC以及较高的SOM含量,说明盐碱地耕作改良后土壤化学性质的改善驱动了土壤原核生物群落构建过程。此外,耕作过程中肥料的施用也可以在一定程度上缓解盐分对植物的胁迫[33]。有趣的是,土壤硝态氮含量在重度盐渍化农田和重度盐碱荒地中都高于轻度盐渍化土壤,这可能是由于重度盐渍化农田和重度盐碱荒地中作物产量和植被覆盖率低,抑制了植物对硝态氮的吸收和利用;另一方面,过高的盐分条件下,土壤渗透势降低,加剧了土壤中硝态氮的残留。有研究表明,在盐渍土中,土壤盐分抑制了氧化亚氮还原酶的活性,从而增加硝态氮的固持并促进N2O排放[34]。因此,耕作过程对土壤原核生物多样性的提高和群落组成的改变可能有利于土壤中元素的循环和作物的生长。
3.2 土壤盐碱障碍消减过程影响原核生物类群和潜在功能在盐渍化土壤盐碱障碍消减和改良利用过程中,土壤原核生物群落结构发生显著变化。盐渍化农田的原核生物群落绝大多数都是细菌,而盐碱荒地中古菌的相对丰度为26.0%。与细菌相比,古菌具有较高的抵御环境胁迫的能力,大量的古菌在极端环境中存在[35]。盐碱荒地,尤其是盐碱程度较高的荒地,特异性地富集了较高丰度的盐杆菌。盐杆菌是一类嗜盐古菌,通常存在于具有极高盐分的环境中[36]。同时,盐碱荒地也含有较高丰度的拟杆菌。一项基于宏基因组的研究发现,拟杆菌可以通过高表达渗透压应激基因,从而在高盐度样品中富集[37]。基于FAPROTAX方法的功能预测结果发现,与烃类化合物分解、木聚糖分解和光能异养相关微生物的相对丰度在盐碱荒地中显著增加,这可能是由于盐碱荒地中寡营养的条件,导致微生物倾向于利用难分解的有机物或利用光能分解有机物[36]。
与此相反,酸杆菌门、绿弯菌门、浮霉菌门、泉古菌门和黏细菌门在盐碱农田中显著富集。酸杆菌门是一类耐酸细菌,有研究报道其相对丰度与pH呈负相关关系[38],这与盐碱地改良和耕作后土壤较低的pH一致。黏细菌门是一类捕食性细菌,其在盐渍化农田中具有较高的丰度表明盐碱地改良和耕作有利于微食物网的构建,从而有利于土壤中元素周转和能量流动[39]。在科水平上,盐渍化农田主要富集了芽单胞菌、诺卡氏菌、鞘氨醇单胞菌和丛毛单胞菌植物等有益微生物,这可能与农田作物种植和较高植被覆盖度有关。有趣的是,功能预测结果发现高达13.51%的原核生物类群可以进行硝化作用。同时,差异分析结果也显示以亚硝化球菌为代表的氨氧化古菌,以亚硝化单胞菌为代表的氨氧化细菌,和以亚硝酸盐氧化和全程氨氧化为代表的硝化螺菌的相对丰度显著增加。除硝化外,与尿素水解和固氮有关的微生物丰度在盐碱农田中也显著提高。盐碱农田中较高的氮循环微生物丰度可能与氮肥的施用和作物对硝酸盐的利用有关[40]。此外,尽管本研究利用土壤原核生物16S rRNA基因的通用引物进行了群落分析和潜在功能的注释,然而由于样本间理化性质(如盐分含量)差异较大导致DNA提取效率的不同,以及引物扩增和功能预测过程中的偏好性,可能对分析结果产生一定的影响。未来针对土壤微生物宏基因组的研究可以避免引物的偏好性并对盐渍土壤微生物群落功能进行更为稳健的分析。综上,本研究发现盐碱地改良和作物种植导致土壤原核生物从嗜盐和分解难利用碳到氮循环和植物促生等功能过渡,对盐渍土壤生态系统的利用和功能评价具有借鉴意义。
4 结论不同土地利用类型和盐渍化程度对盐碱地原核生物群落多样性和组成具有显著的影响,并受到土壤pH、EC、有机质和容重等理化因子的驱动。从盐渍化荒地改良为盐渍化农田的过程中,富集了氮循环相关和具有潜在植物促生功能的微生物,并降低了嗜盐和耐盐微生物的相对丰度。因此,盐碱地改良治理不仅对扩大后备耕地面积和提高作物生产力有关键作用,对促进土壤养分周转和植物-微生物间的协同互作同样具有重要意义。
| [1] |
Wang Z Q, Zhu S Q, Yu R P, et al. Saline soil in China (In Chinese). Beijing: Science Press, 1993: 1-3. [王遵亲, 祝寿泉, 俞仁培, 等. 中国盐渍土[M]. 北京: 科学出版社, 1993: 1-3.]
(  0) 0) |
| [2] |
Yang J S. Development and prospect of the research on salt-affected soils in China (In Chinese)[J]. Acta Pedologica Sinica, 2008, 45(5): 837-845. DOI:10.3321/j.issn:0564-3929.2008.05.010 [杨劲松. 中国盐渍土研究的发展历程与展望[J]. 土壤学报, 2008, 45(5): 837-845.]
(  0) 0) |
| [3] |
Zhang Y Q, Wang R P, Bai Q Y. Development and change of soil salinization in Hetao irrigation area of Inner Mongolia (In Chinese)[J]. Journal of Irrigation and Drainage, 2018, 37(S1): 118-122. [张义强, 王瑞萍, 白巧燕. 内蒙古河套灌区土壤盐碱化发展变化及治理效果研究[J]. 灌溉排水学报, 2018, 37(S1): 118-122.]
(  0) 0) |
| [4] |
Dendooven L, Alcántara-Hernández R J, Valenzuela-Encinas C, et al. Dynamics of carbon and nitrogen in an extreme alkaline saline soil: A review[J]. Soil Biology & Biochemistry, 2010, 42(6): 865-877.
(  0) 0) |
| [5] |
Cuevas J, Daliakopoulos I N, del Moral F, et al. A review of soil-improving cropping systems for soil salinization[J]. Agronomy, 2019, 9(6): 295. DOI:10.3390/agronomy9060295
(  0) 0) |
| [6] |
Teng Y, Zhang Z X, Si Z J, et al. Impact of vibration-tillage of subsoils on their water retention curves (In Chinese)[J]. Journal of Irrigation and Drainage, 2017, 36(5): 52-58. [滕云, 张忠学, 司振江, 等. 振动深松耕作对不同类型土壤水分特征曲线影响研究[J]. 灌溉排水学报, 2017, 36(5): 52-58.]
(  0) 0) |
| [7] |
Zhou L, Wang Y G, Li Y, et al. Effects of cultivation on soil salinity in upper soil profiles of the saline-alkali land (In Chinese)[J]. Arid Land Geography, 2013, 36(2): 285-291. [周丽, 王玉刚, 李彦, 等. 盐碱荒地开垦年限对表层土壤盐分的影响[J]. 干旱区地理, 2013, 36(2): 285-291.]
(  0) 0) |
| [8] |
Ren X Y, Chen Y Y, Liang X H. Effect of Fenlong tillage on soil nutrients and maize yield in saline land of Ningxia Yinbei irrigation area (In Chinese)[J]. Southwest China Journal of Agricultural Sciences, 2022, 35(5): 1063-1068. [任晓月, 陈彦云, 梁新华. 粉垄耕作对宁夏银北盐碱地土壤养分及玉米产量的影响[J]. 西南农业学报, 2022, 35(5): 1063-1068.]
(  0) 0) |
| [9] |
Wu J Y, Liu J H, Li Q, et al. Effect of tillage techniques and planting depth on saline-alkali tolerance of oats in Inner Mongolia (In Chinese)[J]. Agricultural Research in the Arid Areas, 2009, 27(2): 138-141, 147. [武俊英, 刘景辉, 李倩, 等. 内蒙古地区不同耕作方式与播种深度燕麦耐盐碱性分析[J]. 干旱地区农业研究, 2009, 27(2): 138-141, 147.]
(  0) 0) |
| [10] |
Rath K M, Rousk J. Salt effects on the soil microbial decomposer community and their role in organic carbon cycling: A review[J]. Soil Biology & Biochemistry, 2015, 81: 108-123.
(  0) 0) |
| [11] |
Freedman Z B, Romanowicz K J, Upchurch R A, et al. Differential responses of total and active soil microbial communities to long-term experimental N deposition[J]. Soil Biology & Biochemistry, 2015, 90: 275-282.
(  0) 0) |
| [12] |
Delgado-Baquerizo M, Oliverio A M, Brewer T E, et al. A global atlas of the dominant bacteria found in soil[J]. Science, 2018, 359(6373): 320-325. DOI:10.1126/science.aap9516
(  0) 0) |
| [13] |
Ouyang Y, Norton J M, Stark J M, et al. Ammonia-oxidizing bacteria are more responsive than archaea to nitrogen source in an agricultural soil[J]. Soil Biology & Biochemistry, 2016, 96: 4-15.
(  0) 0) |
| [14] |
Mander C, Wakelin S, Young S, et al. Incidence and diversity of phosphate-solubilising bacteria are linked to phosphorus status in grassland soils[J]. Soil Biology & Biochemistry, 2012, 44(1): 93-101.
(  0) 0) |
| [15] |
Flemming H C, Wuertz S. Bacteria and archaea on Earth and their abundance in biofilms[J]. Nature Reviews Microbiology, 2019, 17(4): 247-260.
(  0) 0) |
| [16] |
He J Z, Zhang L M. Key processes and microbial mechanisms of soil nitrogen transformation (In Chinese)[J]. Microbiology China, 2013, 40(1): 98-108. [贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98-108.]
(  0) 0) |
| [17] |
Kumar Arora N, Fatima T, Mishra J, et al. Halo-tolerant plant growth promoting rhizobacteria for improving productivity and remediation of saline soils[J]. Journal of Advanced Research, 2020, 26: 69-82. DOI:10.1016/j.jare.2020.07.003
(  0) 0) |
| [18] |
Chaparro J M, Sheflin A M, Manter D K, et al. Manipulating the soil microbiome to increase soil health and plant fertility[J]. Biology and Fertility of Soils, 2012, 48(5): 489-499.
(  0) 0) |
| [19] |
Schneider D, Engelhaupt M, Allen K, et al. Impact of lowland rainforest transformation on diversity and composition of soil prokaryotic communities in Sumatra(Indonesia)[J]. Frontiers in Microbiology, 2015, 6: 1339. DOI:10.3389/fmicb.2015.01339
(  0) 0) |
| [20] |
Singh K. Microbial and enzyme activities of saline and sodic soils[J]. Land Degradation & Development, 2016, 27(3): 706-718.
(  0) 0) |
| [21] |
Rath K M, Fierer N, Murphy D V, et al. Linking bacterial community composition to soil salinity along environmental gradients[J]. The ISME Journal, 2019, 13(3): 836-846. DOI:10.1038/s41396-018-0313-8
(  0) 0) |
| [22] |
Hu W G, Hou Q Q, Delgado-Baquerizo M, et al. Continental-scale niche differentiation of dominant topsoil archaea in drylands[J]. Environmental Microbiology, 2022, 24(11): 5483-5497. DOI:10.1111/1462-2920.16099
(  0) 0) |
| [23] |
Wu K N, Zhao R. Soil texture classification and its application in China (In Chinese)[J]. Acta Pedologica Sinica, 2019, 56(1): 227-241. [吴克宁, 赵瑞. 土壤质地分类及其在我国应用探讨[J]. 土壤学报, 2019, 56(1): 227-241.]
(  0) 0) |
| [24] |
Bao S D. Soil and agricultural chemistry analysis (In Chinese). 3rd ed[M]. Beijing: China Agriculture Press, 2000. [鲍士旦. 土壤农化分析[M]. 3版. 北京: 中国农业出版社, 2000.]
(  0) 0) |
| [25] |
Lu R K. Analytical methods for soil and agro-chemistry (In Chinese). Beijing: China Agricultural Science and Technology Press, 2000. [鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000.]
(  0) 0) |
| [26] |
Walters W, Hyde E R, Berg-Lyons D, et al. Improved bacterial 16S rRNA gene(V4 and V4-5)and fungal internal transcribed spacer marker gene primers for microbial community surveys[J]. mSystems, 2015, 1(1): e00009-e00015.
(  0) 0) |
| [27] |
Chen S F, Zhou Y Q, Chen Y R, et al. Fastp: An ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890.
(  0) 0) |
| [28] |
Magoč T, Salzberg S L. FLASH: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507
(  0) 0) |
| [29] |
Callahan B J, McMurdie P J, Rosen M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869
(  0) 0) |
| [30] |
Oksanen J, Kindt R, Legendre P, et al. The vegan package[J]. Community Ecology Package, 2009, 10: 631-637.
(  0) 0) |
| [31] |
Louca S, Parfrey L W, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome[J]. Science, 2016, 353(6305): 1272-1277.
(  0) 0) |
| [32] |
Wong V N L, Dalal R C, Greene R S. Carbon dynamics of sodic and saline soils following gypsum and organic material additions: A laboratory incubation[J]. Applied Soil Ecology, 2009, 41(1): 29-40.
(  0) 0) |
| [33] |
Gao S, Yang J S, Yao R J, et al. Effects of soil amelioration measures mitigating soil salinity and improving crop P uptake in coastal area of north Jiangsu (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(5): 1219-1229. [高珊, 杨劲松, 姚荣江, 等. 改良措施对苏北盐渍土盐碱障碍和作物磷素吸收的调控[J]. 土壤学报, 2020, 57(5): 1219-1229.]
(  0) 0) |
| [34] |
Ghosh U, Thapa R, Desutter T, et al. Saline-sodic soils: Potential sources of nitrous oxide and carbon dioxide emissions?[J]. Pedosphere, 2017, 27(1): 65-75.
(  0) 0) |
| [35] |
Konings W N, Albers S V, Koning S, et al. The cell membrane plays a crucial role in survival of bacteria and archaea in extreme environments[J]. Antonie Van Leeuwenhoek, 2002, 81(1): 61-72.
(  0) 0) |
| [36] |
Shu W S, Huang L N. Microbial diversity in extreme environments[J]. Nature Reviews Microbiology, 2022, 20(4): 219-235.
(  0) 0) |
| [37] |
Pandit A S, Joshi M N, Bhargava P, et al. A snapshot of microbial communities from the Kutch: One of the largest salt deserts in the World[J]. Extremophiles, 2015, 19(5): 973-987.
(  0) 0) |
| [38] |
Liu J J, Sui Y Y, Yu Z H, et al. High throughput sequencing analysis of biogeographical distribution of bacterial communities in the black soils of northeast China[J]. Soil Biology & Biochemistry, 2014, 70: 113-122.
(  0) 0) |
| [39] |
Wang W H, Wang N, Dang K K, et al. Long-term nitrogen application decreases the abundance and copy number of predatory myxobacteria and alters the myxobacterial community structure in the soil[J]. Science of the Total Environment, 2020, 708: 135114.
(  0) 0) |
| [40] |
Wang J, Cheng Y, Cai Z C, et al. Effects of long-term fertilization on key processes of soil nitrogen cycling in agricultural soil: A review (In Chinese)[J]. Acta Pedologica Sinica, 2016, 53(2): 292-304. [王敬, 程谊, 蔡祖聪, 等. 长期施肥对农田土壤氮素关键转化过程的影响[J]. 土壤学报, 2016, 53(2): 292-304.]
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


