2. 浙江农林大学环境与资源学院, 杭州 311300;
3. 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所), 南京 210008;
4. 浙江省耕地质量与肥料管理总站, 杭州 310020
2. College of Environment and Resources, Zhejiang A&F University, Hangzhou 311300, China;
3. State Key Laboratory of Soil & Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
4. Zhejiang Cultivated Land Quality and Fertilizer Administration Station, Hangzhou 310020, China
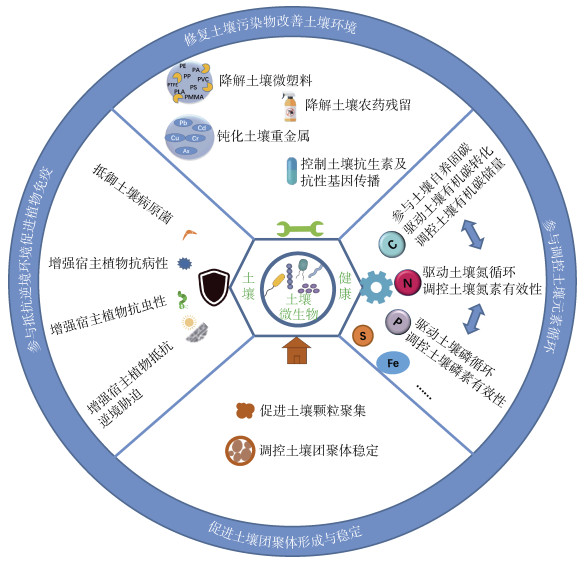
土壤是植物生产的载体,同时也是生态环境的基本要素。土壤健康包括能够维持农业生态系统服务功能的一系列生物、化学和物理的土壤特性,是维持农业生产发展可持续性以及保障粮食安全的关键。长期以来,以单一种植、化肥和农药、机械化和灌溉为特征的集约化农业实践在保障粮食生产的同时对土壤健康产生了极大的威胁。目前,土壤健康状况评估以及可持续性管理正成为全球关注的焦点和热点[1-3]。土壤健康不仅仅是土壤物理、化学特性及生物学特性的量化产物,还代表土壤生命体的运转状态,依赖于作物生产、土壤碳固持与气候调节、营养供给与循环、生物多样性、污染控制等主要功能的维持[4](图 1)。这些功能均表现为在非生物土壤环境影响下,相互作用的生物多样性所提供的各种生物过程的集合体。
|
注:微生物参与多种土壤元素循环,本文仅以碳、氮、磷为主要论述内容。 Note: Microorganisms participate in various soil element cycles. This paper mainly discusses their roles in the carbon, nitrogen and phosphorus cycle. 图 1 微生物影响土壤健康的主要功能框架 Fig. 1 Main functional framework of microbial involvement in soil health regulation |
近年来,随着对土壤微生物在有机质积累、养分循环和植物生长等方面作用的深入认知,研究者们逐渐意识到微生物在土壤生态系统功能中的重要性。土壤微生物在养分转化过程起着土壤养分循环“调配器”的作用,控制土壤养分循环的方向、养分元素化合物种类和交换通量[5]。土壤微生物参与调控温室气体的排放以及土壤污染物的无害化降解,对于维持农田生态环境功能具有重要作用。土壤微生物群落和多样性调控土壤生态系统的多功能性,进而影响土壤本身的抵抗力和恢复力,对维持土壤肥力和可持续性生产至关重要[6]。因此,土壤微生物参与土壤生态功能、环境功能、免疫功能协同调控土壤健康,是维持土壤健康的核心与关键[7]。
量化微生物与土壤生态系统功能之间关系,揭示微生物介导的土壤功能如何调控土壤健康,是评估和管理土壤健康的一项重要任务。同时,利用多组学技术以及学科交叉挖掘和操纵土壤微生物群落来改善土壤环境、促进土壤健康,是定向培育健康土壤,提高农业可持续生产能力的目标。本文从微生物调控土壤元素循环、参与土壤团聚体形成与稳定、参与土壤抵抗逆境环境、降解土壤污染物以及利用微生物预测土壤健康等方面系统综述了土壤微生物影响土壤健康的作用机制,并对未来微生物介导的土壤功能与土壤健康研究方向及重点作出了展望,以期为健康土壤的维持与培育,促进农业生产的可持续发展提供理论支持。
1 土壤微生物参与调控土壤碳循环 1.1 微生物参与土壤有机碳的分解损失与同化固存土壤中丰富的有机碳是土壤健康的重要指标和保证,土壤微生物通过调控有机碳的同化与矿化分解影响土壤净碳平衡。土壤胞外酶是土壤微生物介导土壤有机碳矿化的主要方式,土壤中微生物来源的酶对土壤有机底物催化分解产生的CO2高达土壤呼吸的1/2。微生物生长环境与自身的化学计量不平衡匹配是土壤胞外酶驱动碳分解的主要因素,例如氮素施用通过调节胞外酶活性(Extracellular enzyme activity,EEA)影响土壤有机碳(Soil organic carbon,SOC)分解速率[8]。此外,微生物个体的生理及群体生态特征直接调控整个群落的功能性状,进而影响土壤碳矿化[9]。土壤微生物碳泵理论指出,微生物通过胞外酶“体外修饰”不断将不易利用的有机底物剪切为小分子易吸收利用的底物后,继续通过“体内周转”使微生物残留物迭代持续积累,促进一系列包括微生物死亡残体和部分代谢产物在内的有机物质的形成,最终导致此类化合物稳定于土壤中形成微生物的“续埋效应”(Entombing Effect)[10]。有研究表明微生物的“续埋效应”对土壤有机碳贡献率高达50%~80%[11]。微生物残体对有机碳库的潜在贡献受微生物群体的主导地位调控[12],其中真菌残体对土壤有机碳的贡献(30.0%)高于细菌残体(15.5%)[13]。微生物残体的产生受三个生理性状控制:微生物生长速率(Microbial growth rate,MGR)、微生物碳利用效率(Carbon use efficiency,CUE)以及微生物生物量周转率(Microbial biomass turnover rate,MTR)。有研究表明,气候变化、农业管理措施等因素引起的SOC的变化均与CUE有关[14-15]。微生物的多样性以及组成则是影响土壤微生物CUE的关键因子,更多样化的群落具有更高的碳利用效率,同时受微生物个体代谢途径及活性差异影响,土壤微生物群落组成、CUE及SOC之间也存在显著相关性[16]。在农业土壤中,革兰氏阳性(G+)和革兰氏阴性(G–)细菌群体使用不同的途径来代谢葡萄糖,进而导致不同CUE[17]。但是,目前微生物类群对CUE及SOC的贡献仍然存在争议,例如有研究表明以真菌为主的微生物群落具有相对较高的CUE和SOC[16],然而也有研究表明微生物群落的差异对CUE的影响不大[18]。因此,未来仍需继续破解微生物个体或群体如何通过同化代谢驱动细胞残体及土壤有机碳积累的贡献机制。
1.2 微生物自养固碳土壤中的自养微生物是CO2转化为有机碳的重要触发器,在提高有机碳库累积中发挥关键作用。据估计自养微生物在森林、旱地和水稻表层(0~20 cm)土壤的碳输入量可分别达到142~194、128~174和536~731 kg·hm–2·a–1,且其固定的碳约有74%可以进入到腐殖质等稳定的土壤有机碳库中[19],对土壤稳定性碳组分的提升具有较大潜力[20]。土壤微生物通过还原磷酸戊糖循环(Calvin循环)、还原柠檬酸循环(rTCA循环)、羟基丙酸-羟基丁酸循环(3-HP/4-HB循环)、二羧酸-羟基丁酸循环(DC/4-HB循环)、3-羟基丙酸循环(3-HP循环)和还原乙酰基-辅酶a途径(WL途径)等多条固碳途径进行CO2同化(表 1)[21]。近年来,基因组学的应用在固碳途径标记基因的挖掘和相关微生物类群分布及其与固碳效率的关系研究不断突破。例如,Calvin循环中cbbL/cbbM基因,rTCA通路中aclA以及korA/B基因,4-HB循环中的hcd基因,3-HP/4-HB循环中atoB基因已被广泛作为分子标记反映固碳能力差异[22-23]。土壤自养固碳效率不仅受固碳微生物的丰度、多样性调控[23-24],还与自养微生物共生网络的生态集群高度相关[25],且受到土壤水分、pH、全磷、EC和C:N等非生物因素调控[25-26]。虽然影响土壤微生物固定二氧化碳能力的关键环境因子已引起关注,但是由于土壤的高度异质性,不同生境关键固碳微生物类群以及在复杂环境中的具体作用和贡献还存在争议,仍需进一步研究。
|
|
表 1 土壤中常见的微生物固定二氧化碳的主要途径[21,23] Table 1 Main CO2 fixation pathways of CO2 fixing microorganisms common in soils[21, 23] |
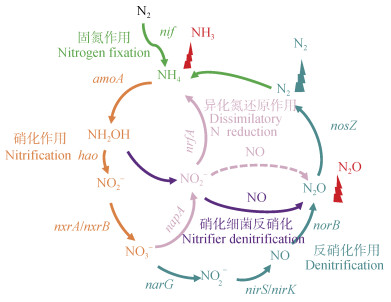
氮有效性是调节农业生产的重要因素之一,土壤中氮素转化过程对土壤氮素有效性和氮肥利用效率具有重要影响。目前,农业生态系统生物固氮(Biological nitrogen fixation,BNF)量每年约为50~70 Tg N[27]。豆科植物和根瘤菌共生固氮是主要来源[28],另一类是来源于不形成根瘤的联合固氮,此外是蓝细菌(Cyanobacteria)、褐球固氮菌(Azotobacter chroococcum)和梭菌(Clostridium prazmowski)参与的自生固氮[29]。优势固氮菌的演替是生物固氮变化的主要因素,高氮肥用量导致根瘤菌和豆科植物之间的共生关系瓦解,抑制土壤中的生物固氮功能[30],而通过基因工程技术对固氮酶基因nif调控网络关键节点进行基因编辑可以优化外源氮存在条件下微生物的固氮能力[31]。同时,外源施用有机物料通过提升土壤有机质含量有利于固氮菌生长,也可以促进土壤生物固氮[32],因此通过施肥方式或基因工程调控土壤微生物群落可以增加生物固氮。此外,固氮菌的接种可以促进土壤固氮,但是固氮菌与土著菌群落的竞争、宿主-共生兼容性以及确保植物整个生命周期的固氮能力仍然是一个挑战。生物固氮作用将大气中的N2转化为NH3后,通过氨氧化古菌及细菌执行的氨氧化以及硝化细菌执行的亚硝酸盐氧化作用将NH3转化为NO3–[33](图 2)。这些硝酸盐可以通过径流或淋洗流失,或者通过反硝化作用转化为N2。然而硝化过程中铵盐的不完全氧化和反硝化过程中亚硝酸盐的不完全还原均会产生副产物——强效温室气体N2O的释放[34]。氨氧化古菌(AOA)利用每摩尔氨产生的N2O低于氨氧化细菌(AOB)[35],因此,AOA与AOB的生态位分异会影响N2O排放,通过农业管理措施提高AOA:AOB的比率对于降低土壤的净N2O排放量具有重要作用。此外,附加微生物和非生物过程的复杂网络也会影响N2O排放。反硝化微生物可以产生N2O,而携带和表达N2O还原酶(由nosZ编码)的微生物可以将N2O还原为N2,N2O的排放降低与较高的nosZ基因丰度有关[36],通过农业措施优化并提升nosZⅡ微生物分支丰度是减少农业土壤N2O的一个重要措施[37]。异化硝态氮还原(DNRA)将硝态氮还原为铵态氮保留在土壤生态系统中,在低硝酸盐和高碳氮比的条件下,更有利于DNRA[38]。因此,调控复杂的微生物氮转化网络可为健康土壤可持续氮素管理提供途径(图 2)。
|
注:不同颜色箭头表示不同的氮素循环过程,箭头上标记基因表示该周转过程中关键酶的标记功能基因。 Note: The arrows with different colors represent the nitrogen cycle processes. And the genes on the arrows are the functional genes encoding key enzymes in nitrogen cycling. 图 2 微生物介导土壤中氮素循环的主要过程[28] Fig. 2 Main process of microbial mediation in soil nitrogen cycling[28] |
土壤中磷的有效性对土壤健康和植物生长至关重要。不同于氮元素,土壤磷元素常以多种有机和无机的形式存在且大多被固定于土壤矿物中,难以被作物直接吸收利用,限制了作物的产量。微生物在土壤磷素循环中发挥着重要作用,对土壤磷素有效性起着调节作用[39]。其中,参与土壤磷循环的微生物主要负责三类功能(表 2):(1)无机磷溶解。在富含矿物磷的土壤中溶解过程是微生物磷周转的主要驱动因素。微生物主要通过有机酸的分泌、NH4+同化作用等伴随pH下降,酸化微生物细胞和周围环境促进无机磷的溶解与利用,且分泌的有机酸还可以与无机磷酸盐结合的阳离子螯合增溶无机磷酸盐[40]。例如,gcd是一种膜结合型醌蛋白葡萄糖脱氢酶,可以氧化醛糖,通过产生葡萄糖酸螯合与磷酸盐结合的阳离子,从而达到增溶无机磷使磷酸盐为植物所用[41]。此外,ppx基因编码的外聚磷酸酶在无机聚磷酸盐降解成磷酸盐中也具有重要作用[42]。(2)有机磷矿化。含有酸性磷酸酶(phoC、phoN、ophA、olpA)、碱性磷酸酶(phoD、phoA、phoX)、植酸酶(appA)和C-P裂解酶(phnJ)等酶编码基因的微生物在土壤中具有较高矿化有机磷的能力。土壤中的碱性磷酸酶微生物主要以phoD为主,且在缺磷土壤中phoD基因丰度增加,碱性磷酸酶活性提高。含有phoD功能性微生物群落中的稀有物种可能是矿化有机磷、缓解缺磷胁迫的关键微生物[43]。此外,土壤不稳定碳、pH和化学计量比也会影响农田土壤中phoD基因的丰度和微生物群落组成[44-45],通过调控碱性磷酸酶的产生驱动土壤有机磷的有效化。(3)磷吸收转运。pit、pstA、pstB和ugpB/C基因的存在可以使微生物能够有效地利用无机磷并固定化为微生物磷,例如编码高亲和力磷酸盐特异性转运体(pst)和低亲和力无机磷酸盐转运体的基因(pit),分别在磷贫瘠或磷充足条件下对促进微生物对磷的固定至关重要[46],且编码高亲和力磷吸收的微生物基因的丰度与土壤磷的低生物有效性之间存在正相关关系[47]。长期氮输入降低微生物对磷的溶磷和矿化能力,而长期磷输入改变微生物的功能分布有利于微生物的固磷[44],从微生物角度考虑有机磷的矿化、无机磷的活化以及微生物的固定化,可以为可持续农业生态系统中磷循环的调控提供新的视角。
|
|
表 2 微生物介导的土壤磷素转化主要过程及相关基因[48-49] Table 2 Microbial mediated soil P cycling process and involved functional genes[48-49] |
土壤团聚体支持根系生长、抵抗土壤侵蚀、具备碳储存和持水能力,良好的团聚土壤结构是确保土壤健康运行的基础。一方面土壤团聚体是各类微生物的微生境,不同粒级团聚体的物理化学特性均存在差异,直接影响其中的微生物的分布、数量、多样性及功能[50];另一方面微生物也是土壤团聚体形成的重要生物因素[51]。首先,土壤有机质是促进土壤团聚体以及土壤结构形成和稳定的一个主要因素,而微生物及其死亡残体是土壤有机碳的重要组成成分[52],表明微生物、大型生物和植物与土壤物理特性密切相关。有研究发现,用田间土壤接种灭菌土壤显著提高了初始结构不良土壤的团聚体稳定性[53],证明了土壤微生物“建造房子”的重要作用。土壤微生物通过多种方式促进土壤团聚体的形成:(1)微生物细胞依靠自身带有的电荷借助静电引力使土壤颗粒彼此连接;(2)微生物分泌物胞外聚合物(Extracellular polymeric substances,EPSs)将土壤颗粒黏结在一起;(3)真菌菌丝和放线菌菌丝将土壤颗粒机械地缠绕在一起[54]。由于微生物在团聚体中的异质性分布,微生物种群各司其职而又相互协同调控团聚体的形成与分布,通常认为真菌更有利于大团聚体的形成,而细菌及其代谢产物更有利于微团聚体的形成。细菌和真菌分泌的胞外多糖在土壤微团聚体稳定中发挥重要作用[55]。细菌分泌的胞外聚合物,作为微米尺度聚集物的黏合剂促进了微土壤团聚体的形成[56],而真菌尤其是丛枝菌根菌丝可以通过交联和嵌套在微米到毫米尺度上将微小的土壤颗粒团聚在一起,产生的胞外聚合物(如多糖)作为临时黏合剂,参与形成稳定的大团聚体[57]。矿物颗粒包裹黏合剂有利于大团聚体内部微团聚体的形成,限制了微团聚体内有机物的分解并减缓了进一步的周转[58]。此外,丛枝菌根真菌产生球囊霉素,在土壤稳定、碳储存和土壤团聚体稳定性中发挥重要作用[59]。由于土壤微生物对土壤团聚体形成的不同贡献,土壤颗粒团聚机制之间也有明显的互补潜力,生物多样性以及主要分类群的物种相互作用是土壤团聚体形成与稳定的关键[51]。虽然细菌和真菌在土壤团聚体形成和稳定中的作用已经建立,但应用微生物改善土壤结构的研究较少。因此,加强微生物生物多样性及其相互作用在调控土壤团聚中的应用,可以为可持续的土壤管理提供理论依据和技术支持。
4 微生物参与抵抗逆境环境促进植物免疫土壤微生物在抵御生物和非生物胁迫中发挥重要作用(图 3)。土壤中的有益微生物可通过生态位和营养的竞争、拮抗物质的产生、寄生、捕食、诱导植物系统抗性以及干扰病原微生物致病信号等方式提高土壤的抗病能力[60]。芽孢杆菌属(Bacillus spp.)和假单胞菌属(Pseudomonas spp.)是最著名的两类生防细菌,主要通过可溶性抑菌物质如抗生素、环脂肽、表面活性剂、胞外多羟基丁酸聚合酶以及产生挥发性有机物等生防因子拮抗、抑制或直接杀害病原物。噬菌体既能通过“专性猎杀”高效裂解病原菌、原生动物,也能利用选择性捕食作用直接降低病原菌的丰度。同时,土著微生物的多样性以及多样化微生物对资源的高效利用是抑制病原菌侵染的关键机制[61-63],竞争能力强的微生物群体形成相互制衡的环境,共同抵御病原体的入侵,降低土传病害的发生[64]。此外,土壤微生物相互作用形成的复杂的、相互连接的微生物网络,会影响互惠、协同、腐生和寄生模式中不同相互依赖成分的组成和功能,对土壤和植物健康产生直接或间接的影响[65]。例如,向土壤中引入的芽孢杆菌使其与土著假单胞菌种群形成互作关系,协同增强多物种的根系定殖,形成益生“菌团”能够有效抵御病原菌侵染植物[66]。因此,创建土壤免疫的“生物疗法”精准调控土壤微生物组,对增加群落中相互作用的有益菌群丰度,减少土壤病原菌的数量,为土壤生物肥力改善、免疫防线构建以及土壤健康的改善提供了新的管理视角。

|
图 3 微生物介导土壤免疫机制模式图 Fig. 3 Model diagram of microbial mediated soil immunity mechanism |
土壤微生物是植物抵抗地上害虫的关键组成部分。有研究表明,向灭菌基质中接种土悬液可显著提升拟南芥叶片对粉纹夜蛾(Trichoplusia ni)幼虫的抗性,证实了土壤中的微生物对植物抗虫性能的贡献[67]。土壤微生物可以直接或者通过植物介导的抗性机制间接影响昆虫取食。来源于土壤的植物内生菌数量会直接影响昆虫对植物叶片的取食,例如,球孢白僵菌(Beauveria bassiana)和绿僵菌(Metarhizium anisopliae)的定殖可以促进植物生长和抗虫性[68];木霉(Trichoderma)等内生真菌定殖可以帮助植物抵抗洋葱蓟马(Thrips tabaci)[69]。此外,根际微生物可以诱导植物的系统抗性,调控植物的表型间接影响昆虫取食,例如,土壤中真菌群落组成的差异影响韧皮部汁液中氨基酸的浓度,间接影响蚜虫取食[70];有机种植下,番茄叶蝉的数量可以减少36%~50%,主要依赖于有机种植下土壤微生物群落改变导致的植物水杨酸积累的影响[71]。因此,土壤或根际微生物的组成及多样性及其与植物的相互作用,对植物的健康生长和病虫害的敏感性降低起重要作用。
5 微生物修复土壤污染物改善土壤环境 5.1 微生物降解土壤微塑料农业土壤中的微塑料(小于5 mm)主要来源于人为的土壤管理,如污水灌溉、污泥应用、农膜覆盖、有机肥的施用等。微塑料进入土壤并在土壤中积累后,改变微生物群落组成、群落周转速率以及代谢功能[72],直接或间接地影响土壤生态系统功能与物质循环。例如,微塑料的分解提供了额外的C,微生物群落代谢状态的改变可能会刺激原生SOM的周转从而影响土壤碳循环[73],微塑料聚合体仅由C、H和O组成,微塑料残留在土壤中可能会诱导土壤中必需的营养元素(例如N、P)的微生物固定化,影响土壤养分循环[74]。生物可降解微塑料显著提高包括脂肪代谢、降解和生物合成等脂质代谢相关功能的微生物丰度[72]。此外,微塑料降解过程中产生的代谢产物会引起土壤环境变化,例如聚羟基丁酸羟基戊酸共聚酯(Poly β-hydroxybutyrate–co-β-hydroxy valerate,PHBV)降解过程中释放出大量的3-羟基丁酸,造成土壤酸化而引起植物中毒[75]。土壤中微塑料残留物或碎片会破坏土壤团聚体以及孔隙连续性[76],而一些颗粒或粉末则很容易填补土壤孔隙空间,同样会改变土壤孔隙度进而改变土壤持水性能[77]。孔隙度和水循环的变化会改变土壤氧通量,改变土壤微生物栖息环境,进而影响土壤中厌氧或好氧微生物的丰度[78]。此外,微塑料的疏水特性使其在土壤中作为疏水性持久有机污染物的载体,吸附土壤重金属与抗生素,成为抗生素抗性基因和潜在致病菌的“热区”,导致严重的复合污染[79]。同时微塑料降解过程中产生的颜料、增塑剂、阻燃剂等物质,在各种环境过程中迁移和传播产生复杂的污染和毒性作用,对土壤健康以及人类的健康造成潜在的威胁。
利用微生物对土壤微塑料的降解功能,有助于恢复土壤健康水平。微生物胞外酶和自由基的分泌可以将高分子的聚合物剪切成小分子低聚物、二聚体或单体,进入微生物细胞内经过同化作用、矿化作用代谢氧化为H2O以及CO2、N2等气体,从而实现微塑料的无害降解[80-81]。通常,微塑料的易降解程度决定微生物降解效率,聚烯烃类的降解效率远低于聚酯类。一些真菌类群在特定情况下可以几乎完全降解聚酯类微塑料(如聚ε-己内酯、聚乳酸、聚3-羟基丁酸酯),而聚烃类如PE在一定时间仅降解约10%~30%[82]。对于聚酯类的降解,微生物主要通过水解酶作用于含氧基团[83],而烯烃类中强C-C骨架通常需要通过氧化作用,将极性基团引入到碳骨架中,增强微生物对微塑料的表面附着,然后降解氧化的微塑料[84]。Aspergillus caespitosus、Phialophora alba、Paecilomyces variotii、Aspergillus terreus、Alternaria alternate和Eupenicillium hirayamae产生漆酶(Laccase)、锰过氧化物酶(Mn peroxidase enzyme,MnP)和木质素过氧化物酶(Lignin peroxidase,LiP)等快速降解低密度聚乙烯薄膜(Low density polyethylene,LDPE)[85]。此外,微生物类群对于微塑料的降解能力也存在差异,真菌对微塑料的降解效果优于细菌。增加土壤中真菌易利用的碳源增强真菌活性,或者加入培养的真菌均会明显加快聚氨酯塑料的降解速度[86]。由于塑料的疏水性,土壤真菌的菌丝通过产生疏水剂附着在微塑料表面[87],同时真菌菌丝的穿透力促进了在塑料际的初始定殖。细菌定殖取决于细胞表面的疏水性,且大多数细菌需要一个与塑料表面的接触点来通过细胞分裂定殖[88]。子囊菌、担子菌和接合菌是在微塑料降解中发挥着重要作用的真菌类群,变形菌门、厚壁菌门、放线菌门和拟杆菌门是参与微塑料降解的主要细菌类群[85]。利用潜在的降解微生物和酶来修复微塑料污染土壤,不产生二次毒性,对于土壤健康水平的维持与修复至关重要。
5.2 微生物钝化土壤重金属重金属一旦进入土壤环境,很难通过化学方法或生物降解反应从土壤中去除。土壤重金属积累超过食物安全利用限值时会威胁人类或动物健康,因此土壤中重金属的修复对于土壤健康和生物有机体健康至关重要。传统的修复方法包括物理化学修复和工程修复,但是会造成二次污染并对土壤特性产生负面影响。相反,土壤中的部分微生物对重金属污染环境具有极强的适应能力,具备耐受或固定重金属的能力,对降低土壤重金属的生物有效性具有重要作用,利用微生物钝化土壤中的重金属是一种高效、经济和环境友好的重金属修复技术。
微生物与金属之间的相互作用包括生物吸附、生物固定、微生物对金属离子的氧化还原以及微生物EPSs与金属离子的配位络合等(图 4)。土壤微生物能与土壤中的重金属离子通过络合作用及配位作用形成化合键吸附土壤重金属,降低土壤重金属的生物可利用率和生物毒性[89],例如假单胞菌(Pseudomonas)、肠杆菌(Enterobacter)、芽孢杆菌(Bacillus)和微球菌(Micrococcus cohn)等对重金属有出色的吸附能力[90]。微生物可通过多种途径多部位螯合重金属离子,一些细胞壁表面的部分化学基团可直接络合重金属离子将其包裹在细胞壁内[91],与细胞表面官能团复合或沉淀在细胞表面[92]。微生物固定依赖于微生物的新陈代谢伴随着能量消耗,通过脂类过氧化、复合物渗透、载体协助、离子泵等运输机制,将土壤中的重金属离子固定在微生物体内,降低生物有效性。例如,从镉污染农田土中分离鉴定出一株镉抗性细菌假单胞菌B7,该细菌通过碳酸酐酶CadW提高细菌对镉离子的抗性,且能够与镉离子特异性结合从而将镉离子固定在细胞内部,培养后可完全去除溶液中的镉离子[93]。EPSs和汞、铅及砷元素可发生配位络合作用,形成稳定的络合物降低重金属的生物有效性[94]。此外,微生物参与氧化还原反应,改变重金属的价态,进而影响其移动性或毒性。例如,土壤中大部分Cr3+以沉淀的形式存在,相对而言Cr6+稳定,毒害程度大。在好氧条件下水溶性的有毒Cr6+可以被Coryne bacterium以及其他微生物还原为水溶性较差的Cr3+,且土壤胶体对Cr3+具有强烈的吸附作用,在土壤中难以再迁移[95]。微生物在稻田土壤中介导砷的解毒作用,决定了砷在土壤中的行为和对水稻的可利用性。在淹水的水稻土中,土壤As(Ⅴ)还原成As(Ⅲ),增加其移动性和水稻砷的有效性。在水稻土中分离出一株属于食酸菌属的细菌ST3,该细菌能在厌氧条件下将硝酸根作为电子受体使As(Ⅲ)被无氧氧化成As(Ⅴ),降低了稻田土壤可溶性总As的浓度[96]。此外,微生物还可以通过改变土壤物理、化学、生物学性质降低土壤重金属的有效性,例如产聚-γ谷氨酸(Poly γ-glutamic acid,γ-PGA)细菌菌株提高镉污染土壤滤液中γ-PGA的产生量、pH、鞘氨醇单胞菌(Sphingomonas)和芽孢杆菌(Bacillus)等的相对丰度(24%~30%),从而固定化镉、降低土壤镉的有效性[97]。

|
图 4 微生物介导土壤重金属钝化的主要机制 Fig. 4 The main mechanism of microbial mediation in soil heavy metal passivation |
农药对作物增产和农业管理贡献巨大,但其带来的环境污染以及对非靶标生物的潜在风险不容忽视。研究表明灭菌处理土壤中吡虫啉、啶虫脒和噻虫嗪等农药残留降解速率明显低于非灭菌土壤,微生物在新烟碱类杀虫剂的降解过程中起到了决定性的作用[98]。作为农药降解与消除的主要驱动力,微生物主要通过酶促反应、非酶促反应等功能降解土壤中残留的农药。酶促反应是指微生物以靶标农药为底物,通过分泌的胞外酶或者产生胞内酶直接作用于农药,将农药完全降解或分解成分子量较小的无毒或毒性较小的化合物的过程。而非酶促反应指的是微生物通过代谢改变农药的环境离子浓度、pH等物理和化学性质,从而间接促使农药降解的过程。酶促反应是微生物降解农药的主要形式。例如,枯草芽孢杆菌YB1菌株主要通过分泌锰结合ABC转运酶、植物过氧化氢酶和乙偶姻脱氢酶E1等3种酶实现对烟嘧磺隆的降解[99]。此外,微生物分泌的农药降解相关的五类加氧酶类(黄素依赖型单加氧酶、细胞色素P450单加氧酶、Rieske非血红色铁加氧酶、Fe(Ⅱ)/a-酮戊二酸依赖性双加氧酶以及环裂解双加氧酶)利用分子氧(O2)作为末端电子受体[100],实现对农药高效率、低成本、彻底降解。利用微生物参与的不同酶促反应,构建多种微生物联盟通过协同代谢、分工合作较单一的微生物菌群更有利于有毒化合物的完全降解,提高农药降解效率。例如,微生物群通过微生物间的协同代谢,可以有效提高对双对氯苯基三氯乙烷(Dichloro-Diphenyl-Trichloroethane,DDT)的生物降解[101]。通过富集的方法筛选出以Sinomonas、Pandoraea和Burkholderia-Caballeronia-Paraburkholderia为优势菌属的混合菌群能够高度耐受乙酰甲胺磷,并且能够利用乙酰甲胺磷作为唯一碳、氮、磷源生长,进而高效降解乙酰甲胺磷及其中间产物甲胺磷[102]。因此,微生物联合将是促进农药生物降解的有利途径,特别是结构顽固或毒性强的农药。
5.4 微生物参与调控抗生素抗性基因的传播抗生素的过度使用导致土壤抗生素污染严重威胁土壤和人类健康。牛粪、鸡粪、猪粪和污水污泥是农田土壤中抗生素的主要污染源,磺胺类、四环素类、氟喹诺酮类是农田土壤中主要的抗生素种类[103]。积累在土壤中的抗生素会选择性地诱杀靶标微生物类群,而非靶标微生物会间接获得大量资源,导致土著微生物群落组成失衡,破坏土壤生态功能。同时,土壤中大量存在的抗生素作为一种环境选择压力会引发非靶标微生物群体抗生素耐药基因的进化,进而导致抗生素抗性的产生。作为一类新型的生物污染物,抗生素抗性基因(Antibiotics resistance genes,ARGs)会在不同环境介质中持久性残留、转移和扩散,而ARGs繁殖受水平基因转移(Horizontal gene transfer,HGT)和微生物群落组成的支配。调控土壤微生物及动物的多样性是管理抗性基因流的一项重要途径[104],土壤微生物多样性的丧失会加剧ARGs的传播[105]。噬菌体接种可裂解宿主释放ARGs进一步降解降低土壤中的抗生素抗性细菌(Antibiotics resistance bacteria,ARB)和ARGs的丰度,表明利用噬菌体免疫疗法可以控制ARGs的累积和传播[106]。然而也有研究表明,噬菌体DNA也是土壤中的ARGs的重要宿主,其作为ARGs的增殖载体也不容忽视。因此,噬菌体免疫疗法与土壤抗生素抗性基因的安全控制还需要进一步研究。农业衍生抗生素现在被广泛认为是全球ARGs增加的关键驱动因素。从源头上降低抗生素的积累是维持土壤健康的关键,也是目前控制抗生素以及抗性基因污染较为有效的手段,例如,减少饲料中抗生素的使用降低畜禽粪便中抗生素的含量,或者将粪肥炭化或酸化处理促进抗生素的降解,能够有效降低土壤抗性基因丰度,并抑制抗性基因的水平转移[107-108]。此外,筛选微生物高效降解菌株和基因工程菌的研究是降解土壤中抗生素的主要研究方向,从土壤中筛选出的Achromobacter xylosoxidans及Bacillus subtilis可以有效降解土霉素、四环素和金霉素[109]。P. chrysosporium和P. sanguineus混合培养情况下可以通过联合氧化酶系统的产生完全降解环丙沙星、诺氟沙星、磺胺甲恶唑[110]。然而,目前由于土著微生物复杂性与竞争性影响接种微生物的定殖,关于抗生素降解的研究仅集中在培养体系中,且少有研究将降解菌应用于土壤中。因此,从“截源控流”方面控制抗性基因在土壤环境中的传播仍需要更多的研究与实践。
6 利用土壤微生物组预测土壤健康土壤健康综合评价体系均需要同时监测大量土壤指标,分析项目成本高昂,在实际生产中不易推行。土壤微生物是土壤环境的敏感表征,是土壤特性的丰富信息来源。土壤微生物群落直接或间接对土壤环境变化做出反应,例如土壤pH、盐度、土传病害、土壤污染等。鉴于其对环境变化的敏感性与指示性,且土壤微生物组可以以一种高通量、经济有效的方式进行检测,尝试利用微生物组预测土壤健康水平,已经成为目前研究的热点。机器学习是基于微生物群落数据的预测指标开发运用最为广泛的方法,最初主要用于人类微生物组,以确定是否可以使用特定的微生物类群概况来诊断疾病状态[111]。目前微生物组信息作为环境健康综合状况的预测指标也逐步获得认可,例如,细菌群落能够指示流域扰动的程度以及地下水铀、硝态氮等污染状况[112],在预测环境污染和生态系统功能中具有很大的潜力。在土壤生态系统中,特定微生物类群与土壤变量之间表现出很强的相关性,利用微生物群落数据可以用来预测物理化学状况的变化[113],如土壤可溶性有机碳[114]、土壤养分含量、容重和pH[115]。在对于土壤健康综合评估与预测的研究中,微生物群落对土壤质量综合指数以及土壤健康综合指数预测的准确率分别达到50%~95%和80%[115-116],对枯萎病发病和健康土壤判断准确率高于80%[117],证明了利用土壤微生物群落预测土壤健康的可行性,但是目前对于土壤健康的评估尚未有统一标准,因此对于不同生态系统或土地利用方式间预测准确率存在较大变异。土壤发病率与具有预测价值的类群有明显的生态关联,例如属于Sphingomonadaceae、Hyphomicrobiaceae、Koribacteraceae和Xanthomonadaceae的细菌类群,属于Fusarium和Mycothermus的真菌类群对于枯萎病发病土壤具有重要的指示作用[117],此类生物标志物的识别对于土壤健康的早期预警具有重要指示作用,而健康土壤生物标志物的识别对于恢复和维持土壤健康具有重要作用,例如属于KD4-96、Nitrososphaeraceae,Pyrinomonadaceae的细菌类群的生态分布与评估的土壤健康关系相匹配[116]。生物标志物对利用合理的农业管理措施或微生物组工程技术定向调控目标土壤微生物群落、提高土壤健康水平,具有重要指示作用[118]。在模型训练与预测中,多数以扩增序列变体(Amplicon sequence variants,ASV)或者操作分类单元(Operational taxonomic units,OTU)等分类水平上最准确,然而大多数对模型准确性至关重要的ASV或OTU属于未分类的或者不可培养的群体,限制了广泛推断模型潜在生态基础的能力。因此,研究健康土壤生物标志物的组成与潜在功能、及其调控土壤健康的机制,对于土壤微生物菌剂的研发及应用,健康土壤的定向培育以及可持续发展具有重要意义。
7 展望土壤健康是土壤多种功能过程的集合体,土壤微生物是养分有效化的“转化器”、环境污染物的“净化器”以及生态平衡的“稳定器”,是土壤功能的主要驱动者。充分利用微生物优化土壤内部功能,对维持土壤健康以及土壤生态系统的可持续发展具有重要意义,但目前土壤微生物介导的土壤功能与土壤健康的研究仍处于起步阶段,未来还应加强以下方面的研究:1)强化土壤微生物多功能研究。土壤健康是由多种微生物介导的多个功能驱动的一个动态平衡过程,一旦动态平衡被打破,会降低土壤的抵抗力,打破土壤免疫防线,威胁土壤健康。未来需加强微生物互作、功能与土壤健康的耦合研究,明确土壤微生物及其功能在调控土壤健康中发挥的作用及其贡献,最大限度提高养分利用率,增强土壤免疫功能,降低污染物的可利用性,最大化土壤内部调节能力,强化其在土壤生态系统中的作用,提升土壤健康。2)加强健康土壤微生物定向培育与调控技术研究针对性的提升土壤健康水平。对于健康土壤微生物定向调控,需解析不同生境及环境下健康土壤微生物组成与功能特征,利用多组学技术、机器学习模型深入揭示微生物组及其功能与土壤健康之间的关系,建立标志土壤健康的关键生物学指标,构建健康土壤微生物菌群库。通过合成生物学或者基因工程技术,定向构建改善土壤健康的微生物种群,依据不同土壤健康问题,针对性地靶向调控,将诸如土壤免疫、污染物降解、土壤固碳等特定功能强化的微生物群作为“一剂良药”定向移植并使之成功定殖在土壤中,实现土壤微生物组的精准调控,优化土壤功能,是健康土壤培育的一个重要研究方向。目前,微生物组移植已经成功用于改变植物微生物菌群组成,在提升植物养分利用及抗病能力方面表现出了巨大潜力,还需继续加强健康土壤核心微生物组的挖掘及其在生产中的应用,以更大范围内实现生态功能、污染修复功能以及免疫功能土壤微生物组的精准调控,提升土壤健康,维持农业可持续生产。
| [1] |
Zhang J L, Zhang J Z, Shen J B, et al. Soil health and agriculture green development: Opportunities and challenges (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(4): 783-796. [张俊伶, 张江周, 申建波, 等. 土壤健康与农业绿色发展: 机遇与对策[J]. 土壤学报, 2020, 57(4): 783-796.]
(  0) 0) |
| [2] |
Zhang J Z, Li Y Z, Li Y, et al. Advances in the indicator system and evaluation approaches of soil health (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(3): 603-616. [张江周, 李奕赞, 李颖, 等. 土壤健康指标体系与评价方法研究进展[J]. 土壤学报, 2022, 59(3): 603-616.]
(  0) 0) |
| [3] |
Li X Z, Luo Y M, Hou D Y. The Indicators, framework and procedures for soil health: a critical review (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(3): 617-625. [李烜桢, 骆永明, 侯德义. 土壤健康评估指标、框架及程序研究进展[J]. 土壤学报, 2022, 59(3): 617-625.]
(  0) 0) |
| [4] |
Li Y Z, Zhang J Z, Jia J Y, et al. Research progresses on farmland soil ecosystem multifunctionality (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(5): 1177-1189. [李奕赞, 张江周, 贾吉玉, 等. 土壤生态系统多功能性研究进展[J]. 土壤学报, 2022, 59(5): 1177-1189.]
(  0) 0) |
| [5] |
Cai Z C. Discussion on the strategies for development of the subdiscipline of soil fertility and soil nutrient cycling for the 14th five-year plan (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(5): 1128-1136. [蔡祖聪. 浅谈"十四五"土壤肥力与土壤养分循环分支学科发展战略[J]. 土壤学报, 2020, 57(5): 1128-1136.]
(  0) 0) |
| [6] |
Chen Q L, Cui H L, Su J Q, et al. Antibiotic resistomes in plant microbiomes[J]. Trends in Plant Science, 2019, 24(6): 530-541. DOI:10.1016/j.tplants.2019.02.010
(  0) 0) |
| [7] |
Zhu Y G, Peng J J, Wei Z, et al. Linking the soil microbiome to soil health (In Chinese)[J]. Scientia Sinica: Vitae, 2021, 51(1): 1-11. [朱永官, 彭静静, 韦中, 等. 土壤微生物组与土壤健康[J]. 中国科学: 生命科学, 2021, 51(1): 1-11.]
(  0) 0) |
| [8] |
Jian S Y, Li J W, Chen J, et al. Soil extracellular enzyme activities, soil carbon and nitrogen storage under nitrogen fertilization: A meta-analysis[J]. Soil Biology & Biochemistry, 2016, 101: 32-43.
(  0) 0) |
| [9] |
Wang C, Morrissey E M, Mau R L, et al. The temperature sensitivity of soil: Microbial biodiversity, growth, and carbon mineralization[J]. The ISME Journal, 2021, 15(9): 2738-2747. DOI:10.1038/s41396-021-00959-1
(  0) 0) |
| [10] |
Liang C, Zhu X F. The soil microbial carbon pump as a new concept for terrestrial carbon sequestration (In Chinese)[J]. Scientia Sinica: Terrae, 2021, 51(5): 680-695. [梁超, 朱雪峰. 土壤微生物碳泵储碳机制概论[J]. 中国科学: 地球科学, 2021, 51(5): 680-695.]
(  0) 0) |
| [11] |
Liang C, Amelung W, Lehmann J, et al. Quantitative assessment of microbial necromass contribution to soil organic matter[J]. Global Change Biology, 2019, 25(11): 3578-3590. DOI:10.1111/gcb.14781
(  0) 0) |
| [12] |
Wang B R, An S S, Liang C, et al. Microbial necromass as the source of soil organic carbon in global ecosystems[J]. Soil Biology & Biochemistry, 2021, 162: 108422.
(  0) 0) |
| [13] |
Zhao X C, Tian P, Liu S G, et al. Mean annual temperature and carbon availability respectively controlled the contributions of bacterial and fungal residues to organic carbon accumulation in topsoil across China's forests[J]. Global Ecology and Biogeography, 2023, 32(1): 120-131. DOI:10.1111/geb.13605
(  0) 0) |
| [14] |
Wang C, Qu L R, Yang L M, et al. Large-scale importance of microbial carbon use efficiency and necromass to soil organic carbon[J]. Global Change Biology, 2021, 27(10): 2039-2048. DOI:10.1111/gcb.15550
(  0) 0) |
| [15] |
Wu J J, Cheng X L, Liu G H. Increased soil organic carbon response to fertilization is associated with increasing microbial carbon use efficiency: Data synthesis[J]. Soil Biology & Biochemistry, 2022, 171: 108731.
(  0) 0) |
| [16] |
Kallenbach C M, Frey S D, Grandy A S. Direct evidence for microbial-derived soil organic matter formation and its ecophysiological controls[J]. Nature Communications, 2016, 7(1): 13630. DOI:10.1038/ncomms13630
(  0) 0) |
| [17] |
Wu W C, Dijkstra P, Hungate B A, et al. In situ diversity of metabolism and carbon use efficiency among soil bacteria[J]. Science Advances, 2022, 8(44): eabq3958. DOI:10.1126/sciadv.abq3958
(  0) 0) |
| [18] |
Feng X H, Qin S Q, Zhang D Y, et al. Nitrogen input enhances microbial carbon use efficiency by altering plant–microbe–mineral interactions[J]. Global Change Biology, 2022, 28(16): 4845-4860. DOI:10.1111/gcb.16229
(  0) 0) |
| [19] |
Xiao K Q, Ge T D, Wu X H, et al. Metagenomic and 14C tracing evidence for autotrophic microbial CO2 fixation in paddy soils[J]. Environmental Microbiology, 2021, 23(2): 924-933. DOI:10.1111/1462-2920.15204
(  0) 0) |
| [20] |
Chen H, Wang F, Kong W D, et al. Soil microbial CO2 fixation plays a significant role in terrestrial carbon sink in a dryland ecosystem: A four-year small-scale field-plot observation on the Tibetan Plateau[J]. Science of the Total Environment, 2021, 761: 143282. DOI:10.1016/j.scitotenv.2020.143282
(  0) 0) |
| [21] |
Thauer R K. Microbiology. A fifth pathway of carbon fixation[J]. Science, 2007, 318(5857): 1732-1733. DOI:10.1126/science.1152209
(  0) 0) |
| [22] |
Yuan H Z, Ge T D, Chen C Y, et al. Significant role for microbial autotrophy in the sequestration of soil carbon[J]. Applied and Environmental Microbiology, 2012, 78(7): 2328-2336. DOI:10.1128/AEM.06881-11
(  0) 0) |
| [23] |
Huang Q, Huang Y M, Wang B R, et al. Metabolic pathways of CO2 fixing microorganisms determined C-fixation rates in grassland soils along the precipitation gradient[J]. Soil Biology & Biochemistry, 2022, 172: 108764.
(  0) 0) |
| [24] |
Liu H M, Hai X, An K R, et al. Effects of different fertilization regimes on community structure diversity of CO2-assimilating bacteria in maize field of fluvo-aquic soil in North China (In Chinese)[J]. Ecology and Environmental Sciences, 2022, 31(4): 715-722. [刘红梅, 海香, 安克锐, 等. 不同施肥措施对华北潮土区玉米田土壤固碳细菌群落结构多样性的影响[J]. 生态环境学报, 2022, 31(4): 715-722.]
(  0) 0) |
| [25] |
Li Z W, Tong D, Nie X D, et al. New insight into soil carbon fixation rate: The intensive co-occurrence network of autotrophic bacteria increases the carbon fixation rate in depositional sites[J]. Agriculture, Ecosystems & Environment, 2021, 320: 107579.
(  0) 0) |
| [26] |
Zhao K, Kong W D, Wang F, et al. Desert and steppe soils exhibit lower autotrophic microbial abundance but higher atmospheric CO2 fixation capacity than meadow soils[J]. Soil Biology & Biochemistry, 2018, 127: 230-238.
(  0) 0) |
| [27] |
Herridge D F, Peoples M B, Boddey R M. Global inputs of biological nitrogen fixation in agricultural systems[J]. Plant and Soil, 2008, 311(1): 1-18.
(  0) 0) |
| [28] |
Klimasmith I M, Kent A D. Micromanaging the nitrogen cycle in agroecosystems[J]. Trends in Microbiology, 2022, 30(11): 1045-1055. DOI:10.1016/j.tim.2022.04.006
(  0) 0) |
| [29] |
Xie Z B, Zhang Y H, Wang H. Advances and perspectives in paddy biological nitrogen fixation (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(3): 540-546. [谢祖彬, 张燕辉, 王慧. 稻田生物固氮研究进展及方向[J]. 土壤学报, 2020, 57(3): 540-546.]
(  0) 0) |
| [30] |
Lin Y X, Ye G P, Liu D Y, et al. Long-term application of lime or pig manure rather than plant residues suppressed diazotroph abundance and diversity and altered community structure in an acidic Ultisol[J]. Soil Biology & Biochemistry, 2018, 123: 218-228.
(  0) 0) |
| [31] |
Bloch S E, Clark R, Gottlieb S S, et al. Biological nitrogen fixation in maize: Optimizing nitrogenase expression in a root-associated diazotroph[J]. Journal of Experimental Botany, 2020, 71(15): 4591-4603. DOI:10.1093/jxb/eraa176
(  0) 0) |
| [32] |
Charyulu P B B N, Rao V R. Influence of carbon substrates and moisture regime on nitrogen fixation in paddy soils[J]. Soil Biology & Biochemistry, 1981, 13(1): 39-42.
(  0) 0) |
| [33] |
Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9
(  0) 0) |
| [34] |
Hink L, Lycus P, Gubry-Rangin C, et al. Kinetics of NH3-oxidation, NO-turnover, N2O-production and electron flow during oxygen depletion in model bacterial and archaeal ammonia oxidisers[J]. Environmental Microbiology, 2017, 19(12): 4882-4896. DOI:10.1111/1462-2920.13914
(  0) 0) |
| [35] |
Bakken L R, Frostegård Å. Emerging options for mitigating N2O emissions from food production by manipulating the soil microbiota[J]. Current Opinion in Environmental Sustainability, 2020, 47: 89-94. DOI:10.1016/j.cosust.2020.08.010
(  0) 0) |
| [36] |
You L C, Ros G H, Chen Y L, et al. Global meta-analysis of terrestrial nitrous oxide emissions and associated functional genes under nitrogen addition[J]. Soil Biology & Biochemistry, 2022, 165: 108523.
(  0) 0) |
| [37] |
Wang W Y, Hou Y T, Pan W H, et al. Continuous application of conservation tillage affects in situ N2O emissions and nitrogen cycling gene abundances following nitrogen fertilization[J]. Soil Biology & Biochemistry, 2021, 157: 108239.
(  0) 0) |
| [38] |
Coskun D, Britto D T, Shi W M, et al. Nitrogen transformations in modern agriculture and the role of biological nitrification inhibition[J]. Nature Plants, 2017, 3(6): 17074. DOI:10.1038/nplants.2017.74
(  0) 0) |
| [39] |
Yao Q M, Li Z, Song Y, et al. Community proteogenomics reveals the systemic impact of phosphorus availability on microbial functions in tropical soil[J]. Nature Ecology & Evolution, 2018, 2(3): 499-509.
(  0) 0) |
| [40] |
Sharma S B, Sayyed R Z, Trivedi M H, et al. Phosphate solubilizing microbes: Sustainable approach for managing phosphorus deficiency in agricultural soils[J]. SpringerPlus, 2013, 2(1): 587. DOI:10.1186/2193-1801-2-587
(  0) 0) |
| [41] |
Rodríguez H, Fraga R, Gonzalez T, et al. Genetics of phosphate solubilization and its potential applications for improving plant growth-promoting bacteria[J]. Plant and Soil, 2006, 287(1): 15-21.
(  0) 0) |
| [42] |
Song H, Dharmasena M N, Wang C, et al. Structure and activity of PPX/GppA homologs from Escherichia coli and Helicobacter pylori[J]. The FEBS Journal, 2020, 287(9): 1865-1885. DOI:10.1111/febs.15120
(  0) 0) |
| [43] |
Wei X M, Hu Y J, Razavi B S, et al. Rare taxa of alkaline phosphomonoesterase-harboring microorganisms mediate soil phosphorus mineralization[J]. Soil Biology & Biochemistry, 2019, 131: 62-70.
(  0) 0) |
| [44] |
Dai Z M, Liu G F, Chen H H, et al. Long-term nutrient inputs shift soil microbial functional profiles of phosphorus cycling in diverse agroecosystems[J]. The ISME Journal, 2020, 14(3): 757-770. DOI:10.1038/s41396-019-0567-9
(  0) 0) |
| [45] |
Huang Y L, Dai Z M, Lin J H, et al. Labile carbon facilitated phosphorus solubilization as regulated by bacterial and fungal communities in Zea mays[J]. Soil Biology & Biochemistry, 2021, 163: 108465.
(  0) 0) |
| [46] |
Oliverio A M, Bissett A, McGuire K, et al. The role of phosphorus limitation in shaping soil bacterial communities and their metabolic capabilities[J]. mBio, 2020, 11(5): e01718-20.
(  0) 0) |
| [47] |
Bergkemper F, Schöler A, Engel M, et al. Phosphorus depletion in forest soils shapes bacterial communities towards phosphorus recycling systems[J]. Environmental Microbiology, 2016, 18(6): 1988-2000. DOI:10.1111/1462-2920.13188
(  0) 0) |
| [48] |
Lu J L, Jia P, Feng S W, et al. Remarkable effects of microbial factors on soil phosphorus bioavailability: A country-scale study[J]. Global Change Biology, 2022, 28(14): 4459-4471. DOI:10.1111/gcb.16213
(  0) 0) |
| [49] |
Park Y, Solhtalab M, Thongsomboon W, et al. Strategies of organic phosphorus recycling by soil bacteria: Acquisition, metabolism, and regulation[J]. Environmental Microbiology Reports, 2022, 14: 3-24. DOI:10.1111/1758-2229.13040
(  0) 0) |
| [50] |
Wilpiszeski R L, Aufrecht J A, Retterer S T, et al. Soil aggregate microbial communities: Towards understanding microbiome interactions at biologically relevant scales[J]. Applied and Environmental Microbiology, 2019, 85(14): e00324-19.
(  0) 0) |
| [51] |
Lehmann A, Zheng W S, Rillig M C. Soil biota contributions to soil aggregation[J]. Nature Ecology & Evolution, 2017, 1(12): 1828-1835.
(  0) 0) |
| [52] |
Zhu X F, Jackson R D, DeLucia E H, et al. The soil microbial carbon pump: From conceptual insights to empirical assessments[J]. Global Change Biology, 2020, 26(11): 6032-6039. DOI:10.1111/gcb.15319
(  0) 0) |
| [53] |
Merino-Martín L, Stokes A, Gweon H S, et al. Interacting effects of land use type, soil microbes and plant traits on aggregate stability[J]. Soil Biology & Biochemistry, 2021, 154: 108072.
(  0) 0) |
| [54] |
Coban O, De Deyn G B, van der Ploeg M. Soil microbiota as game-changers in restoration of degraded lands[J]. Science, 2022, 375(6584): abe0725. DOI:10.1126/science.abe0725
(  0) 0) |
| [55] |
Rashid M I, Mujawar L H, Shahzad T, et al. Bacteria and fungi can contribute to nutrients bioavailability and aggregate formation in degraded soils[J]. Microbiological Research, 2016, 183: 26-41. DOI:10.1016/j.micres.2015.11.007
(  0) 0) |
| [56] |
Deng J Z, Orner E P, Chau J F, et al. Synergistic effects of soil microstructure and bacterial EPS on drying rate in emulated soil micromodels[J]. Soil Biology & Biochemistry, 2015, 83: 116-124.
(  0) 0) |
| [57] |
Olagoke F K, Bettermann A, Nguyen P T B, et al. Importance of substrate quality and clay content on microbial extracellular polymeric substances production and aggregate stability in soils[J]. Biology and Fertility of Soils, 2022, 58(4): 435-457. DOI:10.1007/s00374-022-01632-1
(  0) 0) |
| [58] |
Liu Y L, Wang P, Wang J K. Formation and stability mechanism of soil aggregates: Progress and prospect (In Chinese)[J]. Acta Pedologica Sinica, 2023, 60(3): 627-643. [刘亚龙, 王萍, 汪景宽. 土壤团聚体的形成和稳定机制: 研究进展与展望[J]. 土壤学报, 2023, 60(3): 627-643.]
(  0) 0) |
| [59] |
Rillig M C. Arbuscular mycorrhizae, glomalin, and soil aggregation[J]. Canadian Journal of Soil Science, 2004, 84(4): 355-363. DOI:10.4141/S04-003
(  0) 0) |
| [60] |
Wei Z, Shen Z Z, Yang T J, et al. From suppressive soil to rhizosphere immunity: Towards an ecosystem thinking for soil-borne pathogen control (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(4): 814-824. [韦中, 沈宗专, 杨天杰, 等. 从抑病土壤到根际免疫: 概念提出与发展思考[J]. 土壤学报, 2021, 58(4): 814-824.]
(  0) 0) |
| [61] |
Irikiin Y, Nishiyama M, Otsuka S, et al. Rhizobacterial community-level, sole carbon source utilization pattern affects the delay in the bacterial wilt of tomato grown in rhizobacterial community model system[J]. Applied Soil Ecology, 2006, 34(1): 27-32. DOI:10.1016/j.apsoil.2005.12.003
(  0) 0) |
| [62] |
Mendes L W, Mendes R, Raaijmakers J M, et al. Breeding for soil-borne pathogen resistance impacts active rhizosphere microbiome of common bean[J]. The ISME Journal, 2018, 12(12): 3038-3042. DOI:10.1038/s41396-018-0234-6
(  0) 0) |
| [63] |
Hu J, Wei Z, Friman V P, et al. Probiotic diversity enhances rhizosphere microbiome function and plant disease suppression[J]. mBio, 2016, 7(6): e01790-16.
(  0) 0) |
| [64] |
Wei Z, Yang T J, Friman V P, et al. Trophic network architecture of root-associated bacterial communities determines pathogen invasion and plant health[J]. Nature Communications, 2015, 6(1): 8413. DOI:10.1038/ncomms9413
(  0) 0) |
| [65] |
Zhang N, Nunan N, Hirsch P R, et al. Theory of microbial coexistence in promoting soil–plant ecosystem health[J]. Biology and Fertility of Soils, 2021, 57(7): 897-911. DOI:10.1007/s00374-021-01586-w
(  0) 0) |
| [66] |
Tao C Y, Li R, Xiong W, et al. Bio-organic fertilizers stimulate indigenous soil Pseudomonas populations to enhance plant disease suppression[J]. Microbiome, 2020, 8(1): 137. DOI:10.1186/s40168-020-00892-z
(  0) 0) |
| [67] |
Badri D V, Zolla G, Bakker M G, et al. Potential impact of soil microbiomes on the leaf metabolome and on herbivore feeding behavior[J]. New Phytologist, 2013, 198(1): 264-273. DOI:10.1111/nph.12124
(  0) 0) |
| [68] |
Jaber L R, Enkerli J. Effect of seed treatment duration on growth and colonization of Vicia faba by endophytic Beauveria bassiana and Metarhizium brunneum[J]. Biological Control, 2016, 103: 187-195. DOI:10.1016/j.biocontrol.2016.09.008
(  0) 0) |
| [69] |
Muvea A M, Meyhöfer R, Subramanian S, et al. Colonization of onions by endophytic fungi and their impacts on the biology of Thrips tabaci[J]. PLoS One, 2014, 9(9): e108242. DOI:10.1371/journal.pone.0108242
(  0) 0) |
| [70] |
Kos M, Tuijl M A B, de Roo J, et al. Species-specific plant–soil feedback effects on above-ground plant–insect interactions[J]. Journal of Ecology, 2015, 103(4): 904-914. DOI:10.1111/1365-2745.12402
(  0) 0) |
| [71] |
Blundell R, Schmidt J E, Igwe A, et al. Organic management promotes natural pest control through altered plant resistance to insects[J]. Nature Plants, 2020, 6(5): 483-491. DOI:10.1038/s41477-020-0656-9
(  0) 0) |
| [72] |
Sun Y Z, Duan C X, Cao N, et al. Biodegradable and conventional microplastics exhibit distinct microbiome, functionality, and metabolome changes in soil[J]. Journal of Hazardous Materials, 2022, 424: 127282. DOI:10.1016/j.jhazmat.2021.127282
(  0) 0) |
| [73] |
Huang Y, Zhao Y R, Wang J, et al. LDPE microplastic films alter microbial community composition and enzymatic activities in soil[J]. Environmental Pollution, 2019, 254: 112983. DOI:10.1016/j.envpol.2019.112983
(  0) 0) |
| [74] |
Sander M. Biodegradation of polymeric mulch films in agricultural soils: Concepts, knowledge gaps, and future research directions[J]. Environmental Science & Technology, 2019, 53(5): 2304-2315.
(  0) 0) |
| [75] |
Yang W W, Cheng P, Adams C A, et al. Effects of microplastics on plant growth and arbuscular mycorrhizal fungal communities in a soil spiked with ZnO nanoparticles[J]. Soil Biology & Biochemistry, 2021, 155: 108179.
(  0) 0) |
| [76] |
Rillig M C, Lehmann A. Microplastic in terrestrial ecosystems[J]. Science, 2020, 368(6498): 1430-1431. DOI:10.1126/science.abb5979
(  0) 0) |
| [77] |
Wan Y, Wu C X, Xue Q, et al. Effects of plastic contamination on water evaporation and desiccation cracking in soil[J]. Science of the Total Environment, 2019, 654: 576-582. DOI:10.1016/j.scitotenv.2018.11.123
(  0) 0) |
| [78] |
Rubol S, Manzoni S, Bellin A, et al. Modeling soil moisture and oxygen effects on soil biogeochemical cycles including dissimilatory nitrate reduction to ammonium (DNRA)[J]. Advances in Water Resources, 2013, 62: 106-124. DOI:10.1016/j.advwatres.2013.09.016
(  0) 0) |
| [79] |
Zhu D, Ma J, Li G, et al. Soil plastispheres as hotspots of antibiotic resistance genes and potential pathogens[J]. The ISME Journal, 2022, 16(2): 521-532. DOI:10.1038/s41396-021-01103-9
(  0) 0) |
| [80] |
Muhonja C N, Makonde H, Magoma G, et al. Biodegradability of polyethylene by bacteria and fungi from Dandora dumpsite Nairobi-Kenya[J]. PLoS One, 2018, 13(7): e0198446. DOI:10.1371/journal.pone.0198446
(  0) 0) |
| [81] |
Park S Y, Kim C G. Biodegradation of micro-polyethylene particles by bacterial colonization of a mixed microbial consortium isolated from a landfill site[J]. Chemosphere, 2019, 222: 527-533. DOI:10.1016/j.chemosphere.2019.01.159
(  0) 0) |
| [82] |
Zhang X Y, Li Y, Ouyang D, et al. Systematical review of interactions between microplastics and microorganisms in the soil environment[J]. Journal of Hazardous Materials, 2021, 418: 126288. DOI:10.1016/j.jhazmat.2021.126288
(  0) 0) |
| [83] |
Teuten E L, Saquing J M, Knappe D R U, et al. Transport and release of chemicals from plastics to the environment and to wildlife[J]. Philosophical Transactions: Biological Sciences, 2009, 364(1526): 2027-2045. DOI:10.1098/rstb.2008.0284
(  0) 0) |
| [84] |
Wei R, Zimmermann W. Microbial enzymes for the recycling of recalcitrant petroleum-based plastics: How far are we?[J]. Microbial Biotechnology, 2017, 10(6): 1308-1322. DOI:10.1111/1751-7915.12710
(  0) 0) |
| [85] |
You X X, Wang S, Li G, et al. Microplastics in the soil: A review of distribution, anthropogenic impact, and interaction with soil microorganisms based on meta-analysis[J]. Science of the Total Environment, 2022, 832: 154975. DOI:10.1016/j.scitotenv.2022.154975
(  0) 0) |
| [86] |
Cosgrove L, McGeechan P L, Handley P S, et al. Effect of biostimulation and bioaugmentation on degradation of polyurethane buried in soil[J]. Applied and Environmental Microbiology, 2010, 76(3): 810-819. DOI:10.1128/AEM.00534-09
(  0) 0) |
| [87] |
Sánchez C. Fungal potential for the degradation of petroleum-based polymers: An overview of macro- and microplastics biodegradation[J]. Biotechnology Advances, 2020, 40: 107501. DOI:10.1016/j.biotechadv.2019.107501
(  0) 0) |
| [88] |
Tribedi P, Sil A K. Cell surface hydrophobicity: A key component in the degradation of polyethylene succinate by Pseudomonas sp. AKS2[J]. Journal of Applied Microbiology, 2014, 116(2): 295-303. DOI:10.1111/jam.12375
(  0) 0) |
| [89] |
Mosa K A, Saadoun I, Kumar K, et al. Potential biotechnological strategies for the cleanup of heavy metals and metalloids[J]. Frontiers in Plant Science, 2016, 7: 303.
(  0) 0) |
| [90] |
Ayangbenro A S, Babalola O O. A new strategy for heavy metal polluted environments: A review of microbial biosorbents[J]. International Journal of Environmental Research and Public Health, 2017, 14(1): 94. DOI:10.3390/ijerph14010094
(  0) 0) |
| [91] |
Liu Y G, Feng B Y, Fan T, et al. Study on the biosorption of heavy metals by fungi (In Chinese)[J]. Journal of Hunan University: Natural Sciences, 2008, 35(1): 71-74. [刘云国, 冯宝莹, 樊霆, 等. 真菌吸附重金属离子的研究[J]. 湖南大学学报: 自然科学版, 2008, 35(1): 71-74.]
(  0) 0) |
| [92] |
Bai J, Yang X H, Du R Y, et al. Biosorption mechanisms involved in immobilization of soil Pb by Bacillus subtilis DBM in a multi-metal-contaminated soil[J]. Journal of Environmental Sciences, 2014, 26(10): 2056-2064. DOI:10.1016/j.jes.2014.07.015
(  0) 0) |
| [93] |
Wu S J, Zhou Z J, Zhu L, et al. Cd immobilization mechanisms in a Pseudomonas strain and its application in soil Cd remediation[J]. Journal of Hazardous Materials, 2022, 425: 127919. DOI:10.1016/j.jhazmat.2021.127919
(  0) 0) |
| [94] |
Song W J, Yang Y Y, Liang X J, et al. Influence of metals and metalloids on the composition and fluorescence quenching of the extracellular polymeric substances produced by the polymorphic fungus Aureobasidium pullulans[J]. Applied Microbiology and Biotechnology, 2020, 104(16): 7155-7164. DOI:10.1007/s00253-020-10732-7
(  0) 0) |
| [95] |
Goyal N, Jain S C, Banerjee U C. Comparative studies on the microbial adsorption of heavy metals[J]. Advances in Environmental Research, 2003, 7(2): 311-319. DOI:10.1016/S1093-0191(02)00004-7
(  0) 0) |
| [96] |
Zhang J, Chen J, Wu Y F, et al. Oxidation of organoarsenicals and antimonite by a novel flavin monooxygenase widely present in soil bacteria[J]. Environmental Microbiology, 2022, 24(2): 752-761. DOI:10.1111/1462-2920.15488
(  0) 0) |
| [97] |
Wang X H, Dong G Y, Liu X W, et al. Poly-γ-glutamic acid-producing bacteria reduced Cd uptake and effected the rhizosphere microbial communities of lettuce[J]. Journal of Hazardous Materials, 2020, 398: 123146. DOI:10.1016/j.jhazmat.2020.123146
(  0) 0) |
| [98] |
Song C, Zhou Y Q, Li Y Q, et al. Residue degradation and influencing factors of three neonicotinoids insecticides in soil (In Chinese)[J]. Chinese Journal of Pesticide Science, 2016, 18(6): 738-744. [宋超, 周杨全, 李义强, 等. 三种新烟碱类杀虫剂在土壤中的残留降解及影响因子[J]. 农药学学报, 2016, 18(6): 738-744.]
(  0) 0) |
| [99] |
Zhang Z. Study on the degradation of nicosulfuron by Bacillus subtilis YB1 and Aspergillus niger YF1[D]. Baoding, Hebei: Hebei Agricultural University, 2019.[张哲. 枯草芽孢杆菌YB1和黑曲霉YF1菌株对烟嘧磺隆的降解作用研究[D]. 河北保定: 河北农业大学, 2019.]
(  0) 0) |
| [100] |
Cheng M G, Chen D, Parales R E, et al. Oxygenases as powerful weapons in the microbial degradation of pesticides[J]. Annual Review of Microbiology, 2022, 76: 325-348. DOI:10.1146/annurev-micro-041320-091758
(  0) 0) |
| [101] |
Purnomo A S, Sariwati A, Kamei I. Synergistic interaction of a consortium of the brown-rot fungus Fomitopsis pinicola and the bacterium Ralstonia pickettii for DDT biodegradation[J]. Heliyon, 2020, 6(6): e04027. DOI:10.1016/j.heliyon.2020.e04027
(  0) 0) |
| [102] |
Lin Z Q, Pang S M, Zhou Z, et al. Novel pathway of acephate degradation by the microbial consortium ZQ01 and its potential for environmental bioremediation[J]. Journal of Hazardous Materials, 2022, 426: 127841. DOI:10.1016/j.jhazmat.2021.127841
(  0) 0) |
| [103] |
Wu J, Wang J Y, Li Z T, et al. Antibiotics and antibiotic resistance genes in agricultural soils: A systematic analysis[J]. Critical Reviews in Environmental Science and Technology, 2023, 53(7): 847-864. DOI:10.1080/10643389.2022.2094693
(  0) 0) |
| [104] |
Zhu D, Chen Q L, Ding J, et al. Antibiotic resistance genes in the soil ecosystem and planetary health: Progress and prospect (In Chinese)[J]. Scientia Sinica: Vitae, 2019, 49(12): 1652-1663. [朱冬, 陈青林, 丁晶, 等. 土壤生态系统中抗生素抗性基因与星球健康: 进展与展望[J]. 中国科学: 生命科学, 2019, 49(12): 1652-1663.]
(  0) 0) |
| [105] |
Chen Q L, An X L, Zheng B X, et al. Loss of soil microbial diversity exacerbates spread of antibiotic resistance[J]. Soil Ecology Letters, 2019, 1(1): 3-13.
(  0) 0) |
| [106] |
Ye M, Su J Q, An X L, et al. Silencing the silent pandemic: Eliminating antimicrobial resistance by using bacteriophages[J]. Science China Life Sciences, 2022, 65(9): 1890-1893. DOI:10.1007/s11427-022-2182-6
(  0) 0) |
| [107] |
Zhou X, Qiao M, Su J Q, et al. Turning pig manure into biochar can effectively mitigate antibiotic resistance genes as organic fertilizer[J]. Science of the Total Environment, 2019, 649: 902-908. DOI:10.1016/j.scitotenv.2018.08.368
(  0) 0) |
| [108] |
Lin H, Sun W C, Yu Q G, et al. Acidic conditions enhance the removal of sulfonamide antibiotics and antibiotic resistance determinants in swine manure[J]. Environmental Pollution, 2020, 263: 114439. DOI:10.1016/j.envpol.2020.114439
(  0) 0) |
| [109] |
He Y J, Zhou K P, Rao Y X, et al. Environmental risks of antibiotics in soil and the related bioremediation technologies (In Chinese)[J]. Chinese Journal of Biotechnology, 2021, 37(10): 3487-3504. [何玉洁, 周凯萍, 饶怡璇, 等. 土壤中抗生素的环境风险及污染土壤的生物修复技术[J]. 生物工程学报, 2021, 37(10): 3487-3504.]
(  0) 0) |
| [110] |
Gao N, Liu C X, Xu Q M, et al. Simultaneous removal of ciprofloxacin, norfloxacin, sulfamethoxazole by co-producing oxidative enzymes system of Phanerochaete chrysosporium and Pycnoporus sanguineus[J]. Chemosphere, 2018, 195: 146-155. DOI:10.1016/j.chemosphere.2017.12.062
(  0) 0) |
| [111] |
Poore G D, Kopylova E, Zhu Q Y, et al. Microbiome analyses of blood and tissues suggest cancer diagnostic approach[J]. Nature, 2020, 579(7800): 567-574. DOI:10.1038/s41586-020-2095-1
(  0) 0) |
| [112] |
Lau K E M, Washington V J, Fan V, et al. A novel bacterial community index to assess stream ecological health[J]. Freshwater Biology, 2015, 60(10): 1988-2002. DOI:10.1111/fwb.12625
(  0) 0) |
| [113] |
Hermans S M, Buckley H L, Case B S, et al. Bacteria as emerging indicators of soil condition[J]. Applied and Environmental Microbiology, 2016, 83(1): e02826-16.
(  0) 0) |
| [114] |
Thompson J, Johansen R, Dunbar J, et al. Machine learning to predict microbial community functions: An analysis of dissolved organic carbon from litter decomposition[J]. PLoS One, 2019, 14(7): e0215502. DOI:10.1371/journal.pone.0215502
(  0) 0) |
| [115] |
Hermans S M, Buckley H L, Case B S, et al. Using soil bacterial communities to predict physico-chemical variables and soil quality[J]. Microbiome, 2020, 8(1): 79. DOI:10.1186/s40168-020-00858-1
(  0) 0) |
| [116] |
Wilhelm R C, van Es H M, Buckley D H. Predicting measures of soil health using the microbiome and supervised machine learning[J]. Soil Biology & Biochemistry, 2022, 164: 108472.
(  0) 0) |
| [117] |
Yuan J, Wen T, Zhang H, et al. Predicting disease occurrence with high accuracy based on soil macroecological patterns of Fusarium wilt[J]. The ISME Journal, 2020, 14(12): 2936-2950. DOI:10.1038/s41396-020-0720-5
(  0) 0) |
| [118] |
Toju H, Peay K G, Yamamichi M, et al. Core microbiomes for sustainable agroecosystems[J]. Nature Plants, 2018, 4(5): 247-257. DOI:10.1038/s41477-018-0139-4
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


