抗生素的发现是人类医学史上的重大事件之一,在预防和控制人类和动物疾病过程中占据了举足轻重的地位。抗生素作为一把双刃剑,一方面可以治病救人;另一方面,其滥用又会导致细菌耐药性的产生和传播[1],严重威胁全球公共健康。据报道,牲畜和人类消耗抗生素的大约30%~90%可通过粪便和尿液释放到环境中[2],其产生的选择性压力使环境中抗生素抗性基因(Antibiotic resistance genes,ARGs)含量迅速增加。全球每年因抗生素耐药性病原体感染死亡的人口高达70多万人[3]。若不加以控制,预计到2050年,每年将有1 000万人直接死于微生物耐药性病原体[4],抗生素抗性已成为21世纪人类健康面临的重大挑战[5]。
土壤是ARGs的重要存储库,也是抗生素抗性产生、传播和演变的介质[6]。土壤环境中,抗生素耐药菌及ARGs广泛存在,且与人类致病菌关联紧密[7-8]。除了一些土壤微生物可能存在的固有抗性外[9],土壤中的ARGs主要来源于再生废水灌溉[10-11]、粪肥施用[12]及污水污泥施用[13]等。这些人为活动一方面使ARGs通过转座子、质粒和整合子等可移动遗传元件[14-15]发生水平转移,另一方面还可通过施加选择性压力,使土壤中耐抗生素细菌和ARGs大量增殖[16-17],进而加剧了土壤ARGs污染。研究表明,抗生素及其ARGs在根际土壤中的富集,可促进土壤菌群中的耐药性向植物菌群传播[18-19];同时,人类活动的影响也会加剧ARGs从非致病性宿主向病原体的水平基因转移[20]。ARGs的迁移和转化行为将对人体健康产生巨大威胁。因此,土壤ARGs污染问题仍需重点关注。
尽管世界各国均在持续关注各种环境介质中ARGs污染现状、来源归趋、迁移转化和消减策略等方面的研究,但目前仅有少量有关近十年内水环境中ARGs污染状况的计量分析文章,尚缺乏有关土壤ARGs相关研究的计量整合分析。因此,本研究基于Web of Science数据库,通过文献计量学方法,对2013—2022年土壤ARGs相关文献的文献发表数量年变化、引用次数年变化、文献发表国家/地区及机构等进行分析,帮助读者认识近十年各国学者对于土壤ARGs的关注程度;通过2013—2022年土壤ARGs相关文献的关键词突现和共现分析,使读者了解该领域的研究热点及研究趋势,以期为未来该领域的相关研究提供参考和思路。
1 数据来源与方法 1.1 数据来源基于文献计量学方法,以Web of Science数据库作为信息来源,于2022年10月6日收集数据,分析土壤ARGs研究进展及热点。文献检索主题词设定为“Antibiotic resistance genes”和“Soil”,文献类型不限,时间跨度为2013—2022年。检索后共得到5 360个结果。在所有结果中,首先根据论文的标题和摘要进行初步筛选;对于无法通过标题和摘要明确判断的文献,再通过检查已下载的文献全纪录内容进行二次筛选,确保其主要研究内容为土壤ARGs,最终获得627篇有效文献。将筛选得到的结果以纯文本文件形式导出,并进一步分析。导出信息包括发表期刊、标题、作者姓名、发表年份、关键词、摘要及所引用的参考文献等在内的所有文献的全部信息。
1.2 研究方法根据最终检索结果,利用Origin 2017软件对文献类别、年发文数量变化、文献年被引次数变化及研究方向变化进行分析作图。VOSviewer可通过文献数据的连接和集群,构建关于不同知识领域的相应图谱,以提供相关清晰的可视化信息[21]。本研究首先利用VOSviewer软件中的合作网络(Co-authorship)分析,对文献丛属国家、机构进行分析。再通过VOSviewer软件中的共现(Co-occurrence)分析,对关键词进行处理,最终得到相应可视化图谱,用于分析探讨该研究领域重要关键词及其共现关系。最后根据CiteSpace的关键词突现分析,探讨土壤ARGs研究进展及热点。
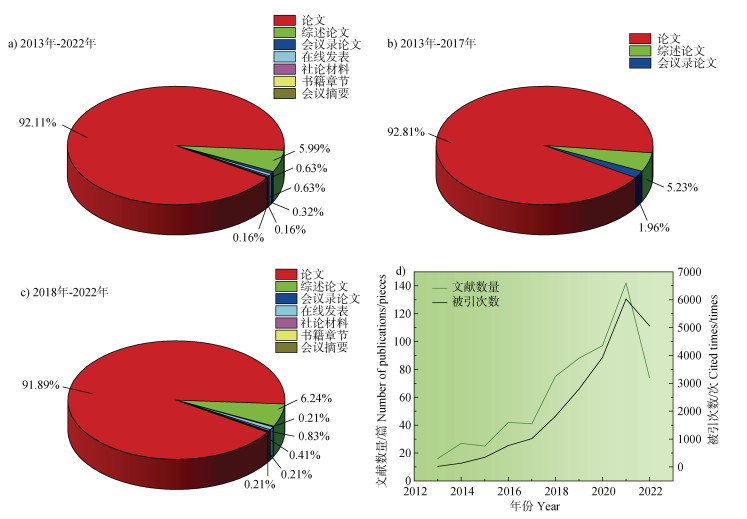
2 结果与讨论 2.1 文献发表类型根据Web of Science数据库的检索结果,对本研究得到的文献类型进行分析,结果如图 1a-图 1c所示。由图 1a可知,土壤ARGs相关文献类型可以分为7类,分别为:论文、综述论文、在线发表、会议录论文、社论材料、会议摘要和书籍。在前5年(2013—2017年)的相关文献中(图 1b),该领域的文献类型仅有3类,分别为论文(141篇,92.81%)、综述论文(8篇,5.23%)及会议录论文(3篇,1.96%);而后5年(2018—2022年)中该领域相关文献类型增加至7类(图 1c),分别为:论文(442篇,91.89%)、综述论文(30篇,6.24%)、在线发表(4篇,0.83%)、社论材料(2篇,0.41%)、书籍(1篇,0.21%)、会议摘要(1篇,0.21%)及会议录论文(1篇,0.21%)。结果表明,2013—2022年的各类文献发表数量均有增加,尤其论文发表数量大幅上涨,后5年发表数量超过前5年3倍。
|
图 1 土壤抗生素抗性基因相关研究的发表类型(a-c)和年发文数量及被引次数变化(d),数据收集于2022年10月6日 Fig. 1 The types(a-c) and the number of publications and cited times(d) of publications related to soil antibiotic resistance genes collected on October 6th, 2022 |
总体上,2013—2022年间论文发表数量占比最大(图 1a),可达到总数的92.11%。其次是综述论文,占比5.99%,剩余5类占比较少。论文和综述论文占总发文量的98.11%,可以很好地反映土壤ARGs的研究热点及进展。因此,下面的分析主要针对以上两类文献。
2.2 文献年发表数量图 1d显示了2013—2022年近10年间土壤ARGs相关文献发表数量年变化。由图可知,2013—2017年文献发表数量增长较平缓,平均年增长量为3篇。表明前5年土壤ARGs相关研究发展较缓慢。而2018—2021年发文数量迅速上升,平均年增长量为17篇。截至2022年9月年最高发文量是在2021年,达到142篇。2022年9个月发文量(74篇)也均超过2013—2017年每年发文量。结果表明,近5年土壤ARGs污染问题已得到世界各地科研人员的关注,该领域也进入快速发展时期,并逐渐成为研究热点。
2.3 论文被引情况文献被引次数年变化可以反映相应研究领域的发展,而单篇文献的被引次数可以作为评价其影响力和质量的指标。图 1d为2013—2022年土壤ARGs相关文献被引次数年变化。从图中可以明显看出近10年被引次数稳步上升,呈指数增长趋势。有效文献的总被引次数为22 123次,其中非自引次数高达17 481次,每篇平均被引次数为35.28次。这一结果表明,土壤ARGs污染问题相关研究具有较高质量和影响力,已引起世界各地研究人员的高度重视。
表 1总结了2013—2022年近10年间土壤ARGs相关文献被引次数最高的5篇论文。发表在Nature上的文章对18种农业和草地土壤中的抗生素进行了功能宏基因组选择,证明了细菌群落组成是土壤ARGs含量的主要决定因素[8],该文章以653次被引位居第一。而被引次数第二和第三的研究文章均提出在田间施用污水污泥堆肥可能导致大量的ARGs在土壤中扩散[22-23],警示人们污水污泥在回收利用之前应做好安全处理,避免造成二次污染。此外,科研人员不仅关注了ARGs在土壤中存在的主要因素及其来源,还研究了ARGs最终对人体健康产生的影响。Jechalke等[24]在Trends in Microbiology上发表的文章中指出,大量ARGs的出现可能会促进多耐药人类病原体的产生,并对抗生素治疗细菌感染过程产生影响。不仅如此,Marti等[25]还指出土壤中的ARGs可能通过蔬菜进入人体,进而对人体健康产生威胁。由表 1可知,5篇被引次数较高的论文均发表于2013—2016年,说明2017年以前有关土壤ARGs的研究为其今后发展奠定了坚实的基础。
|
|
表 1 2013—2022年被引次数最高的5篇论文 Table 1 Top 5 most cited papers from 2013 to 2022 |
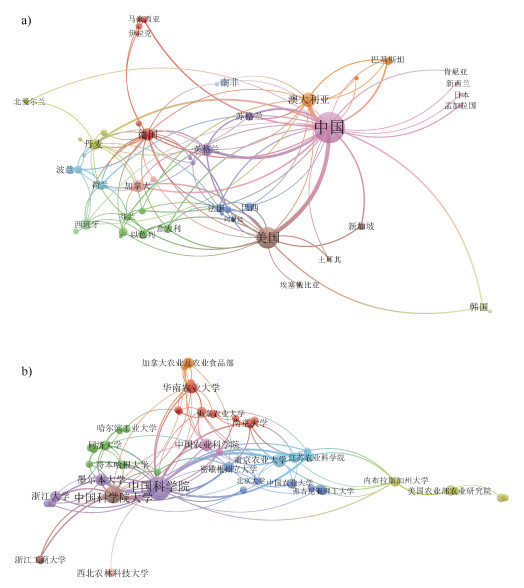
网络图可以提供某些研究领域具有影响力的国家/地区和机构及合作密切度的相关信息,从而推动世界各地科研人员建立合作关系。图 2为土壤ARGs相关研究发文国家/地区及机构的分析结果。图中节点大小代表发文数量,节点越大代表发文数量越多。线条粗细代表各国家/地区或各机构之间合作程度,线条越粗代表合作越密切。
|
图 2 论文发表国家(a)和研究机构(b)网络图 Fig. 2 Network diagram of countries(a) and research institutions(b) |
本研究中,与土壤ARGs相关的627篇文献分别发表于63个国家,发文数量前5的国家分别为中国(385篇),占61.40%;美国(127篇),占20.26%;澳大利亚(45篇),占7.18%;加拿大(27篇),占4.31%;德国(27篇),占4.31%。以上5个国家在土壤ARGs领域研究中占据重要地位。由图 2a可知,中国与27个国家均有合作,总关联度(Total link strength,TLS)值高达148。其中,与澳大利亚合作最密切,其次为美国。中国与澳大利亚和美国的合作发文量达到总发文量的47.30%。此外,美国和澳大利亚与其他国家/地区的合作也较为密切,TLS值分别为71和48,且二者与中国的合作最密切。
世界范围内共有780家机构为土壤ARGs研究做出了贡献,其中具有突出贡献的3家机构如图 2b所示。中国科学院位居第一,发文164篇,占据总发文量的26.16%。其次为中国科学院大学,发文77篇,占12.28%。中国科学院城市环境研究所位列第三,共发文73篇,占11.64%。此外,各机构之间也建立了不同程度的合作关系。中国科学院与140家机构建立了合作关系,TLS高达331,合作机构数量位居第一,与墨尔本大学、南京农业大学和中国农业科学院合作最为密切。而中国科学院大学与国内的多家机构,如中国农业科学院、福建农林大学和浙江大学在该领域合作频繁。总体来看,致力于土壤ARGs相关研究机构主要分布于国内,国外研究机构的活跃度相对较低。
2.5 文献关键词图 3a是根据土壤ARGs相关文献的关键词统计结果,分析关键词和热点密集度,结合颜色光谱获得的密度可视化图。图中黄色代表热点区域,深蓝色代表关注度相对较低的区域。由图 3a可知,密度较高的关键词为“抗生素抗性基因”、“土壤”、“抗生素”、“粪便”、“细菌群落”及“水平基因转移”等。图 3a中出现了多种抗生素类型,其中“四环素”和“磺胺嘧啶”为主要研究的抗生素类型;同时,受关注较多的关键词还包括“动物粪便”、“堆肥”及“粪肥施用”等。这一结果说明,人们对ARGs的认识和定量、源解析、微生物对ARGs的响应及其环境行为关注度较高。

|
图 3 2013—2022年土壤抗生素抗性基因相关研究的关键词密度可视化图(a)及研究方向变化(b) Fig. 3 Keywords density visualization(a) and changing research directions(b) of soil antibiotic resistance gene-related studies from 2013 to 2022 |
如图 3b所示,2013—2022年土壤ARGs相关研究中出现频次最高的6个研究方向分别为:生态环境科学(414篇,66.03%)、工程学(114篇,18.18%)、微生物学(107篇,17.06%)、农业(43篇,6.86%)、生物技术与应用微生物学(35篇,5.58%)及公共环境与职业健康(25篇,3.99%)。其中,环境科学与生态学领域相关研究的文章发表数量最多,发文量总体呈逐年升高的趋势;文献发表数量排名第二的研究方向是工程学,微生物学紧随其后;生物技术与应用微生物学领域的年发文量虽然不高,但近十年每年基本均有涉及;2015年后农业领域也逐渐开始土壤ARGs相关研究,说明农业生产活动与ARGs的产生和传播关系密切。除此之外,2017年在公共环境和职业健康领域也出现了土壤ARGs相关文章的发表,说明研究人员对土壤AGRs的研究远未止步于其对环境的影响,其与人类健康风险的关联也越来越受重视。
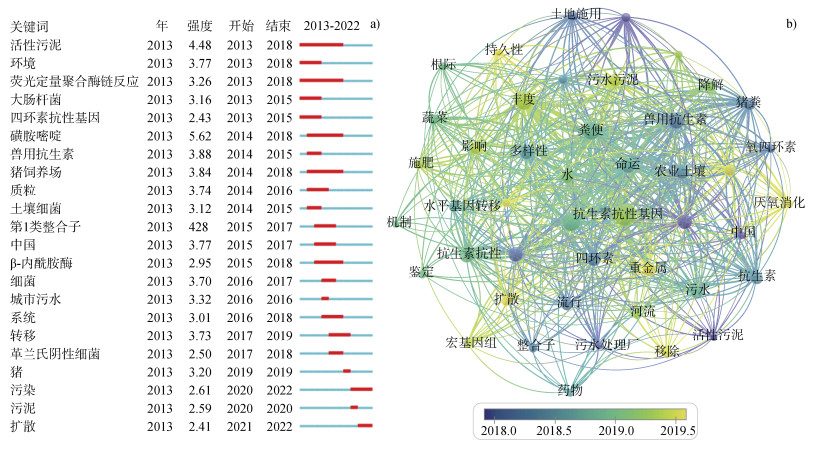
2.7 研究热点及发展趋势关键词与文章主题密切相关,关键词出现的频次越高,说明相关主题的研究程度越高[26]。关键词突现可以体现不同时期研究热点的变化,分析该领域潜在的发展趋势。图 4a为土壤ARGs相关研究的关键词突现。图中共显示了22个关键词,蓝色片段表示所有文章的时间跨度,红色片段表示相应突现关键词从出现到结束的年份。而关键词共现网络可以直观显示出研究热点随时间变化情况,进而分析相应领域研究的发展趋势。本研究分析了2013—2022年近10年间土壤ARGs相关研究的关键词,并通过VOSviewer筛选出现次数在20次以上的关键词,获得土壤ARGs关键词共现网络(图 4b)。图中节点大小代表相应关键词出现的频次,节点越大表明出现频次越高。节点及连线颜色随时间推移发生相应变化,其中紫色节点代表较早提出的关键词,颜色越接近黄色代表关键词提出的时间越晚。根据2013—2022年近十年突现指标(图 4a)及关键词共现(图 4b),对近10年来土壤ARGs领域的研究热点与发展趋势进行归纳总结。
|
图 4 土壤抗生素抗性基因相关研究的关键词突现(a)及共现网络图(b)(关键词出现次数≥ 20) Fig. 4 The keywords with citation bursts (a) and co-existence network (b) (occurrence times of keywords ≥ 20) of soil antibiotic resistance gene-related studies |
从图 4a和图 4b可以看出,“四环素抗性”、“磺胺嘧啶”、“β-内酰胺酶”、“革兰氏阴性细菌”及“荧光定量聚合酶链反应”是2018年前关注较多的关键词。其中,四环素类抗生素被广泛用作疾病治疗药物。美国食品和药物管理局的一份报告指出,2012年美国用于牲畜治疗药物和饲料补充的四环素类抗生素高达5.90×106 kg,占所有抗生素种类的41%[27]。在我国,2013年磺胺类抗生素的生产量就达到12.12万吨,其中磺胺嘧啶是使用较多的抗生素之一[28]。尽管在大多数环境介质中检测到的四环素等抗生素的含量在最低抑制浓度范围内,但其可对细菌暴露环境产生选择压力,进而导致ARGs的产生和传播[29]。此外,有研究证实,葡萄球菌[30]及包括肺炎克雷伯菌[31]在内的革兰氏阴性杆菌[32]在含有抗生素的环境中可以通过与β-内酰胺酶相关的信号通路对抗生素产生抗性。
除了认识ARGs产生的原因,掌握其在土壤环境中的含量也至关重要。随着分子生物技术的不断发展,基于荧光定量聚合酶链反应(Real-time quantitative polymerase chain reaction,qPCR)和宏基因组学技术等方法已被广泛应用于检测土壤等环境介质微生物群中的ARGs。qPCR作为一种快速、灵敏且具有特异性的定量ARGs的方法[33],不仅可以鉴定同源DNA标记,并追踪环境中相关抗性基因的传播扩散行为,还能通过评估不同环境样本中ARGs随时间变化情况来检测生物对抗生素的降解能力[34]。Wang等[35]在南极洲特拉诺瓦湾的两个研究站附近采集了62个土壤样本,采用qPCR技术分析检测到8种ARGs。Zhao等[36]通过qPCR技术分析后发现,鸡粪施用会显著增加土壤ARGs丰度。虽然qPCR技术被众多学者广泛应用,但该方法也具有一定局限性。一方面,它在通常情况下一次仅能检测几种ARGs;另一方面,在检测之前需要了解目标基因的相关信息,以便设计引物。因此,qPCR法通常用于检测已知的ARGs或与已知序列具有同源性的基因[37]。土壤微生物的多样性导致目前仍有众多未被识别的ARGs,2019年后发表的文献中多次出现的“宏基因组学技术”被认为是改善这种状况的有效方法,为土壤环境中ARGs的定量开辟了一条新的途径[38]。宏基因组学技术还可以完善目前已有的或构建新的宏基因组文库,从而为将来进行基因比对提供有力参考。Zheng等[39]分析了全球范围内1 088个土壤宏基因组样本,检测到558个ARGs,并指出农业土壤中的ARGs丰度高于非农业土壤。Li等[40]则通过宏基因组学技术探究了农业土壤内Cu和Zn等重金属对ARGs转移的影响。此外,“大肠杆菌”也是2018年以前重点关注的关键词。在众多微生物中,大肠杆菌作为模式菌,是与ARGs密切相关的微生物类型。一项全球监测数据表明,导致尿路感染的大肠杆菌对几乎所有用于治疗细菌感染的抗生素均有耐药性[41]。在世界卫生组织专家研讨会中,大肠杆菌也曾被称为监测耐药性的良好指标[42]。
2.7.2 土壤中ARGs的源解析畜禽粪便是土壤中ARGs的主要来源。土壤拥有地球上四分之一的生物多样性,是微生物栖息的重要场所。土壤多样性中蕴藏着ARGs或其前体,可能促进抗生素抗性的出现和扩散[43]。一项调查指出,中国每年抗生素产量高达2.1亿kg,其中46.1%用于畜牧业[44],用于动物疾病治疗和提高畜牧业产量。但有研究证实,动物体内的大部分抗生素以母体物质或具有活性的代谢物通过粪便排出体外[45-46]。而动物粪便作为抗生素耐药菌、ARGs及病原菌的储存库[47]施用于土壤后,会使土壤微生物获得抗性基因,进而显著提高土壤菌群内ARGs丰度[48-49]。因此,包括“猪饲养场”在内的动物养殖场产生的粪便就成为了土壤中ARGs的主要来源。以上研究与2018—2019年多次提到的“兽用抗生素”、“粪便”、“土地应用”、“农业土壤”、“细菌”等关键词相对应。
此外,污水和污泥也是土壤ARGs的重要来源。经过污水处理厂的二级和三级处理过程后,污水中的抗生素残留可以得到有效去除。但有研究表明,污水处理厂中种类繁多的营养物质、抗生素及其衍生物的存在,为抗生素耐药菌的生长和生存提供了有利条件,并有利于抗性基因横向迁移[50]。而且部分污水处理厂的最终排水中仍检测到了高浓度的ARGs,如果这部分污水用于污水回灌则会大大增加ARGs在土壤环境中传播的风险[51]。尽管“污水处理厂”是2018年前关注的热点,但由图 4b可知,与之密切相关的“污水污泥”在2019年后仍为重点关注对象。污水污泥含有丰富的营养物质,经堆肥化处理后常被作为有机肥料施用于土壤[52]。但多项研究证明,污水污泥内蕴藏着丰富的抗生素耐药菌及ARGs,被施用于土壤后会使ARGs丰度增加[53-55]。由此可知,污水处理厂的污水、污泥均是土壤中ARGs的重要来源。
2.7.3 土壤中ARGs的传播扩散与归趋从图 4a可知,随着时间的推移,人们对土壤ARGs的研究更加深入。2017年出现了突变关键词“转移”;2020年出现了“可移动遗传元件”。由图 4b可知,2019年以后发表的文章中还多次提到“施肥”、“水平基因转移”、“微生物群落”、“重金属”以及“蔬菜”等关键词,说明对土壤环境中ARGs的传播扩散及其归趋等相关探讨已成为研究热点。
以往研究发现,ARGs与微生物群落密切相关[56]。而作为微生物耐药组的一部分,ARGs及其宿主细菌可随施肥或堆肥施用过程而释放至土壤环境中[57]。进入土壤的ARGs会发生转移,主要包括垂直基因转移(细菌宿主繁殖)和水平基因转移(移动遗传元件)[58-59],这些迁移过程会促进ARGs在土壤环境内的传播扩散,加重土壤ARGs污染。土壤环境条件是影响ARGs迁移扩散过程的重要因素。Yang等[60]通过冗余分析评估了5种土壤理化性质(总氮、总磷、有机质、pH和含水量)与310种ARGs丰度的关系。结果表明,34种ARGs与不同的土壤理化性质相关。水分蒸发可导致不同土层的盐积累不同。最近的一项研究指出,ARGs的丰度自土壤深层到表层呈现逐渐减少的趋势,该现象说明盐度作为一种非生物胁迫可改变ARGs在土壤环境中的分布,影响其迁移扩散过程[61]。此外,土壤中抗生素残留会影响抗性基因的筛选和传播。这种影响取决于抗生素的半衰期及其生物利用度,而生物利用度在一定程度上又取决于土壤理化性质[62]。Chen等[63]指出,较小的土壤基质势对四环素在孔隙水中扩散的阻碍或较低渗透压对大肠杆菌吸收四环素的抑制可能会导致土壤吸附四环素的生物利用度降低,进而影响ARGs的筛选。
此外,土壤中其他污染物(重金属、微塑料及多环芳烃等)的存在会导致携带ARGs细菌的相对丰度升高[62]。Chen等[64]采集了石化厂不同多环芳烃污染水平的三种土壤,并对其进行宏基因组分析。结果表明,与两种多环芳烃污染较轻的土壤相比,ARGs在污染最严重土壤中的丰度升高了大约15倍。而且多环芳烃污染土壤中富集的大多数ARGs没有被质粒携带,表明它们在细菌之间迁移的可能性很低。同时,可生物降解微塑料(聚丁二酸丁二醇酯)的存在也被认为增加了土壤中ARGs的丰度和多样性[65]。而传统微塑料则会抑制ARGs在土壤中的扩散[66]。近期研究表明,除了抗生素滥用带来的选择压力,土壤中重金属等污染物也与抗生素抗性具有显著相关性[67]。据报道,细菌对重金属的防御机制往往与对抗生素的耐药机制类似,重金属含量升高也会间接诱导细菌细胞获得抗生素耐药性[68]。在盐碱地中,ARGs丰度就与Pb、Zn和Cd等重金属浓度呈正相关[69]。此外,部分重金属如Cu、Ag、Cr和Zn等还可加速ARGs的水平转移,从而促进耐抗生素潜在病原体的发展和传播[70]。由此可知,土壤中其他污染物的存在不仅会改变ARGs丰度,还会影响ARGs在土壤中的传播扩散。
ARGs不仅会在土壤环境内部传播扩散,还可能从土壤转移到作物的微生物群落,再通过食物链进入人体,进而使人体内微生物耐药性增加[71]。Zhang等[72]指出,土壤施用有机肥后,生菜根内生菌及叶内生菌对抗生素抗性显著增加,而且在生菜根和叶内均检测到了ARGs。Cerqueira等[73]也发现,在商业化经营的田地收集的番茄叶片和果实中,ARGs的检出量占其在根或土壤中检出量的1%~10%。此外,胡萝卜在施用有机肥的土壤中生长后,在其表皮内检测到ARGs的积累,而且部分ARGs亚型出现了向块茎内部迁移的现象。这对于不去皮食用胡萝卜的动物和人类而言,面临的耐药性风险将会显著增加[74]。因此,ARGs在土壤-植物系统中的传播扩散是其进入人体的潜在途径,应引起人们的重点关注。
在近十年对土壤ARGs的研究中,初期是认识和量化ARGs,随后是土壤ARGs的源解析及其与微生物内在联系的挖掘。随着研究的不断深入,ARGs在土壤中的传播扩散及其归趋的探讨成为近几年的研究热点。但总体来看,对ARGs在土壤中的传播阻控及削减技术的研究仍然不足。因此,面对严峻的土壤ARGs污染问题,该领域的研究仍需进一步深入。
3 结论在过去十年中,土壤ARGs研究领域飞速发展,年发文数量和被引频次快速且持续上涨。我国在该领域的研究一直处于领先地位,且与世界多国合作密切,国际学术影响力较高。论文关键词分析表明,该领域重点关注的抗生素类型主要为四环素和磺胺嘧啶;而在众多土壤微生物类型中,大肠杆菌是一直备受关注的与土壤ARGs最相关的微生物类型。关键词突变分析和共现网络分析均表明,ARGs的定性定量、源解析及其在土壤环境中的传播扩散和归趋逐步成为该领域重点关注的研究课题。面对土壤ARGs污染这一全球性公共健康问题,世界各国或组织应通力合作采取措施对其加以控制。未来研究可在“全健康”的理念下,发展成熟完善的监测系统,做到对土壤ARGs污染全方位的持续监测;建立数学模型,评估土壤ARGs的环境风险及对人体健康的影响;关注土壤ARGs源解析工作,争取做到以预防为主的源头控制;而对于已进入土壤环境的ARGs,深入探讨其传播扩散机制及影响因素,为ARGs削减技术研究奠定坚实基础。
| [1] |
Baym M, Stone L K, Kishony R. Multidrug evolutionary strategies to reverse antibiotic resistance[J]. Science, 2016, 351(6268): aad3292. DOI:10.1126/science.aad3292
(  0) 0) |
| [2] |
Wang H X, Wang N, Wang B, et al. Antibiotics in drinking water in Shanghai and their contribution to antibiotic exposure of school children[J]. Environmental Science & Technology, 2016, 50(5): 2692-2699.
(  0) 0) |
| [3] |
Feng M B, Ye C S, Zhang S Q, et al. Bisphenols promote the conjugative transfer of antibiotic resistance genes without damaging cell membrane[J]. Environmental Chemistry Letters, 2022, 20(3): 1553-1560. DOI:10.1007/s10311-022-01397-x
(  0) 0) |
| [4] |
Li H Z, Yang K, Liao H, et al. Active antibiotic resistome in soils unraveled by single-cell isotope probing and targeted metagenomics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(40): e2201473119.
(  0) 0) |
| [5] |
Zhao C X, Xin L Q, Xu X K, et al. Dynamics of antibiotics and antibiotic resistance genes in four types of kitchen waste composting processes[J]. Journal of Hazardous Materials, 2022, 424: 127526. DOI:10.1016/j.jhazmat.2021.127526
(  0) 0) |
| [6] |
Martínez J L. Antibiotics and antibiotic resistance genes in natural environments[J]. Science, 2008, 321(5887): 365-367. DOI:10.1126/science.1159483
(  0) 0) |
| [7] |
Forsberg K J, Reyes A, Wang B, et al. The shared antibiotic resistome of soil bacteria and human pathogens[J]. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761
(  0) 0) |
| [8] |
Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616. DOI:10.1038/nature13377
(  0) 0) |
| [9] |
D'Costa V M, King C E, Kalan L, et al. Antibiotic resistance is ancient[J]. Nature, 2011, 477(7365): 457-461. DOI:10.1038/nature10388
(  0) 0) |
| [10] |
Cytryn E. The soil resistome: The anthropogenic, the native, and the unknown[J]. Soil Biology & Biochemistry, 2013, 63: 18-23.
(  0) 0) |
| [11] |
Chen C Q, Li J, Chen P P, et al. Occurrence of antibiotics and antibiotic resistances in soils from wastewater irrigation areas in Beijing and Tianjin, China[J]. Environmental Pollution, 2014, 193: 94-101. DOI:10.1016/j.envpol.2014.06.005
(  0) 0) |
| [12] |
Fang H, Wang H F, Cai L, et al. Prevalence of antibiotic resistance genes and bacterial pathogens in long-term manured greenhouse soils as revealed by metagenomic survey[J]. Environmental Science & Technology, 2015, 49(2): 1095-1104.
(  0) 0) |
| [13] |
Czatzkowska M, Harnisz M, Korzeniewska E, et al. The impact of antimicrobials on the efficiency of methane fermentation of sewage sludge, changes in microbial biodiversity and the spread of antibiotic resistance[J]. Journal of Hazardous Materials, 2021, 416: 125773. DOI:10.1016/j.jhazmat.2021.125773
(  0) 0) |
| [14] |
Schlüter A, Szczepanowski R, Pühler A, et al. Genomics of IncP-1 antibiotic resistance plasmids isolated from wastewater treatment plants provides evidence for a widely accessible drug resistance gene pool[J]. FEMS Microbiology Reviews, 2007, 31(4): 449-477. DOI:10.1111/j.1574-6976.2007.00074.x
(  0) 0) |
| [15] |
Wang F H, Qiao M, Su J Q, et al. High throughput profiling of antibiotic resistance genes in urban park soils with reclaimed water irrigation[J]. Environmental Science & Technology, 2014, 48(16): 9079-9085.
(  0) 0) |
| [16] |
Udikovic-Kolic N, Wichmann F, Broderick N A, et al. Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(42): 15202-15207.
(  0) 0) |
| [17] |
Berendonk T U, Manaia C M, Merlin C, et al. Tackling antibiotic resistance: The environmental framework[J]. Nature Reviews Microbiology, 2015, 13(5): 310-317. DOI:10.1038/nrmicro3439
(  0) 0) |
| [18] |
Pan M, Chu L M. Fate of antibiotics in soil and their uptake by edible crops[J]. Science of the Total Environment, 2017, 599/600: 500-512. DOI:10.1016/j.scitotenv.2017.04.214
(  0) 0) |
| [19] |
Wang J H, Wang L J, Zhu L S, et al. Antibiotic resistance in agricultural soils: Source, fate, mechanism and attenuation strategy[J]. Critical Reviews in Environmental Science and Technology, 2022, 52(6): 847-889. DOI:10.1080/10643389.2020.1835438
(  0) 0) |
| [20] |
Zhang Z Y, Zhang Q, Wang T Z, et al. Assessment of global health risk of antibiotic resistance genes[J]. Nature Communications, 2022, 13(1): 1553. DOI:10.1038/s41467-022-29283-8
(  0) 0) |
| [21] |
Kasavan S, Yusoff S, Fakri M F R, et al. Plastic pollution in water ecosystems: A bibliometric analysis from 2000 to 2020[J]. Journal of Cleaner Production, 2021, 313: 127946. DOI:10.1016/j.jclepro.2021.127946
(  0) 0) |
| [22] |
Su J Q, Wei B, Ou-Yang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363.
(  0) 0) |
| [23] |
Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92/93: 1-10. DOI:10.1016/j.envint.2016.03.026
(  0) 0) |
| [24] |
Jechalke S, Heuer H, Siemens J, et al. Fate and effects of veterinary antibiotics in soil[J]. Trends in Microbiology, 2014, 22(9): 536-545. DOI:10.1016/j.tim.2014.05.005
(  0) 0) |
| [25] |
Marti R, Scott A, Tien Y C, et al. Impact of manure fertilization on the abundance of antibiotic-resistant bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest[J]. Applied and Environmental Microbiology, 2013, 79(18): 5701-5709. DOI:10.1128/AEM.01682-13
(  0) 0) |
| [26] |
Yan K, Lou J, Wang H Z, et al. Research of contaminated sites based on knowledge graph analysis and its development trend (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(5): 1234-1245. [严康, 楼骏, 汪海珍, 等. 污染场地研究现状与发展趋势: 基于知识图谱的分析[J]. 土壤学报, 2021, 58(5): 1234-1245.]
(  0) 0) |
| [27] |
Chen Z Y, Zhang Y J, Gao Y Z, et al. Influence of dissolved organic matter on tetracycline bioavailability to an antibiotic-resistant bacterium[J]. Environmental Science & Technology, 2015, 49(18): 10903-10910.
(  0) 0) |
| [28] |
Zhou A X. Research on the migration and transformation mechanism and remediation technique of sulfa antibiotics in phreatic water[D]. Changchun: Jilin University, 2015.[周爱霞. 潜水中磺胺抗生素迁移转化机理及修复技术研究[D]. 长春: 吉林大学, 2015.]
(  0) 0) |
| [29] |
Zhang Y J, Boyd S A, Teppen B J, et al. Role of tetracycline speciation in the bioavailability to Escherichia coli for uptake and expression of antibiotic resistance[J]. Environmental Science & Technology, 2014, 48(9): 4893-4900.
(  0) 0) |
| [30] |
Archer G L, Bosilevac J M. Signaling antibiotic resistance in staphylococci[J]. Science, 2001, 291(5510): 1915-1916. DOI:10.1126/science.1059671
(  0) 0) |
| [31] |
Jiang W, Yang W J, Liu P, et al. Antibiotic resistance of Klebsiella pneumoniae through β-arrestin recruitment-induced β-lactamase signaling pathway[J]. Experimental and Therapeutic Medicine, 2018, 15(3): 2247-2254.
(  0) 0) |
| [32] |
Walker G T, Rockweiler T J, Kersey R K, et al. Analytical performance of multiplexed screening test for 10 antibiotic resistance genes from perianal swab samples[J]. Clinical Chemistry, 2016, 62(2): 353-359. DOI:10.1373/clinchem.2015.246371
(  0) 0) |
| [33] |
Aydin S, Ince B, Ince O. Development of antibiotic resistance genes in microbial communities during long-term operation of anaerobic reactors in the treatment of pharmaceutical wastewater[J]. Water Research, 2015, 83: 337-344. DOI:10.1016/j.watres.2015.07.007
(  0) 0) |
| [34] |
Aydin S, Ince B, Ince O. Assessment of anaerobic bacterial diversity and its effects on anaerobic system stability and the occurrence of antibiotic resistance genes[J]. Bioresource Technology, 2016, 207: 332-338. DOI:10.1016/j.biortech.2016.01.080
(  0) 0) |
| [35] |
Wang F, Stedtfeld R D, Kim O S, et al. Influence of soil characteristics and proximity to Antarctic research stations on abundance of antibiotic resistance genes in soils[J]. Environmental Science & Technology, 2016, 50(23): 12621-12629.
(  0) 0) |
| [36] |
Zhao X, Wang J H, Zhu L S, et al. Environmental analysis of typical antibiotic-resistant bacteria and ARGs in farmland soil chronically fertilized with chicken manure[J]. Science of the Total Environment, 2017, 593/594: 10-17. DOI:10.1016/j.scitotenv.2017.03.062
(  0) 0) |
| [37] |
Fouhy F, Stanton C, Cotter P D, et al. Proteomics as the final step in the functional metagenomics study of antimicrobial resistance[J]. Frontiers in Microbiology, 2015, 6: 172.
(  0) 0) |
| [38] |
Willms I M, Grote M, Kocatürk M, et al. Novel soil-derived beta-lactam, chloramphenicol, fosfomycin and trimethoprim resistance genes revealed by functional metagenomics[J]. Antibiotics: Basel, Switzerland, 2021, 10(4): 378.
(  0) 0) |
| [39] |
Zheng D S, Yin G Y, Liu M, et al. Global biogeography and projection of soil antibiotic resistance genes[J]. Science Advances, 2022, 8(46): eabq8015. DOI:10.1126/sciadv.abq8015
(  0) 0) |
| [40] |
Li N, Chen J G, Liu C, et al. Cu and Zn exert a greater influence on antibiotic resistance and its transfer than doxycycline in agricultural soils[J]. Journal of Hazardous Materials, 2022, 423: 127042. DOI:10.1016/j.jhazmat.2021.127042
(  0) 0) |
| [41] |
Bryce A, Hay A D, Lane I F, et al. Global prevalence of antibiotic resistance in paediatric urinary tract infections caused by Escherichia coli and association with routine use of antibiotics in primary care: Systematic review and meta-analysis[J]. BMJ: Clinical Research Ed, 2016, 352: i939.
(  0) 0) |
| [42] |
Vikesland P J, Pruden A, Alvarez P J J, et al. Toward a comprehensive strategy to mitigate dissemination of environmental sources of antibiotic resistance[J]. Environmental Science & Technology, 2017, 51(22): 13061-13069.
(  0) 0) |
| [43] |
Wang F, Fu Y H, Sheng H J, et al. Antibiotic resistance in the soil ecosystem: A One Health perspective[J]. Current Opinion in Environmental Science & Health, 2021, 20: 100230.
(  0) 0) |
| [44] |
Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440.
(  0) 0) |
| [45] |
Chen Z Q, Wang Y, Wen Q X. Effects of chlortetracycline on the fate of multi-antibiotic resistance genes and the microbial community during swine manure composting[J]. Environmental Pollution, 2018, 237: 977-987. DOI:10.1016/j.envpol.2017.11.009
(  0) 0) |
| [46] |
Menz J, Olsson O, Kümmerer K. Antibiotic residues in livestock manure: Does the EU risk assessment sufficiently protect against microbial toxicity and selection of resistant bacteria in the environment?[J]. Journal of Hazardous Materials, 2019, 379: 120807. DOI:10.1016/j.jhazmat.2019.120807
(  0) 0) |
| [47] |
Heuer H, Smalla K. Manure and sulfadiazine synergistically increased bacterial antibiotic resistance in soil over at least two months[J]. Environmental Microbiology, 2007, 9(3): 657-666. DOI:10.1111/j.1462-2920.2006.01185.x
(  0) 0) |
| [48] |
Sun Y M, Qiu T L, Gao M, et al. Inorganic and organic fertilizers application enhanced antibiotic resistome in greenhouse soils growing vegetables[J]. Ecotoxicology and Environmental Safety, 2019, 179: 24-30. DOI:10.1016/j.ecoenv.2019.04.039
(  0) 0) |
| [49] |
Zhu D, Xiang Q, Yang X R, et al. Trophic transfer of antibiotic resistance genes in a soil detritus food chain[J]. Environmental Science & Technology, 2019, 53(13): 7770-7781.
(  0) 0) |
| [50] |
Yuan Q B, Guo M T, Yang J. Fate of antibiotic resistant bacteria and genes during wastewater chlorination: Implication for antibiotic resistance control[J]. PLoS One, 2015, 10(3): e0119403. DOI:10.1371/journal.pone.0119403
(  0) 0) |
| [51] |
Nnadozie C F, Kumari S, Bux F. Status of pathogens, antibiotic resistance genes and antibiotic residues in wastewater treatment systems[J]. Reviews in Environmental Science and Bio/Technology, 2017, 16(3): 491-515. DOI:10.1007/s11157-017-9438-x
(  0) 0) |
| [52] |
Kirchmann H, Börjesson G, Kätterer T, et al. From agricultural use of sewage sludge to nutrient extraction: A soil science outlook[J]. Ambio, 2017, 46(2): 143-154. DOI:10.1007/s13280-016-0816-3
(  0) 0) |
| [53] |
Urra J, Alkorta I, Mijangos I, et al. Application of sewage sludge to agricultural soil increases the abundance of antibiotic resistance genes without altering the composition of prokaryotic communities[J]. Science of the Total Environment, 2019, 647: 1410-1420. DOI:10.1016/j.scitotenv.2018.08.092
(  0) 0) |
| [54] |
Markowicz A, Bondarczuk K, Cycoń M, et al. Land application of sewage sludge: Response of soil microbial communities and potential spread of antibiotic resistance[J]. Environmental Pollution: 1987, 2021, 271: 116317.
(  0) 0) |
| [55] |
Chen M L, An X L, Liao H, et al. Viral community and virus-associated antibiotic resistance genes in soils amended with organic fertilizers[J]. Environmental Science & Technology, 2021, 55(20): 13881-13890.
(  0) 0) |
| [56] |
Liu Y W, Feng Y, Cheng D M, et al. Dynamics of bacterial composition and the fate of antibiotic resistance genes and mobile genetic elements during the co-composting with gentamicin fermentation residue and lovastatin fermentation residue[J]. Bioresource Technology, 2018, 261: 249-256. DOI:10.1016/j.biortech.2018.04.008
(  0) 0) |
| [57] |
Bai Y, Xu R, Wang Q P, et al. Sludge anaerobic digestion with high concentrations of tetracyclines and sulfonamides: Dynamics of microbial communities and change of antibiotic resistance genes[J]. Bioresource Technology, 2019, 276: 51-59. DOI:10.1016/j.biortech.2018.12.066
(  0) 0) |
| [58] |
Gao Q, Dong Q, Wu L W, et al. Environmental antibiotics drives the genetic functions of resistome dynamics[J]. Environment International, 2020, 135: 105398. DOI:10.1016/j.envint.2019.105398
(  0) 0) |
| [59] |
Zhang S, Wang Y, Song H L, et al. Copper nanoparticles and copper ions promote horizontal transfer of plasmid-mediated multi-antibiotic resistance genes across bacterial Genera[J]. Environment International, 2019, 129: 478-487. DOI:10.1016/j.envint.2019.05.054
(  0) 0) |
| [60] |
Yang F, Zhang F L, Li H P, et al. Contribution of environmental factors on the distribution of antibiotic resistance genes in agricultural soil[J]. European Journal of Soil Biology, 2021, 102: 103269. DOI:10.1016/j.ejsobi.2020.103269
(  0) 0) |
| [61] |
Tan L, Wang F, Liang M M, et al. Antibiotic resistance genes attenuated with salt accumulation in saline soil[J]. Journal of Hazardous Materials, 2019, 374: 35-42. DOI:10.1016/j.jhazmat.2019.04.020
(  0) 0) |
| [62] |
Smalla K, Cook K, Djordjevic S P, et al. Environmental dimensions of antibiotic resistance: Assessment of basic science gaps[J]. FEMS Microbiology Ecology, 2018, 94(12): fiy195.
(  0) 0) |
| [63] |
Chen Z Y, Zhang W, Wang G, et al. Bioavailability of soil-sorbed tetracycline to Escherichia coli under unsaturated conditions[J]. Environmental Science & Technology, 2017, 51(11): 6165-6173.
(  0) 0) |
| [64] |
Chen B W, He R, Yuan K, et al. Polycyclic aromatic hydrocarbons(PAHs) enriching antibiotic resistance genes(ARGs) in the soils[J]. Environmental Pollution, 2017, 220: 1005-1013. DOI:10.1016/j.envpol.2016.11.047
(  0) 0) |
| [65] |
Song R P, Sun Y Z, Li X F, et al. Biodegradable microplastics induced the dissemination of antibiotic resistance genes and virulence factors in soil: A metagenomic perspective[J]. Science of the Total Environment, 2022, 828: 154596. DOI:10.1016/j.scitotenv.2022.154596
(  0) 0) |
| [66] |
Sun M M, Ye M, Jiao W T, et al. Changes in tetracycline partitioning and bacteria/phage-comediated ARGs in microplastic-contaminated greenhouse soil facilitated by sophorolipid[J]. Journal of Hazardous Materials, 2018, 345: 131-139. DOI:10.1016/j.jhazmat.2017.11.036
(  0) 0) |
| [67] |
Gupta S, Graham D W, Sreekrishnan T R, et al. Heavy metal and antibiotic resistance in four Indian and UK rivers with different levels and types of water pollution[J]. Science of the Total Environment, 2022, 857: 159059.
(  0) 0) |
| [68] |
Baker-Austin C, Wright M S, Stepanauskas R, et al. Co-selection of antibiotic and metal resistance[J]. Trends in Microbiology, 2006, 14(4): 176-182. DOI:10.1016/j.tim.2006.02.006
(  0) 0) |
| [69] |
Wang L J, Wang J H, Wang J, et al. Soil types influence the characteristic of antibiotic resistance genes in greenhouse soil with long-term manure application[J]. Journal of Hazardous Materials, 2020, 392: 122334. DOI:10.1016/j.jhazmat.2020.122334
(  0) 0) |
| [70] |
Zhang Y, Gu A Z, Cen T Y, et al. Sub-inhibitory concentrations of heavy metals facilitate the horizontal transfer of plasmid-mediated antibiotic resistance genes in water environment[J]. Environmental Pollution, 2018, 237: 74-82. DOI:10.1016/j.envpol.2018.01.032
(  0) 0) |
| [71] |
Sheng H J, Wang F, Xiang L L, et al. Environmental behavior and control of antibiotic resistance genes in soil – A review (In Chinese)[J]. Acta Pedologica Sinica, 2023, 60(1): 39-49. [生弘杰, 王芳, 相雷雷, 等. 土壤中抗生素抗性基因的环境行为与阻控研究进展[J]. 土壤学报, 2023, 60(1): 39-49.]
(  0) 0) |
| [72] |
Zhang Y J, Hu H W, Chen Q L, et al. Transfer of antibiotic resistance from manure-amended soils to vegetable microbiomes[J]. Environment International, 2019, 130: 104912. DOI:10.1016/j.envint.2019.104912
(  0) 0) |
| [73] |
Cerqueira F, Matamoros V, Bayona J, et al. Antibiotic resistance genes distribution in microbiomes from the soil-plant-fruit continuum in commercial Lycopersicon esculentum fields under different agricultural practices[J]. Science of the Total Environment, 2019, 652: 660-670. DOI:10.1016/j.scitotenv.2018.10.268
(  0) 0) |
| [74] |
Mei Z, Xiang L L, Wang F, et al. Bioaccumulation of Manure-borne antibiotic resistance genes in carrot and its exposure assessment[J]. Environment International, 2021, 157: 106830. DOI:10.1016/j.envint.2021.106830
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


