2. 中国科学院大学, 北京 100049;
3. 南京师范大学地理科学学院, 南京 210046
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. School of Geography Science, Nanjing Normal University, Nanjing 210046, China
尽管全球变暖的驱动力仍然存在一定的争议,但全球变暖的事实已经成为世界各国政府和学术界的共识。例如,CO2作为最重要的温室气体,其浓度从工业革命前280 ppm增加至目前的408 ppm,被认为对全球气候变暖的贡献率高达66%[1]。CH4是仅次于CO2的全球第二大温室气体,尽管大气CH4浓度远低于CO2,但其增温潜势却是CO2的近28倍[2],非CO2温室气体CH4减排计划是各国政府应对全球气候变化的重要战略,也是我国2030年前实现碳达峰、2060年前实现碳中和的重要研究内容。
全球每年排放CH4约550~594 Tg[3],稻田是重要的CH4排放源,占人为CH4排放量的8%[2]。然而,已有研究表明,稻田厌氧层产生的CH4在进入大气之前,约30%~80%已经被微生物氧化[4],而好氧甲烷氧化菌被认为是减少稻田CH4排放、缓减全球变暖的唯一生物过滤器。事实上,甲烷氧化细菌(Methane-Oxidizing Bacteria,MOB)的研究历史已有上百年,是一类以CH4为唯一碳源和能源的微生物[5]。根据其细胞形态、内膜结构、代谢途径差异可分为类型Ⅰ(type Ⅰ)和类型Ⅱ(type Ⅱ)两大类群,分别属于γ-变形菌纲和α-变形菌纲[6],并且均广泛分布于水稻土中[7-8]。然而,甲烷氧化菌分离过程中极易被污染,特别是可培养菌株在很大程度上难以反映原位土壤真实环境。据此,基于标靶基因的分子生态技术成为甲烷氧化菌研究的重要策略。目前已知的甲烷氧化细菌几乎均含有甲烷单加氧酶编码基因pmoA[9],基于pmoA基因的甲烷氧化细菌分类与经典的16S rRNA基因系统发育进化关系基本一致,因此,pmoA基因结合16S rRNA基因高通量测序被广泛应用于甲烷氧化菌的分子生态学研究[10-11]。
未来大气CO2浓度升高(eCO2)可能通过影响水稻生长和土壤微生物过程而影响稻田CH4排放通量。一般认为,eCO2导致稻田CH4排放量最高可提升约40%[12-13]。然而,最近的研究发现eCO2的正反馈效应随着处理年限的增加呈下降趋势。例如,FACE实验(Free Air CO2 Enrichment)第一年增加了69.4%的CH4排放量,而第二、第三年则分别降至44.0%和25.6%[14];FACE处理13~15年之后,CH4排放量较对照处理减少了11%~54%[15]。产生这些不一致结果的主要原因可能是:CH4排放通量由产生与氧化两个过程决定,分别由产甲烷菌和甲烷氧化菌两种功能微生物主导。eCO2一方面促进植物光合作用,增加土壤有机碳输入,为产甲烷菌提供更多碳源,从而促进CH4产生;另一方面,eCO2也可能通过增加植物根系生物量和通气组织,使得根系泌氧能力增强,促进好氧甲烷氧化菌活性而增加甲烷氧化量[16]。因此,土壤类型、水稻品种、秸秆还田、灌溉管理和施肥等环境因子均有可能影响产甲烷菌或甲烷氧化菌[17-21],进而导致大气CO2升高对CH4排放的影响规律不同。通过定量分析土壤中产甲烷菌和甲烷氧化菌的数量,有研究发现eCO2处理可以同时提高产甲烷菌和甲烷氧化菌数量,而随着处理时间增加,eCO2对甲烷氧化菌的促进效应增强,从而逐步加强了CH4的氧化消耗,降低了eCO2对CH4的增排效应[14]。然而,有关eCO2影响甲烷氧化的微生物过程机制报道较少,甲烷氧化微生物组成和活性的变化规律仍然没有明确的结论。尽管有研究结果显示eCO2使得水稻土中的主导甲烷氧化菌类群由类型Ⅱ转变为类型Ⅰ[22],eCO2对类型Ⅱ甲烷氧化菌的相对丰度有显著影响[23],但是单一的微生物群落结构变化与环境因素之间的相关性分析,可能无法真实反映其中的内在联系。而稳定性同位素示踪甲烷氧化菌核酸DNA则能够在微生物分类地位与功能之间建立直接的联系,在一定程度上更加准确地反映甲烷氧化菌及其功能对大气CO2浓度升高的适应规律。
本研究依托中国FACE水稻田实验平台,以好氧甲烷氧化功能基因pmoA以及细菌16S rRNA基因为靶标,通过实时荧光定量PCR、稳定性同位素核酸探针DNA-SIP结合高通量测序技术,研究大气CO2浓度升高条件下水稻土甲烷氧化速率和活性微生物的变化规律,期望为合理制定CH4减排措施、有效应对全球气候变化提供科学依据。
1 材料与方法 1.1 土壤样本采集水稻土样本采集于中国FACE水稻田实验平台,该实验平台建于2004年,位于江苏省扬州市宗村良种场(32°35′5′′N,119°42′0′′E)。当地典型耕作方式为单季稻-麦轮作方式。FACE田间实验区由六个对边距为12.5 m的八角形实验圈构成,其中三个圈中的大气CO2浓度较大气CO2浓度升高200± 40 μL·L–1(在本研究中称为FACE圈),另三个圈为正常大气条件下的CO2浓度(对照圈Ambient)[18,24]。
本研究于2008年水稻收获后,分别采集FACE圈和对照圈表层0~10 cm土壤样本。在实验室对土壤进行晾干处理,研磨过2 mm筛,混合均匀,调节水分含量为田间持水量的60%(即25%左右),用于甲烷氧化微宇宙培养实验。
1.2 13C-CH4稳定性同位素核酸探针(DNA-SIP)基于13C-CH4的稳定性同位素核酸探针DNA-SIP实验流程如下图所示(图 1)。准确称取混合均匀的土样(FACE圈和对照圈Ambient)5.0 g,均匀铺置于120 mL血清瓶底部,血清瓶用丁基橡胶塞密封并用铝盖锁紧,从每个瓶中抽出1.2 mL空气后注入1.2 mL高纯CH4,使瓶内CH4浓度为1%(10 000 μL·L–1)左右。设置13CH4标记组和非标记12CH4对照组,分别注入13CH4(99% 13C原子丰度,Cambridge Isotope Laboratory)或12CH4,并在瓶内气体平衡后测定初始CH4浓度(GC,Agilent 7890,USA)。将所有处理在30 ℃条件下避光静置培养,每隔12 h抽取顶空气体1 mL测定CH4浓度。每个处理设置两组平行,一组样品在甲烷氧化活性最强(消耗60%~70%,36 h)结束培养,另一组在CH4被全部消耗后(100 μL·L–1以下,72 h)结束培养。培养结束后打开瓶塞,将土样全部取出并保存于–20 ℃,用于DNA提取和分子生物学分析。
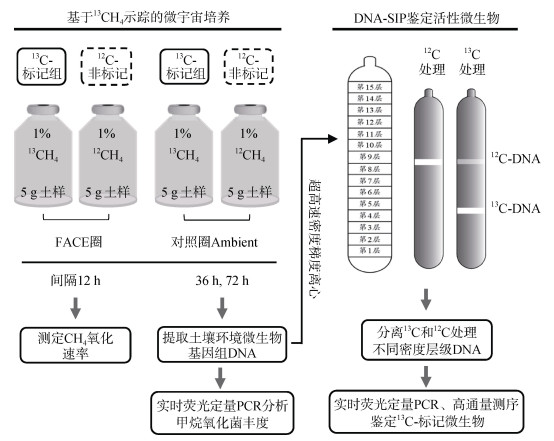
|
图 1 13CH4稳定性同位素核酸探针(DNA-SIP)研究大气CO2浓度升高影响水稻土好氧甲烷氧化的实验设置与流程 Fig. 1 Experimental design of DNA-based stable isotope probing(DNA-SIP)of methane-oxidizing bacteria under elevated atmospheric CO2 in paddy soil |
分别称取培养后的水稻土各0.5 g,包括36 h和72 h两个时间点的标记组和非标记组土壤样本,共计24个,使用FastDNA® Spin Kit for Soil(MP Biomedicals)试剂盒,根据说明书提供的操作步骤提取土壤微生物基因组DNA,并将其溶解于100 μL无菌水。通过微量紫外分光光度计(NanoDrop ND-1000)测定DNA浓度和纯度。
将上述得到的总DNA样品进行超高速密度梯度离心,实验操作参考已有研究[25-26]。关键步骤包括:将2.0 μg DNA与CsCl溶液均匀混合形成初始浮力密度为1.725 g·mL–1的离心液,并转移至5.1 mL的超高速离心管中,随后置于贝克曼超高速离心机中,20 ℃条件下45 000 r·min–1(190 000 ×g)离心44 h。离心结束后将离心管中的溶液由下至上均分为15个不同浮力密度的DNA溶液,分别收集至1.5 mL无菌离心管中,并通过PEG-6000将DNA与溶液中的氯化铯介质分离,进一步采用70%乙醇清洗DNA,最后将其溶于30 μL无菌水中,-20 ℃保存,用于标记DNA(13C-DNA)的鉴定和活性微生物组成分析。
1.3 实时荧光定量PCR与高通量测序针对超高速离心后得到的不同浮力密度层级的DNA样本,使用引物A189f/mb661r[11]定量甲烷氧化功能基因pmoA的丰度。定量PCR反应体系20 μL包括:10 μL SYBR Premix Ex Taq(Takara),上游和下游引物(10 µmol·L–1)各0.5 μL,1 μL DNA模版(DNA-SIP获得的各浮力密度层级的DNA浓度范围在10 ng·μL–1以下,直接用于PCR反应)和8 μL无菌水。pmoA基因扩增的反应条件:95℃,3 min;30 ×(94 ℃,30 s;54.5 ℃,30 s;72 ℃,45 s);72 ℃,10 min。PCR产物经熔解曲线和琼脂糖电泳验证,确认其专一性和有效性。所有扩增效率在80%~100%范围之内,R2为0.990~0.999(CFX96 Optical Real-Time Detection System,Bio-Rad,Laboratories Inc.,Hercules,CA,USA)。最后通过分析pmoA基因绝对量在不同浮力密度DNA中的分布规律来鉴别甲烷氧化菌的13C-DNA。
对13C-DNA样品进行高通量测序,分析其中的微生物组成。首先使用带有不同标签(Tag)的细菌16S rRNA基因通用引物515F/907R(Tag-GTGCCAG CMGCCGCGG/Tag-CCGTCAATTCMTTTRAGTTT)进行PCR扩增[27],PCR产物经试剂盒(DNA Fragment Purification Kit)纯化后溶解在适量的Tris-EDTA缓冲液中,并且测定DNA浓度。最后将DNA样品等质量混合后进行454高通量测序(Roche GS-FLX)。下机数据首先使用mothur(http://www.mothur.org/)进行质控,以25分为质量控制阈值,保留长度为200~600的序列,质控不允许有模糊碱基;根据扩增时所用的标签提取样品信息,获得每个样品的高质量序列。用RDP Multi-Classifier(https://rdp.cme.msu.edu/)对序列进行物种分类注释,cutoff设置为0.5。
1.4 数据处理采用IBM SPSS Statistics 19.0(SPSS Inc.,Cary,NC,USA)统计软件进行数据分析,采用单因素方差(One-way ANOVA)法对FACE圈和对照圈Ambient的甲烷氧化速率、甲烷氧化菌丰度进行统计显著性差异检验,P < 0.05为统计显著性。采用Origin 2021进行图表绘制。
2 结果 2.1 大气CO2浓度升高对水稻土甲烷氧化速率的影响在初始CH4浓度10 000 μL·L–1条件下培养水稻土,通过测定CH4氧化曲线发现,FACE圈土壤在前36 h消耗了瓶中77%的CH4,对照圈Ambient土壤仅消耗了63%的CH4。尽管两种土壤均能在72 h内将初始添加的CH4几乎全部消耗完(最终浓度小于100 μL·L–1),但FACE圈土壤的CH4下降速率高于对照圈(图 2a)。FACE圈在前36 h的甲烷氧化速率最高达301.8 nmol·g–1 d.w.s·h–1,显著高于对照圈Ambient土壤(图 2b)。实时荧光定量pmoA基因发现,FACE圈土壤的甲烷氧化菌丰度为6.1×106~8.8×106 copies·g–1 d.w.s,而且在CH4氧化36 h和72 h后均高于对照圈Ambient,增幅约为1.1倍~1.2倍(图 2c)。这些结果表明大气CO2浓度升高可能刺激了甲烷氧化菌生长并提升了甲烷氧化速率。
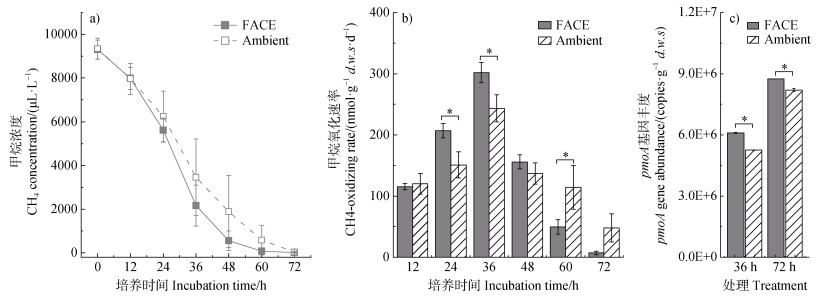
|
注:FACE指CO2浓度较正常大气条件升高(200 ± 40)μL·L–1的实验处理,Ambient指CO2浓度为正常大气条件的实验处理,下同。*表示FACE和Ambient处理之间具有显著性差异,P < 0.05。 Note: FACE indicates the treatment under elevated atmospheric CO2((200 ± 40)μL·L–1), Ambient indicates the treatment under ambient CO2. * indicates significant differences between the FACE and Ambient treatment, P < 0.05. 图 2 大气CO2浓度升高对水稻土甲烷氧化速率和MOB数量的影响 Fig. 2 Methane-oxidizing rates and MOB abundances under elevated atmospheric CO2(FACE)and ambient CO2(Ambient)in paddy soil |
经过36 h和72 h甲烷氧化后,分别提取土壤总DNA样本进行DNA-SIP超高速离心,获得不同CsCl密度层级DNA后,对甲烷氧化菌的pmoA基因定量。以每个处理中pmoA基因拷贝数最大的层级为参照,求得每一层拷贝数与参照的比值,并以此为纵坐标,同时以超高速密度梯度离心后不同DNA层级的浮力密度值为横坐标,得到pmoA基因在不同DNA层的比例分布图(图 3)。结果表明:与12CH4对照处理相比,所有13C-CH4标记处理中的pmoA基因均集中分布在浮力密度较大的层级(重层,第5~8层),而12C-CH4对照处理中的pmoA基因集中分布在轻层(第12~13层),在重层的丰度非常低。表明土壤甲烷氧化过程,甲烷氧化菌同化了大量13C-CH4并且合成13C-DNA,在超高速离心过程中被分离至重层。
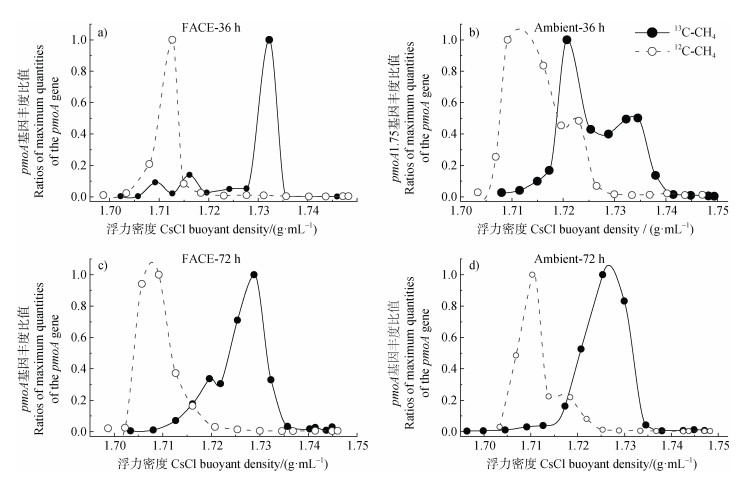
|
图 3 甲烷氧化功能基因pmoA在不同浮力密度DNA层级中的分布规律 Fig. 3 Quantitative distribution of pmoA gene copy numbers across the entire buoyant density gradients of the fractionated DNA from FACE and Ambient treatments incubated with 13CH4 or 12CH4 |
针对FACE圈和对照圈土壤重层DNA进行高通量测序分析,得到222 753条有效序列,其中包括77 936条甲烷氧化菌序列,占比35.0%(表 1)。甲烷氧化菌在重层13C-DNA中的相对丰度较高,表明甲烷氧化菌细胞分裂繁殖并同化了大量13C-CH4。FACE圈13C-CH4标记处理36 h后,甲烷氧化菌在第5层DNA(F5)中的相对丰度达到57.3%,72 h后最高达到64.8%(F6),Ambient处理36 h和72 h后最高达到60.3%和62.8%。此外,甲烷氧化菌在重层DNA中的相对丰度高低与其标记程度以及其他微生物的丰度有关,甲烷氧化菌之外的其他微生物通过同化13C-CH4氧化过程中的中间代谢产物被标记,也可能与甲烷氧化过程紧密关联。
|
|
表 1 13C-DNA的16S rRNA基因高通量测序结果汇总 Table 1 Summary of pyrosequencing results using a universal primer of 515F-907R targeting the 16S rRNA genes of the microbial community in 13C-DNA fractions |
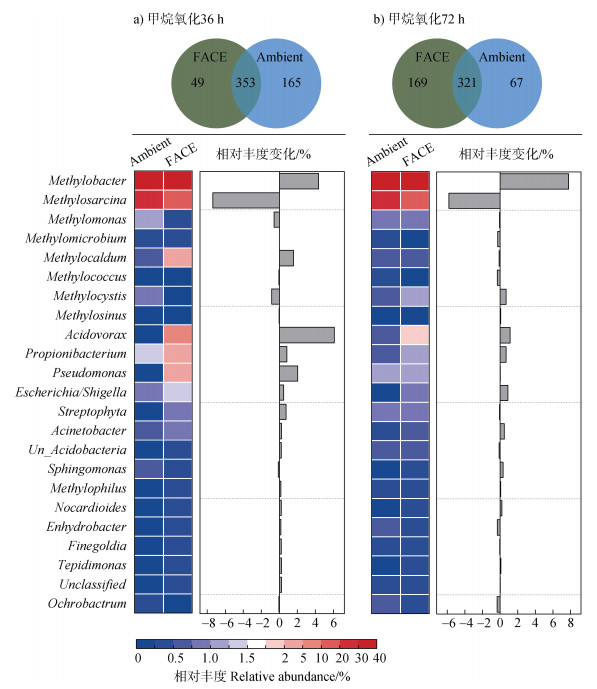
针对重层DNA中的甲烷氧化菌进行物种组成分析结果表明:经过36 h和72 h培养后,FACE圈和对照圈Ambient土壤中绝大多数活性的13C-甲烷氧化菌均属于类型Ⅰ,其相对丰度占总甲烷氧化菌比例高于88%,主导类群为甲基杆菌属Methylobacter和甲基八叠球菌属Methylosarcina(图 4)。进一步分析表明:甲烷氧化36 h后,FACE圈土壤中的Methylobacter属相对丰度超过62%,高于对照圈Ambient(图 4a);甲烷氧化72 h后,FACE土壤中Methylobacter属相对丰度为56.8%~65.5%,仍然高于Ambient(图 4b)。相反,对照圈Ambient土壤中Methylosarcina属的相对丰度较FACE圈明显增加,在培养36 h和72 h后分别增加11.1%和4.7%。因此,大气CO2浓度升高极可能刺激了Methylobacter属的活性,一定程度上抑制了Methylosarcina属的活性。
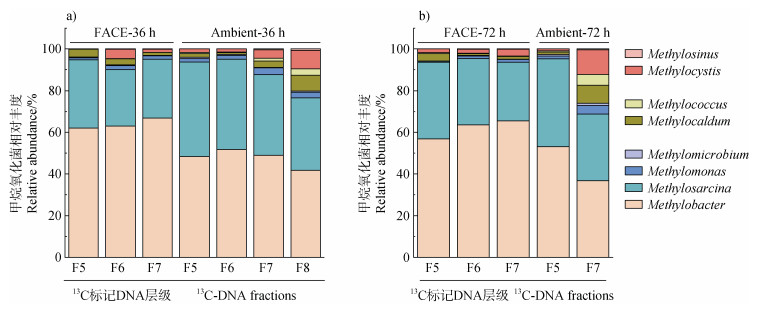
|
图 4 13C-CH4标记处理中重层DNA(13C-DNA)的甲烷氧化菌组成 Fig. 4 Phylogenetic affiliation of MOB in 13C-DNA fractions of 13C-CH4-labeled treatments |
CO2浓度升高不仅影响了活性甲烷氧化菌,也可能通过甲烷氧化代谢产物,如甲醇、甲醛和甲酸等影响其他微生物。土壤培养36 h后,FACE圈13C-DNA中共检测到402个细菌属,其中有353个属和对照圈Ambient相同,而49个属是FACE圈特有的细菌属(图 5a);甲烷氧化72 h后,FACE圈特有的细菌属增至169个(图 5b)。此外,FACE圈显著刺激了食酸菌属Acidovorax、丙酸杆菌属Propionibacterium和假单胞菌属Pseudomonas的生长活性,其相对丰度在36 h后分别较对照圈Ambient增加了6.1%、0.8%和2.0%。这些微生物在大气CO2浓度升高条件下具有更高的生长活性,可能优先利用13C-CH4甲烷氧化过程中产生的中间代谢产物,因此在FACE圈13C-DNA中的比例增加。
|
注:Venn图表示甲烷氧化36 h和72 h后,FACE圈和对照Ambient圈13C-DNA中共有、特有的活性微生物数量(属水平);热图表示FACE圈和Ambient圈13C-DNA中活性MOB和其他主导细菌的相对丰度;柱状图表示这些微生物丰度在FACE和Ambient之间的差值。 Note: Venn diagrams show the taxa distribution(genus level)in 13C-DNA of FACE and Ambient treatments after 36 and 72 hours of methane oxidation, respectively. The heat map displays the relative abundance of MOB and other abundant genera in 13C-DNA. The bar graph displays the changes in bacterial relative abundance in FACE than Ambient treatment. 图 5 大气CO2浓度升高条件下甲烷氧化36 h(a)和72 h(b)后活性甲烷氧化菌和其他主导细菌的相对丰度变化 Fig. 5 Changes in the relative abundance of active MOB and other abundant bacteria between FACE and Ambient treatments.(a)and(b)represents the changes in 36 h and 72 h of methane oxidation, respectively |
利用13C-CH4稳定性同位素核酸探针技术和高通量测序,本研究发现大气CO2浓度升高情景下,水稻土甲烷氧化潜力增强,甲烷氧化菌Methylobacter属活性增加,而Methylosarcina属活性减弱。这一研究结果规避了以往研究单纯相关性分析的内在缺陷[19,22-23]。例如,由于环境中微生物可能处于休眠状态以及游离DNA的存在,DNA水平的物种相对丰度或者数量改变并不能完全代表功能活性的变化。然而,稳定性同位素核酸探针技术完全依赖于细胞的分离繁殖,能够在关键生态过程与微生物物种之间建立直接联系。本研究通过DNA-SIP证明了eCO2对水稻土中类型Ⅰ甲烷氧化菌Methylobacter属生长繁殖的刺激作用,为研究大气CO2浓度升高情景下微生物的适应规律提供了明确证据。值得注意的是,不同生态系统和环境条件下,eCO2对甲烷氧化菌的影响可能不尽相同。例如,有研究报道eCO2降低了水稻根系中类型Ⅱ甲烷氧化菌甲基孢囊菌属Methylosinus的相对丰度[23],而增加了沼泽湿地类型Ⅱ甲烷氧化菌的丰度[28]。此外,甲烷氧化速率在不同生境中对大气CO2浓度升高的响应规律也有不同。本研究中,FACE圈水稻土甲烷氧化速率显著高于对照圈Ambient土壤,而在一些草地[29]和森林[30]等旱地土壤中,eCO2会导致CH4吸收潜力降低。其原因可能是受到水分、温度等环境条件的影响,而且由于旱地土壤中存在高亲和力大气甲烷氧化微生物类群,它们对eCO2的响应可能与传统甲烷氧化菌(类型Ⅰ、类型Ⅱ)不同,但其内在的适应机制,仍需整合分析甲烷氧化菌的遗传进化规律并深入研究。
大气CO2浓度升高可能通过影响土壤性质,进而改变甲烷氧化菌生态位并影响其群落结构。已有大量研究报道分析了大气eCO2升高条件下水稻土的碳、氮、磷、钾以及微量元素的含量变化规律,发现CO2浓度升高可提升土壤有机质含量[31-32]、降低可溶性氮含量[33],同时,碳、氮、磷、微量元素等的固存或活化还会受到养分之间的协助关系、生态化学计量比及植物生长等复杂因素的影响[34-35]。甲烷氧化菌是一类特异的功能微生物,几乎所有甲烷氧化菌都以CH4为唯一碳源和能源,因此,一般认为CH4和O2浓度是影响甲烷氧化过程的关键因素。然而,近年来其他营养元素或金属元素对甲烷氧化菌生理生态过程的影响也得到广泛关注。例如,最近的研究发现eCO2条件下甲烷氧化菌组成的变化与土壤溶解性有机碳和无机氮的含量有关[22]。在森林土壤中,土壤有机碳也可能通过改变土壤团聚体、孔隙度等改变土壤微域中CH4和O2浓度,进而影响微生物甲烷氧化潜力[36]。此外,不同类型甲烷氧化菌的生理特征存在显著差异[37],类型Ⅰ甲烷氧化菌在底物(如CH4和O2浓度,氮素营养)充分的条件下更容易获得生长优势,而类型Ⅱ甲烷氧化菌更能在环境胁迫条件下保持生长活性。在水稻土中,eCO2很可能导致了更多的有机碳输入,促进土壤厌氧微域形成并产生更多CH4,然而,eCO2也可能促进植物根系生长并分泌更多氧气,刺激类型Ⅰ甲烷氧化菌生长。本研究中发现FACE圈95%以上的活性MOB均属于类型Ⅰ,而对照圈Ambient水稻土中则栖息了一部分类型Ⅱ甲烷氧化菌。例如,土壤培养72 h后的13C-DNA中类型Ⅱ甲烷氧化菌占比13%(图 4)。值得注意的是,eCO2促进类型Ⅰ甲烷氧化菌Methylobacter属的活性,然而降低了Methylosarcina属的活性。这可能与氮素条件有关,eCO2增加土壤有机碳输入的同时减少了可溶性氮含量,使得土壤C/N比升高,使微生物处于氮素限制条件下,此时具有固氮潜力的微生物类群即可获得生长优势。前人研究发现,Methylobacter属的甲烷氧化菌株具有固氮潜力,而目前已知的Methylosarcina属的菌株均不具备固氮功能基因[38]。未来通过DNA和RNA水平的稳定性同位素探针技术分析,有可能准确解析Methylosarcina属与Methylobacter属对大气CO2浓度升高的生理代谢差异以及碳、氮耦合机制。
水稻土甲烷氧化过程极有可能激发了系列微生物级联反应,并在土壤有机碳周转过程中发挥了重要作用。每克水稻土中蕴藏的微生物细胞可高达10亿,其微生物多样性和功能极为复杂,单一功能微生物的变化通常耦合其他相关类群的共同响应。甲烷氧化菌作为一类专性自养微生物,通过碳源利用或者其他营养元素的摄取而与其他异养微生物类群产生联系[39],促进整体微生物群的功能发挥。本研究通过稳定性同位素示踪复杂微生物群落13C-DNA得到了明确证据。例如大气CO2浓度升高显著刺激了非甲烷氧化菌Acidovorax和Pseudomonas的生长活性(图 5)。这些微生物在生长过程中利用了更多的13C-CH4来源有机碳,与甲烷氧化菌的生长繁殖密切相关。因此,在研究温室气体CH4减排过程中,甲烷氧化菌与其他微生物之间的偶联关系是一个亟需关注的重要内容。并且,随着基因组、转录组等先进技术的快速发展,将为更精细的分类学和时空变异尺度下研究微观尺度的生命过程及其对宏观层的全球变化影响提供技术支撑。
4 结论大气CO2浓度升高刺激了水稻土好氧甲烷氧化活性。类型Ⅰ甲烷氧化菌Methylobacter属和Methylosarcina属是水稻土的主导活性甲烷氧化菌类群,前者在大气CO2浓度升高条件下生长繁殖活性增强,而后者的生长活性减弱。大气CO2浓度升高同时刺激了多种非甲烷氧化微生物生长活性,与CH4转化过程密切相关。
| [1] |
WMO. Greenhouse gas bulletin-No. 15: The state of greenhouse gases in the atmosphere based on global observations through 2018[R]. WMO Greenhouse Gas Bulletin, Switzerland, 2019. https://library.wmo.int/doc_num.php?explnum_id=10100.
(  0) 0) |
| [2] |
IPCC. Climate change 2014: Synthesis report. Pachauri R K, Meyer L A. Contribution of Working Groups Ⅰ, Ⅱ and Ⅲ to the Fifth Assessment Report of the Intergovernmental Panel on Climate Change[R]. Geneva, Switzerland: IPCC, 2014, 151.
(  0) 0) |
| [3] |
Saunois M, Stavert A R, Poulter B, et al. The global methane budget 2000–2017[J]. Earth System Science Data, 2020, 12(3): 1561-1623. DOI:10.5194/essd-12-1561-2020
(  0) 0) |
| [4] |
Conrad R. Importance of hydrogenotrophic, aceticlastic and methylotrophic methanogenesis for methane production in terrestrial, aquatic and other anoxic environments: A mini review[J]. Pedosphere, 2020, 30(1): 25-39. DOI:10.1016/S1002-0160(18)60052-9
(  0) 0) |
| [5] |
Dedysh S N, Knief C, Dunfield P F. Methylocella species are facultatively methanotrophic[J]. Journal of Bacteriology, 2005, 187(13): 4665-4670. DOI:10.1128/JB.187.13.4665-4670.2005
(  0) 0) |
| [6] |
Hanson R S, Hanson T E. Methanotrophic bacteria[J]. Microbiological Reviews, 1996, 60(2): 439-471. DOI:10.1128/mr.60.2.439-471.1996
(  0) 0) |
| [7] |
Stein L Y. The long-term relationship between microbial metabolism and greenhouse gases[J]. Trends in Microbiology, 2020, 28(6): 500-511. DOI:10.1016/j.tim.2020.01.006
(  0) 0) |
| [8] |
Knief C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker[J]. Frontiers in Microbiology, 2015, 6: 1346.
(  0) 0) |
| [9] |
Murrell J C, McDonald I R, Bourne D G. Molecular methods for the study of methanotroph ecology[J]. FEMS Microbiology Ecology, 1998, 27(2): 103-114. DOI:10.1111/j.1574-6941.1998.tb00528.x
(  0) 0) |
| [10] |
McDonald I R, Bodrossy L, Chen Y, et al. Molecular ecology techniques for the study of aerobic methanotrophs[J]. Applied and Environmental Microbiology, 2008, 74(5): 1305-1315. DOI:10.1128/AEM.02233-07
(  0) 0) |
| [11] |
Costello A M, Lidstrom M E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments[J]. Applied and Environmental Microbiology, 1999, 65(11): 5066-5074. DOI:10.1128/AEM.65.11.5066-5074.1999
(  0) 0) |
| [12] |
van Groenigen K J, Osenberg C W, Hungate B A. Increased soil emissions of potent greenhouse gases under increased atmospheric CO2[J]. Nature, 2011, 475(7355): 214-216. DOI:10.1038/nature10176
(  0) 0) |
| [13] |
Liu S W, Ji C, Wang C, et al. Climatic role of terrestrial ecosystem under elevated CO2: A bottom-up greenhouse gases budget[J]. Ecology Letters, 2018, 21(7): 1108-1118. DOI:10.1111/ele.13078
(  0) 0) |
| [14] |
Qian H Y, Jin Y G, Chen J, et al. Acclimation of CH4 emissions from paddy soil to atmospheric CO2 enrichment in a growth chamber experiment[J]. The Crop Journal, 2022, 10(1): 140-146. DOI:10.1016/j.cj.2021.03.017
(  0) 0) |
| [15] |
Yu H Y, Zhang G B, Xia L L, et al. Elevated CO2 does not necessarily enhance greenhouse gas emissions from rice paddies[J]. Science of the Total Environment, 2022, 810: 152363. DOI:10.1016/j.scitotenv.2021.152363
(  0) 0) |
| [16] |
Schrope M K, Chanton J P, Allen L H, et al. Effect of CO2 enrichment and elevated temperature on methane emissions from rice, Oryza sativa[J]. Global Change Biology, 1999, 5(5): 587-599. DOI:10.1111/j.1365-2486.1999.00252.x
(  0) 0) |
| [17] |
Yue J, Shi Y, Zheng X H, et al. The influence of free-air CO2 enrichment on microorganisms of a paddy soil in the rice-growing season[J]. Applied Soil Ecology, 2007, 35(1): 154-162. DOI:10.1016/j.apsoil.2006.05.005
(  0) 0) |
| [18] |
Wang S Y, Han L, Shi Y, et al. Effects of FACE on population and activities of methanogen and methanotroph in paddy soil (In Chinese)[J]. Soils, 2006, 38(6): 768-773. DOI:10.3321/j.issn:0253-9829.2006.06.018 [王殳屹, 韩琳, 史奕, 等. FACE对水稻土产甲烷菌和甲烷氧化菌种群及其活性的影响[J]. 土壤, 2006, 38(6): 768-773.]
(  0) 0) |
| [19] |
Yan C, Xu J, Zhong W H, et al. Effect of elevated CO2 on methanotrophs in the rhizosphere of rice plant (In Chinese)[J]. Acta Ecologica Sinica, 2013, 33(6): 1881-1888. [严陈, 许静, 钟文辉, 等. 大气CO2浓度升高对稻田根际土壤甲烷氧化细菌丰度的影响[J]. 生态学报, 2013, 33(6): 1881-1888.]
(  0) 0) |
| [20] |
Qian H Y, Chen J, Zhu X C, et al. Intermittent flooding lowers the impact of elevated atmospheric CO2 on CH4 emissions from rice paddies[J]. Agriculture, Ecosystems & Environment, 2022, 329: 107872.
(  0) 0) |
| [21] |
Qian H Y, Huang S, Chen J, et al. Lower-than-expected CH4 emissions from rice paddies with rising CO2 concentrations[J]. Global Change Biology, 2020, 26(4): 2368-2376. DOI:10.1111/gcb.14984
(  0) 0) |
| [22] |
Liu X, Shen L D, Yang W T, et al. Effect of elevated atmospheric CO2 concentration on the activity, abundance and community composition of aerobic methanotrophs in paddy soils[J]. Applied Soil Ecology, 2022, 170: 104301. DOI:10.1016/j.apsoil.2021.104301
(  0) 0) |
| [23] |
Liu J M, Han J J, Zhu C W, et al. Elevated atmospheric CO2 and nitrogen fertilization affect the abundance and community structure of rice root-associated nitrogen-fixing bacteria[J]. Frontiers in Microbiology, 2021, 12: 628108. DOI:10.3389/fmicb.2021.628108
(  0) 0) |
| [24] |
Liu G, Han Y, Zhu J G, et al. Rice-wheat rotational FACE platform Ⅰ. System structure and control (In Chinese)[J]. Chinese Journal of Applied Ecology, 2002, 13(10): 1253-1258. DOI:10.3321/j.issn:1001-9332.2002.10.012 [刘钢, 韩勇, 朱建国, 等. 稻麦轮作FACE系统平台Ⅰ. 系统结构与控制[J]. 应用生态学报, 2002, 13(10): 1253-1258.]
(  0) 0) |
| [25] |
Jia Z J, Conrad R. Bacteria rather than Archaea dominate microbial ammonia oxidation in an agricultural soil[J]. Environmental Microbiology, 2009, 11(7): 1658-1671. DOI:10.1111/j.1462-2920.2009.01891.x
(  0) 0) |
| [26] |
Jia Z J. Principle and application of DNA-based stable isotope probing-A review (In Chinese)[J]. Acta Microbiologica Sinica, 2011, 51(12): 1585-1594. [贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用[J]. 微生物学报, 2011, 51(12): 1585-1594.]
(  0) 0) |
| [27] |
Stubner S. Enumeration of 16S rDNA of Desulfotomaculum lineage 1 in rice field soil by real-time PCR with SybrGreenTM detection[J]. Journal of Microbiological Methods, 2002, 50(2): 155-164. DOI:10.1016/S0167-7012(02)00024-6
(  0) 0) |
| [28] |
Lee S H, Megonigal P J, Kang H. How do elevated CO2 and nitrogen addition affect functional microbial community involved in greenhouse gas flux in salt marsh system[J]. Microbial Ecology, 2017, 74(3): 670-680. DOI:10.1007/s00248-017-0960-8
(  0) 0) |
| [29] |
Dijkstra F A, Morgan J A, Follett R F, et al. Climate change reduces the net sink of CH4 and N2O in a semiarid grassland[J]. Global Change Biology, 2013, 19(6): 1816-1826. DOI:10.1111/gcb.12182
(  0) 0) |
| [30] |
Dubbs L L, Whalen S C. Reduced net atmospheric CH4 consumption is a sustained response to elevated CO2 in a temperate forest[J]. Biology and Fertility of Soils, 2010, 46(6): 597-606. DOI:10.1007/s00374-010-0467-7
(  0) 0) |
| [31] |
Chen D, Yu H Y, Zou L Y, et al. Effects of elevated atmospheric CO2 concentration on the stability of soil organic carbon in different layers of a paddy soil (In Chinese)[J]. Chinese Journal of Applied Ecology, 2018, 29(8): 2559-2565. [陈栋, 郁红艳, 邹路易, 等. 大气CO2浓度升高对不同层次水稻土有机碳稳定性的影响[J]. 应用生态学报, 2018, 29(8): 2559-2565.]
(  0) 0) |
| [32] |
Hu Z K, Chen X Y, Yao J N, et al. Plant-mediated effects of elevated CO2 and rice cultivars on soil carbon dynamics in a paddy soil[J]. New Phytologist, 2020, 225(6): 2368-2379. DOI:10.1111/nph.16298
(  0) 0) |
| [33] |
Ma H L, Zhu J G, Xie Z B, et al. Effect of FACE(free air carbon-dioxide enrichment)on soluble C, N and P in soil during rice growing (In Chinese)[J]. Soils, 2004, 36(4): 392-397. DOI:10.3321/j.issn:0253-9829.2004.04.011 [马红亮, 朱建国, 谢祖彬, 等. 开放式空气CO2浓度升高对水稻土壤可溶性C、N和P的影响[J]. 土壤, 2004, 36(4): 392-397.]
(  0) 0) |
| [34] |
Wang X Z, Zhang H J, Sun W, et al. Effect of elevated atmospheric CO2 on paddy soil nitrogen content during rice season (In Chinese)[J]. Chinese Journal of Applied Ecology, 2010, 21(8): 2161-2165. [王小治, 张海进, 孙伟, 等. 大气CO2浓度升高对稻田土壤氮素的影响[J]. 应用生态学报, 2010, 21(8): 2161-2165.]
(  0) 0) |
| [35] |
Ren S R, Zhu J G, Li H X, et al. Effect of free-air CO2 enrichment(FACE)on microelements in paddy soil (In Chinese)[J]. Ecology and Environment, 2007, 16(3): 982-986. DOI:10.3969/j.issn.1674-5906.2007.03.055 [任思荣, 朱建国, 李辉信, 等. 大气CO2浓度升高对稻田土壤中微量元素的影响[J]. 生态环境, 2007, 16(3): 982-986.]
(  0) 0) |
| [36] |
Lee J, Oh Y, Lee S T, et al. Soil organic carbon is a key determinant of CH4 sink in global forest soils[J]. Nature Communications, 2023, 14(1): 3110. DOI:10.1038/s41467-023-38905-8
(  0) 0) |
| [37] |
Ho A, Kerckhof F M, Luke C, et al. Conceptualizing functional traits and ecological characteristics of methane-oxidizing bacteria as life strategies[J]. Environmental Microbiology Reports, 2013, 5(3): 335-345. DOI:10.1111/j.1758-2229.2012.00370.x
(  0) 0) |
| [38] |
Kalyuzhnaya M G, Stolyar S M, Auman A J, et al. Methylosarcina lacus sp. nov., a methanotroph from Lake Washington, Seattle, USA, and emended description of the genus Methylosarcina[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(6): 2345-2350. DOI:10.1099/ijs.0.63405-0
(  0) 0) |
| [39] |
Oshkin I Y, Beck D A, Lamb A E, et al. Methane-fed microbial microcosms show differential community dynamics and pinpoint taxa involved in communal response[J]. The ISME Journal, 2015, 9(5): 1119-1129. DOI:10.1038/ismej.2014.203
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


