2. 江西省农业科学院土壤肥料与资源环境研究所, 国家红壤改良工程技术研究中心, 南昌 330200;
3. 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所), 南京 210008;
4. 中国科学院东北地理与农业生态研究所, 长春 130102
2. National Engineering & Technology Research Center for Red Soil Improvement, Soil and Fertilizer & Resources and Environment Institute, Jiangxi Academy of Agricultural Sciences, Nanchang 330200, China;
3. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
4. Northeast Institute of Geography and Agroecology, Chinese Academy of Sciences, Changchun 130102, China
土壤磷素存量很高,然而仅有0.1%能直接被植物吸收利用[1]。我国70%以上耕地土壤处于缺磷状态。解磷菌(Phosphate-solubilizing bacteria,PSB)是土壤磷循环中的重要一员,通过分泌有机酸、质子或者释放磷酸酶、植酸酶等,促使土壤难溶性或不溶性磷转化为易于被植物吸收的有效磷[2]。接种解磷菌可提高土壤磷素营养和作物产量,是一项理想的、环境友好且应用前景广阔的农业措施[3]。自20世纪中叶起,围绕环境高效解磷菌的筛选开展了大量研究,但进展有限,最基础的难题之一是传统稀释平板法不能获得土壤中解磷菌群落的全面信息,无法准确评价土壤中具有解磷能力的微生物种类和数量。
传统固体培养基富集分离过程依赖单个菌落的挑选和传代纯化,而菌落挑选具有主观性和随机性,会遗漏很多数量较低的稀有解磷菌[4];在连续传代培养过程中,培养基成分、传代次数及培养时间等因素,亦可能导致特定生理偏好的解磷菌更具生长优势,随着代次增加,丰度较低的稀有菌被易生长的优势菌所淘汰,进而遗漏一些可能在环境中发挥重要功能的解磷菌,以致无法准确评估土壤中解磷菌群落组成全貌。
此外,固体和液体培养基富集培养过程中,微生物细胞在氧气利用和营养获取方面也存在明显不同,可培养菌群在连续传代培养过程中可能产生差异。比如,固体培养基表面生长的菌落易于获取空气中的氧气,但上层细胞只能通过下层细胞或细胞间隙获取营养;而液体培养基内微生物细胞更易获取周围营养,但仅有液体表面微生物接触氧气,液体培养基内部c微生物生长受到较低氧气浓度的限制[4-5]。目前,固体和液体培养基对连续传代富集过程中可培养解磷菌多样性的影响鲜有报道。
高通量测序技术的快速发展,为系统评价连续传代过程中土壤可培养菌群变化规律提供重要技术支撑[6]。在固体和液体培养基连续传代富集过程中,通过收集固体平板或液体培养基中所有可培养微生物菌落/体,针对可培养微生物及其原位环境微生物总DNA,测序16S rRNA基因序列,并与数据库中纯菌株进行系统发育同源性分析,即可研究不同代次的可培养微生物多样性变化,明确可培养微生物种类和比例。运用上述方法,针对固氮菌,利用特殊培养基富集可培养固氮菌,发现传统分离方法遗漏的固氮菌属占比高达95.2%[5]。那么,利用特定培养基针对土壤解磷菌进行传代富集培养和测序分析,则可能明确土壤中可培养解磷菌的种类和比例,以及在不同培养基和代次的富集规律差异。
自然界解磷细菌种类繁多。目前已获得较多纯培养解磷菌株[2,7-10],为评估土壤中可培养解磷菌多样性提供了有效已知解磷菌资源库。解磷机制涉及多种基因,目前认知有限,主要集中在编码碱性磷酸酶的系列基因。其中,phoD基因被认为是土壤中分布最广、最重要的碱性磷酸酶编码基因之一[11],具有phoD基因微生物群落亦可被视为重要的已知解磷菌资源。FunGene(Functional gene)功能基因数据库收录了大量phoD基因序列及相应物种注释信息[12],为全面认知土壤解磷菌资源提供又一重要参考。
掌握环境中解磷菌的可培养特性及种质资源多样性,能够为解磷菌资源进一步开发和利用提供借鉴。基于以上,本研究利用不同解磷菌选择培养基,通过连续传代富集培养技术,获得可培养的第一代、第二代和第三代解磷菌富集物,再通过高通量测序技术进行可培养解磷菌的组成和多样性分析,进一步与供试土壤解磷菌群落和已知解磷菌数据库进行比较,以定量评价不同分类水平下土壤可培养解磷菌的多样性、比例和富集规律。
1 材料与方法 1.1 供试土壤供试土壤采集自江西新余县,为河流冲击性母质发育的冲积性水稻土。水稻收获后,采集0~20 cm耕层土壤,清除石块和植物残体等,过2 mm筛,混合均匀,于4 ℃保存。土壤理化性质:pH 5.61,铵态氮2.59 mg·kg–1,硝态氮1.25 mg·kg–1,全碳20.67 g·kg–1,全氮2.23 g·kg–1,有效磷10.30 mg·kg–1。
1.2 可培养解磷菌的富集 1.2.1 培养基的配制固体无机磷培养基(Solid-IPM)主要成分包括:葡萄糖2.0 g,硫酸铵0.1 g,氯化钠0.06 g,硫酸镁0.06 g,硫酸亚铁0.006 g,硫酸锰0.006 g,碳酸钙1.0 g,氯化钾0.06 g,磷酸三钙1.0 g,琼脂3.6 g,加去离子无菌水200 mL。固体有机磷培养基(Solid-OPM)中,除用0.06 g卵磷脂代替磷酸三钙外,其余成分和用量与Solid-IPM相同。将两种培养基加入适当氢氧化钠调节pH至7.0-7.5。
液体无机磷培养基(Liquid-IPM)和有机磷培养基(Liquid-OPM)除不加入琼脂外,其余配方与上述固体培养基相同。所有培养基置于高压灭菌锅中115 ℃灭菌30 min。待固体培养基稍冷却后,在超净台制备无菌平板。
1.2.2 解磷菌的富集策略称取土壤样品1.0 g,在100 mL无菌水中溶解,加入5.0 g无菌玻璃珠,以200 r·min–1在30 ℃下震荡30 min,制备土壤悬液。本实验研究对象为可培养解磷菌,特指在无机磷和有机磷培养基中富集的微生物集合,可被认为是所有“潜在”可分离纯培养的解磷菌株。
利用固体培养基的富集培养:吸取100 μL土壤悬液,均匀涂布在培养基表面,在30 ℃培养箱中倒置培养5 d,再将平板取出,在超净台里吸取10 mL无菌水将培养基表面菌落分多次冲洗至50 mL无菌离心管中,制成第一代富集菌液,取1 mL提取DNA。同时吸取100 μL菌液,使用平板涂布法接种传代培养,培养5 d后,同样以第一代菌落冲洗方式,将第二代菌落冲洗至50 mL无菌离心管中,制成第二代富集菌液。重复上述方法,制成第三代富集菌液,并提取DNA。每代设置4次重复。
利用液体培养基的富集培养:吸取100 μL土壤悬液,接种至100 mL无菌液体培养基,将培养瓶放入30 ℃培养箱中,震荡培养7 d,获得第一代富集菌液。将100 mL菌液以4 000×g离心10 min,弃上清,加入10 mL无菌水振荡混匀,浓缩为第一代富集菌液,取1 mL提取DNA。同时吸取100 μL第一代富集菌液,接种至新100 mL液体培养基中,传代培养7 d,采用上述相同方法浓缩为第二代富集菌液。依此类推,进行第三代解磷菌培养及富集菌液制备。每代设置3次重复。
1.3 解磷菌的多样性分析 1.3.1 微生物DNA提取供试土壤DNA采用FastDNA® Spin Kit for Soil(MP Bio)试剂盒提取。称取0.5 g土壤3份,用于土壤DNA提取,最终将DNA溶解于100 μL DES缓冲液,于–20 ℃保存。可培养解磷菌富集物DNA采用E.Z.N.A.® Bacterial DNA Kit(Omega)试剂盒进行提取。最后加入50 μL Elution Buffer,过滤收集DNA,于–20 ℃保存。
1.3.2 IIIumina Miseq高通量测序分析开展高通量测序分析微生物多样性变化,探究解磷菌富集规律。由上海派森诺生物科技股份有限公司扩增土壤样品和富集物中细菌16S rRNA基因(引物515F/907R)并建库,在Illumina Miseq测序平台上对其双端测序。根据barcode划分样本,移除barcode序列。针对16S rRNA基因,通过QIIME2过程进行质控、去燥、拼接、去嵌合体,获得非singletons ASVs特征序列和丰度表。根据默认参数,以Greengenes数据库(Release 13.8,http://greengenes.secondgenome.com/)(DeSantis et al.,2006)为参考进行物种注释。最后,在各分类水平下,分析样品检测到的所有物种分类信息(包括分类单元数量和分类单元对应的相对丰度)。相对丰度,是以样本中各分类水平各类型微生物序列数除以序列总数来表示。通过在线工具(https://bioinformatics.psb.ugent.be/webtools/Venn/)生成venn图,比较样本间共有微生物和特有微生物类型。高通量测序概况如表 1所示。为全面评估可培养解磷菌的多样性,避免低估或遗漏数量不占优势的解磷菌类群,后续将重复样品所获序列合并分析。测序原始数据已上传至NCBI数据库,序列号PRJNA1030136。
|
|
表 1 土壤和富集物样品的高通量测序结果 Table 1 High throughput sequencing results of soil and enrichments |
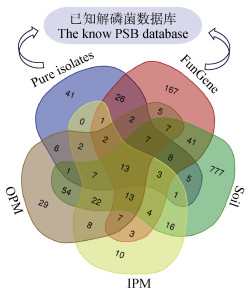
通过对文献报道已分离培养的解磷菌株[2,7-10]以及FunGene数据库含有phoD基因的解磷菌进行检索和整理,并定义为已知解磷菌数据库作为参考(表 2)。其中,(1)根据以往文献检索到除真菌外已知解磷菌125属;(2)针对FunGene数据库,按照设定参数(mimimumHMM Coverage=97%),获得9682条phoD基因和相应分类注释信息,除去冗余,整理出已知解磷菌(除真菌外)309属。以上两部分共有解磷菌62属,因此,构建已知解磷菌数据库共372属。
|
|
表 2 本研究构建的已知解磷菌数据库 Table 2 The known phosphate-solubilizing bacteria (PSB) database retrieved from previous pure isolates and the FunGene repository |
可培养“潜在”解磷菌比例的计算:分别获得(1)土壤DNA中微生物分类单元数。直接提取土壤DNA,高通量测序微生物16S rRNA基因,并在门、纲、目、科和属水平进行注释,获得土壤中所有微生物的系统发育信息和分类单元总数。(2)富集物DNA中微生物分类单元数。根据前述分析方法,分别获得无机磷和有机磷培养基中第一代、第二代或/和第三代中菌落富集物中可培养解磷菌的分类信息和分类单元数。后者除以前者,即可得到不同微生物分类水平下土壤可培养“潜在”解磷菌的比例。
可培养已知解磷菌比例的计算:将富集物DNA和土壤DNA中微生物在属水平分别与已知解磷菌数据库中注释信息进行比对,确定富集物和土壤中包含已知解磷菌属水平分类单元数,前者除以后者,得到属水平下已知解磷菌的可培养比例。
可培养解磷菌富集率的计算:基于16S rRNA基因分类,首先在属水平下,分别获得富集物DNA中所有可培养“潜在”解磷菌的相对丰度;其次获得土壤DNA中相应解磷菌属的相对丰度;前者除以后者,即可得到。富集率 > 1表示该菌属在培养中得到了富集。
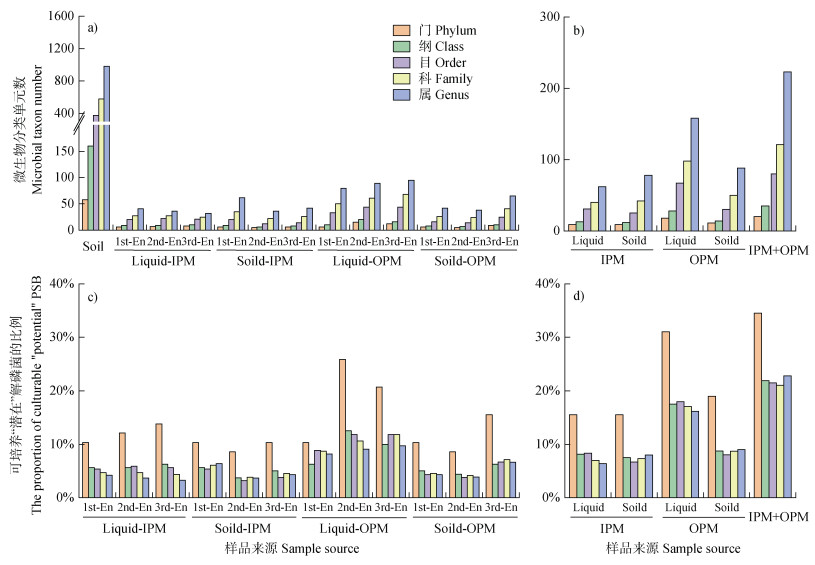
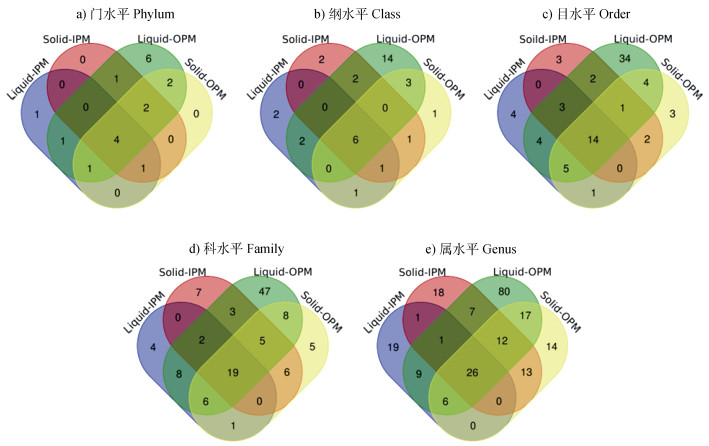
2 结果 2.1 不同培养基和代次中可培养“潜在”解磷菌多样性和比例供试土壤本底微生物共检测58门、160纲、373目、575科和979属(图 1a)。不同培养基和代次来源培养富集物(IPM+OPM)中共检测20门、35纲、80目、121科和223属(图 1c),即可培养“潜在”解磷菌在门、纲、目、科和属水平分别占供试土壤本底微生物的34.5%、21.9%、21.5%、21.0%和22.8%(图 1d)。在同一分类水平,不同培养基富集到“潜在”解磷菌分类单元数差异较大。OPM富集“潜在”解磷菌种类数普遍高于IPM,且在液体OPM中最多,而在液体IPM最少(图 1b)。对于不同培养基和代次来源培养富集物而言,可培养“潜在”解磷菌在门水平的占比最高(8.62%~25.9%),而在其他分类水平上的占比仅为3.22%~12.5%(图 1c)。综上可见,IPM和OPM均定向富集了相当数量“潜在”解磷菌。四种培养基富集共有解磷菌分类单元数由高到低依次为:属(26属)、科(19科)、目(14目)、纲(6纲)、门(4门),共有程度高低依次为:门(21.0%)、目(18.4%)、纲(17.1%)、科(15.7%)、属(11.7%)(图 2)。
|
注:分组样品的微生物分类单元数,由组内所有样品检测到的共有和特有微生物分类单元数求和得到。 Note: The microbial taxon number of group samples was obtained by summing the number of shared and specific microbial taxa detected in all samples in the group. 图 1 不同微生物分类水平下,可培养“潜在”解磷菌的分类单元数(a,b)和在土壤本底微生物中所占的比例(c,d) Fig. 1 The taxon number (a, b) and proportion (c, d) of culturable "potential" phosphate-solubilizing bacteria (PSB) in soil at different microbial taxonomic levels |
|
图 2 不同培养基中可培养“潜在”解磷菌间的共现规律(韦恩图) Fig. 2 Co-occurrence of the culturable "potential" PSB in different media at different microbial taxonomic levels (Venn diagram) |
如图 3所示,在属水平,供试土壤中检测到已知解磷菌110属,在土壤微生物群落中的占比为11.2%。IPM富集物检测到“潜在”解磷菌112属,占土壤微生物群落11.4%,其中包括已知解磷菌56属,在富集物群落中占比为50.0%;IPM对土壤已知解磷菌的培养比例可达50.9%。OPM富集物中检测到“潜在”解磷菌185属,占土壤微生物群落18.9%,包括已知解磷菌71属,在富集物群落中占比为38.4%;OPM对土壤已知解磷菌的培养比例可达64.5%。上述表明,OPM富集已知解磷菌多样性高于IPM。
|
图 3 不同培养基中可培养“潜在”解磷菌与已知解磷菌数据库在属分类水平的多样性比较 Fig. 3 Diversity comparison of culturable "potential" PSB in different media and the known PSB database at the genus level |
IPM+OPM共检测到“潜在”解磷菌223属(图 3),包括已知解磷菌83属,占土壤已知解磷菌群落75.5%,至少约24.5%(27属)土壤已知解磷菌未被富集或遗漏。IPM和OPM间还检测到相同已知解磷菌44属(图 3)。此外,富集物中还检测到大量未被检索的“潜在”解磷菌,亦可称之为未知解磷菌,共计140属,在可培养“潜在”解磷菌群落中占62.8%,在IPM和OPM富集物群落中分别占50.0%(56属)和61.6%(114属)。
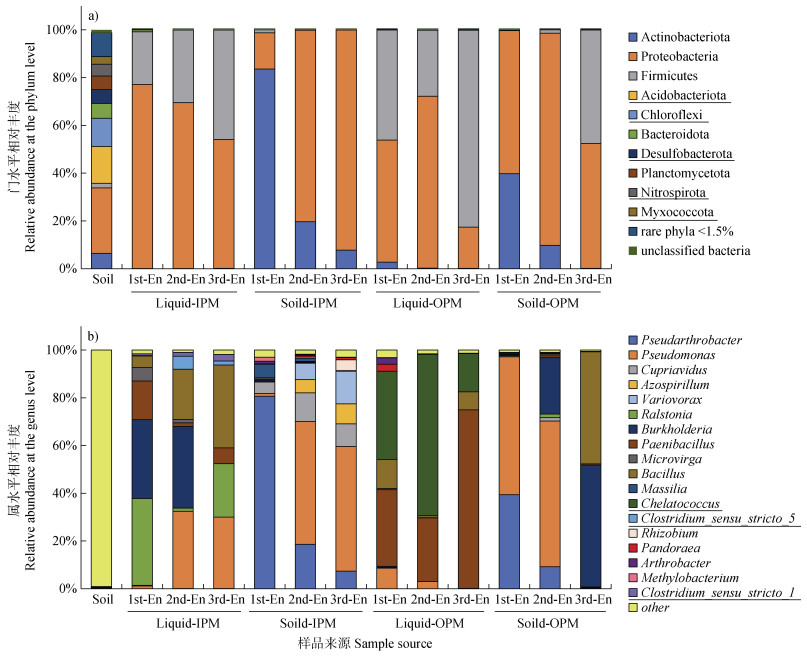
2.3 不同培养基和代次中可培养“潜在”解磷菌群落组成比较由图 4a可知,在门水平,土壤中优势微生物高低为:Proteobacteria(27.45%)、Acidobacteriota(15.38%)、Chloroflexi(11.9%)、Actinobacteriota(6.40%)、Bacteroidota(6.14%)、Planctomycetota(5.68%)、Nitrospirota(4.98%),以上类群共占整体微生物群落的77.93%。不同培养基和代次来源富集物中优势解磷菌主要隶属于Proteobacteria、Actinobacteriota和Firmicutes。液体IPM和OPM富集微生物主要有Proteobacteria和Firmicutes,两者之和分别达99.11%~99.89%和97.20%~99.79%。固体IPM富集微生物主要有Proteobacteria和Actinobacteriota,两者总和高于98.76%。固体OPM富集微生物门主要有Proteobacteria、Actinobacteriota和Firmicutes,三者总和高于99.97%。
|
注:图中标有下划线微生物,为可富集、但尚未被检索的“潜在”解磷菌门或属。 Note: The underlined microorganisms are the "potential" PSB which were enriched but have not yet been searched in the known PSB database. 图 4 土壤本底和培养基富集的微生物在门(a)和属水平(b)的相对丰度 Fig. 4 Microbial relative abundances in soil and enrichments at the phylum (a) and genus level (b) |
在属水平富集的优势微生物(保留任意代次培养富集物中微生物相对丰度 > 1%的属),如图 4b所示。不同培养基富集优势微生物属差别较大。液体IPM主要富集了Ralstonia、Pseudomonas、Burkholderia、Bacillus、Paenibacillus等。固体IPM富集的优势微生物,同样包括Pseudomonas,除此之外,还主要包括Pseudarthrobacter、Cupriavidus、Variovorax、Azospirillum、Massilia等。液体OPM富集的优势微生物,除Pseudomonas、Paenibacillus和Bacillus三属同液体IPM相似,还有Chelatococcus。与固体IPM相比,固体OPM同样富集了较多Pseudarthrobacter、Pseudomonas和Cupriavidus,此外,Burkholderia和Bacillus在传代培养时竞争优势逐渐增加。以上优势微生物中,Chelatococcus、Pandoraea、Arthrobacter三属仅在培养富集物而未在土壤中被检测。
2.4 不同培养基和代次中可培养“潜在”解磷菌的富集规律在门水平,微生物在不同培养基中富集规律(相对丰度随传代次数的变化)不同(图 5a-图 5c)。Proteobacteria普遍具有富集现象,尤其在OPM中均先增加后减少(图 5a)。在两种固体培养基中,Actinobacteriota都在第一代培养时富集率最高,随传代次数增加,富集率不断下降;而在两种液体培养基中未被明显富集(图 5b)。在连续传代培养过程中,Firmicutes在OPM和液体IPM中富集程度逐渐增加,第三代时富集率最高可达45倍,而在固体IPM中没有被明显富集(图 5c)。
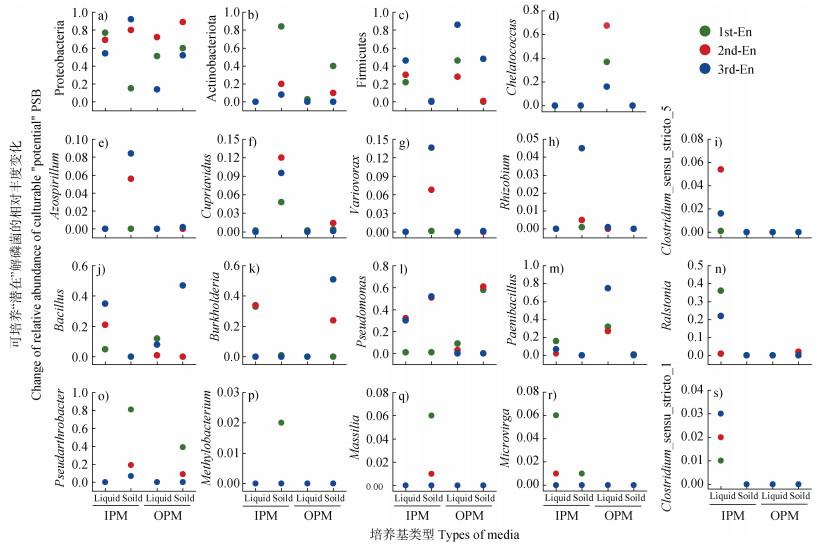
|
注:图a-c为不同微生物在门水平的相对丰度随代次的变化;图d-s为不同微生物在属水平的相对丰度随代次的变化。 Note:Fig. a-c represent the relative abundance changes of different microbes at the phylum level;Fig. d-s indicate the relative abundance changes of different microbes at the genus level. 图 5 不同培养基中可培养“潜在”解磷菌相对丰度随传代次数的变化 Fig. 5 Changes of relative abundance of culturable "potential" PSB in different media with generations |
在属水平上,不同培养基中各属微生物的富集规律随传代次数的变化趋势差异较大(图 5d-图 5s)。Azospirillum、Cupriavidus、Variovorax和Rhizobium在固体IPM中富集率明显高于其他三种培养基,且随传代次数增加而增加,但在其他三种培养基中变化较小。Clostridium_sensu_stricto_1、Clostridium_sensu_stricto_5、Ralstonia、Microvirga在液体IPM中富集率明显高于其他三种培养基。Pseudarthrobacter、Methylobacterium和Massilia在固体IPM传代培养中,富集率随传代次数增加而不断降低;在固体OPM传代培养中,富集率亦表现出下降趋势,但变化范围较小;在其他两种液体培养基中富集率较低。Bacillus和Burkholderia富集率在固体OPM明显高于固体IPM,且在固体OPM中随传代次数增加而增加,在固体IPM中变化较小。Paenibacillus富集率在两种液体培养基中高于其他两种固体培养基,且在液体OPM中富集率最高。Chelatococcus只在液体OPM而不是IPM上具生长优势。Pseudomonas在固体培养基中的富集现象明显强于液体培养基;在IPM中富集规律一致,富集率随传代次数增加而增加。
3 讨论针对551个解磷菌株分析发现Proteobacteria、Firmicutes和Actinobacteriota是自然界最为丰富的解磷菌门[7],与本研究结果高度一致,证明在本实验条件下对土壤解磷菌群落进行了有效富集,为可培养解磷菌多样性评估提供了保障。将所有三代培养基中可培养解磷菌综合分析表明,在属水平下,土壤可培养“潜在”解磷菌仅占土壤总细菌群落11.2%,其中83属已被报道具有解磷生理或特征基因,占土壤已知解磷菌群落75.5%,因此,相当比例(24.5%)土壤解磷菌未被培养或被遗漏。通过改良培养基配方和改变培养策略,可能有效恢复未培养类群[13-14],有望进一步提高土壤可培养解磷菌的多样性。
富集培养过程是一个复杂的混菌培养过程,微生物之间的相互作用尚不清楚。本研究中尽管将所有可培养纯菌株的集合定义为可培养“潜在”解磷菌,但不能完全排除个别微生物为非解磷菌或者必须通过互利共生的非解磷菌。如,IPM和OPM中分别有56属和114属“潜在”解磷菌尚未在已知解磷菌数据库中被检索,这些可能是代时较长、生长缓慢的“非优势”解磷菌,亦可能是利用解磷菌溶解的磷或其他生长因子而生长的“非”解磷菌。通过优化解磷培养基和富集策略,获得这些与未知解磷菌16S rRNA基因高度同源的纯菌株将具有重要意义。
培养机成分和代次显著影响可培养解磷菌群落组成和多样性变化(图 4b)。一般解磷菌可通过分泌无机酸或有机酸溶解无机磷或分泌磷酸酶、植酸酶等溶解有机磷[15]。然而,不同解磷菌分泌酸和酶的组成和活力区别很大,导致溶磷活性和生长繁殖能力也存在巨大差异。因此,具有不同磷源偏好或生长代谢特点的土壤可培养解磷菌呈现不同富集特征,如:Azospirillum、Cupriavidus、Variovorax等在固体IPM而不是OPM上富集强烈(图 5e-图 5g),表明以上各属能够强烈溶解无机磷生长[16-17];Chelatococcus和Paenibacillus在液体OPM而不是IPM上具生长优势(图 5d,图 5m),表明其具有解有机磷的能力;Bacillus、Burkholderia、Pseudomonas和Pseudarthrobacter在IPM和OPM中均强烈富集(图 5j-图 5l,图 5o),表明以上各属同时具备溶解无机磷和有机磷的生理途径和较强竞争优势,在一些研究中已得到证实[18-19]。本研究只在属水平探究了不同解磷菌的生长特点,而相同种属或同一菌株在不同环境条件下的解磷能力及机理也可能不同[17-20],相关研究值得进一步探讨。
固体和液体培养基分别富集了不同解磷菌类群。本研究中以IPM为例,固体IPM选择性富集了Azospirillum、Cupriavidus、Variovorax、Rhizobium、Pseudarthrobacte、Methylobacterium和Massilia;而液体LPM富集了更多Clostridium_sensu_stricto_1、Clostridium_sensu_stricto_5、Ralstonia、Microvirga;Paenibacillus在液体培养基中富集程度也高于固体培养基。造成这一差异的原因可能包括氧气、营养物、琼脂、附着物等因素。固体培养基中菌落与空气充分接触,氧气浓度高(约为21%),而液体培养基中溶解氧浓度受到一定限制,但营养物浓度更加均匀。已有报道表明Paenibacillus和Clostridium具有更强适应氧气胁迫能力[21-22]。此外,固体培养基由于加入琼脂,形成了固体的平板表面,为微生物提供了附着物,有利于可培养解磷菌群落形成由胞外多聚基质联合的微生物被膜[23],进而改变解磷菌群落之间的群体感应过程,从而影响解磷菌及其共生微生物的生理生态行为。
固体培养基均对Pseudarthrobacter和Pseudomonas具有较好富集效果(Pseudarthrobacter:IPM第一代80.65%;OPM第三代39.36%;Pseudomonas:IPM第二、三代51.47%~52.23%;OPM第一、二代57.82~61.09%)。Pseudomonas是最有效的解磷菌属和植物促生菌属之一,不仅能够提高有效磷含量,还能通过产生吲哚乙酸、非共生或共生N2固定、抑制植物病原微生物、产生抗生素、合成关键酶等方式,促进植物生长和发育[24]。Pseudarthrobacter是2016年从Arthrobacter属划分出来的新属[25],在系统发育上与Arthrobacter十分接近。少量研究表明Pseudarthrobacter属菌株具有营养多功能性及多种抗胁迫特性,能够参与物质循环和有机物/污染物降解[26-29]。目前已报道的5个Pseudarthrobacter菌株基因组中均检测到phoD[28-30],但其解磷能力大小尚未可知。
Bacillus亦是土壤中优势解磷菌属,国内外已对其有较多研究,主要集中在属内解磷菌株的筛选与鉴定、已分离菌株的解磷机制及应用效果等[18-19]。本研究利用液体无机磷和固体有机磷培养基,通过连续三次传代培养获得较多Bacillus富集物(液体IPM第三代34.72%;固体OPM第三代46.97%),为Bacillus属解磷菌进一步筛选和研究提供思路。
以phoD基因为标靶的解磷菌,可作为已分离解磷菌纯菌资源库外的重要补充,是当前掌握环境解磷菌资源的重要途径。目前,FunGene数据库所包含的解磷菌中有62属在纯培养条件下被证实具有溶磷功能,其余247属仅检测到phoD基因;而已知解磷菌纯菌数据库中亦有63属未在FunGene数据库被检索。产生这一现象可能由于:(1)早期解磷菌研究主要采用传统微生物培养方法,未对phoD等相关基因进行分析,数据库不够全面。例如,早前曾报道Azospirillum纯菌株能够产生葡萄糖酸而导致Ca3(PO4)2降解[16],但未在FunGene数据库中检索到该属phoD基因。(2)除phoD外,解磷菌存在其他溶磷基因[8],如碱性磷酸酶编码基因phoA、phoX,酸性磷酸酶编码基因phoC、acpA,植酸酶编码基因phyA、phyC等,葡萄糖酸产生相关基因gcd、pqqC等,这些基因研究较少,无完善数据库可检索。例如,刘彩凤等[31]通过phoX基因检测到水库沉积物中Chelatococcus为优势解磷菌之一,但本研究中Chelatococcus phoD基因未被检索。(3)分子分析过程(DNA提取、PCR扩增、文库构建、高通量测序等),均可能引入误差,产生一些稀有序列变异体,进而被注释到已知或未知分类属单元[5]。正如本研究中检测到多数“未知解磷菌”属相对丰度 < 0.1%。
4 结论培养基类型和传代次数较大程度影响土壤可培养解磷菌多样性评估。利用四种培养基经连续三次传代培养,可培养“潜在”解磷菌在土壤土著细菌群落中的占比在门水平最高(8.62%~25.9%),而在其他分类水平上仅为3.22%~12.5%。与已知解磷菌数据库进行比较,四种培养基连续传代三次对土壤已知解磷菌属的培养比例为75.5%,表明至少约24.5%土壤已知解磷菌属未被富集或遗漏。培养基还富集到大量尚未被证实具有解磷功能或基因的未知解磷菌,可能成为未来定向分离培养的重要解磷菌资源。不同培养基和代次富集了优势解磷菌门,主要包括Proteobacteria、Actinobacteriota和Firmicutes;而在属水平下,不同培养基和代次富集了具有明显生理代谢特征差异的解磷菌属。
| [1] |
Zhu J, Li M, Whelan M. Phosphorus activators contribute to legacy phosphorus availability in agricultural soils: A review[J]. Science of the Total Environment, 2018, 612: 522-537. DOI:10.1016/j.scitotenv.2017.08.095
(  0) 0) |
| [2] |
Sarmah R, Sarma A K. Phosphate solubilizing microorganisms: A review[J]. Communications in Soil Science and Plant Analysis, 2023, 54(10): 1306-1315. DOI:10.1080/00103624.2022.2142238
(  0) 0) |
| [3] |
Alori E T, Glick B R, Babalola O O. Microbial phosphorus solubilization and its potential for use in sustainable agriculture[J]. Frontiers in Microbiology, 2017, 8: 971. DOI:10.3389/fmicb.2017.00971
(  0) 0) |
| [4] |
Wen C L, Cao W W, Tang X L, et al. High-throughput sequencing technology based research on the changes of culturable bacteria communities in soil during serial enrichment (In Chinese)[J]. Journal of Ecology and Rural Environment, 2023, 39(1): 123-135. [文昌丽, 曹伟伟, 唐雪莲, 等. 基于高通量测序的连续传代富集土壤可培养菌菌群变化规律研究[J]. 生态与农村环境学报, 2023, 39(1): 123-135.]
(  0) 0) |
| [5] |
Yang L J, Jia Z J. History and definition of the "99% unculturability paradigm" – Case analysis with nitrogen-fixing bacteria (In Chinese)[J]. Acta Microbiologica Sinica, 2021, 61(4): 903-922. [杨丽洁, 贾仲君. "99%难培养"微生物的概念与初步评价: 以固氮菌为例[J]. 微生物学报, 2021, 61(4): 903-922.]
(  0) 0) |
| [6] |
Quail M A, Kozarewa I, Smith F, et al. A large genome center's improvements to the illumina sequencing system[J]. Nature Methods, 2008, 5(12): 1005-1010. DOI:10.1038/nmeth.1270
(  0) 0) |
| [7] |
Kour D, Rana K L, Kaur T, et al. Biodiversity, current developments and potential biotechnological applications of phosphorus-solubilizing and-mobilizing microbes: A review[J]. Pedosphere, 2021, 31(1): 43-75. DOI:10.1016/S1002-0160(20)60057-1
(  0) 0) |
| [8] |
Rawat P, Das S, Shankhdhar D, et al. Phosphate-solubilizing microorganisms: Mechanism and their role in phosphate solubilization and uptake[J]. Journal of Soil Science and Plant Nutrition, 2021, 21(1): 49-68. DOI:10.1007/s42729-020-00342-7
(  0) 0) |
| [9] |
Li M, Teng Z D, Zhu J, et al. Research advances in heavy metal contaminated soil remediation by phosphate solubilizing microorganisms (In Chinese)[J]. Acta Ecologica Sinica, 2018, 38(10): 3393-3402. [李敏, 滕泽栋, 朱静, 等. 解磷微生物修复土壤重金属污染研究进展[J]. 生态学报, 2018, 38(10): 3393-3402.]
(  0) 0) |
| [10] |
Lu X M, Shang K, Liu X, et al. Diversity characteristics of organic phosphate-solubilizing bacteria in surface sediments of the Yangtze River setuary (In Chinese)[J]. Advances in Marine Science, 2019, 37(3): 495-507. DOI:10.3969/j.issn.1671-6647.2019.03.013 [鲁小曼, 尚琨, 刘欣, 等. 长江口表层沉积物中可培养有机解磷菌多样性特征[J]. 海洋科学进展, 2019, 37(3): 495-507.]
(  0) 0) |
| [11] |
Tan H, Barret M, Mooij M J, et al. Long-term phosphorus fertilisation increased the diversity of the total bacterial community and the phoD phosphorus mineraliser group in pasture soils[J]. Biology and Fertility of Soils, 2013, 49(6): 661-672. DOI:10.1007/s00374-012-0755-5
(  0) 0) |
| [12] |
Fish J A, Chai B, Wang Q, et al. Fungene: The functional gene pipeline and repository[J]. Frontiers in Microbiology, 2013, 4: 291.
(  0) 0) |
| [13] |
Lewis W H, Ettema T J G. Culturing the uncultured[J]. Nature Biotechnology, 2019, 37(11): 1278-1279. DOI:10.1038/s41587-019-0300-2
(  0) 0) |
| [14] |
Mu D S, Liang Q Y, Wang X M, et al. Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing[J]. Microbiome, 2018, 6(1): 230. DOI:10.1186/s40168-018-0613-2
(  0) 0) |
| [15] |
Ma K, Wang X C, Xie J H, et al. Research progress of phosphate-solubilizing bacteria in sediments: Distribution, phosphate-solubilizing ability, and functional genes (In Chinese)[J]. Journal of Agricultural Resources and Environment, 2023, 40(2): 280-290. [马凯, 王效昌, 谢嘉慧, 等. 沉积物解磷菌的研究进展: 分布、解磷能力及功能基因[J]. 农业资源与环境学报, 2023, 40(2): 280-290.]
(  0) 0) |
| [16] |
Rodriguez H, Gonzalez T, Goire I, et al. Gluconic acid production and phosphate solubilization by the plant growth-promoting bacterium Azospirillum spp.[J]. Naturwissenschaften, 2004, 91(11): 552-555. DOI:10.1007/s00114-004-0566-0
(  0) 0) |
| [17] |
Shi J J, Zhang L, Jiang F Y, et al. Dual functions of bacteria colonized on AM fungal hyphae–fixing N2 and solubilizing phosphate (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(5): 1289-1298. [石晶晶, 张林, 江飞焰, 等. AM真菌菌丝际细菌具有固氮解磷双重功能[J]. 土壤学报, 2021, 58(5): 1289-1298.]
(  0) 0) |
| [18] |
Hilda R G, Reynaldo F. Phosphate solubilizing bacteria and their role in plant growth promotion[J]. Biotechnology Advances, 1999, 17(4): 319-339.
(  0) 0) |
| [19] |
Jorquera M A, Crowley D E, Marschner P, et al. Identification of β-propeller phytase-encoding genes in culturable Paenibacillus and Bacillus spp. from the rhizosphere of pasture plants on volcanic soils[J]. FEMS Microbiology Ecology, 2011, 75(1): 163-172. DOI:10.1111/j.1574-6941.2010.00995.x
(  0) 0) |
| [20] |
Wang G H, Zhao Y, Zhou D R, et al. Review of phosphate - solubilizing microorganisms (In Chinese)[J]. Ecology and Environment, 2003, 12(1): 96-101. DOI:10.3969/j.issn.1674-5906.2003.01.024 [王光华, 赵英, 周德瑞, 等. 解磷菌的研究现状与展望[J]. 生态环境学报, 2003, 12(1): 96-101.]
(  0) 0) |
| [21] |
Morvan C, Folgosa F, Kint N, et al. Responses of clostridia to oxygen: From detoxification to adaptive strategies[J]. Environmental Microbiology, 2021, 23(8): 4112-4125. DOI:10.1111/1462-2920.15665
(  0) 0) |
| [22] |
Szaniawski M A, Spivak A M. Recurrent Paenibacillus infection[J]. Oxford Medical Case Reports, 2019, 2019(5): omz034.
(  0) 0) |
| [23] |
McDougald D, Rice S A, Barraud N, et al. Should we stay or should we go: Mechanisms and ecological consequences for biofilm dispersal[J]. Nature Reviews Microbiology, 2012, 10(1): 39-50. DOI:10.1038/nrmicro2695
(  0) 0) |
| [24] |
Zaidi A, Khan M, Ahemad M, et al. Plant growth promotion by phosphate solubilizing bacteria[J]. Acta Microbiologica et Immunologica Hungarica, 2009, 56: 263-284. DOI:10.1556/AMicr.56.2009.3.6
(  0) 0) |
| [25] |
Busse H J. Review of the taxonomy of the genus Arthrobacter, emendation of the genus Arthrobacter sensu lato, proposal to reclassify selected species of the genus Arthrobacter in the novel genera Glutamicibacter gen. Nov., Paeniglutamicibacter gen. Nov., Pseudoglutamicibacter gen. Nov., Paenarthrobacter gen. Nov. And Pseudarthrobacter gen. Nov., and emended description of Arthrobacter roseus[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 9-37. DOI:10.1099/ijsem.0.000702
(  0) 0) |
| [26] |
Chen F, Chen Y, Chen C, et al. High-efficiency degradation of phthalic acid esters (PAEs) by Pseudarthrobacter defluvii e5: Performance, degradative pathway, and key genes[J]. Science of the Total Environment, 2021, 794: 148719. DOI:10.1016/j.scitotenv.2021.148719
(  0) 0) |
| [27] |
Han S R, Kim B, Jang J H, et al. Complete genome sequence of Arthrobacter sp. Pamc25564 and its comparative genome analysis for elucidating the role of cazymes in cold adaptation[J]. BMC Genomics, 2021, 22(1): 403. DOI:10.1186/s12864-021-07734-8
(  0) 0) |
| [28] |
Kallimanis A, Labutti K M, Lapidus A, et al. Complete genome sequence of Arthrobacter phenanthrenivorans type strain (Sphe3)[J]. Standards in Genomic Sciences, 2011, 4(2): 123-130. DOI:10.4056/sigs.1393494
(  0) 0) |
| [29] |
Zhang X J, Zhang G Z, Yang H T. Genomics basis of Arthrobacter spp. environmental adaptability-A review (In Chinese)[J]. Acta Microbiologica Sinica, 2016, 56(4): 570-577. [张新建, 张广志, 杨合同. 节杆菌环境适应性的基因组学研究进展[J]. 微生物学报, 2016, 56(4): 570-577.]
(  0) 0) |
| [30] |
Park M K, Park Y J, Kim M C, et al. Complete genome sequence of Pseudarthrobacter sp. Nibrbac000502771 isolated from shooting range soil in Republic of Korea[J]. Korean Journal of Microbiology, 2019, 55(4): 419-421.
(  0) 0) |
| [31] |
Liu C F, She C X, Su Y P, et al. Effects of micro-nanometer aeration on bacterial and phosphorus functional bacterial communities in reservoir sediments (In Chinese)[J]. Chinese Journal of Environmental Engineering, 2022, 16(4): 1340-1352. [刘彩凤, 佘晨兴, 苏玉萍, 等. 微纳米曝气对水库沉积物细菌及磷功能菌群落的影响[J]. 环境工程学报, 2022, 16(4): 1340-1352.]
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


