2. 华南农业大学农学院, 广州 510642
2. College of Agriculture, South China Agricultural University, Guangzhou 510642, China
土壤微生态中的微生物群落(细菌、真菌、古菌和病毒)在生物地球化学循环,农业可持续化和植物生产等方面发挥着重要作用。以往研究多集中在不同生态系统中细菌和真菌的群落组成和多样性上[1],而土壤中病毒对生态系统影响的研究较少,尤其是对深层陆地生物圈的病毒知之甚少。土壤中含有极高丰度的病毒,预计其数量约为4.88×1030个VLPs,超过海洋中病毒数量(1.29×1030个VLPs)[2],土壤病毒组可能是地球上最大的遗传多样性库,并且在土壤生态系统中具有不可代替的作用[3-4]。土壤病毒中,绝大部分由噬菌体组成,噬菌体与细菌之间的互作关系,是土壤病毒研究的重点[5]。
土壤中的病毒,尤其是噬菌体可以携带辅助代谢基因(Auxiliary Metabolic Genes,AMGs),进而影响宿主生物被膜的形成[6-7]或参与宿主碳、硫、磷代谢[4,8-10]。其中碳循环相关基因[11]是目前的研究热点,韩丽丽课题组[12]在土壤病毒组中挖掘出137个碳水化合物活性酶(Carbohydrate active enzymes,CAZymes)基因,发现病毒能够通过携带CAZymes来影响生态系统的碳利用。目前AMGs已在多个土壤生态系统中被鉴定出[13],并认为在生物地球化学循环中起到重要的作用,但大多数AMGs在塑造土壤细菌的生态和进化轨迹方面的作用仍不清楚。
目前多数土壤病毒生态学的研究侧重于在自然生态系统中进行,如沙漠和永冻土等,而对农田等受到人工干预的土壤生态系统研究较少[14]。农业生态系统不仅具有经济效益,为人类提供了大量食物,而且还通过无机氮固定和甲烷合成等过程影响全球生态系统。其中,稻田系统是最重要的农业系统,但对稻田系统中病毒群及病毒-细菌互作的相关研究仍然较少[15-16]。与水生和其他陆地生态系统不同,稻田生态系统处于一般干湿循环的过程中[17]。在这个干湿循环的过程中,水稻土具有特殊的氧化还原过程,影响养分循环利用以及微生物介导的土壤有机质(Soil organic matter,SOM)周转[18],同时也会影响土壤团聚体之间的连通性,提高土壤中微生物群的接触机会。最近的研究表明,土壤含水量和SOM的变化会影响病毒-细菌的相互作用,进而影响土壤中病毒和细菌群落的组成[19-20]。
鉴于稻田系统的复杂性和重要性,亟需开发出用于该系统中病毒-细菌互作的研究方法和相关数据库。本研究从公共数据库中收集稻田土壤相关宏基因组数据,挖掘并注释其中的病毒相关序列和基因,构建稻田土壤病毒数据库,为后续水稻-微生物互作等研究奠定基础。
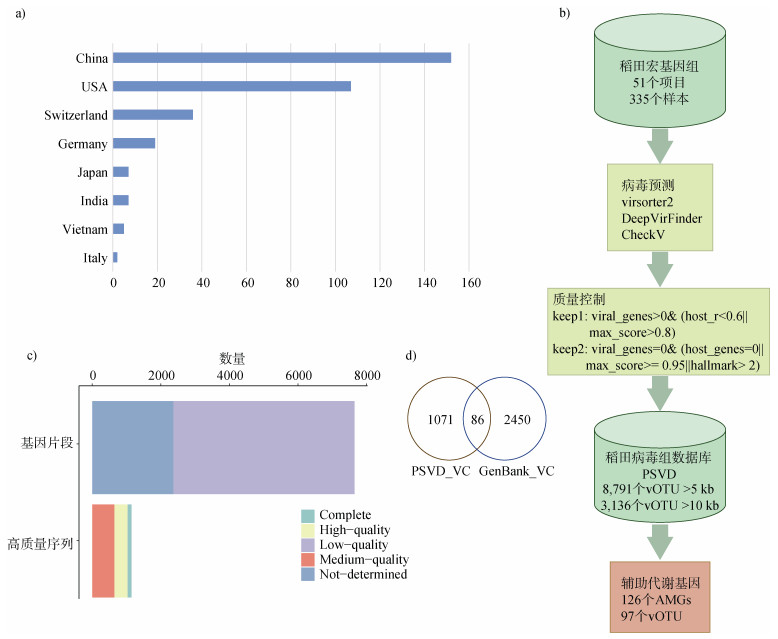
1 材料与方法 1.1 稻田土壤宏基因组数据下载及整理在PubMed上使用关键词“rice soils metagenomic”检索近10年内的文献,并且在欧洲核苷酸档案数据库(European Nucleotide Archive,ENA)中以分类号为1672641 rice paddy metagenome、939928 rhizosphere metagenome和410658 soil metagenome下载所有项目的信息表,通过逐一手动筛选,获得53个水稻宏基因组项目,并收集项目内所有样品的SRA号(合计344个),之后使用prefetch进行下载,使用fasterq-dump进行文件解析,获得fastq文件。最终经过数据质控和病毒序列筛选后,仅51个项目、总计335个样本符合要求。
1.2 病毒序列识别使用MEGAHIT[21]进行序列组装,参数设置为--presets meta-large --k-min 27。对获得的样品contig序列采用两种病毒序列识别软件进行同步筛选,分别为DeepVirFinder[22](参数为p value小于0.05)和VirSorter2[23](参数为--include-groups dsDNAphage,RNA,ssDNA--min-length 1000--min-score 0.5)。之后汇总两个软件的病毒序列预测结果,使用ChcekV[24]对预测结果进行剪切去除细菌污染。之后参考Sullivan Lab的病毒筛选条件(doi:dx.doi.org/10.17504/protocols.io.bwm5pc86)并进行调整,具体筛选条件如下:
保留条件1:viral_genes大于0 and(host_r小于0.6 or max_score大于0.8)
保留条件2:viral_genes等于0 and(host_genes等于0 or max_score大于或等于0.95 or hallmark大于2)
丢弃条件1:viral_genes等于0 and host_genes大于1
丢弃条件2:viral_genes等于0 and host_genes等于1 and contig_length小于10 000
丢弃条件3:viral_genes大于0 and(host_r大于0.6 or max_score小于0.8)
其中,viral_genes,host_genes和host_r由CheckV提供;max_score为DeepVirFinder或Virsorter2的病毒性得分;hallmark为Virsorter2判定序列所含有病毒标记的基因数量。
保留所有满足保留条件的序列和不满足丢弃条件的序列。最后对序列集合进行去重,使用CheckV自带的aniclust.py脚本进行去(参数为--min_ani 95--min_tcov 85),获得非冗余病毒数据集(即,vOTUs)。
使用Prodigal从非冗余病毒数据集中注释开放阅读框(Open Reading Frames,ORFs),参数为:-p mata -g 11 -c,并对ORFs基于eggNOG-mapper进行基因注释,具体参数为:--go_evidence non-electronic--cpu 20 --override。最后使用R进行可视化。
病毒序列在eggNOG注释结果中存在溶原相关基因,如:整合酶(integrase)、转座酶(transposase)、重组酶(recombinase)、细胞溶素原(lysogen)和切除酶(excisionase),则被认定为溶原病毒。
数据库存储在https://github.com/lzk98/PSVD上。
1.3 病毒序列分类和宿主预测病毒聚类分析使用vConTACT2[25]对非冗余病毒序列和GenBank上所有病毒序列(2023年4月)进行聚类,获得聚类病毒簇(viral cluster,VC)信息。病毒分类使用PhaGCN2[26]进行,使用其默认数据库(2022年8月)。病毒序列比对使用BLASTn进行,对比NCBI的核酸数据库(Nucleotide Sequence Database,NT)中的病毒子数据库(txid:10239,下载时间为2022年11月)。末端酶大亚基(large terminase subunit,TerL)系统发育树的构建分别基于Terminase_3(PF04466)、GpA_ATPase(PF05876)、Terminase_6N(PF03237)和TerL_ATPase(PF03354)四个结构域进行构建。简而言之,经过MAFFT多序列比对[27],trimAl序列对齐剪切[28],FastTree构建系统发育树[29],最后使用TVBOT[30]进行美化。
宿主信息使用RefSeq-spacers数据库与病毒序列进行匹配预测,其中RefSeq-spacers数据库构建参考前人的构建方法[31],简而言之,下载RefSeq中细菌和古菌的基因组数据集合(2022年06月),使用CRT v1.2和PILER v1.06识别CRISPR阵列,再汇总二者的结果。使用BLASTn进行比对,参数为blastn-short -word_size 18 -dust no -evalue 1e-5,过滤“bit score大于45”的比对结果[31-32]。
1.4 辅助代谢基因的鉴定辅助代谢基因注释主要基于DRAM[33]软件进行。简而言之,首先通过VirSorter2,参数为--prep-for-dramv,运行所有已识别的vOTU序列以生成affi-contigs.tab文件,然后使用默认数据库进行注释。最后根据DRAM-v注释提取出的结果,选择具有辅助评分小于4和基因描述的推定AMGs。遵循ter Horst等[34]报告的方法,保守起见,对注释输出进行了过滤,以提高AMG鉴定的可信度,主要是去除与核苷酸代谢、有机氮、糖基转移酶和核糖体蛋白相关的基因类别。此外,如果推定的AMG位于两个病毒标志基因或病毒样基因之间或附近,则该基因在后续分析中被认为是高置信度的病毒AMG[35]。每个编码AMG的重叠群的基因组图使用chiplot(https://www.chiplot.online/)基于DRAM-v的注释结果完成可视化。
AMG序列的进化溯源使用BLASTp检索NR数据库,使用MAFFT进行多序列比对[27],FastTree构建系统发育树[29],最后使用TVBOT[30]进行美化,通过系统发育树了解该基因的进化情况。AMG三维结构预测使用Phyre2在线网站进行。
2 结果与讨论 2.1 稻田土壤病毒数据库构建收集到的51个稻田宏基因组相关项目,合计335个样本,其中来自中国的测序数据最多,占45.37%,其次为美国,占31.94%(图 1a)。按图 1b所示的病毒组分析流程进行稻田土壤病毒数据库的构建与分析。具体而言,通过VirSorter2、DeepVirFinder和CheckV进行病毒序列挖掘,获得54万条病毒序列,再通过自定义的筛选标准过滤后,获得约37万条病毒序列,去冗余处理,余下约29万条非冗余的病毒序列,其中序列长度大于等于5 kb和10 kb的数量分别为8 791和3 136条。
|
a)样本来源的地理分布;b)数据库构建流程;c)序列完整性分布情况;d)稻田土壤病毒数据库与已知病毒的vConTACT2聚类情况。 a)geographical distribution of sample sources; b)database construction process; c)sequence integrity distribution; d)vConTACT2 clustering of the PSVD with known viruses. 图 1 稻田土壤病毒数据库构建 Fig. 1 Construction of the Paddy Soil Viruses Database |
在大于5 kb序列的基础上构建稻田土壤病毒数据库(Paddy Soil Viruses Database,PSVD),该数据库由8 791个病毒vOTU构成(合计138 Mbp),并且由168 940个ORFs组成。vOTU序列长度主要分布在5 kb~50 kb之间(96.05%)。进一步统计稻田土壤病毒数据库中病毒序列完整性情况,被认为具有中等质量完整、高等质量完整和完全完整的分别有642、390和113条序列的(图 1c),合计占比13.02%(1 145/8 791),在这些具有较高序列完整性(中等质量以上)的序列中,多数长度依旧分布在5 kb~50 kb之间(80.70%),其中有43条vOTU可能为巨噬菌体(序列长度大于200 kb)。通过vConTACT2对病毒群聚类发现,稻田病毒中存在大量与已知病毒不同的VC簇(图 1d),仅86个VC簇与已知的病毒相同,92.57%(1 071/1 157)的稻田土壤病毒VC簇是独有的。因此,PSVB很好弥补了当前土壤病毒库的空缺。
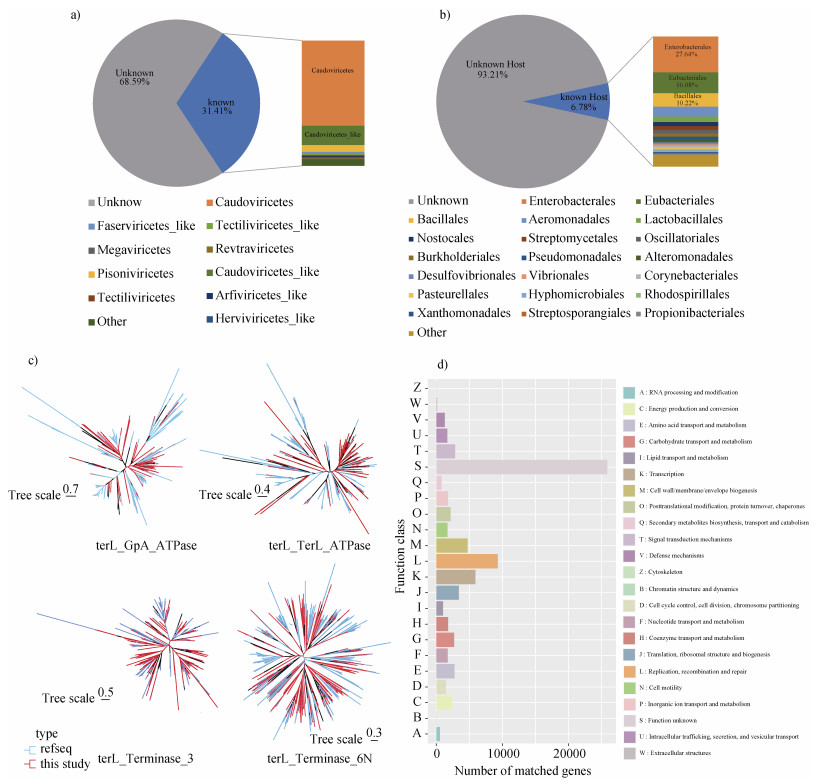
2.2 稻田病毒的未知性和多样性在对稻田土壤病毒数据库中的序列进行分类地位预测的过程中,发现只有部分病毒序列(31.41%)能够预测出物种分类地位信息,包括了76个已知的病毒科,分属于20个病毒纲,其中纲水平分类主要为有尾病毒纲(Caudoviricetes)占全部序列的83.45%(2 304/2 761),而科水平分类又以Peduoviridae科和螺旋病毒科(Herelleviridae)为主(图 2a)。Caudoviricetes基本由噬菌体组成,说明噬菌体是稻田土壤病毒圈中重要的成员。同时,还预测出56个与己知病毒群具有一定关系的新病毒类群,包括615个病毒vOTU,该类群使用_like后缀标识(图 2a)。接着,将序列与NCBI的NT数据库进行比对,只有部分序列(255/8 791)能够比对上,并且所比对上的序列多数是未经验证的宏基因组组装病毒序列。最后,基于TerL的系统发育树(图 2c)和大部分预测的病毒ORFs未能得到注释的结果(图 2d),进一步说明稻田土壤病毒群在很大程度上是未知的。进一步研究稻田土壤病毒群是否与其他环境病毒一样,可能通过感染微生物来驱动生物地球化学循环,进而影响生态系统。通过自主构建的RefSeq-spacers数据库进行宿主预测分析表明,8 791个vOTU中的597个病毒序列预测出假定的宿主(图 2b)。预测出的宿主主要分布在11个细菌门,其中变形菌门(Proteobacteria)的肠杆菌目(Enterobacterales)和厚壁菌门(Bacillota)的真杆菌目(Eubacteriales)与芽孢杆菌目(Bacillales)的数量最多,该结论与稻田土壤系统中细菌物种组成相类似[36-37]。同时说明,稻田土壤中存在许多未被表征的病毒,其基因组和侵染特性均有待进一步挖掘。如此高比例的未知病毒序列和基因,进一步阐明了稻田土壤病毒组的多样性和未知性。
|
a)分类预测;b)宿主预测;c)末端酶结构域系统发育树;d)基因注释。 a) classification prediction; b) host prediction; c) terminase phylogenetic tree; d) gene annotation. 图 2 PSVD中病毒序列 Fig. 2 Virus sequences in PSVD |
土壤中的许多病毒编码辅助代谢基因(AMGs),参与到宿主代谢过程中,从而影响生物地球化学循环并促进宿主对环境的适应。本研究评估了稻田土壤病毒AMGs对生物地球化学循环的影响,以加深对稻田土壤病毒的生态位特化和生态功能的了解。
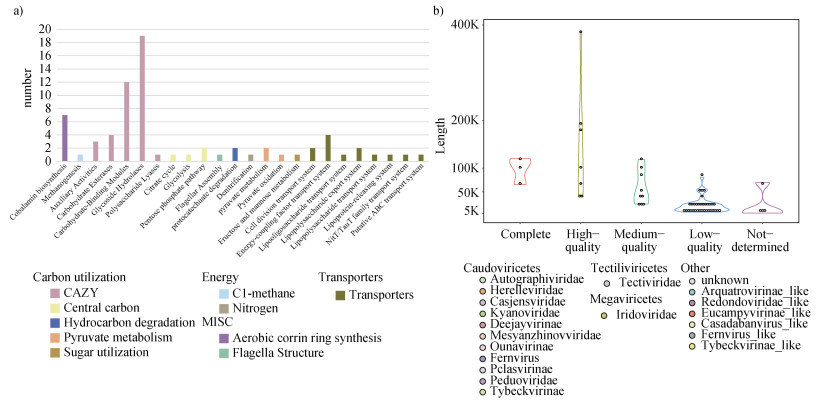
在手动筛选前,DRAM-v预测了561个假定的AMGs,随后参考Pratama等[35]的方法和标准,手动筛选了DRAM-v软件的注释结果,共获得126个AMGs,分布在97个vOTU序列上,参与多种代谢途径,如碳代谢、氮代谢、光合作用和生物素合成等。其中碳源利用模块数量最多,达到73.02%(92/126)。根据预测的AMGs周围是否具有病毒基因再进行筛选,共获得72个高置信度的AMGs,仅占全部AMGs的半数(57.14%,72/126),其代谢模块的数量分布情况与前者相似(图 3a)。高置信度的AMGs分布在57个vOTU上,其中有尾噬菌体纲的病毒占据多数,其余为不具有尾部结构的Tectiliviricetes纲dsDNA噬菌体和具有较大基因组的Megaviricetes纲巨型病毒(图 3b)。对病毒的宿主进行分析,发现其属于Shewanella、Methylomonas、Klebsiella、Geobacillus、Enterobacter、Clostridium、Citrobacter和Bacillus属。在所有高置信度的AMGs中,CAZymes模块数量最多,为39个(表 1),其中又以糖苷水解酶(Glycoside Hydrolases,GH)和碳水化合物结合模块(Carbohydrate-Binding Modules,CBM)模块的数量最多,分别为19和12个,合计占比79.45%。
|
图 3 稻田土壤病毒中辅助代谢基因的分布情况及其序列表征 Fig. 3 Distribution of auxiliary metabolic genes and their sequence characterization in viruses of paddy soils |
|
|
表 1 碳水化合物活性酶基因统计表 Table 1 Statistics of carbohydrate-active enzyme genes |
病毒AMGs还可能参与了核心碳循环,如糖酵解、丙酮酸代谢、戊糖途径和三羧酸循环,能够影响稻田系统中细菌的能量代谢。值得注意的是,有两个AMGs(vOTU303-cat_1_21和vOTU590-cat_2_12)注释为4-羧基粘内酯脱羧酶(4-carboxymuconolactone decarboxylase),能使苯甲酸盐降解,参与芳香族化合物的降解,该基因的存在能够提高宿主细菌对被石油污染土壤的耐受能力。同时vOTU551-cat_2_17编码的AMGs被注释为砷酸还原酶(arsenate reductase),能够提高稻田系统对重金属污染的修复能力,虽然该AMGs并非高置信度,但可以说明其具有一定潜力。还有些稻田土壤的病毒AMGs(vOTU325-cat_1_25)通过编码乙酰辅酶A合成酶(acetyl-CoA synthetase),参与包括甲烷合成、糖酵解和丙酮酸代谢在内的多种代谢途径。此外,还有一些AMGs编码编码亚硝酸还原酶和钴硫合酶,分别参与了亚硝酸盐降解和钴胺素(维生素B12)生物合成。
病毒携带的AMGs说明了稻田土壤病毒是参与生物地球化学循环的重要组成部分,并且对环境土壤改善也起到一定的调节作用。
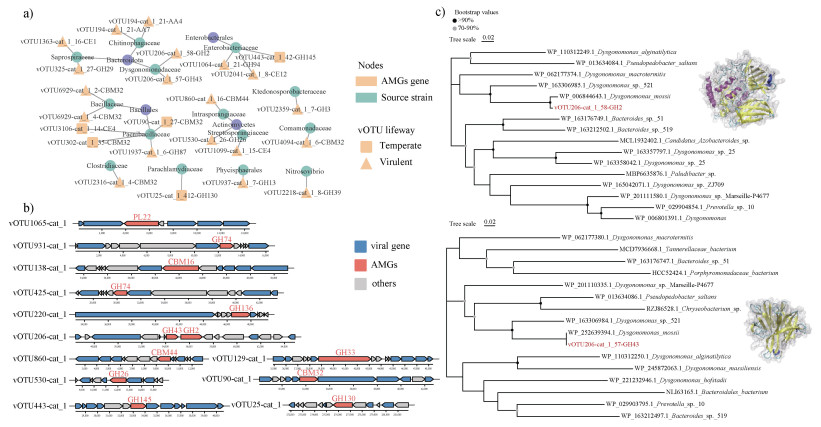
2.4 碳水化合物活性酶基因的多样性稻田病毒群中含有丰富的CAZymes基因,而病毒AMGs通常通过水平基因转移从宿主中获得[38]。在本次预测出的CAZymes基因中有13个来源于具有较高完整性病毒序列(病毒序列完整性大于50%,图 4a),分别属于糖苷水解酶、多糖裂解酶和碳水化合物结合模块。其中PL22蛋白与果胶酶同源,能使果胶骨架裂解,而GH74、GH43、GH2、GH26和GH130蛋白与纤维素酶同源,能够水解半纤维。这些病毒碳水化合物代谢酶可能有助于稻田系统中多糖的降解。此外还有一种病毒AMGs编码GH13,参与淀粉水解过程。
|
图 4 稻田土壤病毒中碳水化合物活性酶基因中部分高置信基因簇(a)、基因来源情况(b)及vOTU206序列中GH43和GH2系统发育树(c) Fig. 4 Partial high-confidence gene clusters of carbohydrate-active enzyme genes in paddy soil virus (a), gene origin status (b), and phylogenetic tree of GH43 and GH2 in vOTU206 sequences (c) |
为预测这些CAZymes基因(表 1)的潜在来源,对上述CAZymes基因进行BLASTp比对和系统发育分析,其中有24个高置信度CAZymes基因能够预测出假定的基因来源宿主(图 4b),其余AMGs基因在系统发育树构建过程中,与多种宿主来源的基因混杂在一起,不能明确基因的宿主来源情况。
稻田病毒群中的CAZymes基因主要来源于厚壁菌门的芽孢杆菌目(Bacillales)、拟杆菌门(Bacteroidota)、假单胞菌门的肠杆菌目(Enterobacterales)和放线菌门的放线菌纲(Actinomycetes)中,该结果与前人在稻田中细菌群落丰度描述的结果一致[16],该研究发现稻田系统中,变形杆菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)和拟杆菌门(Bacteroidetes)是五个丰度最高的门,这意味着病毒CAZymes基因可能在稻田群落中广泛存在。
在能够预测出宿主的CAZymes基因中,CBM32表现出较高的多样性,其分布在伯克氏菌目的Ramlibacter和芽孢杆菌目的Paenibacillus,Lysinibacillus和Clostridium。值得注意的是,有5个AMGs基因(包括2个CBM32)来源于假定的溶原病毒(噬菌体)序列(图 4b中椭圆矩形节点),推测该类基因的存在可能参与到细菌代谢过程中,能够增强宿主细菌的碳水化合物合成功能。
在具有较高序列完整性的病毒中,序列vOTU206含有两个高置信度的糖苷水解酶基因(图 4a),分别是糖苷水解酶家族43(GH43)和β-半乳糖苷酶(GH2)。GH43中的大多数酶利用芳基糖苷作为底物,同时显示α-L-阿拉伯呋喃糖苷酶和β-D-木糖苷酶活性,而GH2的主要天然底物是乳糖,可将乳糖转化为半乳糖和葡萄糖(水解)或异乳糖(半乳糖基转移)。通过系统发育分析发现(图 4c),这两个糖苷水解酶基因均来自拟杆菌门(Bacteroidota),而根据蛋白质三维结构模型,预测其存在催化复合糖的水解的能力。
此外,稻田病毒群中还含有水解碳水化合物的酯类(CE)相关的酶(表 1),如多糖脱乙酰基酶(polysaccharide deacetylase,CE4)和α/β水解酶(CE1)等。其中,携带多糖脱乙酰基酶的病毒具有两种不同的宿主来源,分别是放线菌和芽孢杆菌,进一步说明了稻田病毒群中CAZymes的来源多样性。
目前研究发现,病毒AMGs与土壤中核心碳代谢和土壤有机质降解有密切的关系,其具有与“自上而下”的病毒裂解作用相反地调控土壤系统碳循环的能力,即病毒通过AMGs基因加强或赋予宿主菌的碳代谢功能,也被称为“自下而上”的调控方式[39]。AMGs参与代谢多种具有复杂特性的碳水化合物[20],其中检测最广泛的是CAZymes基因。目前对红树林系统中病毒组的研究中发现,编码CAZymes基因的病毒可能有助于红树林沉积物中多糖的降解[40]。而CAZymes基因不仅仅在宏基因组数据中被表征,也出现在噬菌体分离株中,如枯草芽孢噬菌体SP32,φNIT10和SPG1中均含有GH32,该基因是由水平基因转移从乳酸芽孢杆菌(Sporolactobacillus laevolacticus)原始祖先宿主中获得,其具有左旋果糖转移酶的功能[41]。
本研究在稻田土壤中发现多种CAZymes基因,包括了低聚糖氧化酶、乙酰木聚糖酯酶、果胶乙酰酯酶、β-甘露聚糖酶、α-淀粉酶、鼠李糖水解酶、β-半乳糖苷酶、纤维素酶和β-葡萄糖苷酶等,可能参与到纤维素、乳糖、鼠李糖、淀粉和葡萄糖等多种糖类物质的代谢过程中。这意味着含有CAZymes基因的病毒群体可能具有在稻田土壤进行有机碳分解的潜在作用,值得注意的是,尽管病毒AMGs在不同的土壤环境中共享,但CAZymes基因会在局部尺度上表现出生态位划分。例如,因土壤中碳源底物构成的差异,导致了红树林土壤中具有参与海藻糖降解的GH37和GT20,而在稻田土壤系统中并不存在[40,42],说明不同土壤系统中病毒参与全球碳循环过程也可能存在差异。虽然稻田土壤病毒,特别是噬菌体,携带大量碳水化合物的催化基因,但目前仅有少部分的病毒CAZymes基因被验证参与碳代谢[41,43]。总之,关于稻田土壤病毒AMGs如何参与到土壤碳代谢过程还需要进一步的研究。
3 结论本研究使用ENA公开数据,构建了迄今为止最大的稻田土壤病毒数据库(PSVD),该数据库由8 791个病毒vOTU组成,有31.41%的序列能够预测出物种分类地位信息,这些病毒序列分属于76个已知的病毒科,其中1 145个vOTU是具有较高完整性的病毒序列。与已知的病毒序列比对发现,稻田土壤中存在很多未被表征的病毒群系,该数据库的发布将极大的填补稻田病毒组研究的不足,并为之后稻田病毒组的相关研究奠定基础。根据对稻田土壤病毒中所携带的AMGs,尤其是CAZymes进行分析,该基因主要来源于稻田土壤中常见的细菌病毒类型,说明含有该类型基因的病毒(噬菌体)可能广泛存在于稻田群落中,可能对该系统中的碳循环起到潜在的作用。
| [1] |
Sun A Q, Jiao X Y, Chen Q L, et al. Microbial communities in crop phyllosphere and root endosphere are more resistant than soil microbiota to fertilization[J]. Soil Biology & Biochemistry, 2021, 153: 108113.
(  0) 0) |
| [2] |
Cobián Güemes A G, Youle M, Cantú V A, et al. Viruses as winners in the game of life[J]. Annual Review of Virology, 2016, 3(1): 197-214. DOI:10.1146/annurev-virology-100114-054952
(  0) 0) |
| [3] |
Han L L, Cao M M, Bi L, et al. Research advances and application prospect of soil viruses (In Chinese)[J]. Science & Technology Review, 2022, 40(3): 75-86. [韩丽丽, 曹苗苗, 毕丽, 等. 土壤病毒的研究进展与应用前景[J]. 科技导报, 2022, 40(3): 75-86.]
(  0) 0) |
| [4] |
Wang G H, Liu J J, Zhu D, et al. A review of researches on viruses in soil – Advancement and challenges (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(6): 1319-1332. [王光华, 刘俊杰, 朱冬, 等. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319-1332.]
(  0) 0) |
| [5] |
Gao R C, Hu M, Li F B, et al. Research progress and ecological function of phages in soil (In Chinese)[J]. Journal of South China Agricultural University, 2022, 43(5): 1-11. [高瑞川, 胡敏, 李芳柏, 等. 土壤噬菌体的研究进展及生态功能解析[J]. 华南农业大学学报, 2022, 43(5): 1-11.]
(  0) 0) |
| [6] |
Erez Z, Steinberger-Levy I, Shamir M, et al. Communication between viruses guides lysis-lysogeny decisions[J]. Nature, 2017, 541(7638): 488-493. DOI:10.1038/nature21049
(  0) 0) |
| [7] |
Yang M Y, Chen H F, Huang Q L, et al. Characterization of the novel phage vB_VpaP_FE11 and its potential role in controlling Vibrio parahaemolyticus biofilms[J]. Viruses, 2022, 14(2): 264. DOI:10.3390/v14020264
(  0) 0) |
| [8] |
Roux S, Brum J R, Dutilh B E, et al. Ecogenomics and potential biogeochemical impacts of globally abundant ocean viruses[J]. Nature, 2016, 537(7622): 689-693. DOI:10.1038/nature19366
(  0) 0) |
| [9] |
Zhan Y C, Chen F. Bacteriophages that infect marine roseobacters: Genomics and ecology[J]. Environmental Microbiology, 2019, 21(6): 1885-1895. DOI:10.1111/1462-2920.14504
(  0) 0) |
| [10] |
Han L, Yu D, Bi L, et al. Distribution of soil viruses across China and their potential role in phosphorous metabolism[J]. Environmental Microbiome, 2022, 17. DOI:10.1186/s40793-022-00401-9
(  0) 0) |
| [11] |
Bi L, Yu D T, Du S, et al. Diversity and potential biogeochemical impacts of viruses in bulk and rhizosphere soils[J]. Environmental Microbiology, 2021, 23(2): 588-599. DOI:10.1111/1462-2920.15010
(  0) 0) |
| [12] |
Cao M M, Liu S Y, Bi L, et al. Distribution characteristics of soil viruses under different precipitation gradients on the Qinghai-Tibet plateau[J]. Frontiers in Microbiology, 2022, 13: 848305. DOI:10.3389/fmicb.2022.848305
(  0) 0) |
| [13] |
Santos-Medellin C, Zinke L A, ter Horst A M, et al. Viromes outperform total metagenomes in revealing the spatiotemporal patterns of agricultural soil viral communities[J]. The ISME Journal, 2021, 15(7): 1956-1970. DOI:10.1038/s41396-021-00897-y
(  0) 0) |
| [14] |
Ruan C J, Xiong G Z, Niu X Y, et al. Preliminary analysis of the virus metagenome in a farmland black soil of northeast China (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(5): 1447-1456. [阮楚晋, 熊广州, 牛欣尧, 等. 一个东北农田黑土样品宏病毒组的初步分析[J]. 土壤学报, 2022, 59(5): 1447-1456.]
(  0) 0) |
| [15] |
Li Y T, Sun H, Yang W C, et al. Dynamics of bacterial and viral communities in paddy soil with irrigation and urea application[J]. Viruses, 2019, 11(4): 347. DOI:10.3390/v11040347
(  0) 0) |
| [16] |
Wang Y, Liu Y, Wu Y, et al. Heterogeneity of soil bacterial and bacteriophage communities in three rice agroecosystems and potential impacts of bacteriophage on nutrient cycling[J]. Environmental Microbiome, 2022, 17. DOI:10.1186/s40793-022-00410-8
(  0) 0) |
| [17] |
Yu G Y. Discussion on water-saving irrigation cultivation technology of rice in Northern China (In Chinese)[J]. Agricultural Machinery and Agronomy, 2023, 54(6): 75-77. DOI:10.3969/j.issn.1672-3872.2023.06.022 [于广洋. 浅谈北方水稻节水灌溉栽培技术[J]. 南方农机, 2023, 54(6): 75-77.]
(  0) 0) |
| [18] |
Wei L, Ge T D, Zhu Z K, et al. Comparing carbon and nitrogen stocks in paddy and upland soils: Accumulation, stabilization mechanisms, and environmental drivers[J]. Geoderma, 2021, 398: 115121. DOI:10.1016/j.geoderma.2021.115121
(  0) 0) |
| [19] |
Williamson K E, Radosevich M, Wommack K E. Abundance and diversity of viruses in six Delaware soils[J]. Applied and Environmental Microbiology, 2005, 71(6): 3119-3125. DOI:10.1128/AEM.71.6.3119-3125.2005
(  0) 0) |
| [20] |
Wu R, Davison M R, Nelson W C, et al. DNA viral diversity, abundance, and functional potential vary across grassland soils with a range of historical moisture regimes[J]. mBio, 2021, 12(6): e259521.
(  0) 0) |
| [21] |
Li D H, Liu C M, Luo R B, et al. MEGAHIT: An ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J]. Bioinformatics, 2015, 31(10): 1674-1676. DOI:10.1093/bioinformatics/btv033
(  0) 0) |
| [22] |
Ren J, Song K, Deng C, et al. Identifying viruses from metagenomic data using deep learning[J]. Quantitative Biology, 2020, 8(1): 64-77. DOI:10.1007/s40484-019-0187-4
(  0) 0) |
| [23] |
Guo J R, Bolduc B, Zayed A A, et al. VirSorter2: A multi-classifier, expert-guided approach to detect diverse DNA and RNA viruses[J]. Microbiome, 2021, 9(1): 37. DOI:10.1186/s40168-020-00990-y
(  0) 0) |
| [24] |
Nayfach S, Camargo A P, Schulz F, et al. CheckV assesses the quality and completeness of metagenome-assembled viral genomes[J]. Nature Biotechnology, 2021, 39(5): 578-585. DOI:10.1038/s41587-020-00774-7
(  0) 0) |
| [25] |
Bin Jang H, Bolduc B, Zablocki O, et al. Taxonomic assignment of uncultivated prokaryotic virus genomes is enabled by gene-sharing networks[J]. Nature Biotechnology, 2019, 37(6): 632-639. DOI:10.1038/s41587-019-0100-8
(  0) 0) |
| [26] |
Jiang J Z, Yuan W G, Shang J Y, et al. Virus classification for viral genomic fragments using PhaGCN2[J]. Briefings in Bioinformatics, 2023, 24(1): bbac505. DOI:10.1093/bib/bbac505
(  0) 0) |
| [27] |
Nakamura T, Yamada K D, Tomii K, et al. Parallelization of MAFFT for large-scale multiple sequence alignments[J]. Bioinformatics, 2018, 34(14): 2490-2492. DOI:10.1093/bioinformatics/bty121
(  0) 0) |
| [28] |
Capella-Gutiérrez S, Silla-Martínez J M, Gabaldón T. trimAl: A tool for automated alignment trimming in large-scale phylogenetic analyses[J]. Bioinformatics, 2009, 25(15): 1972-1973. DOI:10.1093/bioinformatics/btp348
(  0) 0) |
| [29] |
Price M N, Dehal P S, Arkin A P. FastTree: Computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular Biology and Evolution, 2009, 26(7): 1641-1650. DOI:10.1093/molbev/msp077
(  0) 0) |
| [30] |
Xie J M, Chen Y R, Cai G J, et al. Tree Visualization By One Table (tvBOT): A web application for visualizing, modifying and annotating phylogenetic trees[J]. Nucleic Acids Research, 2023, 51(W1): W587-W592. DOI:10.1093/nar/gkad359
(  0) 0) |
| [31] |
Nayfach S, Páez-Espino D, Call L, et al. Metagenomic compendium of 189, 680 DNA viruses from the human gut microbiome[J]. Nature Microbiology, 2021, 6(7): 960-970. DOI:10.1038/s41564-021-00928-6
(  0) 0) |
| [32] |
Shkoporov A N, Clooney A G, Sutton T D S, et al. The human gut virome is highly diverse, stable, and individual specific[J]. Cell Host & Microbe, 2019, 26(4): 527-541.e5.
(  0) 0) |
| [33] |
Shaffer M, Borton M A, McGivern B B, et al. DRAM for distilling microbial metabolism to automate the curation of microbiome function[J]. Nucleic Acids Research, 2020, 48(16): 8883-8900. DOI:10.1093/nar/gkaa621
(  0) 0) |
| [34] |
ter Horst A M, Santos-Medellín C, Sorensen J W, et al. Minnesota peat viromes reveal terrestrial and aquatic niche partitioning for local and global viral populations[J]. Microbiome, 2021, 9: 242. DOI:10.1186/s40168-021-01210-x
(  0) 0) |
| [35] |
Pratama A A, Bolduc B, Zayed A A, et al. Expanding standards in viromics: In silico evaluation of dsDNA viral genome identification, classification, and auxiliary metabolic gene curation[J]. Peer J, 2021, 9: e11447. DOI:10.7717/peerj.11447
(  0) 0) |
| [36] |
Tang Z Q, Zhang L Y, He N, et al. Soil bacterial community as impacted by addition of rice straw and biochar[J]. Scientific Reports, 2021, 11(1): 22185. DOI:10.1038/s41598-021-99001-9
(  0) 0) |
| [37] |
Tang Z, Zhang L, He N, et al. Influence of planting methods and organic amendments on rice yield and bacterial communities in the rhizosphere soil[J]. Frontiers in Microbiology, 2022, 13: 918986. DOI:10.3389/fmicb.2022.918986
(  0) 0) |
| [38] |
Sullivan M B, Lindell D, Lee J A, et al. Prevalence and evolution of core photosystem Ⅱ genes in marine cyanobacterial viruses and their hosts[J]. PLoS Biology, 2006, 4(8): e234. DOI:10.1371/journal.pbio.0040234
(  0) 0) |
| [39] |
Trubl G, Jang H B, Roux S, et al. Soil viruses are underexplored players in ecosystem carbon processing[J]. mSystems, 2018, 3(5): e00076-18.
(  0) 0) |
| [40] |
Jin M, Guo X, Zhang R, et al. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7(1): 58. DOI:10.1186/s40168-019-0675-9
(  0) 0) |
| [41] |
Maaroufi H, Levesque R C. Glycoside hydrolase family 32 is present in Bacillus subtilis phages[J]. Virology Journal, 2015, 12: 157. DOI:10.1186/s12985-015-0373-6
(  0) 0) |
| [42] |
Sun M M, Yuan S J, Xia R, et al. Underexplored viral auxiliary metabolic genes in soil: Diversity and eco-evolutionary significance[J]. Environmental Microbiology, 2023, 25(4): 800-810. DOI:10.1111/1462-2920.16329
(  0) 0) |
| [43] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880. DOI:10.1038/s41564-018-0190-y
(  0) 0) |
 2024, Vol. 61
2024, Vol. 61


