2. 南京农业大学植物保护学院, 南京 210095;
3. 江苏海洋大学海洋食品与生物工程学院, 江苏连云港 222005;
4. 生态环境部南京环境科学研究所, 南京 210042
2. College of Plant Protection, Nanjing Agricultural University, Nanjing 210095, China;
3. School of Ocean Food and Biological Engineering, Jiangsu Ocean University, Lianyungang, Jiangsu 222005, China;
4. Nanjing Institute of Environmental Sciences, Ministry of Ecology and Environment, Nanjing 210042, China
毒死蜱是广泛使用的有机磷杀虫剂,其残留是食品安全中的一个重要问题,应用微生物降解是消除其残留的有效方法[1-2]。自20世纪70年代以来,多株以毒死蜱为碳源和能源的降解菌被分离鉴定。Pseudomonas、Bacillus、Arthrobacter、Stenotrophomonas、Sphingomonas等多个属的毒死蜱降解菌在纯培养条件下对毒死蜱表现出良好的降解能力[3],但在实际应用中经常出现接入的降解菌存活率低、降解活性弱等问题[4]。原因之一可能是未考虑到应用降解菌进行实地生物修复的过程属于入侵过程,即要求外来微生物在目标环境的常驻微生物群落中定殖并在该环境中执行降解功能。降解菌与目标环境中常驻微生物之间的相互作用,无论是对抗性的还是协同性的,均会影响降解菌的入侵结果,进而影响其降解功能的发挥[5]。因此,探究污染土壤中土著微生物和毒死蜱降解菌之间的互作关系,对了解土著微生物影响降解菌存活及其降解功能发挥的机理至关重要。
外来微生物的入侵不仅受土壤环境非生物因素如环境温度[6]、资源有效性[7]等的影响,同时也受生物因素如微生物互作关系[8]等的影响。微生物间既存在相互抑制的关系,也存在相互促进的关系[9-11]。研究表明,微生物之间的协同作用可促进环境中的污染物降解[12-13]。例如,阿特拉津降解菌Arthrobacter sp. DNS10与解磷菌Enterobacter sp. P1(无阿特拉津降解能力)共培养条件下阿特拉津降解效果明显高于菌株Arthrobacter sp. DNS10纯培养条件。这说明Enterobacter sp. P1能够通过微生物交互作用来提高降解菌Arthrobacter sp. DNS10代谢阿特拉津的能力[14];地下水中微量污染物2, 6-二氯苯甲酰胺降解菌Aminobacter sp. MSH1用于生物修复时,目的环境中存在促进其矿化2, 6-二氯苯甲酰胺的土著微生物[5]。因此,在污染土壤中有意引入毒死蜱降解菌的背景下,也应关注是否存在降解菌的土著帮手,为降解菌提供生态位或促进降解菌在新环境的适应性,从而提高入侵成功率和毒死蜱降解率。本文以前期从土壤中分离的毒死蜱降解菌Shingopyxis granuli CP-2为材料,从土壤中分离筛选其细菌帮手,建立毒死蜱降解菌土著细菌帮手库,并初步研究CP-2的土著细菌帮手对其降解毒死蜱的影响,结果可为今后毒死蜱降解菌和土著细菌帮手的共接种修复污染提供理论指导和技术支撑。
1 材料与方法 1.1 供试材料 1.1.1 供试菌株毒死蜱降解菌Shingopyxis granuli CP-2由南京农业大学生命科学学院提供,分离自毒死蜱污染土壤。
1.1.2 主要试剂Luria-Bertani(LB)培养基:称取蛋白胨10 g、酵母粉5 g、NaCl 10 g,使用去离子水加以溶解,最终定容至1 000 mL,调节pH至7.0;胰酪大豆胨液体培养基(TSB):称取胰蛋白胨15 g·L–1,大豆蛋白胨5 g·L–1,氯化钠5 g·L–1,使用去离子水溶解,最终定容至1 000 mL,调节pH至7.0;胰酪大豆胨固体培养基(TSA):TSB培养基加15 g·L–1琼脂条;无菌缓冲溶液(SM缓冲液):七水硫酸镁2.0 g·L–1,氯化钠5.8 g·L–1,1 mol·L–1 Tris-盐酸(pH 7.5)50 mL·L–1,2%明胶5 mL·L–1;以上培养基于115 ℃高压灭菌30 min,备用。
毒死蜱标准品(99.9%)购于德国Dr.Eh-renstorfer GmbH公司;乙腈(色谱纯)购于德国Merk公司。
1.2 研究内容及方法 1.2.1 土著细菌分离、纯化与保存从田间采回的土样于4 ℃冰箱保存,用于分离土著细菌。将1 g土样与9 mL SM缓冲溶液30 ℃下在摇床中180 r·min–1振荡30 min。使用无菌水将土壤悬液稀释至10–5~10–6。然后将100 μL土壤悬液均匀地涂布于含有TSA培养基的培养皿。将连续梯度稀释涂布的平板于30 ℃下温育48 h。随后使用无菌牙签从每个样品挑取菌落形态有差异的菌株,分别接入含有100 μL TSB培养基(为TSA的液体)的96孔板中。随后将它们放置在摇床,30 ℃下180 r·min–1培养过夜,加入100 μL 30%甘油并充分混合,然后将其保存于–80 ℃冰箱备用。将保存的菌液在LB平板上划线,共122株土著细菌,编号(1~122)标记后在恒温箱中培养,待平板中细菌完全长出,备用。
1.2.2 分离土著细菌菌种的鉴定土著细菌的分子生物学鉴定采用菌体直接扩增16S rRNA基因的方法。扩增用的16S rRNA基因序列引物为27 F(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492 R(5’-AAGGAGGTGATCCAGCCGCA-3’),浓度为10 pmol·μL–1。反应体系包括:dNTP mix 4 μL、10×PCR缓冲液(含0.75 μL 0.1 mol·L−1 MgCl2)5 μL、无菌水36.5 μL和Taq DNA连接酶(5 U·μL–1)0.5 μL,无菌枪头蘸微量菌体加入反应体系中,混匀。反应程序如下:94 ℃ 5 min,之后进行35个循环,循环条件为94 ℃ 1 min、52 ℃ 1 min 30 s和72 ℃ 1 min,最后72 ℃ 10 min。将PCR扩增产物交由南京擎科生物技术有限公司进行纯化后测序。测序完成获得结果后,登录 http://www.ncbi.nlm.nih.gov在线查询分析,利用Blast软件在基因库(GenBank)中与其他已知序列进行同源性比较,最后得到土著细菌的物种信息。
1.2.3 分离土著细菌系统发育树的构建为明确从土壤中分离的土著细菌之间系统发育关系,采用了MEGA X软件的MUSCLE功能对122株细菌的16S rDNA全长序列进行比对[15]。通过手动检查比对质量,比对中的序列在两端被修整以获得最大的重叠。然后使用通用时间可逆(GTR)+ G + I模型构造了最大似然(ML)树。最后利用EVOLVIEW网络工具(https://evolgenius.info//evolview-v2/)优化系统发育树。为了显示系统发育与土著细菌分类地位之间的关系,将每个菌株的分类状态(门水平)作为热图环添加至系统发育树的外圈。
1.2.4 从分离的土著细菌中筛选毒死蜱降解菌CP-2的帮手从土壤筛选出122株细菌,但由于后期从–80 ℃冰箱将菌株划线活化时,仅有109株细菌存活,因此本实验检测109株土著细菌对毒死蜱降解菌CP-2生长的影响。采用发酵液培养的方法,利用微孔板培养体系测定筛选的土著细菌对毒死蜱降解菌CP-2生长的影响。每株土著细菌先分别在液体LB培养基中培养48 h,然后将培养液用离心机10 000 r·min–1离心10 min,将离心后的上清液用0.22 µm滤膜过滤除菌,得到每株菌的无菌发酵液,备用。将活化后的CP-2在180 r·min–1、30 ℃的摇床中培养过夜,离心调整菌液在600 nm波长处的吸光值(OD600)为1,备用。以每株土著细菌20 µL的发酵液作为处理分别加入事先加好180 µL浓度为20 mg·L–1毒死蜱的10% LB液体培养基的96孔板中,再在每个处理中加入2 µL OD600=1的CP-2悬液,设置阳性对照为加入200 µL浓度为20 mg·L–1毒死蜱的10% LB液体培养基和2 µL CP-2悬液,阴性对照为加入200 µL浓度为20 mg·L–1毒死蜱的10% LB液体培养基,每处理3重复。将96孔板置于180 r·min–1、30 ℃的摇床,培养36 h后检测OD600值,表征CP-2的生长情况。当土著细菌发酵液对CP-2的生长有显著促进作用时,认为该细菌是降解菌的帮手;当土著细菌发酵液显著抑制降解菌生长时,则认为该细菌是降解菌的抑制者。
1.2.5 土著细菌帮手促进CP-2降解毒死蜱的效果研究选择对CP-2促进效果较好的72号土著细菌B72作为其细菌帮手,研究B72菌体及其无菌发酵液促进CP-2降解毒死蜱的效果。将活化后的CP-2和B72分别在LB液体培养基中180 r·min–1、30 ℃摇床过夜培养,调整OD600为1,备用。
B72菌体对CP-2降解毒死蜱的影响相关处理如下:CK处理:3 mL毒死蜱浓度为20 mg·L–1的10% LB培养基;CP-2处理:2 850 µL毒死蜱浓度为20 mg·L–1的10% LB培养基中加入150 µLCP-2菌液;B72处理:2 850 µL毒死蜱浓度为20 mg·L–1的10% LB培养基中加入150 µLB72菌液;CP-2+B72处理:2 850µL毒死蜱浓度为20 mg·L–1的10% LB培养基中加入75 µLCP-2菌液和75 µLB72菌液。
B72的无菌发酵液对CP-2降解毒死蜱的影响相关处理如下:CK和CP-2处理同菌体实验;CP-2+B72无菌发酵液处理:2 550 µL毒死蜱浓度为20 mg·L–1的10% LB培养基中加入150 µLCP-2菌液和300 µLB72的无菌发酵液。每处理4重复。将以上处理分别加入无菌的10 mL玻璃离心管,置于摇床30℃、180 r·min–1避光培养。分别在0、12、24、36、48、60、72 h取样,检测OD600值。随后按照实验室之前的方法[4]在每个时间点提取培养液中的毒死蜱,并检测毒死蜱浓度。
1.3 数据处理试验数据处理使用Excel 2013和SigmaPlot 12.5等统计分析软件。图片处理采用Adobe Illustrator CS6软件。两个处理比较分析方法为t检验(t-test),P < 0.05表示有显著差异。
2 结果 2.1 分离的土著细菌在门水平物种组成为了明确分离土著细菌门水平的分类地位、物种组成和多样性,基于16S rRNA基因对每株菌进行分类鉴定,并用系统发育树呈现结果(图 1)。分析显示,分离的土著细菌主要隶属于4个门,分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobactoeria),分别占细菌总分离数的54.1%、14.8%、15.6%和15.6%。鉴定结果表明,分离的土著细菌覆盖了土壤中通常存在的主要细菌类群。
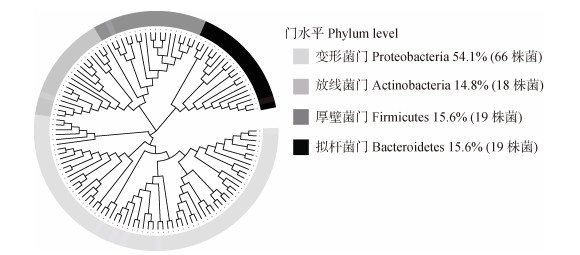
|
图 1 分离土著细菌门水平的物种多样性和分类 Fig. 1 Diversity and classification of isolated indigenous bacteria in the soil at phylum level |
分离的土著细菌中,有41.3%显著抑制毒死蜱降解菌CP-2的生长,17.4%对毒死蜱降解菌CP-2的生长无显著影响,41.3%显著促进毒死蜱降解菌CP-2的生长(图 2a),最后一类菌株被称为毒死蜱降解菌CP-2的细菌帮手。显著抑制CP-2生长的细菌中,来自于变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)的比例分别为36.2%、37.5%、78.9%和18.8%(表 1);对CP-2生长无显著作用的土著细菌中,来自以上四个门的比例分别为12.1%、12.5%、21.1%和37.5%(表 1);CP-2的细菌帮手中,来自以上四个门的比例分别为51.7%、50.0%、0.0%和43.8%(表 1)。
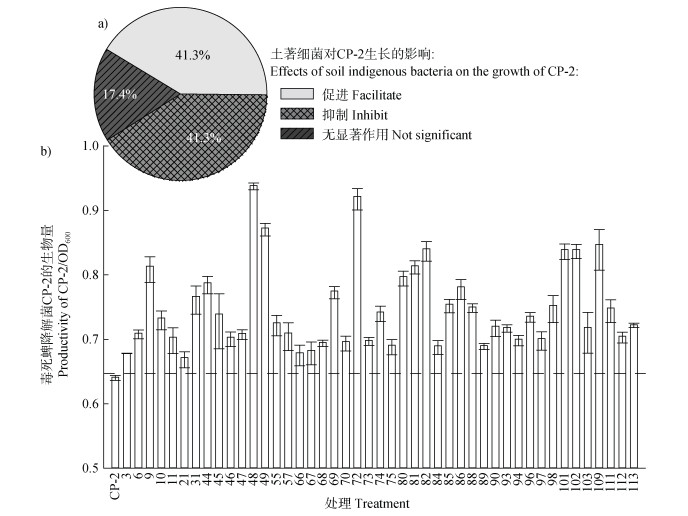
|
注:CP-2为毒死蜱降解菌编号,3~113为土著细菌编号。采用t检验,P < 0.05表示有显著性差异。下同。 Note: CP-2 is the number for chlorpyrifos degrading bacteria, and 3-113 are the numbers for indigenous bacteria. The t-test was used, and P < 0.05 indicated a significant difference. The same as below. 图 2 土著细菌对毒死蜱降解菌CP-2生长的影响(a. 对CP-2生长有不同作用的土著细菌比例;b. 显著促进CP-2生长的土著细菌) Fig. 2 Effects of soil indigenous bacteria on the growth of CP-2(a. Proportion of indigenous bacteria with different effects on CP-2 growth; b. Indigenous bacteria that significantly promote CP-2 growth) |
|
|
表 1 不同门类的土著细菌对CP-2生长有抑制、促进或无作用的细菌所占的比例 Table 1 The proportion of bacterial isolates per phylum whose supernatant showed inhibitory, stimulatory or no effect on CP-2 growth |
所有分离的土著细菌中有45株属于CP-2的帮手(图 2b),其中48号菌、72号菌对毒死蜱降解菌CP-2的促进作用最为明显,本研究将72号细菌作为后续检测影响CP-2降解毒死蜱的帮手,编号为B72。将菌株B72的16S rRNA序列进行同源比对,并构建系统进化树,结果如图 3所示,发现该菌株与赖氨酸芽孢杆菌属(Lysinibacillus sp.)相似度较高,且与耐硼赖氨酸芽孢杆菌(Lysinibacillus xylanilyticus)同源性高达100%,初步鉴定为耐硼赖氨酸芽孢杆菌(Lysinibacillus xylanilyticus)。
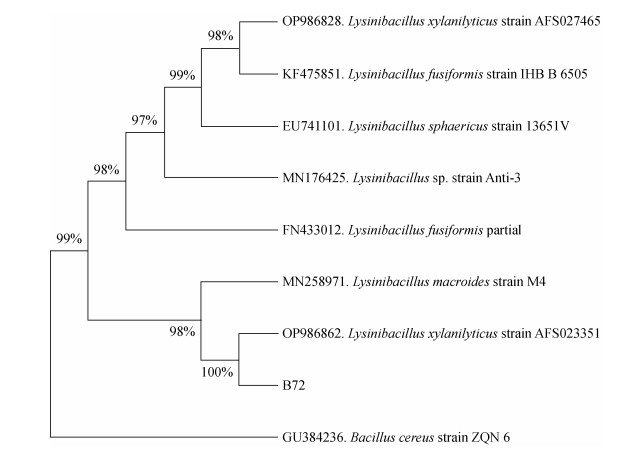
|
图 3 菌株B72的系统进化树 Fig. 3 The phylogenetic relationship of bacterial strain B72 |
通过菌体直接扩增16S rRNA基因的方法,可得到45株毒死蜱降解菌CP-2土著细菌帮手的物种信息(图 4):这45株细菌帮手主要隶属于3个门,分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria),分别占总分离细菌数的66.67%、17.78%和15.56%。拟杆菌门(Bacteroidetes)中未筛选到毒死蜱降解菌CP-2的帮手。这些细菌帮手隶属于4个纲、7个目、13个科和20个属,其中三个丰度比较高的属为假单胞菌属(Bacillus)、不动杆菌属(Acinetobacter)和类芽孢杆菌属(Paenibacillus),丰度分别占总分离细菌数的15.6%、11.1%和11.1%,这三个属的总比例达到了37.8%。
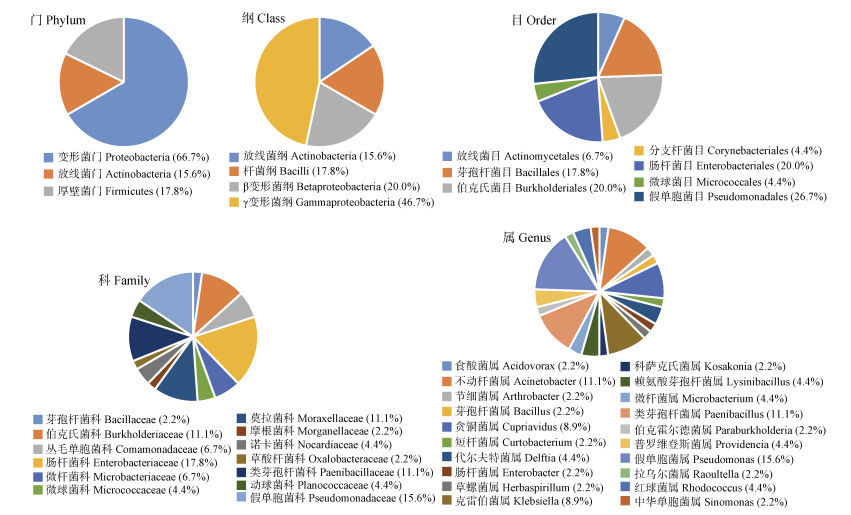
|
图 4 毒死蜱降解菌CP-2细菌帮手库的物种信息 Fig. 4 Species information of the bacteria that significantly promoted CP-2 growth |
将毒死蜱降解菌CP-2和筛选出的土著细菌帮手B72及其无菌发酵液在含浓度为20 mg·L–1毒死蜱的液体培养基中共培养72 h后,分析菌株的生长情况和体系中毒死蜱的浓度。图 5a和图 5c表明,毒死蜱降解菌CP-2和土著细菌帮手B72均在培养12 h后进入平台期,两株菌共培养时生物量高于各自单独培养时的生物量,且B72菌株的无菌发酵液显著促进降解菌CP-2的生长。图 5b和图 5d显示,空白对照处理组(仅加入培养基和毒死蜱)中毒死蜱在0~60 h内基本不降解,培养72 h后培养基中毒死蜱有少量降解。菌株B72处理组中毒死蜱在0~72 h内也基本不降解(图 5b),说明在本实验条件下,菌株B72不具备降解毒死蜱的能力。毒死蜱降解菌CP-2在0~12 h时间段内对毒死蜱的降解速度最快,培养体系中的毒死蜱由20 mg·L–1迅速降至5 mg·L–1左右,12 h后降解菌CP-2降解毒死蜱的速率减缓,培养体系中毒死蜱的浓度由12 h的5 mg·L–1左右到72 h的2 mg·L–1左右(图 5b和图 5d)。与单独加毒死蜱降解菌CP-2的处理相比,加了细菌帮手B72后,0~12 h对毒死蜱的降解速率有所降低,但从24 h开始,到72 h,细菌帮手B72均显著促进CP-2对毒死蜱的降解(图 5b)。与菌体培养实验结果不同,B72菌株的无菌发酵液从12 h开始就显著促进CP-2对毒死蜱的降解(图 5d)。
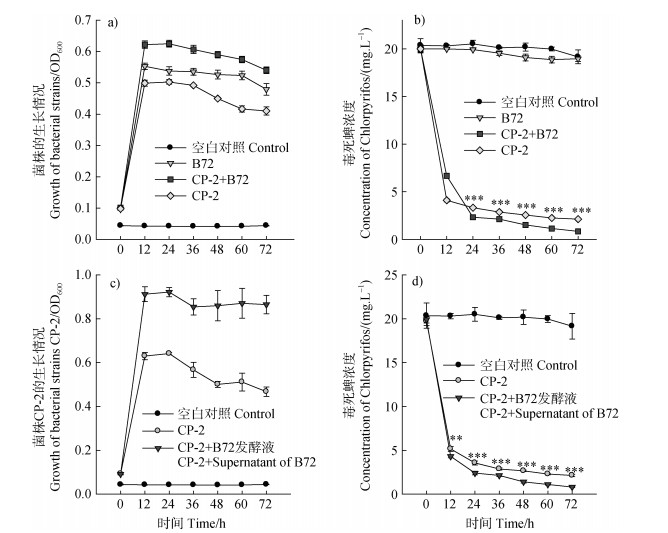
|
注:B72为毒死蜱降解菌CP-2的土著细菌帮手。**、***分别表示P < 0.01和P < 0.001。 Note: B72 is an indigenous bacterial helper of chlorpyrifos degrading bacterium CP-2. **, *** represent P < 0.01 and P < 0.001, respectively. 图 5 菌株B72对CP-2生长(a)和降解毒死蜱的影响(b),以及菌株B72的无菌上清液对CP-2生长(c)和降解毒死蜱的影响(d) Fig. 5 Effects of strain B72 on CP-2 growth (a) and degradation of chlorpyrifos (b), and effects of sterile supernatant of strain B72 on CP-2 growth (c) and degradation of chlorpyrifos (d) |
土壤污染,如重金属、石油烃、微塑料和农药等污染物,对土壤健康造成了严重的威胁[16-18]。众所周知,微生物修复是一种利用生物修复污染土壤的经济、高效且环境友好的方法,已有多种具备高效生物修复能力的细菌、真菌、藻类等微生物物种成功用于降低土壤中有毒污染物的毒性[19-20]。本研究分离的土著细菌主要隶属于4个门,分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),与大部分相关研究相似,筛选的土著细菌覆盖了土壤中通常存在的主要细菌类群[10,21]。这些细菌为后续土壤中毒死蜱或者其他污染物的微生物降解相关研究及应用提供了丰富的菌种资源。
3.2 微生物之间互作关系及其对毒死蜱降解菌降解毒死蜱的影响微生物之间广泛存在便利型的互作关系,微生物可通过交叉喂养[22]、分子通信[23]或生产公共物质[24]等方式来促进彼此的生长。对于细菌之间互惠或共生关系的机制,有研究已经证明主要是因为细菌群落中的成员能够相互利用或受益于彼此产生的代谢产物[11]。例如,蜡状芽孢杆菌产生的肽聚糖可促进若干属于噬细菌-黄杆菌属(Cytophaga-Flavobacterium)的菌株生长[25]。微生物产生的铁载体也可被其他具有铁载体蛋白受体的细菌作为公共物品接受,在环境中获得有限的铁以维持生长和代谢,从而增加群落生物量[26]。毒死蜱降解菌的细菌帮手对其促进机制很可能与某些代谢物有关,因为在本研究中,细菌帮手的无菌上清液促进了降解菌的生长(图 2b和5c)。后续实验中需进一步检测细菌帮手分泌的促进毒死蜱降解菌生长的具体物质。
本研究中毒死蜱降解菌CP-2在0~12 h对20 mg·L–1毒死蜱的降解速率最高,在12 h时降解率达75%以上(图 5b和图 5d),原因之一可能是菌株CP-2在0~12 h处于对数生长期,需要大量的营养物质。在0~12 h内,毒死蜱降解菌的土著细菌帮手菌体本身并未促进反而抑制其对毒死蜱的降解(图 5b),可能由于本研究中所用培养基(10% LB)营养比较贫瘠,前12 h属于两株菌生长的对数期,培养体系中两株菌之间形成激烈的营养竞争关系[27],导致CP-2降解毒死蜱的速率有所减慢。而到后期,体系中培养基的营养物质消耗殆尽,降解菌CP-2可能开始利用其细菌帮手B72产生的物质生长增殖,并继续降解毒死蜱。因此,菌株B72在24 h开始促进降解菌CP-2对毒死蜱的降解(图 5b)。但从本研究可以看出菌株CP-2对20 mg·L–1毒死蜱的降解能力很强,后续实验需要考虑设置更高的浓度,这样土著帮手对降解菌降解能力的促进作用应该会更显著。
应用微生物降解毒死蜱是消除其残留的有效方法[2]。对于微生物降解毒死蜱的相关研究大多数是关于如何筛选出高效毒死蜱降解菌并对其进行鉴定以及优化降解条件来提高降解效率[28-30]。而本研究则使用已经筛选得到的毒死蜱高效降解菌Shingopyxis granuli CP-2作为研究材料,分离出土著微生物,研究它们与降解菌的互作关系,从而筛选出降解菌的土著微生物帮手。相较于使用单一降解菌株对毒死蜱进行生物修复,本研究为毒死蜱降解菌的实际应用提供了一种新思路,即与其土著细菌帮手共接种,提高其生长增殖能力以及降解毒死蜱的能力。但本研究仅检测了毒死蜱降解菌细菌帮手库中的一株——菌株B72对毒死蜱降解菌CP-2降解毒死蜱能力的影响,且仅在严格控制条件的体外试验中进行了此实验。后续研究需要检测土著细菌帮手库中的其他成员对毒死蜱降解菌CP-2降解毒死蜱能力的影响,揭示其中的机理,并在实际土壤中验证室内得到的结果,为后续应用作准备。
4 结论本研究从土壤分离109株土著细菌,隶属于变形菌门、厚壁菌门、拟杆菌门和放线菌门,涵盖了土壤中通常存在的主要细菌类群。基于分离的土著细菌对毒死蜱降解菌CP-2生长的影响,建立由45株土著细菌构成的毒死蜱降解菌CP-2的帮手库,隶属于3个门、13个科和20个属,且筛选的土著细菌帮手显著促进毒死蜱降解菌CP-2降解毒死蜱的能力。本研究中筛选的菌株可为后续土壤中毒死蜱或者其他污染物的微生物降解相关研究及应用提供丰富的菌种资源,研究结果为今后毒死蜱降解菌和土著细菌帮手们的共接种修复污染提供理论指导与技术支撑。
致谢 诚挚地感谢南京农业大学生命科学学院洪青教授团队为本研究提供毒死蜱降解菌Shingopyxis granuli CP-2。
| [1] |
Foong S Y, Ma N L, Lam S S, et al. A recent global review of hazardous chlorpyrifos pesticide in fruit and vegetables: Prevalence, remediation and actions needed[J]. Journal of Hazardous Materials, 2020, 400: 123006. DOI:10.1016/j.jhazmat.2020.123006
(  0) 0) |
| [2] |
Dar M A, Kaushik G, Villarreal-Chiu J F. Pollution status and bioremediation of chlorpyrifos in environmental matrices by the application of bacterial communities: A review[J]. Journal of Environmental Management, 2019, 239: 124-136.
(  0) 0) |
| [3] |
Huang Y H, Zhang W P, Pang S M, et al. Insights into the microbial degradation and catalytic mechanisms of chlorpyrifos[J]. Environmental Research, 2021, 194: 110660. DOI:10.1016/j.envres.2020.110660
(  0) 0) |
| [4] |
Zhan H L, Ge J, Cheng J F, , et al. Effects of endophyte inoculation in rice on the degradation of chlorpyrifos in paddy soil (In Chinese)[J]. Jiangsu Agricultural Sciences, 2020, 48(14): 278-282. [詹红林, 葛静, 程江峰, 等. 内生菌定殖水稻对稻田土壤中毒死蜱降解的影响[J]. 江苏农业科学, 2020, 48(14): 278-282.]
(  0) 0) |
| [5] |
Vandermaesen J, Du S Y, Daly A J, et al. Interspecies interactions of the 2, 6-dichlorobenzamide degrading Aminobacter sp. MSH1 with resident sand filter bacteria: Indications for mutual cooperative interactions that improve BAM mineralization activity[J]. Environmental Science & Technology, 2022, 56(2): 1352-1364.
(  0) 0) |
| [6] |
Wei Z, Huang J F, Yang T J, et al. Seasonal variation in the biocontrol efficiency of bacterial wilt is driven by temperature-mediated changes in bacterial competitive interactions[J]. Journal of Applied Ecology, 2017, 54(5): 1440-1448. DOI:10.1111/1365-2664.12873
(  0) 0) |
| [7] |
Yang T J, Wei Z, Friman V P, et al. Resource availability modulates biodiversity-invasion relationships by altering competitive interactions[J]. Environmental Microbiology, 2017, 19(8): 2984-2991. DOI:10.1111/1462-2920.13708
(  0) 0) |
| [8] |
Li M, Wei Z, Wang J N, et al. Facilitation promotes invasions in plant-associated microbial communities[J]. Ecology Letters, 2019, 22(1): 149-158. DOI:10.1111/ele.13177
(  0) 0) |
| [9] |
Hassani M A, Durán P, Hacquard S. Microbial interactions within the plant holobiont[J]. Microbiome, 2018, 6(1): 58. DOI:10.1186/s40168-018-0445-0
(  0) 0) |
| [10] |
Li M, Pommier T, Yin Y, et al. Indirect reduction of Ralstonia solanacearum via pathogen helper inhibition[J]. ISME Journal, 2022, 16(3): 868-875. DOI:10.1038/s41396-021-01126-2
(  0) 0) |
| [11] |
Gu S H, Wei Z, Shao Z Y, et al. Competition for iron drives phytopathogen control by natural rhizosphere microbiomes[J]. Nature Microbiology, 2020, 5(8): 1002-1010. DOI:10.1038/s41564-020-0719-8
(  0) 0) |
| [12] |
Li M T, Zhao X M, Zhang X F, et al. Biodegradation of 17β-estradiol by bacterial co-culture isolated from manure[J]. Scientific Reports, 2018, 8(1): 3787. DOI:10.1038/s41598-018-22169-0
(  0) 0) |
| [13] |
Xu X H, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5
(  0) 0) |
| [14] |
Jiang Z, Zhang X Y, Wang Z Y, et al. Enhanced biodegradation of atrazine by Arthrobacter sp. DNS10 during co-culture with a phosphorus solubilizing bacteria: Enterobacter sp. P1[J]. Ecotoxicology and Environmental Safety, 2019, 172: 159-166. DOI:10.1016/j.ecoenv.2019.01.070
(  0) 0) |
| [15] |
Kumar S, Stecher G, Li M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096
(  0) 0) |
| [16] |
Xu J M, He L Z, Tang X J, et al. Problems and suggestions on safe utilization of heavy metal (loid)-contaminated farmlands in China (In Chinese)[J]. Acta Pedologica Sinica, 2023, 60(5): 1289-1296. DOI:10.11766/trxb202309030353 [徐建明, 何丽芝, 唐先进, 等. 中国重金属污染耕地土壤安全利用存在问题与建议[J]. 土壤学报, 2023, 60(5): 1289-1296.]
(  0) 0) |
| [17] |
Riedo J, Wettstein F E, Rösch A, et al. Widespread occurrence of pesticides in organically managed agricultural soils-the ghost of a conventional agricultural past?[J]. Environmental Science & Technology, 2021, 55(5): 2919-2928.
(  0) 0) |
| [18] |
Feng X Y, Sun Y H, Zhang S W, et al. Ecological effects of microplastics on soil-plant systems (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(2): 299-313. DOI:10.11766/trxb202007190402 [冯雪莹, 孙玉焕, 张书武, 等. 微塑料对土壤-植物系统的生态效应[J]. 土壤学报, 2021, 58(2): 299-313.]
(  0) 0) |
| [19] |
Löffler F E, Edwards E A. Harnessing microbial activities for environmental cleanup[J]. Current Opinion in Biotechnology, 2006, 17(3): 274-284. DOI:10.1016/j.copbio.2006.05.001
(  0) 0) |
| [20] |
Coban O, de Deyn G B, van der Ploeg M. Soil microbiota as game-changers in restoration of degraded lands[J]. Science, 2022, 375(6584): abe0725. DOI:10.1126/science.abe0725
(  0) 0) |
| [21] |
Xia Q, Rufty T, Shi W. Soil microbial diversity and composition: Links to soil texture and associated properties[J]. Soil Biology and Biochemistry, 2020, 149: 107953. DOI:10.1016/j.soilbio.2020.107953
(  0) 0) |
| [22] |
Pacheco A R, Moel M, Segrè D. Costless metabolic secretions as drivers of interspecies interactions in microbial ecosystems[J]. Nature Communications, 2019, 10(1): 103. DOI:10.1038/s41467-018-07946-9
(  0) 0) |
| [23] |
Striednig B, Hilbi H. Bacterial quorum sensing and phenotypic heterogeneity: How the collective shapes the individual[J]. Trends in Microbiology, 2022, 30(4): 379-389. DOI:10.1016/j.tim.2021.09.001
(  0) 0) |
| [24] |
Kramer J, Özkaya Ö, Kümmerli R. Bacterial siderophores in community and host interactions[J]. Nature Reviews Microbiology, 2020, 18(3): 152-163. DOI:10.1038/s41579-019-0284-4
(  0) 0) |
| [25] |
Peterson S B, Dunn A K, Klimowicz A K, et al. Peptidoglycan from Bacillus cereus mediates commensalism with rhizosphere bacteria from the Cytophaga-Flavobacterium group[J]. Applied and Environmental Microbiology, 2006, 72(8): 5421-5427. DOI:10.1128/AEM.02928-05
(  0) 0) |
| [26] |
Figueiredo A R T, Özkaya Ö, Kümmerli R, et al. Siderophores drive invasion dynamics in bacterial communities through their dual role as public good versus public bad[J]. Ecology Letters, 2022, 25(1): 138-150.
(  0) 0) |
| [27] |
Li M, Pommier T, Yin Y, et al. Resource availability drives bacteria community resistance to pathogen invasion via altering bacterial pairwise interactions[J]. Environmental Microbiology, 2022, 24(12): 5680-5689.
(  0) 0) |
| [28] |
Elzakey E M, El-Sabbagh S M, Eldeen E E N, et al. Bioremediation of chlorpyrifos residues using some indigenous species of bacteria and fungi in wastewater[J]. Environmental Monitoring and Assessment, 2023, 195(6): 779.
(  0) 0) |
| [29] |
Uniyal S, Sharma R K, Kondakal V. New insights into the biodegradation of chlorpyrifos by a novel bacterial consortium: Process optimization using general factorial experimental design[J]. Ecotoxicology and Environmental Safety, 2021, 209: 111799.
(  0) 0) |
| [30] |
Eissa F, Mahmoud H, Massoud O, et al. Biodegradation of chlorpyrifos by microbial strains isolated from agricultural wastewater[J]. Journal of American Science, 2014, 10(3): 98-108.
(  0) 0) |
 2025, Vol. 62
2025, Vol. 62


