2. 江苏省土壤利用与农业可持续发展工程研究中心, 南京 210023;
3. 江苏省地理信息资源开发与利用协同创新中心, 南京 210023
2. Jiangsu Engineering Research Center for Soil Utilization & Sustainable Agriculture, Nanjing 210023, China;
3. Jiangsu Center for Collaborative Innovation in Geographical Information Resource Development and Application, Nanjing 210023, China
抗生素主要是由细菌、霉菌或其他微生物产生的一类次级代谢产物或人工合成的类似物,在致病微生物感染类疾病治疗方面发挥着重要的作用。据估计,2030年全球用于畜禽疾病防治的抗生素使用量将达到20.0万吨,这些抗生素大多不能被完全吸收,而是以原型或代谢物的形式排放至土壤、水体等环境中[1]。研究表明,长期施用富含抗生素或抗性细菌的畜禽粪便能够大幅提高农田土壤中抗生素抗性基因(Antibiotic resistance genes,ARGs)的丰度和多样性,增加ARGs通过食物链向人类传播扩散的风险,并进而对人类健康构成威胁[2]。目前,土壤中的ARGs已成为备受关注的新型生物污染物之一。另一方面,高投入高产出的集约化种植模式在激发土传病原微生物生长和繁殖的同时,降低了土壤微生物多样性和抑病功能,导致作物土传病害频发[3]。Li等[4]研究发现,番茄致病菌青枯菌(Ralstonia solanacearum)入侵能够显著增加根际土壤中ARGs及其转移扩散载体可移动遗传元件(Mobile genetic elements,MGEs)的丰度,表明土传病原菌增殖能够促进土壤ARGs的富集和水平转移,加剧ARGs传播扩散的风险[5]。因此,在集约化种植模式不断推广和发展的背景下,以土传病原菌与ARGs和MGEs叠加互作所形成的土壤生物复合污染已成为制约其绿色可持续发展和阻碍“一体化健康”目标实现的关键。
同步降低土传病原菌和携带ARGs的抗性菌种群数量或相对丰度是实现土壤病原菌和ARGs复合污染有效修复的关键。强还原土壤处理(Reductive soil disinfestation,RSD)是一种依赖大量易分解有机碳输入的土壤处理方法,其具有改善土壤理化性质、杀灭土传病原菌、优化土壤微生物群落结构和协调土壤微生物互作关系等多重作用[6-7]。大量研究表明,RSD处理能够广谱性地杀灭镰刀菌(Fusarium spp.)、丝核菌(Rhizoctonia spp.)、交链孢霉(Alternaria spp.)、青枯菌(R. solanacearum)等土传病原真菌和细菌,大幅降低病原菌在微生物群落中的占比[6,8]。同时,还有一些研究发现,RSD处理能够通过抑制主要ARGs的潜在宿主,从而实现土壤中ARGs的消减[9-10]。但是,RSD处理并不能抑制土壤中所有携带ARGs的抗性菌,因此RSD处理对土壤ARGs的消减作用在很大程度上取决于其宿主类型。此外,采用不同有机物料进行的RSD处理能够驱动形成差异化的土壤微生物群落[11],这表明RSD处理对ARGs宿主的抑制作用还可能与所用有机物料类型有关。所以,RSD处理能否协同消减土壤病原菌与ARGs和MGEs叠加形成的生物复合污染,以及RSD处理对土壤生物复合污染的修复效果是否与所用有机物料类型有关,目前还不得而知。
研究表明,通过畜禽粪便直接输入的抗性细菌所携带的ARGs和通过畜禽粪便中所含抗生素诱导土著微生物产生的ARGs是土壤ARGs的主要来源,但畜禽粪便或其制备的有机肥对土壤ARGs储库和传播扩散的影响与土壤类型密切相关[12-13]。Song等[14]研究发现,有机肥或混有金霉素的有机肥施用对红壤中多药类ARGs相对丰度的增加作用较黑土和潮土显著,且能加剧红壤中病原微生物的传播风险。此外,由于不同土壤类型中初始微生物群落组成和结构存在差异,因此粪肥施用后同一ARGs在不同土壤类型中的宿主也可能存在差异[14-15]。综上,土壤生物复合污染的特征与所用粪肥和土壤类型密切相关,所以RSD处理对土壤生物复合污染的修复效果可能也与土壤类型有关。鉴于此,本研究选取3种典型土壤类型(黑土、红壤和潮土)展开,以具有长期粪肥施用历史且青枯菌与ARGs和MGEs复合污染严重的集约化农田土壤为研究对象,通过设置不同有机物料的RSD处理,探究其对不同土壤类型中生物复合污染的修复效果,旨在寻求一种能够协同消减土壤生物复合污染的方法,为保障土壤生物健康和实现集约化农业的良性可持续发展提供一定的技术支撑。
1 材料与方法 1.1 供试土壤及其复合污染特征供试土壤包括黑土(BS)、红壤(RS)和潮土(FS),分别采集自吉林省吉林市(43°52′N,126°33′E)、江西省吉安市(27°02′N,114°54′E)和江苏省海安市(32°66′N,120°70′E)具有多年粪肥施用历史的农田,其中BS和RS所用粪肥为猪粪,FS所用粪肥为鸡粪。供试土壤均具有严重的青枯菌与ARGs和MGEs复合污染的特征,其中BS中ARGs、MGEs和青枯菌的绝对丰度分别为8.53×109、2.01×109和1.39×107 copies·g–1干土;RS中ARGs、MGEs和青枯菌的绝对丰度分别为9.96×109、2.91×109和2.40×107 copies·g–1干土;FS中ARGs、MGEs和青枯菌的绝对丰度分别为6.96×109、1.42×109和2.34×107 copies·g–1干土(图 1)。此外,供试土壤的基本理化性质存在一定差异,BS:pH 7.55,电导率1 992 μS·cm–1,有机质63.1 g·kg–1,全氮7.65 g·kg–1;RS:pH 7.00,电导率447.0 μS·cm–1,有机质15.4 g·kg–1,全氮1.73 g·kg–1;FS:pH 7.65,电导率335.0 μS·cm–1,有机质11.2 g·kg–1,全氮1.41 g·kg–1[16]。将采集的土壤过2 mm筛后混匀,4 ℃储存备用。
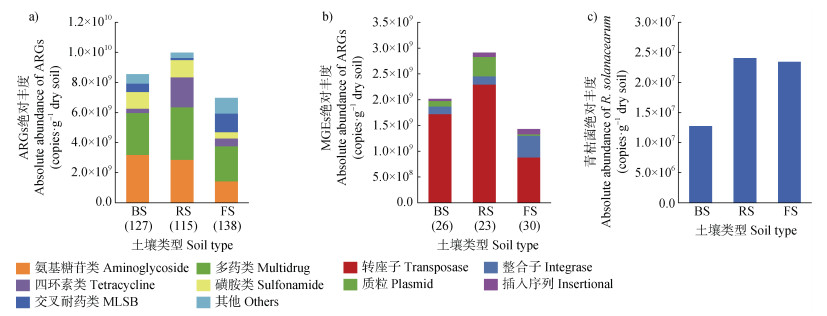
|
注:BS、RS和FS分别表示黑土、红壤和潮土。括号中数字表示ARGs或MGEs基因的数量。下同。 Note: BS, RS, and FS represent black soil, red soil, and fluvo-aquic soil, respectively. The numbers in brackets indicate the number of ARGs or MGEs genes. The same below. 图 1 供试土壤中主要ARGs(a)、MGEs(b)和青枯菌(c)的分布特征 Fig. 1 Distribution characteristics of major ARGs (a), MGEs (b), and R. solanacearum (c) in the tested soils |
3种土壤类型各设置5个培养处理:1)土壤不做处理的对照(CK);2)最大持水量处理(FCK);3)添加1%的乙醇RSD处理(ET);4)添加1%的苜蓿粉(C/N:21.2)RSD处理(AL);5)添加1%的糖蜜(C/N:12.6)RSD处理(MO),每个处理包含3个生物学重复。具体操作如下:称取100 g土壤及对应有机物料置于20丝的自封袋中,充分混匀后加水至最大持水量(CK除外),排尽袋内空气后封口密闭,置于恒温培养箱35 ℃培养30 d。处理结束后,采集各处理的土壤样品,部分保存于4 ℃用于土壤基本理化性质的测定,部分保存于–80 ℃用于土壤DNA提取及定量PCR分析。
1.3 土壤DNA提取称取0.5 g保存于–80 ℃的土壤样品,使用FastDNA®SPIN Kit for Soil(MP Biomedicals,USA)提取土壤DNA,所有DNA样品经DS-11超微量分光光度计(DeNovix,USA)质控后保存于–20 ℃待用。
1.4 荧光定量PCR分析采用荧光定量PCR技术对土壤中细菌、青枯菌、主要ARGs和MGEs的绝对丰度进行分析,扩增反应在QuanStudio 3 Real-Time PCR system(Applied Biosystems,USA)上进行,反应体系(20 μL)包括2× SYBR®Green Pro Taq HS Premix(艾瑞科生物,中国湖南)10 μL,正、反引物(10 μmol·L–1)各1 μL,DNA模板1 μL和dd H2O 7 μL。用于定量细菌16S rRNA(Eub338F/518R)、青枯菌fliC(fliC-f/fliC-r)、氨基糖苷类抗性基因aadA7(aadA7-f/aadA7-r)和aadA21(aadA21-f/aadA21-r)、多药类抗性基因qacH(qacH-f/ qacH-r)和msrE(msrE-f/msrE-r)、四环素类抗性基因tetG(tetG-f/tetG-r)、tetM(tetM-f/tetM-r)和tet36(tet36-f/tet36-r)、磺胺类抗性基因sul1(sul1-f/sul1-r)、MGEs基因intl1(intl1-f/intl1-r)、IS6100(IS6100-f/IS6100-r)和IS26(IS26-f/IS26-r)的引物及扩增条件见表 1。各基因标准曲线的扩增效率为97.26%~102.61%,均符合测定要求。
|
|
表 1 荧光定量PCR所用引物及扩增条件 Table 1 Primers and PCR conditions used in real-time PCR |
采用相对丰度和消减率评估RSD处理对青枯菌、ARGs和MGEs的影响[15]。其中相对丰度是指某个特定基因的拷贝数与16S rRNA基因拷贝数的比值,用于表示该基因占总细菌基因的比例;消减率是指处理前后某一特定基因相对丰度的变化,若值为正数,则表明处理具有消减作用,若值为负数,则表明处理后该特定基因相对丰度增加。具体计算方法如下:
| $ 相对丰度R =\frac{{{C_i}}}{{{C_b}}} $ | (1) |
| $ 相对丰度消减率 = \frac{{{R_0} - {R_i}}}{{{R_0}}} \times 100\% $ | (2) |
式中,Ci为某一特定ARGs或MGEs基因的拷贝数,Cb为16S rRNA基因的拷贝数;R0为对照组某一特定基因的相对丰度,Ri则为处理后该基因的相对丰度。
单因素方差分析(One-way ANOVA)配合Duncan新复极差法(Duncan’s multiple range test)检验同一土壤不同处理间均值差异的显著性(P < 0.05)。采用双因素方差分析检验土壤类型、处理方式及其交互作用对青枯菌、ARGs和MGEs相对丰度影响差异的显著性。主坐标分析(Principal coordinate analysis,PCoA)用于比较不同处理中青枯菌、ARGs和MGEs相对丰度上的差异(Bray-Curtis距离),并采用置换多元方差分析检验不同处理方式之间所有基因相对丰度差异的显著性。采用Pearson相关性分析法对各土壤类型中青枯菌的相对丰度与ARGs和MGEs的相对丰度、ARGs相对丰度与MGEs相对丰度、以及青枯菌、ARGs、MGEs的相对丰度与土壤理化性质之间的相关性进行检验。所有统计分析均由R软件(v4.3.2)和IBM SPSS Statistics 26统计软件完成。
2 结果 2.1 RSD处理对黑土中青枯菌、ARGs和MGEs相对丰度的影响PCoA分析结果(图 2a)表明,不同处理对BS中青枯菌、ARGs和MGEs的污染特征具有显著(P < 0.001)影响,其中RSD处理(ET、AL和MO)的作用较FCK处理显著,且ET、AL和MO处理之间存在一定的差异。与CK相比,ET和AL处理均能显著(P < 0.05)降低BS中青枯菌的相对丰度,其中ET处理的消减率达到71.7%,显著(P < 0.05)高于AL处理的43.3%,而FCK和MO处理对其无显著影响(表 2和图 2b)。由表 2可知,AL和MO处理均能在一定程度上降低BS中总ARGs的相对丰度,但较CK处理的差异并不显著,其消减率分别为14.8%和22.9%(图 2b)。与CK相比,FCK、AL和MO处理均能显著(P < 0.05)降低BS中氨基糖苷类ARGs(aadA7和aadA21)的相对丰度,其消减率分别为34.2%~56.4%和28.5%~49.9%(表 2和图 2b)。类似地,AL和MO处理均能降低BS中多药类ARGs msrE的相对丰度,其消减率分别为19.1%和18.1%,但不同土壤处理对多药类ARGs qacH的相对丰度均无显著影响(表 2和图 2b)。由表 2可知,不同土壤处理对BS中四环素类ARGs(tetG、tetM和tet36)相对丰度的影响存在差异,其中FCK和MO处理能够显著(P < 0.05)降低tetG基因的相对丰度,AL和MO处理对tet36基因相对丰度的消减效果显著(P < 0.05),而所有处理对tetM基因的相对丰度均无显著影响。与CK相比,AL和MO处理均能显著(P < 0.05)降低BS中磺胺类ARGs sul1的相对丰度,其消减率分别为28.4%和33.2%,而FCK和ET处理对其无显著影响。此外,不同土壤处理对BS中MGEs基因相对丰度的影响各异,RSD和FCK处理均能显著(P < 0.05)降低土壤中转座子IS6100基因的相对丰度,其中RSD处理的消减率为37.6%~43.9%,优于FCK处理的25.4%;与CK相比,ET处理能够显著(P < 0.05)增加土壤中转座子IS26基因的相对丰度,而所有处理对整合子intl1基因的相对丰度均无显著影响(表 2和图 2b)。总体而言,RSD处理对BS中青枯菌、ARGs和MGEs复合污染具有一定的修复效果,且这种修复效果与ARGs、MGEs的基因类型以及RSD处理所用有机物料类型密切相关。
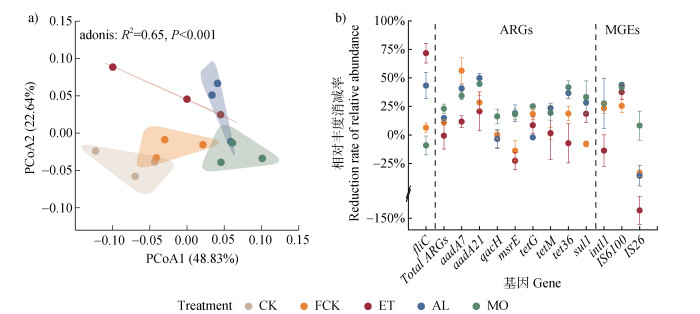
|
注:CK,对照;FCK,最大持水量处理;ET,添加1%酒精的RSD处理;AL,添加1%苜蓿粉的RSD处理;MO,添加1%糖蜜的RSD处理。误差线表示标准误(n = 3)。下同。 Note: CK, control; FCK, maximum water holding capacity treatment; ET, RSD incorporated with 1% ethanol; AL: RSD incorporated with 1% alfalfa; MO, RSD incorporated with 1% molasses. Error bars indicate the standard error of the means of three replicates. The same below. 图 2 不同土壤处理对黑土中青枯菌、ARGs和MGEs污染特征(a)及消减率(b)的影响 Fig. 2 Effects of different soil treatments on the contamination characteristics (a) and reduction rates (b) of R. solanacearum, ARGs, and MGEs in black soil |
|
|
表 2 不同土壤处理对黑土中青枯菌、ARGs和MGEs相对丰度的影响 Table 2 Effects of different soil treatments on the relative abundance of R. solanacearum, ARGs, and MGEs in black soil |
不同土壤处理对RS中青枯菌、ARGs和MGEs污染特征的影响显著(P < 0.001),且不同处理的作用效果存在一定的差异(图 3a)。与CK相比,RSD和FCK处理均能显著(P < 0.05)降低RS中青枯菌的相对丰度,其中RSD处理的消减率可达88.0%~92.3%,显著(P < 0.05)高于FCK处理(59.5%),且ET、AL和MO处理间无显著差异(表 3和图 3b)。由表 3可知,相较于CK处理,AL处理能够显著(P < 0.05)提高RS中总ARGs的相对丰度(1.1×10–1),而其他处理对其无显著影响。RSD和FCK处理均能显著(P < 0.05)降低RS中aadA7基因的相对丰度,其中ET处理的消减作用最强,其消减率达到90.2%,AL、MO和FCK的消减率分别为76.3%、51.3%和34.5%(表 3和图 3b)。与CK相比,不同处理对RS中aadA21基因的相对丰度均无显著影响,但ET和AL处理后aadA21基因的相对丰度显著(P < 0.05)高于FCK和MO处理(表 3)。由表 3可知,不同处理对RS中多药类ARGs(qacH和msrE)相对丰度的影响不同,其中AL处理能够显著(P < 0.05)增加qacH基因的相对丰度,而ET和MO处理能够显著(P < 0.05)增加msrE基因的相对丰度。与CK相比,RSD处理均能显著(P < 0.05)增加RS中tetM基因的相对丰度,而ET处理能够降低tetG和tet36基因的相对丰度,且对tet36基因相对丰度的消减作用达到显著(P < 0.05),其消减率为80.8%(表 3和图 3b)。此外,AL处理能显著(P < 0.05)增加RS中sul1、intl1和IS26基因的相对丰度,而其他处理对三者的相对丰度较CK均无显著影响(表 3)。总之,RSD处理能够有效抑制RS中青枯菌的相对丰度,但对ARGs和MGEs相对丰度的消减作用微弱,其中AL处理对多种ARGs的相对丰度还具有增加作用。
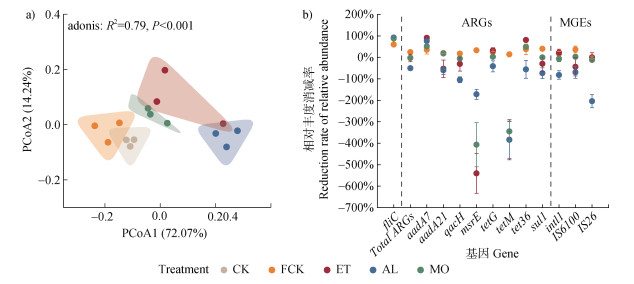
|
图 3 不同土壤处理对红壤中青枯菌、ARGs和MGEs污染特征(a)及消减率(b)的影响 Fig. 3 Effects of different soil treatments on the contamination characteristics (a) and reduction rates (b) of R. solanacearum, ARGs, and MGEs in red soil |
|
|
表 3 不同土壤处理对红壤中青枯菌、ARGs和MGEs相对丰度的影响 Table 3 Effects of different soil treatments on the relative abundance of R. solanacearum, ARGs, and MGEs in red soil |
如图 4a所示,与CK相比,RSD处理均能显著(P < 0.001)改变FS中青枯菌、ARGs和MGEs的复合污染特征,其中复杂有机物料(AL和MO)的处理效果与简单有机物料(ET)存在明显差异。由表 4可知,不同有机物料RSD处理对FS中青枯菌、ARGs和MGEs相对丰度的影响存在差异,而FCK处理对其均无显著影响。与CK相比,RSD处理均能显著(P < 0.05)降低FS中青枯菌的相对丰度,其中ET处理的消减率最高,达到94.2%,高于AL处理的76.1%和MO处理的80.9%(表 4,图 4b)。AL和MO处理能够降低FS中总ARGs的相对丰度,消减率分别为15.0%和20.6%,其中MO处理的消减效果达到显著(P < 0.05),而ET处理后土壤总ARGs的相对丰度较CK显著(P < 0.05)增加(表 4,图 4b)。如表 4所示,ET处理后,FS中aadA7基因的相对丰度降至1.7×10–3,显著(P < 0.05)低于CK和其他处理,其消减率高达81.6%,但aadA21基因的相对丰度显著(P < 0.05)增加至9.6×10–3,而AL和MO处理均能降低FS中aadA7和aadA21基因的相对丰度,其消减率分别为22.0%~29.8%和50.5%~57.9%,但两者间无显著差异(图 4b)。类似地,ET处理能够显著(P < 0.05)增加FS中qacH基因的相对丰度,并显著(P < 0.05)降低msrE基因的相对丰度,消减率可达80.9%,而AL和MO处理均能降低qacH和msrE基因的相对丰度,但较CK差异不显著,qacH和msrE基因的消减率分别为8.6%~20.5%和44.1%~45.6%(表 4,图 4b)。与CK相比,ET处理能够显著(P < 0.05)降低FS中tetG、tetM和tet36基因的相对丰度,其消减率分别达到78.3%、66.9% 和79.4%;而AL和MO处理对tetG和tet36基因相对丰度的消减作用较ET处理弱(表 4,图 4b)。AL和MO处理对FS中sul1基因的相对丰度无显著影响,而ET处理较CK能显著(P < 0.05)提高其相对丰度(表 4)。由表 4可知,RSD处理均能显著(P < 0.05)降低FS中IS6100和IS26基因的相对丰度,其消减率分别为72.5%~76.2%和38.4%~56.2%,且ET处理对IS26基因相对丰度的消减效果显著(P < 0.05)优于MO处理(图 4b,表 4)。此外,与CK相比,ET和AL处理均能显著(P < 0.05)降低FS中intl1基因的相对丰度,其中ET处理的消减率为59.0%,显著高于AL处理(30.5%),而MO处理对其无显著影响(表 4,图 4b)。综合而言,RSD处理对FS中青枯菌、ARGs和MGEs复合污染的修复效果较BS和RS强。
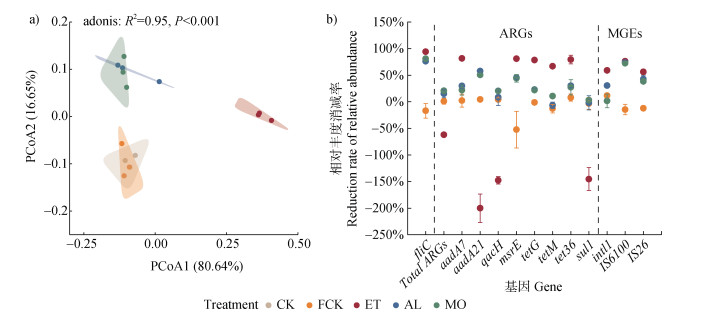
|
图 4 不同土壤处理对潮土中青枯菌、ARGs和MGEs污染特征(a)及消减率(b)的影响 Fig. 4 Effects of different soil treatments on the contamination characteristics (a) and reduction rates (b) of R. solanacearum, ARGs, and MGEs in fluvo-aquic soil |
|
|
表 4 不同土壤处理对潮土中青枯菌、ARGs和MGEs相对丰度的影响 Table 4 Effects of different soil treatments on the relative abundance of R. solanacearum, ARGs, and MGEs in fluvo-aquic soil |
双因素方差分析显示(表 5),除处理方式对IS6100基因相对丰度无显著影响外,处理方式、土壤类型及其交互作用均能显著(P < 0.05)影响土壤中青枯菌、ARGs和MGEs的相对丰度。其中,大部分ARGs(msrE、tetG、tetM、tet36和sul1)和MGEs(intl1、IS6100和IS26)的相对丰度受土壤类型的影响更为显著(F=11.4~755.4,P < 0.001),而处理方式对青枯菌、aadA7、aadA21和qacH基因相对丰度的影响更显著(F=17.1~90.4,P < 0.001)(表 5)。这些结果表明RSD处理对土壤生物复合污染的消减效果与土壤类型和有机物料类型密切相关,尤其是大部分ARGs和MGEs相对丰度的变化受土壤类型的影响更大。
|
|
表 5 土壤类型、处理方式及其交互作用对青枯菌、ARGs和MGEs相对丰度的影响 Table 5 Effects of soil types, treatments, and their interaction terms on the relative abundance of R. solanacearum, ARGs, and MGEs |
由表 6可知,不同土壤类型中青枯菌相对丰度与ARGs和MGEs相对丰度的相关性结果存在显著差异。在BS中,青枯菌相对丰度与sul1和IS6100基因的相对丰度呈正相关,但不显著;而在RS中,青枯菌相对丰度与aadA7和tet36基因的相对丰度呈正相关,其中与aadA7基因的关系极显著(P < 0.01),这表明RSD处理仅能协同消减BS和RS中青枯菌与少量ARGs和MGEs基因形成的复合污染。在FS中,青枯菌相对丰度与大部分ARGs(aadA7、msrE、tetG、tetM和tet36)和MGEs(intl1、IS6100和IS26)的相对丰度呈显著(P < 0.05)正相关关系,表明RSD处理对FS中病原菌与ARGs和MGEs叠加形成的生物复合污染具有较好的协同消减作用。同时,aadA7、msrE、tetG和tet36基因的相对丰度均与MGEs基因的相对丰度呈显著(P < 0.05)正相关,结合大部分RSD处理均能在一定程度上消减aadA7、msrE、tetG和tet36基因的相对丰度,表明RSD处理同样能够有效消减FS中ARGs和MGEs形成的复合污染。
|
|
表 6 不同类型土壤中青枯菌、ARGs和MGEs相对丰度的相关性 Table 6 Pearson correlation between the relative abundance of R. solanacearum, ARGs, and MGEs in different soils |
相关性分析结果表明(图 5),不同类型土壤中青枯菌、ARGs和MGEs的相对丰度与理化性质的相关性存在较大差异。在BS中,土壤pH与IS6100、aadA21、aadA7和tet36基因的相对丰度呈显著(P < 0.05)负相关,而土壤电导率(EC)与青枯菌、sul1和IS6100基因的相对丰度呈显著(P < 0.05)正相关(图 5a)。在RS中,土壤pH和NH4+-N含量与青枯菌和aadA7基因的相对丰度呈显著(P < 0.001)负相关,而与tetM和msrE基因的相对丰度呈显著(P < 0.001)正相关(图 5b)。如图 5c所示,FS的基本理化性质与青枯菌、ARGs和MGEs的相对丰度关系密切,其中土壤EC、AP和NO3–-N含量与青枯菌、大部分ARGs(aadA7、msrE、tetG、tetM和tet36)和MGEs(intl1、IS6100和IS26)的相对丰度均呈显著(P < 0.05)正相关关系,而土壤pH和NH4+-N含量与msrE、IS26、tetG、aadA7、tet36和intl1基因的相对丰度显著(P < 0.05)负相关。此外,土壤pH与青枯菌、tetM和IS6100基因的相对丰度呈显著(P < 0.05)负相关,而土壤NH4+-N含量与aadA21、qacH和sul1基因的相对丰度呈显著(P < 0.05)正相关。上述结果表明,RSD处理对不同土壤类型中生物复合污染的修复效果差异与其基本理化性质高度相关。
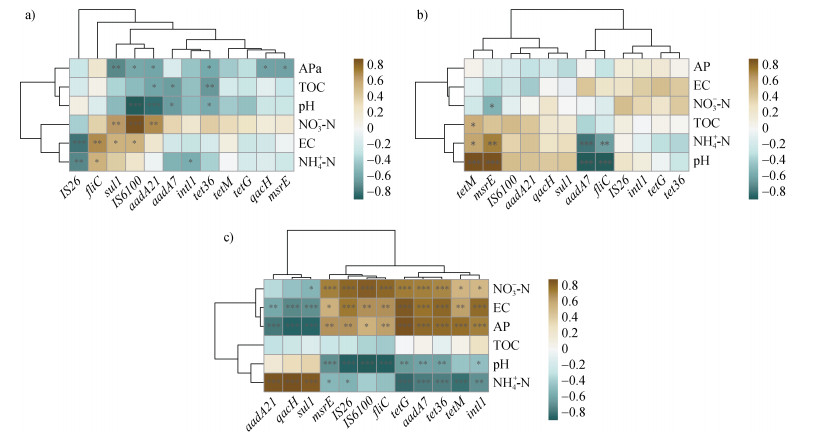
|
图 5 黑土(a)、红壤(b)和潮土(c)中土壤理化性质与青枯菌、ARGs及MGEs相对丰度的相关性分析 Fig. 5 Pearson correlation analyses between soil physicochemical properties and the relative abundance of R. solanacearum, ARGs, and MGEs in black soil (a), red soil (b), and fluvo-aquic soil (c) |
近年来,随着农业集约化尤其是设施农业的快速发展,由病原菌与ARGs和MGEs叠加形成的土壤生物复合污染日趋严重,已威胁到农产品安全与生态系统健康。本研究发现,RSD处理(AL、MO)对BS和FS中总ARGs的相对丰度均具有一定的消减作用,其中AL处理能够显著降低BS中aadA7、aadA21、tet36和sul1基因的相对丰度,消减率为28.4%~49.9%;而MO处理后FS中aadA21和tetG基因的相对丰度显著下降,其消减率为22.1%~50.5%。虽然ET处理对不同土壤类型中总ARGs的相对丰度无消减作用,但其能有效消减特定ARGs的相对丰度,如ET处理能够显著降低RS中aadA7和tet36基因的相对丰度,其消减率分别高达90.2%和80.8%。这与前人的研究结果类似,表明RSD处理能够通过杀灭携带ARGs的抗性菌实现土壤ARGs的快速消减[21]。除此之外,RSD处理对MGEs的相对丰度也具有较好地抑制作用,其中AL处理后,BS中IS6100基因的相对丰度下降幅度为43.9%,而ET处理能够显著降低FS中intl1、IS6100和IS26基因的相对丰度,其消减率可达56.2%~76.2%,表明RSD处理能够有效降低土壤中ARGs的水平转移能力和扩散风险[22]。
作为典型的土壤生物污染物,土传病原细菌青枯菌能够导致多种作物如番茄、马铃薯、茄子、烟草等罹患青枯病,是世界上危害最大的植物病害之一[23]。有研究表明,青枯菌入侵能够促进番茄根际土壤中ARGs和MGEs富集,加剧土壤生物复合污染发生风险[4]。本研究发现RSD处理能够有效降低不同土壤类型(BS、RS和FS)中青枯菌的相对丰度,其消减率为43.3%~94.2%,这与前人的研究结果一致[24-25],即RSD处理能够大幅降低病土中青枯菌的数量及相对丰度。RSD处理杀菌的作用机理主要包括:1)营造的高温、厌氧土壤环境;2)有机物料厌氧分解产生的如小分子有机酸等杀菌物质;3)重建的具有高丰度拮抗菌的健康微生物区系[6,26]。进一步分析发现,土壤中青枯菌的相对丰度与ARGs或MGEs的相对丰度存在一定的偶联关系,如FS中青枯菌的相对丰度与sul1和IS6100基因的相对丰度呈正相关关系,RS中青枯菌的相对丰度与aadA7和tet36基因的相对丰度呈正相关关系。此外,潮土中部分ARGs(aadA7、msrE、tetG和tet36)基因的相对丰度与intl1、IS6100和IS26等MGEs基因的相对丰度呈显著正相关。这些结果表明RSD处理能够协同消减青枯菌与ARGs和MGEs叠加形成的土壤生物复合污染。
3.2 RSD处理对土壤生物复合污染的协同修复效果与土壤类型和有机物料类型密切相关本研究发现,RSD处理能够显著改变BS、RS和FS中的土壤生物复合污染特征,其中FS中青枯菌的相对丰度与aadA7、msrE、tetG、tetM和tet36等ARGs和intl1、IS6100和IS26等MGEs基因的相对丰度呈显著正相关,而BS和RS中青枯菌的相对丰度仅与少量ARGs或MGEs基因的相对丰度存在正相关关系,这表明RSD处理对青枯菌与ARGs和MGEs叠加形成的土壤生物复合污染的修复效果与土壤类型密切相关。进一步分析发现,RSD处理对土壤生物复合污染的消减效果与土壤理化性质关系密切,其中,FS中青枯菌和大部分ARGs和MGEs基因的相对丰度与土壤EC和NO3–-N含量呈正相关关系,而与土壤pH呈负相关关系,这表明RSD处理能够通过改善土壤理化性质实现生物复合污染的协同消减[27-28]。此外,RSD处理后,BS中青枯菌的消减效果明显弱于RS和FS,这可能与BS中青枯菌的绝对丰度较RS和FS低有关。综上,造成不同土壤类型中RSD处理对土壤生物复合污染的消减效果存在差异的原因包括:1)土壤生物复合污染程度不同;2)土壤生物复合污染特征不同,尤其是ARGs和MGEs宿主不同引起的对RSD处理的耐受程度存在差异。
RSD处理对土壤生物复合污染的协同修复效果除受土壤类型的影响外,还受所用有机物料类型的影响。本研究发现采用不同有机物料进行的RSD处理对青枯菌的消减效果存在差异,其中ET处理对RS和FS中青枯菌相对丰度的消减率达到90.2%~94.2%,显著高于AL和MO处理的51.3%~80.9%,这可能与酒精属简单有机物,较苜蓿粉和糖蜜等复杂有机物更易分解,因而能够在相同时间内累积更高浓度的杀菌物质,实现土传病原菌的高效杀灭有关[29]。类似地,有机物料类型能够显著影响RSD处理对ARGs和MGEs的消减效果。在BS中,AL和MO处理对大部分ARGs基因的相对丰度消减率要优于ET处理,其中AL和MO处理对aadA7、aadA21和tet36基因的相对丰度的消减率分别为34.2%~40.6%、44.7%~49.9%和36.6%~41.%,明显高于ET处理的11.6%、20.7%和–7.4%。而在FS中,ET处理对大部分ARGs(aadA7、msrE、tetG、tetM和tet36)和MGEs(intl1、IS6100和IS26)基因相对丰度的消减效果高于AL和MO处理,但其能大幅增加aadA21、qacH和sul1基因的相对丰度,从而导致总ARGs的相对丰度较AL和MO处理高。这一方面与RSD处理对不同ARGs宿主的抑制/促进作用存在物料偏好性有关,另一方面与不同有机物料RSD处理介导的差异化微生物群落对ARGs宿主的间接抑制作用存在差异也有关联[16,30]。此外,同一有机物料对不同土壤类型中相同ARGs的作用效果也存在差异,这可能与同一ARGs基因在不同土壤类型中的宿主存在分异有关。
4 结论RSD处理对土壤病原菌与ARGs和MGEs叠加形成的生物复合污染具有一定的协同消减作用,其协同修复效果受污染程度、土壤类型和有机物料类型的影响,土壤类型对RSD处理消减土壤ARGs和MGEs的影响更为显著,而有机物料类型是影响RSD处理消减青枯菌相对丰度的主要因素。因此,在应用RSD处理技术消减土壤生物复合污染时,应综合考量土壤性质、生物复合污染特征及其污染程度、ARGs和MGEs类型及其宿主特征,以便选择适宜的有机物料类型并制订科学的修复策略,这样才能保障RSD处理对土壤生物复合污染的协同修复效果。
| [1] |
van Boeckel T P, Glennon E E, Chen D, et al. Reducing antimicrobial use in food animals[J]. Science, 2017, 357(6358): 1350-1352.
(  0) 0) |
| [2] |
Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92: 1-10.
(  0) 0) |
| [3] |
Zhang Y J, Ye C, Su Y W, et al. Soil acidification caused by excessive application of nitrogen fertilizer aggravates soil-borne diseases: Evidence from literature review and field trials[J]. Agriculture, Ecosystems & Environment, 2022, 340: 108176.
(  0) 0) |
| [4] |
Li Y C, Deng X H, Zhang N, et al. Rhizosphere suppression hinders antibiotic resistance gene (ARG) spread under bacterial invasion[J]. One Health, 2023, 16: 100481.
(  0) 0) |
| [5] |
Tokuda M, Shintani M. Microbial evolution through horizontal gene transfer by mobile genetic elements[J]. Microbial Biotechnology, 2024, 17(1): e14408.
(  0) 0) |
| [6] |
Cai Z C, Zhang J B, Huang X Q, et al. Application of reductive soil disinfestation to suppress soil-borne pathogens (In Chinese)[J]. Acta Pedologica Sinica, 2015, 52(3): 469-476. DOI:10.11766/trxb201411040554 [蔡祖聪, 张金波, 黄新琦, 等. 强还原土壤灭菌防控作物土传病的应用研究[J]. 土壤学报, 2015, 52(3): 469-476.]
(  0) 0) |
| [7] |
Zhao J, Zhang J Q, Lin Y L, et al. Correlation between the stability and function of soil microbial community driven by reductive soil disinfestation (In Chinese)[J]. Acta Pedologica Sinica, 2024, 61(1): 187-199. DOI:10.11766/trxb202205150254 [赵军, 张晶清, 林于蓝, 等. 强还原土壤处理驱动的微生物群落稳定性与功能的关联性[J]. 土壤学报, 2024, 61(1): 187-199.]
(  0) 0) |
| [8] |
Serrano-Pérez P, Rosskopf E, de Santiago A, et al. Anaerobic soil disinfestation reduces survival and infectivity of Phytophthora nicotianae chlamydospores in pepper[J]. Scientia Horticulturae, 2017, 215: 38-48.
(  0) 0) |
| [9] |
Chen Y L, Yang K J, Ye Y, et al. Reductive soil disinfestation attenuates antibiotic resistance genes in greenhouse vegetable soils[J]. Journal of Hazardous Materials, 2021, 420: 126632.
(  0) 0) |
| [10] |
Chen Y L, Zhang Y H, Xu R S, et al. Effects of anaerobic soil disinfestation on antibiotics, human pathogenic bacteria, and their associated antibiotic resistance genes in soil[J]. Applied Soil Ecology, 2024, 195: 105266.
(  0) 0) |
| [11] |
Liu L L, Kong J J, Cui H L, et al. Relationships of decomposability and C/N ratio in different types of organic matter with suppression of Fusarium oxysporum and microbial communities during reductive soil disinfestation[J]. Biological Control, 2016, 101: 103-113.
(  0) 0) |
| [12] |
Yuan X X, Liang J Y, Fan L X, et al. Effects of manure application on source and transport of antibiotic resistant genes in soil and their affecting factors (In Chinese)[J]. Acta Pedologica Sinica, 2020, 57(1): 36-47. DOI:10.11766/trxb201903110049 [苑学霞, 梁京芸, 范丽霞, 等. 粪肥施用土壤抗生素抗性基因来源、转移及影响因素[J]. 土壤学报, 2020, 57(1): 36-47.]
(  0) 0) |
| [13] |
Zhang J Y, Sui Q W, Tong J, et al. Soil types influence the fate of antibiotic-resistant bacteria and antibiotic resistance genes following the land application of sludge composts[J]. Environment International, 2018, 118: 34-43.
(  0) 0) |
| [14] |
Song T T, Zhu C X, Li B X, et al. Manure application led to higher antibiotic resistance risk in red soil compared with black soil and fluvo-aquic soil[J]. Journal of Hazardous Materials Advances, 2023, 9: 100209.
(  0) 0) |
| [15] |
Tian Q F, He Q S, Lu A X, et al. Relationship between antibiotic resistance genes and microbial communities in farmland soil (In Chinese)[J]. Environmental Chemistry, 2020, 39(5): 1346-1355. [田其凡, 何玘霜, 陆安祥, 等. 农田土壤抗生素抗性基因与微生物群落的关系[J]. 环境化学, 2020, 39(5): 1346-1355.]
(  0) 0) |
| [16] |
Yu W H, Li S, Lin Y L, et al. Effects of dazomet fumigation and reductive soil disinfestation onantibiotic resistance genes in farmland soil (In Chinese)[J]. Acta Pedologica Sinica, 2025, 62(1): 114-126. DOI:10.11766/trxb202309100369 [于文豪, 李舒, 林于蓝, 等. 棉隆熏蒸和强还原处理对农田土壤抗生素抗性基因的影响研究[J]. 土壤学报, 2025, 62(1): 114-126.]
(  0) 0) |
| [17] |
Dorsch M, Lane D, Stackebrandt E. Towards a phylogeny of the genus Vibrio based on 16S rRNA sequences[J]. International Journal of Systematic Bacteriology, 1992, 42(1): 58-63.
(  0) 0) |
| [18] |
Yuan S F, Li M Y, Fang Z Y, et al. Biological control of tobacco bacterial wilt using Trichoderma harzianum amended bioorganic fertilizer and the arbuscular mycorrhizal fungi Glomus mosseae[J]. Biological Control, 2016, 92: 164-171.
(  0) 0) |
| [19] |
Chen L, Xu Y L, Dong X F, et al. Removal of intracellular and extracellular antibiotic resistance genes in municipal wastewater effluent by electrocoagulation[J]. Environmental Engineering Science, 2020, 37(12): 783-789.
(  0) 0) |
| [20] |
Stedtfeld R D, Guo X P, Stedtfeld T M, et al. Primer set 2.0 for highly parallel qPCR array targeting antibiotic resistance genes and mobile genetic elements[J]. FEMS Microbiology Ecology, 2018, 94(9): fiy130.
(  0) 0) |
| [21] |
Duan H J, Yin Y, Wang Y F, et al. Effects of reductive soil disinfestation on potential pathogens and antibiotic resistance genes in soil[J]. Journal of Environmental Sciences, 2025, 150: 373-384.
(  0) 0) |
| [22] |
von Wintersdorff C J H, Penders J, van Niekerk J M, et al. Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer[J]. Frontiers in Microbiology, 2016, 7: 173.
(  0) 0) |
| [23] |
Jiang G F, Wei Z, Xu J, et al. Bacterial wilt in China: History, current status, and future perspectives[J]. Frontiers in Plant Science, 2017, 8: 1549.
(  0) 0) |
| [24] |
Wu C R, Lin W P, Huang F, et al. Effects of anaerobic soil disinfestation on bacterial wilt and soil bacterial community (In Chinese)[J]. Acta Pedologica Sinica, 2018, 55(4): 987-998. DOI:10.11766/trxb201712240611 [伍朝荣, 林威鹏, 黄飞, 等. 土壤厌氧消毒对青枯病的控制及土壤细菌群落结构的影响[J]. 土壤学报, 2018, 55(4): 987-998.]
(  0) 0) |
| [25] |
van Overbeek L, Runia W, Kastelein P, et al. Anaerobic disinfestation of tare soils contaminated with Ralstonia solanacearum biovar 2 and Globodera pallida[J]. European Journal of Plant Pathology, 2014, 138(2): 323-330.
(  0) 0) |
| [26] |
Li Y L, Wang B Y, Chang Y F, et al. Reductive soil disinfestation effectively alleviates the replant failure of Sanqi ginseng through allelochemical degradation and pathogen suppression[J]. Applied Microbiology and Biotechnology, 2019, 103(8): 3581-3595.
(  0) 0) |
| [27] |
Liu Z S, Zhao Y X, Zhang B F, et al. Deterministic effect of pH on shaping soil resistome revealed by metagenomic analysis[J]. Environmental Science & Technology, 2023, 57(2): 985-996.
(  0) 0) |
| [28] |
Xu Y, You G X, Yin J B, et al. Salt tolerance evolution facilitates antibiotic resistome in soil microbiota: Evidences from dissemination evaluation, hosts identification and co-occurrence exploration[J]. Environmental Pollution, 2023, 317: 120830.
(  0) 0) |
| [29] |
Momma N, Momma M, Kobara Y. Biological soil disinfestation using ethanol: Effect on Fusarium oxysporum f. sp. lycopersici and soil microorganisms[J]. Journal of General Plant Pathology, 2010, 76(5): 336-344.
(  0) 0) |
| [30] |
Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616.
(  0) 0) |
 2025, Vol. 62
2025, Vol. 62


