2. 中国科学院大学, 北京 100049;
3. 中国科学院大学南京学院, 南京 211135
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. University of Chinese Academy of Sciences, Nanjing, Nanjing 211135, China
青枯病是由青枯劳尔氏菌(Ralstonia solanacearum)引起的一种全球性的细菌性土传病害,发生于50多个科的200多种植物中,对农业经济发展具有不可忽视的危害性[1]。在众多寄主作物中,青枯菌对番茄作物的侵染尤为普遍[2]。当青枯菌从地下成功入侵番茄植株后,会造成番茄维管束组织的堵塞和损伤,使其无法有效从地下吸收水分和养分,从而引起叶片萎蔫和植株死亡,导致果实的减产甚至绝收[3]。由于番茄青枯病的发生隐匿于地下且发展迅速,给稳定高效的防病技术开发带来了巨大挑战。因此,深入研究青枯菌与寄主植物的互作过程,对于推进病害防控具有重要意义。
根际环境作为青枯菌入侵番茄作物的第一道屏障,很大程度上决定了病原菌的入侵过程与结果。根际是土壤与植物根系交界的微域,孕育了多样化的微生物体,包括细菌、真菌、原生动物和线虫等,大量的研究表明它们对病原菌存活和植物生长具有重要影响[4-5]。细菌群落中,存在着诸多有益微生物,如链霉菌(Streptomyces)、芽孢杆菌(Bacillus)和假单胞菌(Pseudomonas)等,能够通过产生抗生素、抢夺生态位、竞争养分和诱导植物抗性等方式抑制病原菌生长和病害发生[6-7]。真菌中的木霉菌(Trichoderma)和根霉菌(Gliocladium)、青霉菌(Penicillium)、黄曲霉菌(Aspergillus)也被报道对青枯病害有抑制效果[8-9]。此外,捕食性原生动物也会影响植物的健康状态[10],例如肾形虫(Colpoda)能够捕食青枯菌,抑制病害的发生[11]。
上述不同界的微生物在根际并不是相互独立的,它们之间存在着千丝万缕的联系,共同组成了根际复杂的微生物共生网络,与病原菌的入侵过程和植物健康状态紧密相关[12]。荧光假单胞菌(Pseudomonas fluorescens)通过与真菌互作可促进有益微生物的增殖,从而增强植物对根腐病的抵抗力[13]。丛枝菌根真菌(Arbuscular mycorrhizae)与假单胞菌和芽孢杆菌的联合应用能够协同促进植物的系统抗逆性,增强对病害的防效[14]。诸多研究表明,细菌和真菌群落的组成和功能在维持根际生态稳定和植物健康方面起到了重要的作用[5,15]。然而,关于青枯菌入侵下的根际细菌与真菌的互作研究相对较少,对病原菌入侵引起的根际跨界微生物生态变化需要更多的关注和研究。针对此,微生物共现网络分析(Co-occurrence network analysis)是合适的研究工具,其基于物种丰度计算相关性,构建微生物共生网络。通过节点的生物学分类和边的正负属性,能够有效阐释群落物种间的互作模式,帮助破译微生物群落的内部成员关联特性和整体结构特征。
为此,本研究通过采集番茄连作大棚中罹患青枯病与健康植株的根际土壤,利用荧光定量PCR(qPCR)和高通量扩增子测序技术检测根际土壤中细菌和真菌群落丰度和组成,并通过微生物网络关联技术分析细菌与真菌的生态互作关系,从而揭示病原菌入侵下的根际细菌与真菌群落的组成及其互作关联的变异,为番茄根系微生物的病害防控策略提供理论依据。
1 材料与方法 1.1 土壤样品的收集实验地位于江苏省南京市横溪街道南京蔬菜花卉研究所(31°43′N,118°46′E)中的番茄设施大棚。自2016年以来,该设施大棚长期进行单一的番茄栽培,一年两季。本研究于2021年7月收获期采集罹病和健康番茄植株的根际土壤样品。选择了10株出现严重枯萎症状(75%~100%的叶片枯萎或死亡)的番茄植株,以及10株毗邻患病植株而无病害症状的健康植株。将这些罹病和健康植株完整挖出,抖落根围的松散土壤后,小心收集贴附于根际的土壤,并通过2 mm筛网去除植物组织和碎屑。将收集到的根际土壤样品使用Fast DNA Spin试剂盒(MP Biomedicals,上海)进行DNA的提取,随后储存于–80℃冰箱,用于后续的扩增子测序和基因定量分析。
1.2 病原菌Ralstonia solanacearum、细菌和真菌的拷贝数定量检测病原菌R. solanacearum、细菌和真菌的数量通过实时荧光定量PCR技术检测。针对R. solanacearum的特异性扩增引物使用fliC-F(5'-GAACGCCAACGG TGCGAACT-3')和fliC-R(5'-GGCGGCCTTCAGG GAGGTC-3')[16];细菌的特异性引物为338F(5'-ACT CCTACGGGAGGCAGCAG-3')和518R(5'-ATTAC CGCGGCTGCTGG-3')[17];真菌的特异性引物为1737F(5'-GGAAGTAAAAGTCGTAACAAGG-3')和2043R(5'-GCTGCGTTCTTCATCGATGC-3')[18]。DNA样品的扩增采用20 μL的反应体系,包含2 μL的引物、7 μL的ddH2O和10 μL的SYBR Green Ⅰ核酸凝胶染料(Sigma,上海)。扩增反应条件为:95 ℃ 1 min;95 ℃ 15 s,60 ℃ 15 s,72 ℃ 30 s,循环40次;随后95 ℃ 5 s,60 ℃ 1 min;最后升温至95 ℃,持续15 s。在扩增周期结束时检测SYBR荧光信号。每个样品的检测包括三个技术重复。
1.3 细菌与真菌群落的扩增子测序提取的根际土壤DNA分别用于靶向细菌16S V3~V4区域和真菌ITS1区域进行基因扩增子测序。细菌16S rRNA基因的V3~V4区使用引物338F(5'-ACTCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')进行扩增[17];真菌ITS1区使用引物1737F(5'-GGAAGTAAAAGT CGTAACAAGG-3')和2043R(5'-GCTGCGTTC TTCATCGATGC-3')进行扩增[18]。文库的构建包括纯化、定量和标准化,以及后续的上机测序在上海派森诺生物科技股份有限公司的Illumina MiSeq平台上进行。下机的原始数据使用QIIME2软件进行分析,利用DADA2插件对数据进行过滤、去噪、聚类和去除嵌合体[19]。随后,从剩余序列中整合扩增子序列变体(ASVs),并使用SILVA数据库(版本138.1)进行序列比对获得各ASV的分类信息,生成的ASV丰度表供后续的微生物多样性分析[20]。
1.4 数据分析根据测序得到的细菌和真菌ASV数据,进行微生物多样性分析。主坐标分析(PCoA)用于展示样本群落组成的差异性,置换多因素方差分析(PERMANOVA)以及相似性分析(ANOSIM)用以检验组间群落差异的显著性,均通过R语言(版本4.0.2)的Vegan包执行。堆叠图、箱线图、线性拟合的分析和绘图在R语言中利用ggplot2包实现。线性判别效应量分析(Linear discriminant analysis effect size,LEfSe)用于鉴定群落间的关键差异物种,其核心指标线性判别分析分数(Linear discriminant analysis score,LDA score)用以量化物种在不同组间差异中的重要性,阈值设置为3.5,在R语言中利用microeco包执行。使用Cytoscape(版本3.8.2)进行物种网络维恩图的分析和可视化,用以呈现不同微生物组的独特ASV。
微生物的共现网络分析在Cytoscape软件中使用CoNet插件运行。将抽平后的细菌和真菌ASV数据表合并,保留相对丰度大于0.05%的ASV进行相关性分析。相关性计算方法选择斯皮尔曼(Spearman),相关性r阈值设置为0.7,显著性P阈值为0.05,矫正方法采用Benjamini-Hochberg方法。计算得到的总共现网络中,选择其中细菌与真菌节点的连线保留,得到细菌—真菌互作的共现子网络。得到的网络数据利用Gephi(版本0.9.2)软件进行可视化。节点的拓扑属性在R语言中利用psych和rnetcarto包通过计算模块内连通度(Zi)和模块间连通度(Pi)进行评估,节点属性分为4类:网络中心点(Zi > 2.5且Pi > 0.62),模块中心点(Zi > 2.5且Pi < 0.62),连接节点(Zi < 2.5且Pi > 0.62)和外围节点(Zi < 2.5且Pi < 0.62)。
2 结果 2.1 番茄健康状态影响根际细菌和真菌群落的结构与组成qPCR结果表明,罹病和健康番茄根际中存在的病原菌Ralstonia solanacearum数量存在显著差异(Student’s t-test,P < 0.001,图 1a)。罹病状态下,每克根际土壤中存在108至109的青枯菌拷贝数,而健康状态下的每克根际土壤中仅存有约107数量的青枯菌。群落分析的物种多样性结果(图 1b)表明,细菌香农指数(Shannon index)在健康番茄根际中显著高于其在罹病番茄根际中(P < 0.05),而真菌物种丰富度(Richness)则在罹病番茄根际中显著更高(P < 0.05)。PERMANOVA和ANOSIM分析以及基于Bray-curtis距离的细菌群落PCoA结果(表 1,图 1c)表明,罹病番茄与健康番茄的根际细菌群落存在显著差异(PERMANOVA检验统计值F. model = 3.779,P = 0.001;ANOSIM检验统计值R = 0.458,P = 0.002),样品在第一柱坐标成分(PCoA1,解释度:26.8%)上得到了明显的区分。细菌门水平组成上,罹病番茄根际中的变形菌门(Proteobacteria)相对丰度显著高于健康根际,而健康根际中的放线菌门(Actinobacteria)、髌骨菌门(Patescibacteria)、厚壁菌门(Firmicutes)和绿湾菌门(Chloroflexi)的相对丰度水平则显著更高(P < 0.05,图 1d)。在属水平上,病原菌劳尔氏菌属Ralstonia在罹病根际占绝对优势,而健康根际中的Saccharimonadales、芽孢杆菌属(Bacillus)和朱氏杆菌属(Chujaibacter)的相对丰度则显著更高(P < 0.05)。然而,对于真菌群落,罹病和健康的番茄根际群落间无显著差异(F. model = 1.389,P = 0.195;R = 0.054,P = 0.172),样品不能在PCoA图中得到明显的区分(图 1e)。在真菌群落组成上,门水平上以子囊菌门(Ascomycota)为主,属水平上则以淡紫紫霉属(Purpureocillium)和镰刀菌属(Fusarium)为主,但样品变异较大,在罹病和健康根际中无显著差异(P > 0.05,图 1f)。上述结果表明,番茄青枯病发生情况对根际细菌群落的物种多样性和组成具有显著影响,而对真菌的影响较小,仅体现在物种多样性的影响上。
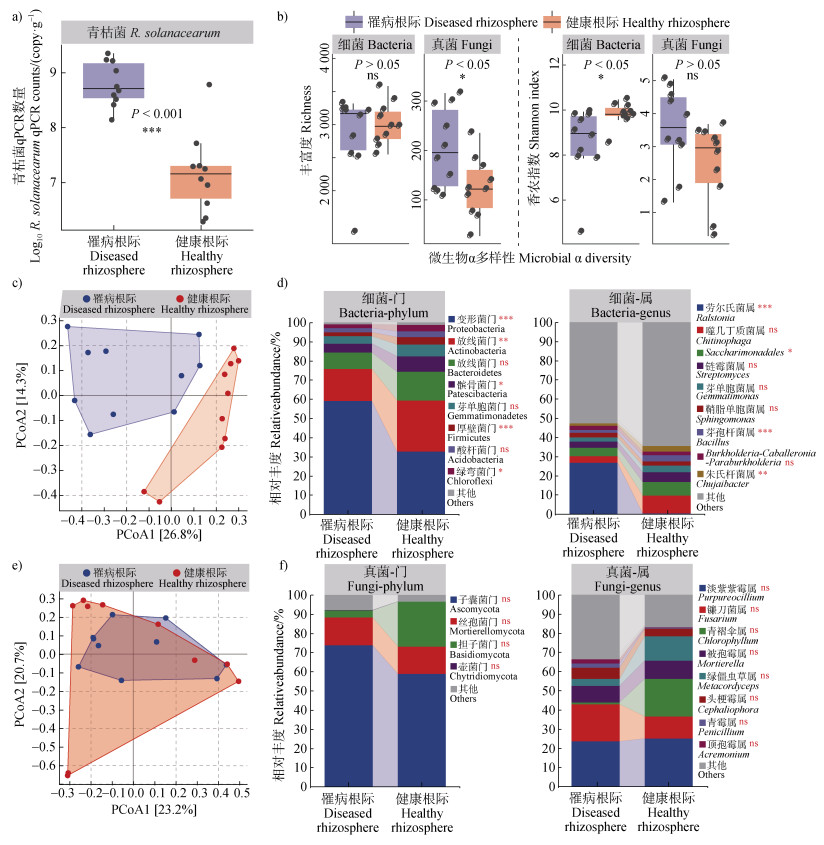
|
注:“*”“**”“***”分别表示差异检验(Student’s t-test)的显著性P值小于0.05、0.01、0.001;“ns”表示差异不显著,P值大于0.05。下同。Note:“*”“**”and “***” indicate that the significance P-value of Student’s t-test is less than 0.05,0.01,and 0.001,respectively;“ns” indicates a non-significant difference with a P-value greater than 0.05. The same as below. 图 1 罹病与健康番茄根际的细菌和真菌群落的结构与组成差异(a. 罹病和健康番茄根际中的R. solanacearum数量;b. 罹病和健康番茄根际中细菌和真菌的α多样性(丰富度和香农多样性指数);罹病和健康番茄根际中细菌(c)和真菌(e)群落主坐标分析(PCoA);罹病和健康番茄根际中细菌(d)和真菌(f)群落的门及属水平组成) Fig. 1 Differences in the structure and composition of bacterial and fungal communities in the rhizosphere of diseased and healthy tomato plants(a. The qPCR amounts of R. solanacearum in the rhizosphere of diseased and healthy tomatoes; b. The α diversity(richness and Shannon diversity index)of bacterial and fungal communities in the rhizosphere soils of diseased and healthy tomatoes; Principal coordinate analysis(PCoA)of bacterial(c)and fungal(e)communities in the rhizosphere of diseased and healthy tomato plants; Bacterial(d)and fungal(f)community composition in the rhizosphere of diseased and healthy tomato plants at the phylum or genus level) |
|
|
表 1 罹病和健康番茄根际细菌和真菌群落的显著性差异检验 Table 1 The significance test of the differences between bacterial and fungal communities in the rhizosphere of diseased and healthy tomato plants |
对于细菌群落进一步分析,通过网络维恩图(图 2a)发现,罹病根际相对于健康根际存在更多特异Proteobacteria门的ASV,数量达到327个,而健康根际中Proteobacteria门下的特异ASV仅有152个;相反,Actinobacteria中特异的ASV则在健康根际中更多,存在191个,高于其在罹病根际中的52个。LEfSe分析有类似的结果(图 2b),罹病根际中富集了Proteobacteria门及其下属的物种,而健康根际中富集的细菌则以Actinobacteria和Firmicutes为主。
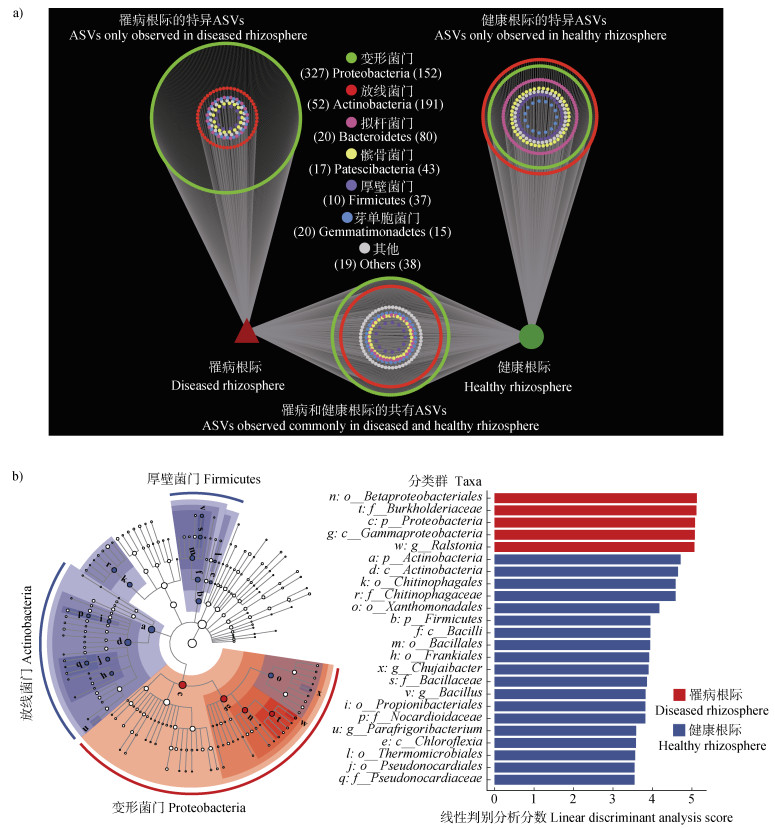
|
图 2 罹病与健康番茄根际细菌的特异物种及富集物种差异(a. 网络韦恩图展示罹病和健康番茄根际中的特异细菌物种;b. 线性判别效应分析(LEfSe)展示罹病和健康番茄根际中的富集细菌) Fig. 2 Differences in specific and enriched bacterial species in the rhizosphere of diseased and healthy tomatoes(a. Venn network diagram showing specific bacterial species in the rhizosphere of diseased and healthy tomato plants; b. Linear discriminant analysis effect size(LEfSe)showing enriched bacterial species in the rhizosphere of diseased and healthy tomato plants) |
荧光定量(qPCR)检测结果表明,罹病番茄根际中的细菌总数量与健康番茄根际相比无显著差异(Student’s t-test,P > 0.05,图 3a)。与细菌相反,真菌在罹病番茄根际中的数量要显著低于其在健康番茄根际中的数量(P < 0.05)。此外,真菌的物种生态位(P < 0.05)和群落生境生态位(P < 0.01)在罹病番茄根际中也处于更低的水平,显著低于健康番茄根际(图 3b)。上述结果表明,在番茄患病状态下,根际环境中真菌的生存空间受到了一定程度的限制。进一步分析真菌总数量和生境生态位与病原菌的关联(图 3c),发现它们均与青枯菌数量呈显著的负相关关系,拟合R2值分别为0.379(P < 0.01)和0.523(P < 0.001),这表明青枯菌的入侵是真菌生存空间降低的重要原因。
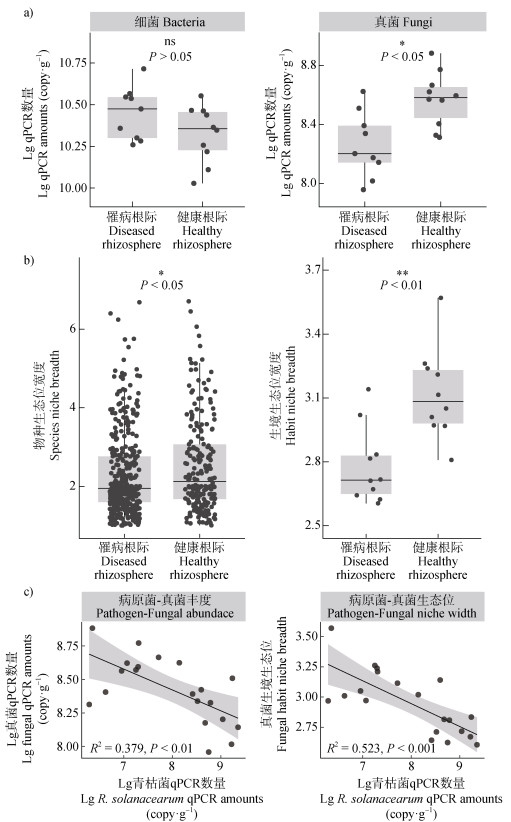
|
图 3 罹病与健康番茄根际的微生物丰度与生态位差异(a. 罹病和健康番茄根际中细菌和真菌的qPCR数量;b. 罹病和健康番茄根际中真菌的物种生态位宽度和群落生境生态位宽度;c. 病原菌qPCR数量与真菌qPCR数量和生境生态位宽度的相关性) Fig. 3 The difference in microbial abundance and niche width in the rhizosphere of diseased and healthy tomatoes(a. The qPCR amounts of bacteria and fungi in the rhizosphere of diseased and healthy tomato plants; b. Species niche breadth and habitat niche breadth of fungi in the rhizosphere of diseased and healthy tomato plants; c. Correlation fitting analysis between pathogen qPCR amounts and fungal qPCR amounts and habitat niche breadth) |
对番茄根际的细菌和真菌群落进行网络整合分析,得到罹病和健康状态下两种不同的网络(图 4a)。罹病的根际网络具有168个节点、725条边,健康根际网络具有156个节点和517条边。在节点分布上,罹病状态下的网络中,真菌节点的数量占总网络节点的48.2%;而在健康状态下,真菌节点数量的占比明显减小,仅占25.6%(图 4b)。对于网络连接,罹病和健康状态下的网络均以正相关连接为主导,占比分别达到70.2%和65.6%。Zi-Pi结果(图 4c)表明,健康根际网络中含有15个连接节点(Connector),其中4个属于Proteobacteria门,3个属于Patescibacteria门,2个属于Ascomycota门;而罹病根际网络中的关键节点则主要为Ascomycota门下物种,含有1个模块中心点(Connector hub)和4个连接节点。进一步关注网络连线(图 4d),发现细菌内部连线(细菌—细菌)、真菌内部连线(真菌—真菌)和细菌—真菌间连线在罹病和健康状态的网络中占不同比例。相较于健康状态,罹病状态下的根际网络中真菌—真菌和细菌—真菌的网络连接占比明显提升。对细菌与真菌子网络(细菌—真菌)深入分析(图 4e),发现罹病状态下子网络具有137个节点,其中46.7%为真菌节点,而健康状态下仅有100个节点,且仅有31%的真菌节点。在细菌—真菌子网络的关联性上(图 4f),罹病状态下的边有281条,高于健康状态下的164条,并且罹病子网络的负相关连接占比达46.3%,要高于其在健康状态下的35.4%,说明病原菌的成功入侵使细菌和真菌的关联性更加复杂,且向负向关系发展。
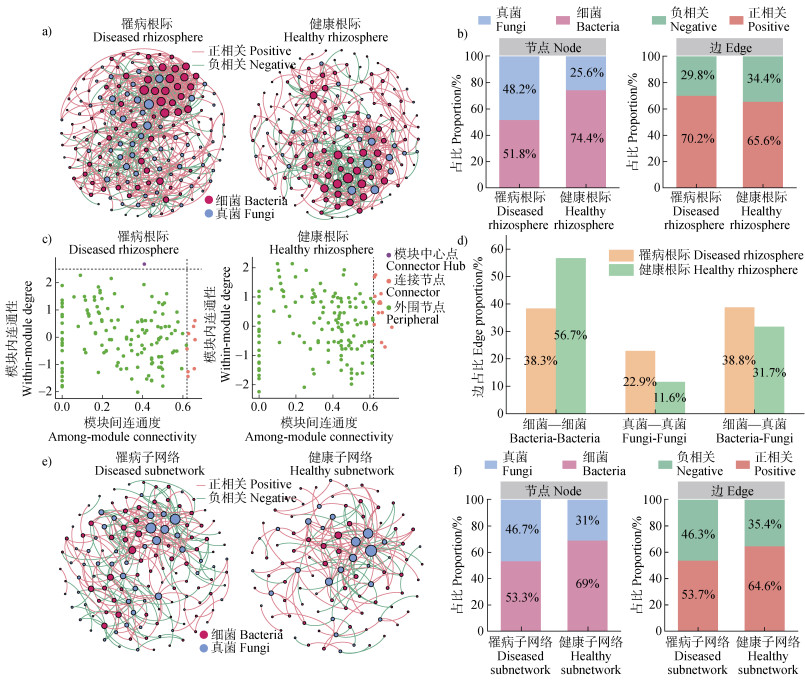
|
图 4 罹病和健康番茄根际的微生物网络分析(a. 罹病和健康番茄根际中的细菌—真菌的整体微生物网络;b. 罹病和健康根际中整体微生物网络中的节点与边的属性占比;c. 罹病和健康根际微生物网络节点的模块内与模块间的连通度(Zi-Pi)图;d. 罹病和健康根际中不同连接方式(细菌—细菌、真菌—真菌、细菌—真菌)在整体微生物网络中的占比;e. 罹病和健康番茄根际中的细菌与真菌互作的子网络;f. 罹病和健康根际中细菌—真菌互作子网络中的节点与边的属性占比) Fig. 4 Network analysis of rhizosphere microbial communities in the diseased and healthy tomato plants(a. Overall bacteria-fungi networks in the rhizosphere of diseased and healthy tomato plants; b. Distribution of node and edge attributes in the microbial networks in the rhizosphere of diseased and healthy tomato plants; c. Within-module degree and among-module connectivity(Zi-Pi)plots of nodes in the diseased and healthy rhizosphere microbial networks; d. Proportions of different connection types(bacteria-bacteria, fungi-fungi, bacteria-fungi)in the overall microbial networks in the rhizosphere of diseased and healthy tomato plants; e. Sub-networks of bacteria-fungi interactions in the rhizosphere of diseased and healthy tomato plants; f. Distribution of node and edge attributes in the bacteria-fungi interaction sub-networks in the rhizosphere of diseased and healthy tomato plants) |
堆叠图表明,罹病和健康状态下的细菌—真菌子网络的节点分类组成上有明显差异(图 5a)。对于细菌节点,罹病状态下Proteobacteria、Actinobacteria和拟杆菌门(Bacteroidetes)的节点数量分别为31、19和12个,而健康状态下Actinobacteria的节点数量最多,存在22个。真菌节点的差异主要体现在子囊菌门(Ascomycota)上,罹病的细菌—真菌子网络存在41个Ascomycota节点,相对应其在健康状态下仅有15个。进一步的节点丰度分析(图 5b)显示,健康状态子网络中的放线菌节点在健康根际群落中有更高的丰度水平(Student’s t-test,P < 0.01)。而对于真菌节点(图 5c),罹病状态子网络中的Ascomycota节点在罹病根际群落中表现出显著更高的丰度(P < 0.001)。上述结果表明,Ascomycota和Actinobacteria分别是罹病和健康状态下细菌—真菌子网络中的关键类群,它们在对应植株状态下的富集驱动了细菌和真菌的关联互作。
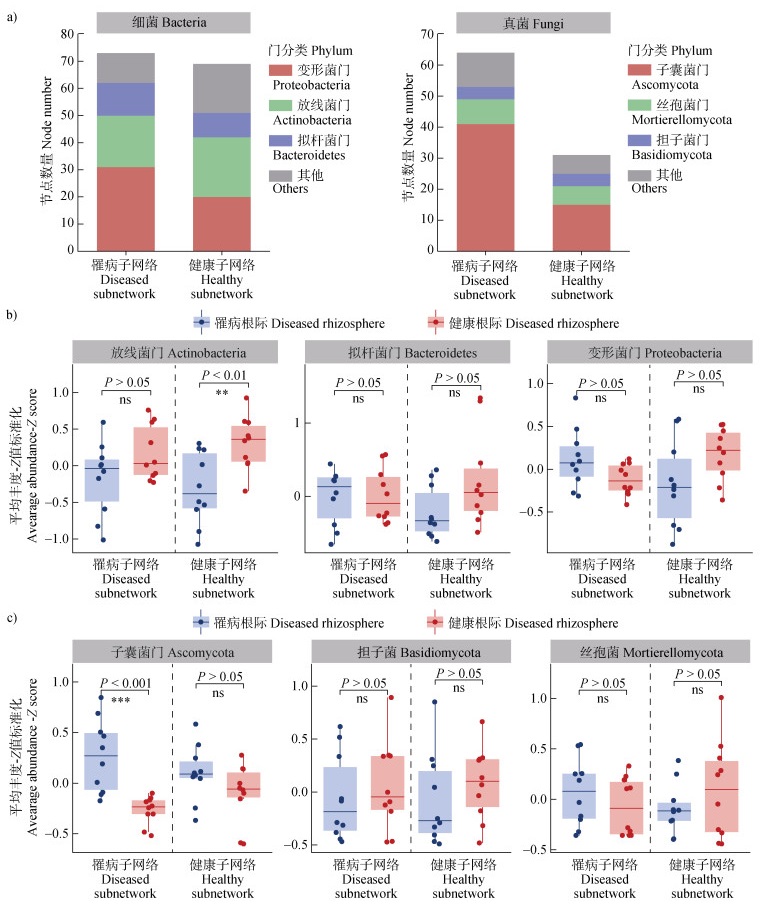
|
图 5 细菌—真菌互作子网络中的节点分类及丰度变化(a. 罹病和健康根际中细菌—真菌互作子网络中细菌和真菌节点的分类学组成;罹病和健康根际中细菌—真菌互作子网络中的不同细菌(b)和真菌(c)门水平节点在不同健康状态下的丰度变化) Fig. 5 Classification and abundance changes of nodes in the bacteria-fungi interaction sub-network(a. Taxonomic composition of bacterial and fungal nodes in the bacteria-fungi interaction sub-network in the rhizosphere of diseased and healthy tomato plants; Changes in the abundance(Z-score)of nodes attributed to different bacterial(b)and fungal(c)phylum in the bacteria-fungi interaction sub-networks in the rhizosphere of diseased and healthy tomato plants) |
进一步的拟合分析以验证子网络中不同门的节点对病原菌入侵的响应。结果(图 6)表明,在罹病根际的细菌—真菌子网络中(图 6a),Ascomycota的节点是对青枯菌入侵响应最强的真菌节点(R2 = 0.393,P < 0.01),且与病原菌数量呈正相关关系,表明病原菌入侵促进了细菌—真菌互作中Ascomycota相关物种的生存优势。而在健康根际的细菌—真菌子网络中(图 6b),Actinobacteria节点与青枯菌关联性最强,与病原菌数量呈现显著的负相关关系(R2 = 0.523,P < 0.001)。上述结果进一步表明,Actinobacteria和Ascomycota分别是病原菌入侵下的根际细菌—真菌互作中的关键细菌和真菌类群,且它们可作为病原菌入侵及植物健康状态的重要指示生物。
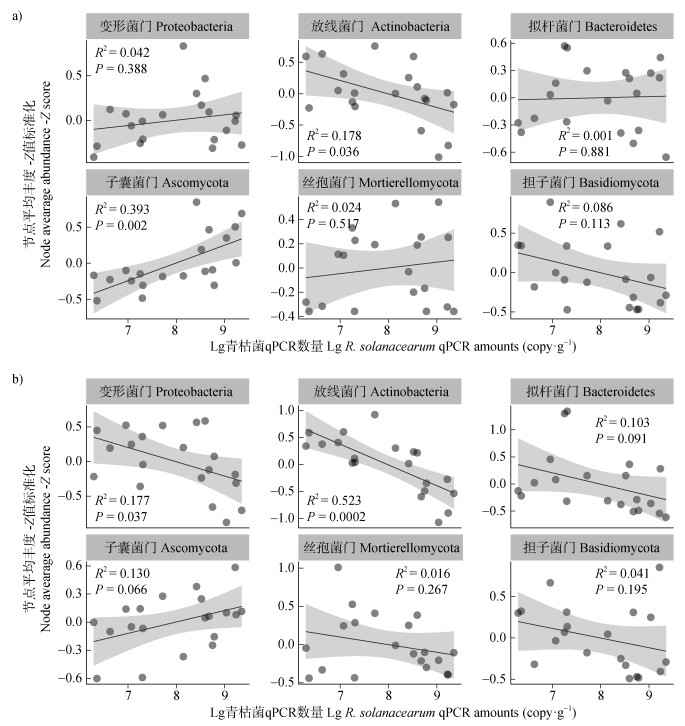
|
图 6 罹病(a)和健康(b)根际细菌—真菌互作子网络中的不同门类节点丰度与病原菌的相关性拟合 Fig. 6 Correlation fitting analysis between the abundance(Z-score)of bacterial and fungal nodes at different phylum levels in the bacteria-fungi interaction sub-network and pathogen qPCR amounts in the rhizosphere of diseased(a)and healthy(b)tomato plants |
青枯菌(R. solanacearum)作为病原细菌,对植株的成功入侵势必引起根际细菌群落的相应变化。本研究中发现,罹病番茄根际中检测到了更多的R. solanacearum数量,且健康番茄根际的细菌多样性更高(图 1a,图 1b),这可能是由于R. solanacearum的大量侵入引起了根际细菌群落的失衡。罹病和健康番茄根际细菌群落的结构与组成也具有显著差异,表明病原菌入侵对根际细菌群落的构成具有重要影响[21-22]。在罹病番茄根际细菌群落中,富集了更多的Proteobacteria的物种,而健康番茄根际中则富集了更多的Actinobacteria和Firmicutes物种(图 2)。Lee等[23]发现Actinobacteria和Firmicutes在根际中的存在是番茄抵御病原菌入侵和维持健康状态的关键。并且,诸多Actinobacteria和Firmicutes门中的物种如Streptomyces和Bacillus均被广泛报道能产生多种具有抗生性的次级代谢产物,常用于农业病害的防控研究与实践中[24-25]。它们的富集可能代表了根际微生物群落的抑病潜力,决定了病原菌是否能够成功侵染番茄寄主。
而真菌群落组成上,尽管罹病和健康番茄根际间无显著性差异(图 1e,图 1f),但在真菌多样性、数量和生态位上表现出了显著差异(图 3),表明R. solanacearum的入侵对真菌群落的整体发展上有较强的干扰。这可能是因为细菌和真菌会共同竞争土壤碳和能量资源[15],而细菌群落整体在内部R. solanacearum大量扩繁的情况下,向外作出资源与空间的侵略和掠夺,从而压缩了真菌在根际环境中的生存空间。
3.2 Actinobacteria和Ascomycota分别是细菌—真菌互作网络中的关键细菌与真菌类群网络分析表明,罹病状态下的细菌—真菌网络结构较健康状态更为复杂,节点数量和连线数量均有所增加(图 4),这可能是由于罹病情况下番茄植株会采用呼叫策略(Cry for help)调节根系分泌物的组成和数量,使得根际环境资源更为复杂,进而增强了微生物的种间互作[26-27]。此外,病原菌入侵压缩了真菌的生态空间,使得真菌相关的竞争作用加剧,这可能解释了网络中真菌节点数量增加,以及真菌相关连线(细菌—真菌,真菌—真菌)占比提高的现象(图 4d)。同时,细菌—真菌子网络中更高比例的负相关连线(图 4f)也表明病原菌的入侵加强了细菌与真菌间的负向竞争作用。
在罹病和健康状态下Ascomycota和Actinobacteria分别是真菌和细菌共现网络中的关键类群(图 5),它们的丰度变化可能是驱动群落结构变化以及影响细菌—真菌互作的主要因素。有研究表明,在植物组织的分解过程中,Ascomycota对植株组织的特异性响应较高,是负责细菌与真菌相互关联的主要物种[28]。而Actinobacteria门的物种则被报道对多种真菌物种具有拮抗能力[26,29]。这些功能特性可能是它们主导网络结构变化的重要原因。进一步的拟合分析(图 6)证明,这种网络变化的驱动力来源于病原菌的入侵。R. solanacearum的入侵使得Ascomycota物种在真菌内部的生态位竞争中占据优势,类似的研究也报道了Ascomycota门下镰刀菌(Fusarium spp.)在罹病植株根际的富集[30]。相反,健康根际子网络的Actinobacteria节点与病原菌呈负相关,说明其可能是抵御病原菌入侵的重要保障。上述结果表明,R. solanacearum不仅通过直接侵染导致病害,还可能通过扰动根际细菌与真菌群落的关键类群与互作结构,间接影响病害的发生。
然而,本研究展现的细菌—真菌网络共现模式虽然从生态学角度上解释了病原菌入侵对根际微生物跨界互作的影响,但其中涉及的具体物种互作机制仍然难以明确,无法通过简单的相关性数值体现。这些种间互作关乎根际微生物群落的功能与稳定,对抵御病原菌的入侵和维持植物健康均起到了尤为重要的作用[5,31]。因此,针对这方面的不足尚需更深入的室内机理研究,从而为病害的防控与技术发展提供更全面的理论基础。
4 结论本研究通过分析罹病和健康番茄根际细菌与真菌群落组成及关联网络结构,揭示了病原菌R. solanacearum入侵对根际微生物群落的复杂影响。结果表明,病原菌入侵会压缩真菌在根际的生存空间,与此同时,病原菌会通过影响Actinobacteria门和Ascomycota门中的关键物种,进而改变细菌和真菌的关联网络。该发现对理解植物病害与根际微生物群落之间的关系具有重要意义,尤其是在揭示病原菌如何通过影响关键微生物节点来改变根际跨界微生物群落方面。未来研究应进一步探讨不同作物系统和环境条件下病原菌入侵对根际微生物群落结构和功能的影响,并探索如何通过调控关键类群微生物来增强植物的抗病性。
| [1] |
Guo J H, Qi H Y, Guo Y H, et al. Biocontrol of tomato wilt by plant growth-promoting rhizobacteria[J]. Biological Control, 2004, 29(1): 66-72.
(  0) 0) |
| [2] |
Wang J, Long S F, Wang Z W, et al. Research progress in controlling tomato bacterial wilt (In Chinese)[J]. China Vegetables, 2020(1): 22-30. [王杰, 龙世芳, 王正文, 等. 番茄青枯病防治研究进展[J]. 中国蔬菜, 2020(1): 22-30.]
(  0) 0) |
| [3] |
Kwak M J, Kong H G, Choi K, et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature Biotechnology, 2018, DOI: 10.1038/nbt.4232.
(  0) 0) |
| [4] |
Boro M, Sannyasi S, Chettri D, et al. Microorganisms in biological control strategies to manage microbial plant pathogens: A review[J]. Archives of Microbiology, 2022, 204(11): 666.
(  0) 0) |
| [5] |
Philippot L, Raaijmakers J M, Lemanceau P, et al. Going back to the roots: The microbial ecology of the rhizosphere[J]. Nature Reviews Microbiology, 2013, 11(11): 789-799.
(  0) 0) |
| [6] |
Singh H B. Management of plant pathogens with microorganisms[J]. Proceedings of the Indian National Science Academy, 2014, 80(2): 443-454.
(  0) 0) |
| [7] |
Ma C, Yang X R, Jiang G F, et al. Research progresses on key factors affecting survival of Ralstonia solanacearum in soils (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(6): 1359-1367. [马超, 杨欣润, 江高飞, 等. 病原青枯菌土壤存活的影响因素研究进展[J]. 土壤学报, 2021, 58(6): 1359-1367.]
(  0) 0) |
| [8] |
Konappa N, Krishnamurthy S, Siddaiah C N, et al. Evaluation of biological efficacy of Trichoderma asperellum against tomato bacterial wilt caused by Ralstonia solanacearum[J]. Egyptian Journal of Biological Pest Control, 2018, 28(1): Article number 63.
(  0) 0) |
| [9] |
Gafur A. Development of biocontrol agents to manage major diseases of tropical plantation forests in Indonesia: A review[J]. Environmental Sciences Proceedings, 2021, 3(1): 11. DOI:10.3390/IECF2020-07907
(  0) 0) |
| [10] |
Wei Z, SongY Q, Xiong W, et al. Soil protozoa: Research methods and roles in the biocontrol of soil-borne diseases (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(1): 14-22. [韦中, 宋宇琦, 熊武, 等. 土壤原生动物——研究方法及其在土传病害防控中的作用[J]. 土壤学报, 2021, 58(1): 14-22.]
(  0) 0) |
| [11] |
Guo S, Jiao Z, Yan Z, et al. Predatory protists reduce bacteria wilt disease incidence in tomato plants[J]. Nature Communications, 2024, 15(1): 829.
(  0) 0) |
| [12] |
Deveau A, Bonito G, Uehling J, et al. Bacterial–fungal interactions: Ecology, mechanisms and challenges[J]. FEMS Microbiology Reviews, 2018, 42(3): 335-352.
(  0) 0) |
| [13] |
Saravanakumar D, Lavanya N, Muthumeena B, et al. Pseudomonas fluorescens enhances resistance and natural enemy population in rice plants against leaffolder pest[J]. Journal of Applied Entomology, 2008, 132(6): 469-479.
(  0) 0) |
| [14] |
Li F X, Duan T Y. Research progress in the control of plant diseases by the combination of fungi and beneficial microorganisms (In Chinese)[J]. Chinese Journal of Grassland, 2021, 43(8): 93-105. [李法喜, 段廷玉. 真菌和有益微生物联合防治植物病害研究进展[J]. 中国草地学报, 2021, 43(8): 93-105.]
(  0) 0) |
| [15] |
Wang C Q, Kuzyakov Y. Mechanisms and implications of bacterial–fungal competition for soil resources[J]. The ISME Journal, 2024, 18(1): wrae073.
(  0) 0) |
| [16] |
Schönfeld J, Heuer H, Van Elsas J D, et al. Specific and sensitive detection of Ralstonia solanacearum in soil on the basis of PCR amplification of fliC fragments[J]. Applied and Environmental Microbiology, 2003, 69(12): 7248-7256.
(  0) 0) |
| [17] |
Mori H, Maruyama F, Kato H, et al. Design and experimental application of a novel non-degenerate universal primer set that amplifies prokaryotic 16S rRNA genes with a low possibility to amplify eukaryotic rRNA genes[J]. DNA Research, 2014, 21(2): 217-227.
(  0) 0) |
| [18] |
Degnan P H, Ochman H. Illumina-based analysis of microbial community diversity[J]. The ISME Journal, 2012, 6(1): 183-194.
(  0) 0) |
| [19] |
Hall M, Beiko R G. 16S rRNA gene analysis with QIIME2[J]. Methods in Molecular Biology, 2018, 1849: 113-129.
(  0) 0) |
| [20] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D596.
(  0) 0) |
| [21] |
Kuang L, Li T, Wang B, et al. Diseased-induced multifaceted variations in community assembly and functions of plant-associated microbiomes[J]. Frontiers in Microbiology, 2023, 14: 1141585.
(  0) 0) |
| [22] |
Liu H, Dong Y H, Shen M C, et al. Characteristics of rhizosphere microbial communities in a disease- suppressive soil of tomato bacterial wilt and its disease-suppressive transmission mechanism (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(4): 1125-1135. [刘洪, 董元华, 申民翀, 等. 番茄青枯病抑病土壤根际微生物群落特征及其抑制性传递机制[J]. 土壤学报, 2022, 59(4): 1125-1135.]
(  0) 0) |
| [23] |
Lee S M, Kong H G, Song G C, et al. Disruption of Firmicutes and Actinobacteria abundance in tomato rhizosphere causes the incidence of bacterial wilt disease[J]. The ISME Journal, 2021, 15(1): 330-347.
(  0) 0) |
| [24] |
Barka E A, Vatsa P, Sanchez L, et al. Taxonomy, physiology, and natural products of Actinobacteria[J]. Microbiology and Molecular Biology Reviews, 2016, 80(1): 1-43.
(  0) 0) |
| [25] |
Palaniyandi S A, Yang S H, Zhang L, et al. Effects of Actinobacteria on plant disease suppression and growth promotion[J]. Applied Microbiology and Biotechnology, 2013, 97(22): 9621-9636.
(  0) 0) |
| [26] |
Huang X Q, Liu S Z, Liu X, et al. Plant pathological condition is associated with fungal community succession triggered by root exudates in the plant-soil system[J]. Soil Biology and Biochemistry, 2020, 151: 108046.
(  0) 0) |
| [27] |
You C, Yang T J, Zhou X G, et al. Research advances on mechanisms and preventions of soil-borne diseases exacerbated by root exudates in continuous cropping systems (In Chinese)[J]. Acta Pedologica Sinica, 2024, 61(5): 1201-1211. [游川, 杨天杰, 周新刚, 等. 连作根系分泌物加剧土传病害的机制和缓解措施研究进展[J]. 土壤学报, 2024, 61(5): 1201-1211.]
(  0) 0) |
| [28] |
Zhao B, Xing P, Wu Q L. Interactions between bacteria and fungi in macrophyte leaf litter decomposition[J]. Environmental Microbiology, 2021, 23(2): 1130-1144.
(  0) 0) |
| [29] |
Sharma P, Aswini K, Prasad J S, et al. Characterization of Actinobacteria from wheat seeds for plant growth promoting traits and protection against fungal pathogens[J]. Journal of Basic Microbiology, 2023, 63(3/4): 439-453.
(  0) 0) |
| [30] |
Li T T, Deng X H, Li R C, et al. Effects of Ralstonia solanacearum infection on soil fungal community diversity (In Chinese)[J]. Biotechnology Bulletin, 2022, 38(10): 195-203. [李婷婷, 邓旭辉, 李若尘, 等. 番茄青枯病发生对土壤真菌群落多样性的影响[J]. 生物技术通报, 2022, 38(10): 195-203.]
(  0) 0) |
| [31] |
Jambon I, Thijs S, Weyens N, et al. Harnessing plant-bacteria-fungi interactions to improve plant growth and degradation of organic pollutants[J]. Journal of Plant Interactions, 2018, 13(1): 119-130.
(  0) 0) |
 2025, Vol. 62
2025, Vol. 62


