2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
阿特拉津(Atrazine,商品名:莠去津)是一种人工合成的三嗪类除草剂,广泛用于玉米、高粱、甘蔗等作物的杂草控制。在农业生产中长期大量施用阿特拉津易导致土壤残留,引起后茬敏感作物的药害[1]。同时,阿特拉津干扰内分泌、神经系统,影响生物多样性[2]、生态系统结构和功能[3],具有人体健康及生态风险[4]。因此,消除农田土壤中残存的阿特拉津是保障粮食安全、生态环境以及人体健康的重要举措。
微生物经脱氯、脱烷基、开环等过程降解阿特拉津[5-6]。已经分离获得多个降解菌株,如Pseudomonas sp. ADP、Arthrobacter aurescens TC1等,它们编码阿特拉津氯水解酶(Atrazine chlorohydrolase,AtzA)或三嗪水解酶(Triazine hydrolase,TrzN)两种在结构、催化机制等方面存在重大差异的脱氯酶[7]。需要指出阿特拉津降解中间产物的毒性可能较母体更高[8],因此,污染物矿化量(即转化为CO2的量)较土壤残留浓度更适宜作为评估其解毒程度的指标。另一方面,土壤微生物高度多样,可能存在复杂降解行为。例如,降解菌和非降解菌通过代谢互养(Cross-feeding)协同降解阿特拉津[9]。通过分析土壤微生物的群落响应,可获得阿特拉津生物降解机制的线索。
不同来源土壤中阿特拉津的降解可能存在明显差异[10],但目前尚缺乏针对土壤类型效应的比较研究。黑土、潮土和红壤广泛分布于我国东北、华北和南方,总计超过全国耕地面积的65%[11-12]。这些地区玉米等作物种植中阿特拉津的使用较为普遍,导致局部土壤阿特拉津残留浓度较高[13]。当前对土壤阿特拉津降解的研究多以单一土壤类型为主[14-16],限于方法、设置等,不同研究结果间难以比较,阿特拉津在不同类型土壤中的转化规律、生态效应尚不清楚。基于此,本研究选择黑土、潮土和红壤区旱地农田土壤,综合采用同位素示踪、微生物群落分析等技术手段研究阿特拉津的降解和微生物效应及其机制,以期为不同土壤类型区合理使用除草剂、有效治理农药残留提供科学依据。
1 材料与方法 1.1 供试土壤供试土壤分别为采自吉林省农安县的黑土(44°4′ N,125°7′ E,均腐土,记为BS)、河南省通许县的潮土(37°27′ N,114°32′ E,雏形土,记为FA)、云南省昌宁县的红壤(24°27′ N,99°38′ E,淋溶土,记为RS)农田。采样时田块均种植玉米,经调查均有使用阿特拉津的历史,但三种土壤均未检出阿特拉津残留。采用五点采样法采集表层0~10 cm土壤,剔除土壤中的砂砾、石块以及动植物残体等杂质,充分混匀,自然风干后过2 mm筛备用。三种土壤的理化性质如表 1所示。
|
|
表 1 供试土壤理化性质 Table 1 Physicochemical properties of the soils |
14C标记阿特拉津购自American Radiolabeled Chemicals,Inc.,12C-阿特拉津(纯度大于97%)购自梯西爱(上海)化成工业发展有限公司,其他所用试剂均为分析纯或以上。
1.3 试验设计设置两组平行试验,采用土壤微宇宙培养方法探究三种土壤中阿特拉津的矿化特征及其微生物响应。第一组试验(试验Ⅰ)用于测定阿特拉津的矿化特征,土壤中阿特拉津终浓度为50 μg·g–1,14C-阿特拉津为2.0×105 DPM(DPM表示每分钟内放射性物质中发生衰变的原子核数量),每周取样测定14CO2的产生量。第二组试验(试验Ⅱ)则仅添加50 μg·g–1的12C-阿特拉津(ATZ),同时设立不添加阿特拉津的空白对照(CK),于培养第28天和第56天进行破坏性取样,用于研究微生物对阿特拉津的响应。土壤微宇宙的设置[15]和阿特拉津的加入[17]参照文献进行,所有处理均设置3个重复,于25 ℃下避光培养,定期通气。
1.4 阿特拉津矿化的测定在试验Ⅰ中,矿化管橡胶塞下悬挂有一个盛有1 mol·L–1 NaOH溶液的2 mL小瓶作为CO2捕集器。每周取出CO2捕集器,并用新鲜的NaOH替换,同时通气15 min。使用液体闪烁计数器(LSC,Beckman-Coulter,美国)测定NaOH溶液中的放射性,通过计算NaOH溶液中检测到的放射性占总14C阿特拉津放射性(2.0×105 DPM)的百分比计算阿特拉津矿化率,即矿化为14CO2的阿特拉津占添加[U-ring-14C]-阿特拉津总量的百分比。
1.5 阿特拉津矿化累积模型及其参数估计使用Gompertz非线性模型[18]对累积矿化量进行拟合,得到相应各个参数的估计值和拟合优度(R2值)。Gompertz模型如下式:
| $ y = a \times \exp [ - \exp (b - cx)] $ | (1) |
式中,y为累积矿化率,%;x为时间,d;a、b、c无明确的生物学意义,因此通过引入A(矿化速率降低为零时的累积矿化率,即最大累积矿化率,%)、μm(最大矿化速率,%·d–1)和λ(滞后时间,d)对方程进行优化,得到累积矿化率y(%)关于培养时间t(d)的方程(2):
| $ y = A \times \exp \left\{ { - \exp \left[ {\frac{{\mu m \times e}}{A}(\lambda - t)} \right] + 1} \right\} $ | (2) |
对试验Ⅱ中包括0 d样品在内的总共45个土壤样品进行微生物群落分析。称取0.50 g土壤,使用FastDNA® SPIN Kit for Soils试剂盒(MP Biomedicals)按其说明进行DNA的提取,1.5%的琼脂糖凝胶电泳检测DNA质量,NanoDrop ND-1000分光光度计测定DNA浓度和纯度。高通量测序委托上海美吉公司在Illumina MiSeq平台上完成,使用Qiime 2以及默认参数去除低质量序列,基于99%序列同一性阈值划分扩增序列变体(ASV),与Silva 132数据库比较确定序列的系统学分类。
1.7 细菌16S rRNA及降解功能基因荧光定量PCR分析采用定量PCR(qPCR)测定细菌16S rRNA和三嗪氯水解酶基因trzN的拷贝数。16S rRNA使用引物:338F-ACTCCTACGGGAGGCAGCAG;518R-ATTACCGCGGCTGCTGG,扩增条件为95 ℃预变性3 min、95 ℃变性30 s、56 ℃退火30 s、72 ℃延伸30 s,共30个循环。trzN引物为:trzN-F- CACC AGCACCTGTACGAAGG;trzN-R-GATTCGAACCA TTCCAAACG,扩增条件为94 ℃预变性4 min、94 ℃变性30 s、53 ℃退火1 min、72 ℃延伸45 s,共计40个循环[19]。
制备qPCR标准品,经梯度稀释制备标准曲线(拷贝数范围102~109 copies·μL–1)。采用SYBR PremixExTaqTM试剂盒(TaKaRa Biomedical Technology)在实时荧光定量PCR扩增仪(耶拿,德国)上进行扩增分析。每个样品重复三次,以无菌水作为阴性对照。细菌16S rRNA基因的扩增效率112.8%,R2为0.997;trzN基因的扩增效率85.4%,R2为0.999。根据熔解曲线评价扩增的特异性。
1.8 数据处理使用SPSS 26对不同处理进行单因素方差分析(ANOVA),并采用邓肯(Duncan)多重比较来判断差异显著性(P < 0.05)。采用R 4.3.3的vegan包计算微生物多样性指数和布雷距离(Bray-Curtis);筛选相对丰度总和高于0.5%、在样品中出现次数大于4的ASV,使用igraph包进行相关性矩阵分析,选择相关系数|r|>0.8且显著性P<0.001的ASV进行网络分析;采用limma包进行差异表达分析,采用ggplot2包绘制气泡图和物种组成堆叠图等,寻找土壤中的响应微生物。
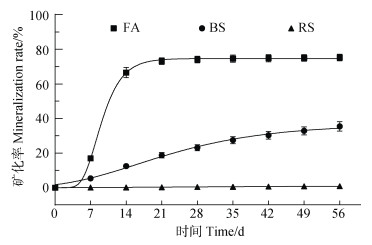
2 结果 2.1 阿特拉津矿化特征同位素示踪显示,三种土壤中阿特拉津的矿化动态存在明显差异(图 1)。潮土中阿特拉津快速矿化,培养前21 d内累计矿化率呈指数快速增长,在21 d时矿化率达73.2%,之后趋缓,直至培养结束矿化率为75.2%。黑土中阿特拉津矿化活性稳定,累积矿化率基本呈线性增长,培养结束时矿化率为35.5%。红壤则缺乏阿特拉津矿化能力,在整个培养期间累积矿化率几乎未变化,培养56 d后矿化率仅为0.810%。
|
注:FA为潮土;BS为黑土;RS为红壤。下同。 Note: FA is fluvo-aquic soil; BS is black soil; RS is red soil. The same below. 图 1 阿特拉津在三种土壤中的累积矿化曲线 Fig. 1 The mineralization dynamics of atrazine in three types of soils |
使用Gompertz非线性模型对土壤中阿特拉津的累积矿化曲线进行拟合,相关参数如表 2所示。三种土壤的拟合优度(R2)均达到0.90以上,潮土和黑土拟合效果相对较好。比较拟合参数可知,潮土最大累积矿化率(A)约为黑土的2倍,约为红壤的74倍,矿化程度显著大于后两者;潮土的μm最高,为黑土的10.6倍,而红壤的μm仅有0.0185,矿化速率极其缓慢;滞后时间(λ)表明,黑土较潮土和红壤更快结束延滞期,而红壤滞后时间最长,约为黑土的4.5倍,为潮土的1.5倍。
|
|
表 2 不同土壤中阿特拉津矿化的模型拟合参数 Table 2 Gompertz model parameters of atrazine mineralization in the three types of soils |
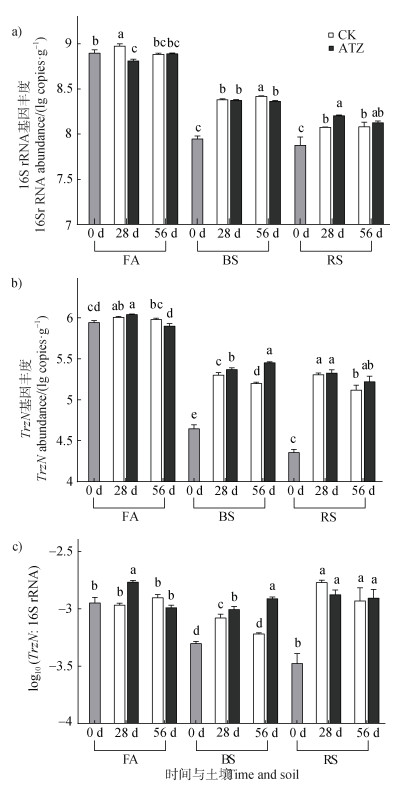
采用qPCR分别对不同培养时期16S rRNA和trzN基因进行定量,以评估土壤细菌及阿特拉津降解菌的数量动态(图 2)。三种土壤中,潮土整体上具有更高的16S rRNA基因拷贝数,但在56 d的培养期间增长幅度不大。而在黑土和红壤中,经培养后16S rRNA基因拷贝数明显增加(图 2a)。
|
注:CK为不添加阿特拉津的对照处理、ATZ为添加阿特拉津的处理;0 d、28 d和56 d培养时间。图中不同小写字母表示相同土壤不同处理间存在统计学差异(P < 0.05)。下同。 Note: CK is the control without atrazine addition, and ATZ represents the treatment of 50 μg·g–1 atrazine. Time is 0 d, 28 d, and 56 d respectively. Different lowercase letters indicated significant differences at the P < 0.05 level among different treatments of the same soil. The same below. 图 2 三种土壤中16S rRNA基因丰度(a)与trzN基因丰度(b)及其比值(c) Fig. 2 The abundances of 16S rRNA genes(a)and trzN genes(b), and their ratio(c)in the three types of soils |
与16S rRNA基因的情况类似,在培养开始时(0 d)潮土中trzN基因的丰度分别是黑土和红壤的20倍和38倍。潮土的FA-ATZ(潮土添加阿特拉津的处理)处理中trzN随时间先增加后减少,且在28 d时FA-ATZ(1.10×106 copies·g–1)与FA-CK(1.02× 106 copies·g–1)无显著差异,但在56 d时FA-ATZ(7.97×105 copies·g–1)显著低于FA-CK(9.56× 105 copies·g–1)。黑土经培养后trzN有大幅增加,且BS-ATZ(黑土添加阿特拉津的处理)在不同培养时间始终显著高于BS-CK(黑土未添加阿特拉津的空白对照)。而在红壤中,经过培养后trzN有显著增长,但RS-CK(红壤未添加阿特拉津的空白对照)和RS-ATZ(红壤添加阿特拉津的处理)两个处理之间无显著差异(图 2b)。
trzN与16S rRNA两个基因拷贝数的比值(trzN:16S rRNA)可近似地反映降解菌在总体细菌中的占比。在潮土中,阿特拉津处理对该比值的效应随时间发生变化。培养第28天时,FA-ATZ处理显著高于FA-CK,而第56天时两者无显著差异,且均低于28 d的FA-ATZ。黑土BS-ATZ处理中该比值始终高于BS-CK,且随培养时间增加幅度不断加大。红壤中,经培养后该比值较初始值有明显增加,但RS-CK和RS-ATZ处理之间无显著差异(图 2c)。
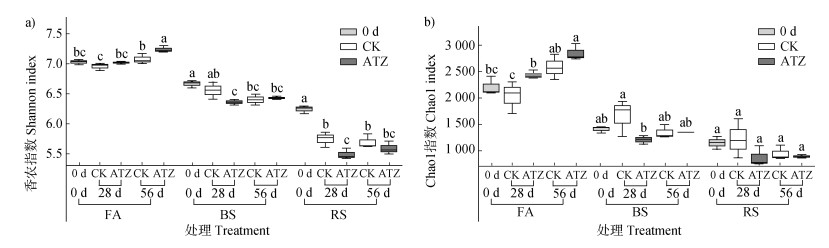
2.3 土壤细菌群落多样性对所有样品开展16S rRNA基因高通量测序,共获得271万条序列,划分为35 138个ASVs,基于此进行细菌多样性分析。整体上,潮土细菌群落的α-多样性始终高于黑土和红壤(图 3)。同一土壤的香农(Shannon)指数(图 3a)和Chao1指数(图 3b)变化趋势类似,但不同土壤间存在差异。在潮土中,FA-ATZ处理中微生物多样性逐渐增加,至第56天显著高于FA-CK处理。红壤则相反,在第28天,阿特拉津处理下微生物多样性均低于对照。在黑土中,28 d时BS-ATZ相较BS-CK较低,56 d时两处理间无显著差异。
|
图 3 三种土壤中细菌群落的α多样性分析(a. 香农指数;b. Chao1指数) Fig. 3 α-diversity of the bacterial community in the three types of soils(a. Shannon index; b. Chao 1 index) |
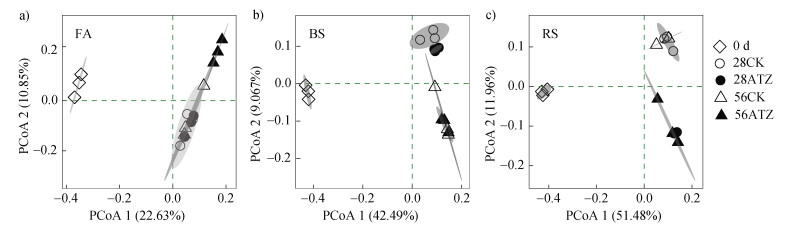
采用基于Bray-Curtis距离的主坐标分析法(PCoA)评估土壤细菌群落结构变化(图 4)。对潮土群落,PCoA1和PCoA2的累计方差解释率为33.5%,培养至28 d的FA-CK、FA-ATZ及56 d的FA-CK相对聚集,而56 d的FA-ATZ与三者距离较远,表明阿特拉津处理在28 d时对潮土中微生物群落结构无显著影响,但在56 d时效应明显(图 4a)。对于黑土群落,PCoA1、PCoA2的累计方差解释率达51.6%,各样品根据培养时间聚为两类,但BS-CK和BS-ATZ处理之间的距离相对较小(图 4b)。红壤中PCoA1与PCoA2合并解释率达63.4%,其群落响应表现出与潮土、黑土明显不同的模式,即根据阿特拉津的添加与否聚为两类,RS-CK与RS-ATZ分别形成两组,阿特拉津对微生物群落结构的影响明显(图 4c)。
|
注:0d代表 0 d样品;28CK代表培养至28 d时不添加阿特拉津的对照处理;28ATZ代表培养至28 d时添加阿特拉津的处理;56CK代表培养至56 d时不添加阿特拉津的对照处理;56ATZ代表培养至56 d时添加阿特拉津的处理。椭圆表示95%置信区间。 Note: 0d indicates the sample of 0 day; 28CK indicates the control without atrazine addition for 28 days; 28ATZ indicates the treatment of 50 μg·g–1 atrazine for 28 days; 56CK indicates the control without atrazine addition for 56 days; 56ATZ indicates the treatment of 50 μg·g–1 atrazine for 56 days. Ellipse represents 95% confidence intervals. 图 4 三种不同农田土壤中细菌群落的主坐标分析(a. 潮土;b. 黑土;c. 红壤) Fig. 4 Principal coordinates analysis of bacterial communities in the three types of soils(a. fluvo-aquic soil, b. black soil, c. red soil) |
为进一步研究阿特拉津对土壤细菌群落结构的影响,针对3种土壤构建共现网络,网络拓扑特征如表 3所示。结果表明阿特拉津对微生物网络的拓扑特性产生了不同影响,具体影响因土壤而异。对于潮土,FA-ATZ网络相对于FA-CK网络的节点、边和平均度并无明显变化,但阿特拉津处理网络中正相关关系达64.7%,较对照中的55.3%有明显增加。红壤中,阿特拉津明显降低了RS-ATZ网络中的边数和平均度,与对照相比网络结构变得简单。黑土的BS-ATZ与BS-CK网络相比,边和平均度均有所减少,但变化幅度小于红壤,同时BS-ATZ网络的正相关系数也有所增加。
|
|
表 3 阿特拉津影响下的土壤微生物网络拓扑特性 Table 3 Topological characteristics of microbial networks in the three soils |
土壤细菌在门水平的物种组成如图 5所示。第一优势菌门为变形菌门(Proteobacteria),潮土和黑土中CK和ATZ均无显著差异(P>0.05);而在红壤中,无论28 d还是56 d,ATZ丰度显著高于CK(P<0.05)。其次为酸杆菌门(Acidobacteriota),其在潮土中无显著差异(P>0.05);黑土中28 d的CK显著低于除0 d外的其他处理(P<0.05);红壤中则是56 d的CK显著高于其他处理(P<0.05)。第三为绿弯菌门(Chloroflexi),无论28 d还是56 d,三种土壤中CK和ATZ均无显著差异(P>0.05)。浮霉菌门(Planctomycetota)在潮土和红壤中28 d的ATZ均显著高于CK(P<0.05)。其他优势菌门包括放线菌门(Actinobacteriota)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadota)、拟杆菌门(Bacteroidota)、黏菌门(Myxococcota)等。
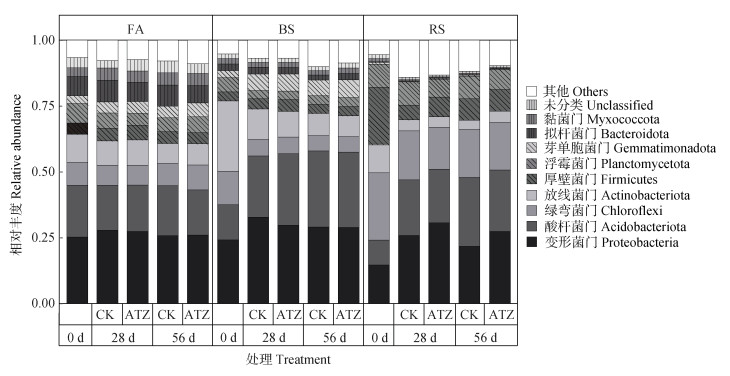
|
图 5 三种农田土壤中细菌群落门水平组成 Fig. 5 Soil bacteria community composition at the phylum level in the three soils |
为进一步揭示细菌群落对阿特拉津的响应差异,选择每种土壤不同培养时间中变化较明显(P<0.05及Log2(fold change) > 1)的前10个属进行分析(图 6),其中,拟节杆菌属(Paenarthrobacter)是潮土中增长幅度最大的物种,其在FA-CK中未检出,而在FA-ATZ处理中平均丰度增加至0.12%。此外,亚硝化单胞菌属(Nitrosomonas)丰度也显著增加,其在FA-ATZ中平均相对丰度为FA-CK的2倍。
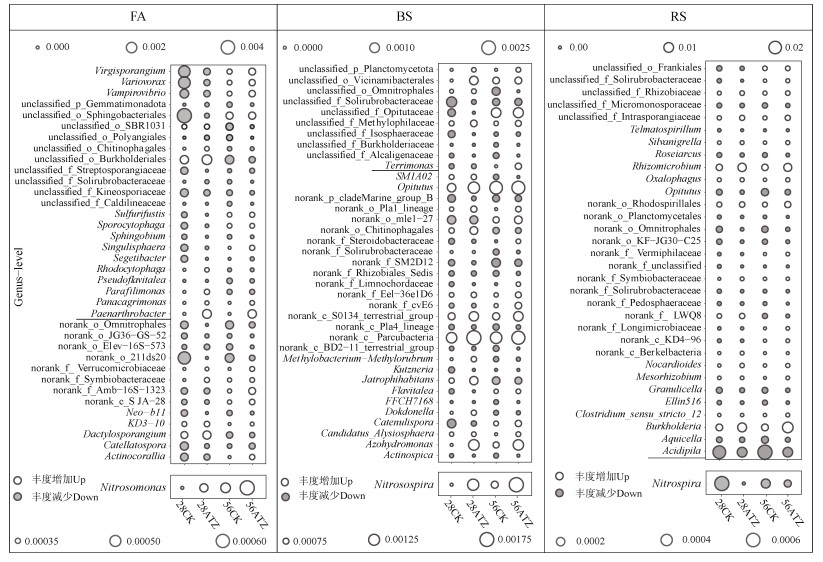
|
注:通过差异分析,在不同土壤及不同培养时间中选择P<0.05、Log2(fold change) > 1前十的属作气泡图。○代表ATZ相较CK丰度显著增加的属(Up);●代表ATZ相较CK丰度显著降低的属(Down)。点的大小对应于相对丰度。 Note: The most abundant top ten genera of P < 0.05 and Log2(fold change) > 1 were selected by differential analysis to create the bubble plot. ○ shows significantly increased genus by ATZ treatment, while ● represents diminished genus.The size of the bubble corresponds to its abundance. 图 6 三种土壤细菌群落差异分析气泡图(属水平) Fig. 6 The bubble plot of bacterial communities in the three soils at the genus level |
黑土加入阿特拉津后,培养后期(56 d)增长幅度最大的是土生单胞菌属(Terrimonas),其在BS-CK中未检出,而BS-ATZ中平均相对丰度增加至0.025%。亚硝化螺菌属(Nitrosospira)相对丰度也有所增加,在BS-ATZ中达0.18%,相较BS-CK增长了2倍。
红壤细菌群落对阿特拉津产生正响应的种属较少,一些优势细菌受阿特拉津影响甚至出现负响应。如酸球菌(Acidipila),在28 d时RS-CK中的相对丰度为2.08%,但在RS-ATZ中降低至1.02%;56 d时则由RS-CK的2.73%降低为RS-ATZ的1.00%。硝化螺旋菌属(Nitrospira)在添加阿特拉津后丰度也受到抑制,前期由0.067%降至0.0054%,后期则由0.024%降低为0.014%。
3 讨论农药与土壤微生物的交互作用分为生物降解和微生物群落胁迫两方面。三种土壤中阿特拉津的降解和细菌群落响应形成鲜明反差,充分说明两者紧密联系,特定情境下可能突出表现为某一方面。
3.1 三种土壤中阿特拉津的生物降解差异及可能原因矿化作用可指示土壤对有机污染物的自净能力。本研究采用Gompertz模型对矿化累积曲线进行拟合,其常用来拟合细菌生长曲线,也可用于解释微生物生产动力学[20]。拟合参数显示三种土壤中阿特拉津的矿化特征差异明显(表 2):潮土具有最高的累积矿化率A和最快的矿化速率μm;红壤的上述参数与潮土形成鲜明对比,指示极低的矿化活性;黑土的矿化特征介于潮土和红壤之间。这种不同类型土壤间有机污染物降解的差异似乎具有普遍性,如黑土和潮土中多环芳烃和抗生素的降解均远快于红壤[21-22]。有研究发现某旱地红壤中阿特拉津矿化率仅为0.33%[15],接近本研究中的测定值0.81%。
阿特拉津的矿化差异与降解菌的数量和活性有关。已知微生物编码两种三嗪氯水解酶基因trzN与atzA。本研究中三种土壤中均检出trzN,但未能成功扩增atzA(图 2b)。atzA基因序列高度保守,由于引物错配导致扩增失败的可能性相对较小[23]。目前的研究发现土壤中阿特拉津降解基因以trzN为主[24],这可能与两者的结构差异有关[7],决定了trzN相较于atzA具有更广泛的底物范围、更高的底物亲和力和动力学参数(Kcat/Km)[25]。
三种土壤中,trzN基因拷贝数由高到低依次为潮土、黑土、红壤(图 2),与土壤矿化活性趋势(图 2b)一致;潮土与黑土的阿特拉津处理中可观察到trzN丰度、trzN:16S rRNA比值随矿化显著增加的趋势(图 2)。前人研究发现,土壤中三嗪类除草剂的降解与trzN丰度密切相关[26]。鉴于此,在本研究中,携带trzN基因的土壤细菌很可能在潮土和黑土的阿特拉津转化中发挥关键作用。
另一方面,红壤虽然缺乏矿化活性,但也可检出丰度水平与黑土类似的trzN基因(图 2b)。这种现象并不罕见[27],可能表明红壤中的trzN基因并不表达为降解活性;亦可能是红壤中缺乏下游降解基因,导致阿特拉津降解不彻底,主要以中间代谢产物状态存在于土壤中。此外,红壤特殊的酸性条件也可能导致阿特拉津有效性偏低而抑制降解:阿特拉津水溶性低(约33 mg·L–1),酸解离常数(pKa)为1.68,在较低pH下电离分数减少,不利于微生物降解[28]。许多研究指出酸性土壤中阿特拉津降解或矿化速率明显低于中性或碱性土壤[29]。董攀月等[15]发现调节红壤pH后阿特拉津矿化率大幅增加。上述研究均表明红壤的低pH条件可能是影响阿特拉津降解率的关键因素。
3.2 潮土、黑土中的阿特拉津降解菌潮土中加入阿特拉津显著促进了拟节杆菌的相对丰度(图 6)。拟节杆菌、节杆菌、假节杆菌是相互关联的一类细菌[30],许多成员具有阿特拉津降解能力,如Arthrobacter aurescens TC1、Paenarthrobacter sp. AT和P. ureafaciens ZY等,它们一般携带trzN、atzB和atzC降解基因,能将阿特拉津降解为无毒的氰尿酸[31-32]。Arbeli和Fuentes[23]从13种农田土壤中分离的83株降解菌中节杆菌占54株,表明这类细菌可能在阿特拉津降解中发挥重要作用。鉴于FA-ATZ处理中拟节杆菌的高度富集,其很可能是潮土中最主要的阿特拉津降解菌。
黑土虽表现出明显的阿特拉津矿化活性,但经物种差异分析并未发现已报道的典型阿特拉津降解菌(图 6)。其可能原因有二,一是降解基因如trzN等一般位于质粒[33],可通过基因水平转移扩散,导致存在许多功能尚未挖掘的降解微生物。二是可能存在共代谢途径,即微生物利用其他碳源生长时伴随着部分污染物的降解[34],此时微生物的丰度不会随阿特拉津发生显著变化,导致无法识别降解菌。
在经阿特拉津处理的潮土和黑土中,观察到硝化细菌如Nitrosomonas、Notrosospira丰度的显著增加(图 6)。阿特拉津分子含5个氮原子,它们在微生物降解过程中将以铵态氮形式释放,可能被硝化细菌利用。黑土中BS-ATZ培养后期高度富集的Terrimonas,是一种潜在的反硝化细菌[35]。Nitrosomonas、Notrosospira、Terrimonas的富集表明,阿特拉津作为含氮底物可能影响土壤氮转化微生物[36]。
3.3 土壤细菌群落对阿特拉津的响应三种土壤中的细菌群落对阿特拉津产生不同响应。潮土中,相较于培养前期,阿特拉津在后期(56 d)显著提高了Shannon指数(图 3a),改变了细菌群落结构(图 4a)。鉴于潮土中阿特拉津快速矿化,在28 d时的响应微生物以拟节杆菌等降解菌为主,对土壤细菌整体结构影响较小。但阿特拉津降解过程中释放出铵态氮等,这可能是培养期间硝化菌富集、细菌群落结构持续变化的可能原因。同时,FA-ATZ共现网络中正相关比例的增加(表 3)说明微生物之间的联系加强、协同合作增加。有研究表明,阿特拉津降解菌和非降解菌间存在代谢互养关系(Cross-feeding),在改善细菌生长的同时促进阿特拉津降解[9,32,37],是潮土中细菌协同降解阿特拉津的又一证据。
红壤中阿特拉津矿化活性低,可以推断加入红壤的阿特拉津主要以未修饰结构或部分修饰中间物的形式存在。PCoA分析显示阿特拉津是影响红壤微生物群落的关键因素(图 4c),RS-ATZ处理中细菌多样性减少、网络破碎化、竞争趋势增加;优势细菌如Acidipila等适应酸性土壤环境的化能异养菌[38]也受到抑制,显示出一定的生态毒性效应。Ren等[39]的研究发现,低剂量多环芳烃就能导致红壤微生物群落的丰富度和多样性降低,群落结构改变。由此观之,红壤中微生物群落易受外界污染物的影响,抵御外界变化的能力较弱,提示阿特拉津污染的生态风险。
相比潮土和红壤,阿特拉津对黑土细菌群落影响较小,培养时间成为决定其群落结构的主要因素(图 4b)。总体而言,黑土细菌群落对阿特拉津污染具有一定的抗性。
4 结论不同类型农田土壤中阿特拉津的矿化动力学和细菌群落响应模式存在较大差异。潮土中矿化活性显著高于黑土和红壤,出现了拟节杆菌、硝化细菌等功能菌的富集,表明其阿特拉津残留风险较低。相较而言,黑土矿化速率稳定但稍低,细菌群落对阿特拉津不敏感,必要时需采取外接菌剂、共代谢底物等措施加快降解进程。红壤则几乎完全缺乏矿化能力,受阿特拉津影响,细菌群落多样性和网络复杂度降低、群落结构改变、优势物种被抑制,表现出明显的生态风险。综上,提示不同类型土壤中阿特拉津残留的治理可参照分区原则,从潮土等较高矿化活性土壤中挖掘降解微生物资源,对红壤等易产生残留的土壤则应重点从源头控制,加强土壤生态系统的保护。
致谢 感谢南京大学季荣教授在同位素实验中给予的协助。
| [1] |
Huang X W, Yang X L, Lin J H, et al. Biochar alleviated the toxicity of atrazine to soybeans, as revealed by soil microbial community and the assembly process[J]. Science of the Total Environment, 2022, 834: 155261. DOI:10.1016/j.scitotenv.2022.155261
(  0) 0) |
| [2] |
Miao H, Xu J M, Yu Y, et al. Toxic effects of atrazine on earthworms in three forest soils (In Chinese)[J]. Soils, 2023, 55(6): 1316-1322. [苗桓, 许加明, 虞悦, 等. 三种森林土壤中莠去津对蚯蚓的毒性效应[J]. 土壤, 2023, 55(6): 1316-1322.]
(  0) 0) |
| [3] |
Fang H, Lian J J, Wang H F, et al. Exploring bacterial community structure and function associated with atrazine biodegradation in repeatedly treated soils[J]. Journal of Hazardous Materials, 2015, 286: 457-465. DOI:10.1016/j.jhazmat.2015.01.006
(  0) 0) |
| [4] |
Zhao H T, Qian H H, Cui J W, et al. Endocrine toxicity of atrazine and its underlying mechanisms[J]. Toxicology, 2024, 505: 153846. DOI:10.1016/j.tox.2024.153846
(  0) 0) |
| [5] |
Udiković-Kolić N, Scott C, Martin-Laurent F. Evolution of atrazine-degrading capabilities in the environment[J]. Applied Microbiology and Biotechnology, 2012, 96(5): 1175-1189. DOI:10.1007/s00253-012-4495-0
(  0) 0) |
| [6] |
Lee T H Y, Ziegler A D, Li C X, et al. Riverine pesticides in an agricultural catchment in northern Thailand: With focus on atrazine and metabolites[J]. ACS ES & T Water, 2024, 4(9): 3758-3772.
(  0) 0) |
| [7] |
Peat T S, Newman J, Balotra S, et al. The structure of the hexameric atrazine chlorohydrolase AtzA[J]. Acta Crystallographica Section D Biological Crystallography, 2015, 71(3): 710-720. DOI:10.1107/S1399004715000619
(  0) 0) |
| [8] |
Bhatti P, Duhan A, Pal A, et al. Ultimate fate and possible ecological risks associated with atrazine and its principal metabolites(DIA and DEA)in soil and water environment[J]. Ecotoxicology and Environmental Safety, 2022, 248: 114299. DOI:10.1016/j.ecoenv.2022.114299
(  0) 0) |
| [9] |
Xu X H, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5
(  0) 0) |
| [10] |
Liu Z Y, Han L X, Zhang X, et al. Core bacteria carrying the genes associated with the degradation of atrazine in different soils[J]. Environment International, 2023, 181: 108303. DOI:10.1016/j.envint.2023.108303
(  0) 0) |
| [11] |
Ren W J, Teng Y, Luo Y M. Research progress and perspective on the pollution process and abatement technology of herbicides in black soil region in northeastern China (In Chinese)[J]. Acta Pedologica Sinica, 2022, 59(4): 888-898. DOI:10.11766/trxb202107090354 [任文杰, 滕应, 骆永明. 东北黑土地农田除草剂污染过程与消减技术研究进展与展望[J]. 土壤学报, 2022, 59(4): 888-898.]
(  0) 0) |
| [12] |
Sun B, Zhu A N, Yao R J, et al. Research progress on barrier remediation technology and productivity enhancement model for fluvo-aquic soil, red soil, and saline-alkali soil (In Chinese)[J]. Acta Pedologica Sinica, 2023, 60(5): 1231-1247. DOI:10.11766/trxb202307210286 [孙波, 朱安宁, 姚荣江, 等. 潮土、红壤和盐碱地障碍消减技术与产能提升模式研究进展[J]. 土壤学报, 2023, 60(5): 1231-1247.]
(  0) 0) |
| [13] |
Dou R N, Sun J T, Deng F C, et al. Contamination of pyrethroids and atrazine in greenhouse and open-field agricultural soils in China[J]. Science of the Total Environment, 2020, 701: 134916. DOI:10.1016/j.scitotenv.2019.134916
(  0) 0) |
| [14] |
Gao T C, Tian H X, Xiang L L, et al. Characteristics of bacterial community and extracellular enzymes in response to atrazine application in black soil[J]. Environmental Pollution, 2024, 343: 123286. DOI:10.1016/j.envpol.2023.123286
(  0) 0) |
| [15] |
Dong P Y, Chen Y Z, Zeng J, et al. Environmental fate and regulation of atrazine in upland red soil under different fertilization regimes (In Chinese)[J]. Soils, 2022, 54(6): 1201-1209. [董攀月, 陈禹竹, 曾军, 等. 不同施肥处理下旱地红壤中阿特拉津的环境归趋及其调控研究[J]. 土壤, 2022, 54(6): 1201-1209.]
(  0) 0) |
| [16] |
Yao B, Gu X J, Shu Y G, et al. Degradation of atrazine and changes in soil biological indices throughout dendroremediation using poplars[J]. Journal of Forestry Research, 2019, 30(6): 2331-2339. DOI:10.1007/s11676-018-0759-6
(  0) 0) |
| [17] |
Wu Y C, Zhu Q H, Zeng J, et al. Effects of pH and polycyclic aromatic hydrocarbon pollution on thaumarchaeotal community in agricultural soils[J]. Journal of Soils and Sediments, 2016, 16(7): 1960-1969. DOI:10.1007/s11368-016-1390-9
(  0) 0) |
| [18] |
Zwietering M H, Jongenburger I, Rombouts F M, et al. Modeling of the bacterial growth curve[J]. Applied and Environmental Microbiology, 1990, 56(6): 1875-1881. DOI:10.1128/aem.56.6.1875-1881.1990
(  0) 0) |
| [19] |
Zhang J P, Liang S, Wang X H, et al. Biodegradation of atrazine by the novel Klebsiella variicola strain FH-1[J]. BioMed Research International, 2019, 2019: 4756579.
(  0) 0) |
| [20] |
Wang J L, Guo X. The Gompertz model and its applications in microbial growth and bioproduction kinetics: Past, present and future[J]. Biotechnology Advances, 2024, 72: 108335. DOI:10.1016/j.biotechadv.2024.108335
(  0) 0) |
| [21] |
Ren G D, Teng Y, Ren W J, et al. Pyrene dissipation potential varies with soil type and associated bacterial community changes[J]. Soil Biology and Biochemistry, 2016, 103: 71-85. DOI:10.1016/j.soilbio.2016.08.007
(  0) 0) |
| [22] |
Shen D H, Gu X, Zheng Y Y, et al. The fate of erythromycin in soils and its effect on soil microbial community structure[J]. Science of the Total Environment, 2022, 820: 153373. DOI:10.1016/j.scitotenv.2022.153373
(  0) 0) |
| [23] |
Arbeli Z, Fuentes C. Prevalence of the gene trzN and biogeographic patterns among atrazine-degrading bacteria isolated from 13 Colombian agricultural soils[J]. FEMS Microbiology Ecology, 2010, 73(3): 611-623.
(  0) 0) |
| [24] |
Douglass J F, Radosevich M, Tuovinen O H. Microbial attenuation of atrazine in agricultural soils: Biometer assays, bacterial taxonomic diversity, and catabolic genes[J]. Chemosphere, 2017, 176: 352-360. DOI:10.1016/j.chemosphere.2017.02.102
(  0) 0) |
| [25] |
Esquirol L, Peat T S, Sugrue E, et al. Chapter Four Bacterial catabolism of s-triazine herbicides: Biochemistry, evolution and application[J]. Advances in Microbial Physiology, 2020, 76: 129-186.
(  0) 0) |
| [26] |
Fernandes A F T, Wang P, Staley C, et al. Impact of atrazine exposure on the microbial community structure in a Brazilian tropical latosol soil[J]. Microbes and Environments, 2020, 35(2): 215596631.
(  0) 0) |
| [27] |
Nousiainen A O, Björklöf K, Sagarkar S, et al. Atrazine degradation in boreal nonagricultural subsoil and tropical agricultural soil[J]. Journal of Soils & Sediments, 2014, 14(6): 1179-1188.
(  0) 0) |
| [28] |
Lin Z, Zhen Z, Ren L, et al. Effects of two ecological earthworm species on atrazine degradation performance and bacterial community structure in red soil[J]. Chemosphere, 2018, 196: 467-475.
(  0) 0) |
| [29] |
Houot S, Topp E, Yassir A, et al. Dependence of accelerated degradation of atrazine on soil pH in French and Canadian soils[J]. Soil Biology and Biochemistry, 2000, 32(5): 615-625.
(  0) 0) |
| [30] |
Busse H J. Review of the taxonomy of the genus Arthrobacter, emendation of the genus Arthrobacter sensu lato, proposal to reclassify selected species of the genus Arthrobacter in the novel Genera Glutamicibacter gen. nov., Paeniglutamicibacter gen. nov., Pseudoglutamicibacter gen. nov., Paenarthrobacter gen. nov. and Pseudarthrobacter gen. nov., and emended description of Arthrobacter roseus[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 9-37.
(  0) 0) |
| [31] |
Li X L, Wu S H, Fan H N, et al. Simultaneous Cr(Ⅵ)reduction and atrazine degradation by an isolated Paenarthrobacter sp. AT (In Chinese)[J]. Acta Scientiae Circumstantiae, 2023, 43(4): 236-242. [李祥龙, 吴尚华, 樊浩楠, 等. 一株类节杆菌协同还原六价铬和降解阿特拉津的研究[J]. 环境科学学报, 2023, 43(4): 236-242.]
(  0) 0) |
| [32] |
Zhao Y, Li X, Li Y Y, et al. Rapid biodegradation of atrazine by a novel Paenarthrobacter ureafaciens ZY and its effects on soil native microbial community dynamic[J]. Frontiers in Microbiology, 2023, 13: 1103168.
(  0) 0) |
| [33] |
Sajjaphan K, Shapir N, Wackett L P, et al. Arthrobacter aurescens TC1 atrazine catabolism genes trzN, atzB, and atzC are linked on a 160-kilobase region and are functional in Escherichia coli[J]. Applied and Environmental Microbiology, 2004, 70(7): 4402-4407.
(  0) 0) |
| [34] |
Hazen T C. Cometabolic bioremediation//Steffan R J. Consequences of microbial interactions with hydrocarbons, oils, and lipids: biodegradation and bioremediation[C]. Cham: Springer International Publishing, 2019: 233—247.
(  0) 0) |
| [35] |
Ren Z Y, Guo H, Jin H K, et al. P, N, and C-related functional genes in SBR system promoted antibiotics resistance gene transmission under polystyrene microplastics stress[J]. Water Research, 2023, 235: 119884.
(  0) 0) |
| [36] |
Dhakar K, Medina S, Ziadna H, et al. Comparative study of bacterial community dynamics in different soils following application of the herbicide atrazine[J]. Environmental Research, 2023, 220: 115189.
(  0) 0) |
| [37] |
Abdelgawad D M, Marei A S, Mansee A H. Managing the efficiencies of three different bacterial isolates for removing atrazine from wastewater[J]. Journal of Environmental Science and Health, Part B, 2022, 57(12): 948-959.
(  0) 0) |
| [38] |
Okamura K, Kawai A, Yamada T, et al. Acidipila rosea gen. nov., sp. nov., an acidophilic chemoorganotrophic bacterium belonging to the Phylum Acidobacteria[J]. FEMS Microbiology Letters, 2011, 317(2): 138-142.
(  0) 0) |
| [39] |
Ren G D, Ren W J, Teng Y, et al. Evh ident bacterial community changes but only slight degradation when polluted with Pyrene in a red soil[J]. Frontiers in Microbiology, 2015, 6: 22.
(  0) 0) |
 2025, Vol. 62
2025, Vol. 62


