氧化亚氮(N2O)是一种强效温室气体,百年时间尺度上,其气候变暖效应是二氧化碳(CO2)的298倍[1]。N2O还是破坏平流层臭氧的主要物质,可在大气中存留长达百年[2]。农业氮肥施用导致农田土壤N2O排放增加,使其成为大气N2O排放的主要来源,威胁生态环境[3]。农业土壤N2O排放所引发的生态环境问题已成为全球关注的焦点。土壤硝化和反硝化过程是氮循环的两个关键过程,它影响土壤氮平衡和生产力。研究土壤N2O的产生过程、影响因素及其微生物作用机制对于寻求土壤N2O减排对策、服务国家“双碳”目标具有重要意义[4-5]。
为保障粮食生产,化学农药被广泛使用。百菌清(Chlorothalonil,CTN),化学名2,4,5,6-四氯间苯二甲腈,化学式为C8Cl4N2,相对分子质量为265.9,是一种广谱性有机氯杀菌剂。百菌清能在酸性、中性和微碱性水溶液中稳定存在,当pH > 9时缓慢降解。其杀菌原理是抑制微生物细胞代谢使其失去活性,可用来防治多种由真菌引起的植物病害,高效低毒和杀菌范围广等优势使其成为农业生产中应用最广泛的农药之一,在作物病害严重时被连续大量喷洒于农田,大大减少了植物病害引起的经济损失[6-7]。据调查,每年用于作物的百菌清高达4.8×106 kg[6]。然而喷施的农药仅有少部分发挥作用,大部分落入土壤中,经常在土壤中检测到百菌清残留物,可能会影响土壤生态系统和氮循环[8]。百菌清主要通过微生物降解进行耗散,半衰期为3 d至6个月,受土壤类型、pH、浓度和施用次数的影响[8-9]。
百菌清进入土壤后能改变土壤微生物的数量和生物多样性[10]。作为非专一性杀菌剂,百菌清不仅抑制病原微生物,还影响非目标微生物的数量及活性[8]。研究表明,百菌清延缓尿素水解,原因是其抑制了硝化和反硝化过程[11]。百菌清由于其杀真菌作用,显著降低产N2O真菌的丰度,导致真菌参与的N2O排放量显著下降[12]。因此百菌清可能通过影响氮循环微生物,进而调控土壤N2O排放。以往研究表明,40倍田间推荐剂量的百菌清抑制酸性土壤N2O排放[13]。然而Su等[14]发现百菌清抑制反硝化过程但促进N2O排放。连续施用百菌清降低亚硝酸还原菌基因(nirK、nirS)丰度,而增加氧化亚氮还原菌基因(nosZ)丰度 [15]。然而,也有研究表明百菌清抑制N2O还原酶活性,但不影响nosZ基因丰度[16]。以往关于土壤氮循环的研究集中在自然因素和氮肥施用上,忽视了农药的作用,且不同土壤条件下百菌清对土壤N2O排放的影响并不一致,作用机制也存在争议,需要进一步深入探究。本研究以三种典型农田土壤进行室内培养实验,探究不同浓度百菌清对N2O排放的影响。该研究对揭示百菌清对土壤氮循环的作用机理、减少N2O排放以及指导农业安全用药和评估其生态环境效应具有重要意义。
1 材料与方法 1.1 供试土壤概况供试土壤采自江苏省内三种农田生态系统,依次编号为S1、S2和S3,分别为黏壤土、黏土、砂质壤土,该地区属于亚热带季风气候。S1和S2土壤采自镇江句容(31°55' N,119°17' E),年平均气温为15.4 ℃,年平均降水量为1 106.5 mm;S3土壤采自盐城大丰(33°19' N,120°45' E),年平均气温为16.0 ℃,年平均降水量为1 045 mm。供试土壤的详细信息如表 1所示。每种类型的土壤样品分别从5个不同的点采集表层(0~20 cm)土壤混合,带回实验室后立即将新鲜土样过筛(2 mm)以去除植物残体和石头,混匀后分成两部分。一部分土壤于4 ℃下保存用于培养试验;另一部分土壤在室内通风处风干,待土样完全风干磨细过筛后常温下储存,用于土壤基本理化性质测定。
|
|
表 1 供试土壤基本理化性质 Table 1 Physicochemical properties of tested soils |
本实验共设置空白对照(CK)、5 mg·kg–1百菌清添加(T5)、10 mg·kg–1百菌清添加(T10)、25 mg·kg–1百菌清添加(T25)四个处理,每个处理3个重复。在农业生产中,5 mg·kg–1百菌清添加量为农药施用推荐剂量,10和25 mg·kg–1分别为2倍和5倍推荐剂量,代表未来累积浓度[7,15,17]。
称取80 g风干土(以干物质量计)于250 mL锥形瓶内,调节土壤含水量至50%田间持水量(WHC)后于25 ℃黑暗条件下预培养7 d。将商业百菌清制剂(75%(质量百分比)活性成分)用超纯水溶解后均匀喷洒于土壤表面,分别使其在土壤中的浓度达到5 mg·kg–1、10 mg·kg–1、25 mg·kg–1,对照组加入等量超纯水,同时每个处理加入尿素(200 mg·kg–1,以N计)作为初始氮源。用去离子水调节土壤含水率至70%WHC,随即称量并记录锥形瓶质量。恒温培养30 d左右,实验期间定期补水。
1.3 土壤理化指标测定土壤铵态氮(NH4+-N)和硝态氮(NO3–-N)采用2 mol·L–1氯化钾浸提(土水质量比1︰5),流动分析仪(Skalar SANplus analyser,Skalar,荷兰)测定。土壤可溶性有机碳(DOC)用超纯水浸提(土水质量比1︰5),总有机碳分析仪(multi N/C 3100/1,analytikjena,德国)测定。土壤全碳(TC)和全氮(TN)含量使用元素分析仪(multi EA 5000,analytikjena,德国)测定;土壤电导率(EC)和pH按照土水质量比1︰2.5浸提,分别用电导率仪和pH计测定;土壤硝化潜势(PNR)和反硝化潜势(PDR)分别采用氯酸盐抑制法和乙炔抑制法测定[18-19]。
1.4 气体采集与分析实验正式开始后每天采集气体,在N2O排放峰值出现后每2~3天采集一次气体,采气时间固定在上午7:00 — 9:00。培养期间,根据土壤N2O峰值出现的时间破坏性采集土壤。通过气相色谱仪(Agilent 7890B,Agilent,美国)测定采集的气体样品浓度。土壤气体排放通量计算公式如下:
| $ F=\rho \cdot \frac{\Delta C}{\Delta T} \cdot \frac{273.15}{273.15+T} \cdot \frac{V}{m} $ | (1) |
式中,F是指气体排放通量,μg·kg–1·h–1;ρ为标准状态下N2O密度,为1.25 kg·m–3;ΔC/ΔT为特定时间密封容器内气体浓度变化,μg·kg–1·h–1;V为锥形瓶内顶部空间体积,m3;m为培养土壤干物质量,kg;T为培养温度,℃。
气体累积排放量计算公式如下:
| $ M = \sum\nolimits_{i = 1}^n {\frac{1}{2}} ({F_i} + {F_{i + 1}}) \cdot 24({t_{i + 1}} -{t_i}) $ | (2) |
式中,M为气体累积排放量,mg·kg–1;t为采样时间,d;i为采样次数。
1.5 DNA提取由DNA提取试剂盒(Dneasy® PowerSoil®kit,QIAGEN,德国)提取土壤样品,具体步骤参考说明书,使用超微量紫外分光光度计(Nanodrop,Thermo Scientific,美国)测定所提样品中DNA浓度和纯度。检测质量合格的样品进行实时荧光定量PCR(Applied-Biosystems Real-Time PCR,Thermo Scientific,美国)分析,反应体系为20 μL,按添加顺序如下:①SYBR® Premix Ex Taq TM(2×),10 μL;②正向引物,0.4 μL;③反向引物,0.4 μL;④ROX参考染料(50×),0.4 μL;⑤DNA或质粒模板,2 μL;⑥ddH2O,6.8 μL。扩增程序如下:①预变性95 ℃,30 s;②循环40次:变性95 ℃,5 s,退火55 ℃,30 s,延伸72 ℃,1 min;③溶解曲线程序:95 ℃,15 s,60 ℃,1 min,95 ℃,15 s,确定扩增产物的特异性。
1.6 数据处理本研究中,数据均以平均值±标准误差(mean±SE)形式表示,n=3。微生物基因丰度的拷贝数均经过对数转换处理,借助SPSS 25软件对数据进行单因素方差分析(ANOVA)和皮尔逊(Pearson)相关性分析。采用最小显著差异法(LSD)进行多重比较,Origin 2021绘图,使用Excel 2016制作表格。通过单因素方差分析评价百菌清浓度对土壤N2O累积排放量、土壤理化性质(DOC、NH4+-N和NO3–-N等)和微生物基因丰度的影响。通过相关分析,确定了百菌清添加下土壤N2O排放与土壤性质(DOC、NH4+-N和NO3–-N等)或微生物基因丰度的关系。
2 结果 2.1 百菌清对土壤N2O排放的影响图 1为培养期间各处理土壤N2O排放通量及累积排放量,在三种土壤中,百菌清添加处理均增加了土壤N2O排放,除S2土壤T5处理及S3土壤T25处理外,其余处理均达显著水平。百菌清添加量对S1和S3土壤N2O累积排放量的促进作用均表现为:T5 > T10 > T25 > CK。S1土壤T5、T10、T25、CK处理下N2O累积排放量分别为4.33、3.00、0.73、0.22 mg·kg–1。与CK相比,T5、T10、T25处理导致N2O排放分别增加1868%、1264%、232%;S3土壤T5、T10、T25、CK处理下N2O累积排放量分别为12.33、8.20、4.59、3.74 mg·kg–1。与CK相比,T5、T10、T25处理导致N2O排放分别增加230%、119%、23%。而在S2土壤中,不同浓度百菌清对N2O促进作用表现为:T25 > T10 > T5 > CK,T25、T10、T5、CK处理下土壤N2O累积排放量分别为2.17、1.19、0.52、0.50 mg·kg–1,与CK相比,T25、T10、T5分别导致N2O排放增加334%、138%、4%。
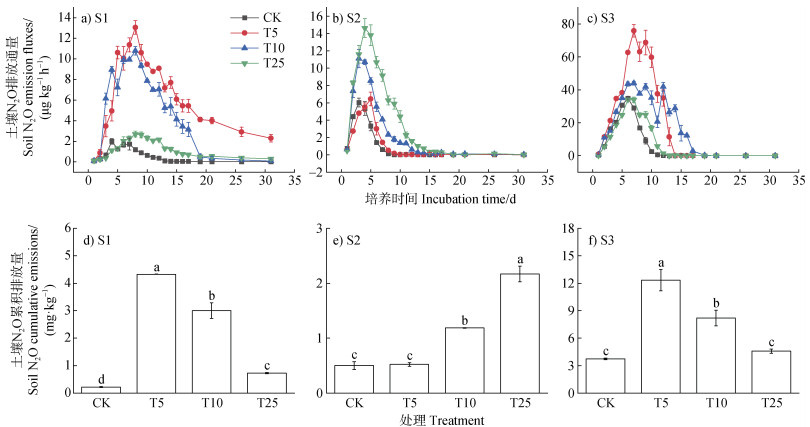
|
注:N2O:氧化亚氮。CK、T5、T10和T25分别为空白对照、5、10和25 mg·kg–1百菌清添加。不同小写字母表示处理间显著差异(P < 0. 05)。下同。 Note:N2O:nitrous oxide. CK,T5,T10 and T25 were control,and 5,10 and 25 mg·kg–1 chlorothalonil was added respectively. Different lowercase letters indicate significant differences between treatments(P < 0.05). The same below. 图 1 菌清添加下土壤N2O排放通量(a~c)和累积排放量(d~f) Fig. 1 Soil N2O emission fluxes(a-c)and cumulative emissions(d-f)under chlorothalonil addition |
通过测定土壤理化指标(表 2,图 2)发现,S2、S3土壤各理化指标对百菌清添加变化明显,而S1土壤部分理化指标对百菌清添加响应不敏感。相较于CK,百菌清添加处理对土壤NH4+-N、pH、EC、TC、TN和硝化潜势的影响无明显规律。但百菌清添加均增加了三种土壤NO3–-N和DOC含量,且降低了土壤反硝化潜势。
|
|
表 2 百菌清添加下土壤理化性质的差异 Table 2 Differences in physicochemical properties of soils under chlorothalonil addition |
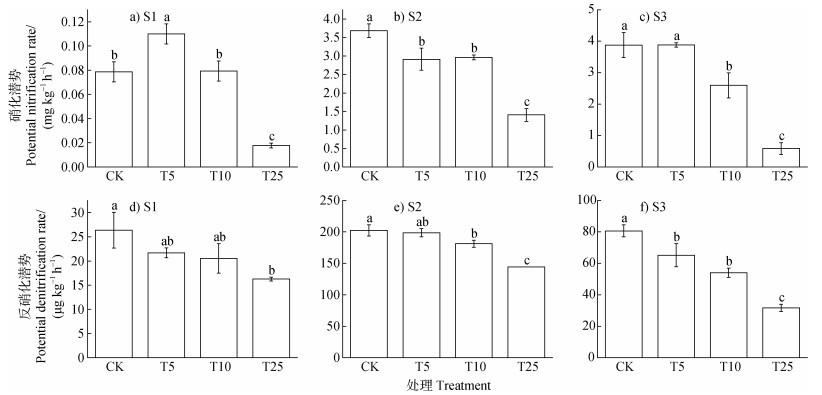
|
图 2 百菌清添加下土壤硝化(a~c)和反硝化潜势(d~f) Fig. 2 Potential nitrification(a-c)and denitrification(d-f)rate under chlorothalonil addition |
对于土壤NH4+-N含量,S1土壤T25处理显著高于CK,S2土壤T10和T25处理显著低于CK,S3土壤T5处理显著高于CK。对于土壤NO3–-N含量,三种土壤中不同浓度百菌清添加处理均显著增加了土壤NO3–-N含量。对于三种土壤DOC含量,百菌清添加均增加土壤DOC含量,相较于CK,S2土壤中百菌清添加处理均达显著水平,S3土壤中T10和T25处理达显著水平。对于土壤pH,与CK相比,百菌清添加对S1土壤pH无显著影响,S2土壤T10和T25处理显著降低了土壤pH,S3土壤T25处理显著增加了土壤pH。百菌清添加对三种土壤EC均无显著影响。对于土壤TC含量,T25处理显著增加了S3土壤TC含量。对于土壤TN含量,与CK相比,S1土壤中T10和T25显著降低了土壤TN含量,S2土壤T25处理显著增加了土壤TN含量。对于土壤硝化潜势,与CK相比,S1土壤中T5处理显著增加了土壤硝化潜势,T25处理显著降低了土壤硝化潜势。S2土壤中三种浓度百菌清添加处理均显著降低了土壤硝化潜势。S3土壤T10和T25处理显著降低土壤硝化潜势。对于土壤反硝化潜势,S1土壤中T25处理显著降低土壤反硝化潜势,S2土壤中T10和T25处理显著降低土壤反硝化潜势,S3土壤中三种浓度百菌清添加处理均显著降低土壤反硝化潜势。
2.3 百菌清对土壤微生物基因丰度的影响通过测定土壤硝化及反硝化微生物基因丰度可发现,如图 3所示,对于土壤硝化相关功能微生物基因丰度,三种浓度百菌清添加处理对S1土壤AOA和AOB基因丰度均无显著影响。与CK相比,三种浓度百菌清添加处理均显著增加了S2土壤AOB基因丰度,而对土壤AOA基因丰度无显著影响。在S3土壤中,T10和T25处理显著增加了土壤AOA和AOB基因丰度,而T5处理无显著影响。对于土壤反硝化相关功能微生物基因丰度,与CK相比,在S1土壤中,除T5处理显著降低了土壤nosZ基因丰度外,其余处理均无显著影响。在S2土壤中,三种浓度百菌清添加对土壤nirK、nirS及nosZ基因丰度均无显著影响。在S3土壤中,T10和T25处理显著增加了土壤nirK、nirS及nosZ基因丰度,而T5处理无显著影响。对于土壤总细菌丰度,与CK相比,T5和T25处理显著增加了S1土壤16S rRNA基因丰度,而T10处理无影响;在S2土壤中,三种浓度百菌清添加均对16S rRNA基因丰度无显著影响;在S3土壤中,T25处理显著增加土壤16S rRNA基因丰度,而对于T5和T10处理均无显著影响。
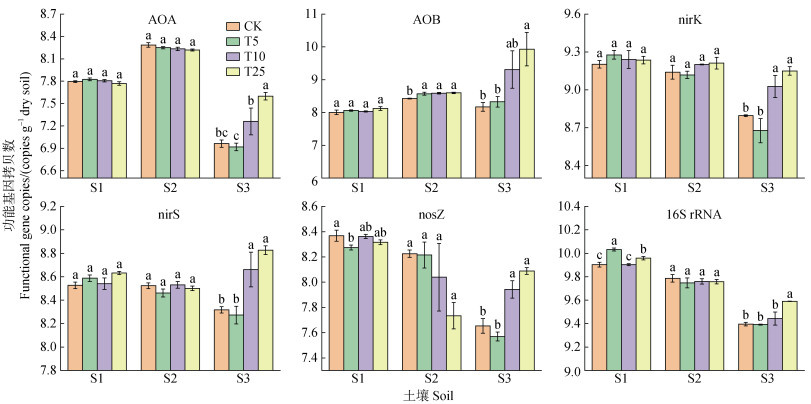
|
注:AOA:氨氧化古菌;AOB:氨氧化细菌;nirK、nirS:亚硝酸还原菌;nosZ:N2O还原菌;16S rRNA:总细菌。不同字母表示同一土壤不同处理间显著差异(P < 0.05)。 Note:AOA:Ammonia-oxidizing archaea;AOB:Ammonia-oxidizing bacteria;nirK,nirS:Nitrite-reducing bacteria;nosZ:N2O reducing bacteria;16S rRNA:Total bacteria. Different letters indicate significant differences between treatments of the same soil(P < 0.05). 图 3 土壤硝化和反硝化功能基因丰度 Fig. 3 Abundances of soil nitrification and denitrification functional gene |
如图 4所示,对N2O排放量与理化性质、基因丰度进行了相关性分析。结果表明,在S1土壤中,N2O排放量与PNR、16S rRNA显著正相关(P < 0.05),与土壤NH4+-N含量显著负相关(P < 0.05)。PNR与AOA显著正相关(P < 0.05),与土壤NH4+-N含量显著负相关(P < 0.05);PDR与土壤TN显著正相关(P < 0.05),与NO3–-N显著负相关(P < 0.05)。在S2土壤中,N2O排放量与土壤DOC、TN含量极显著正相关(P < 0.01),与pH显著负相关(P < 0.05),与NH4+-N、PNR、PDR、nosZ基因丰度极显著负相关(P < 0.01);PNR与AOA显著正相关(P < 0.05),与TN显著负相关(P < 0.05)、与AOB、DOC极显著负相关(P < 0.01);PDR与pH、nosZ显著正相关(P < 0.05),与NH4+-N极显著正相关(P < 0.01),与nirK显著负相关(P < 0.05),与DOC极显著负相关(P < 0.01);AOB与NO3–-N、DOC极显著正相关(P < 0.01),与pH显著负相关(P < 0.05);nosZ与NH4+-N极显著正相关(P < 0.01),与TN显著负相关(P < 0.05)。在S3土壤中,N2O排放量与NH4+-N含量显著正相关(P < 0.05);PNR与NO3–-N、TC显著负相关(P < 0.05),与DOC、AOA、AOB、nirK、nirS、nosZ、16S rRNA极显著负相关(P < 0.01);PDR与NO3–-N、pH、TC、AOA、AOB、nirK、nirS显著负相关(P < 0.05)、与DOC、nosZ、16S rRNA极显著负相关(P < 0.01);AOA与DOC极显著正相关(P < 0.01);AOB与DOC极显著正相关(P < 0.01);nirK与DOC显著正相关(P < 0.05);nirS与DOC显著正相关(P < 0.05);nosZ与DOC极显著正相关(P < 0.01);16S rRNA与DOC、TC显著正相关(P < 0.05)。
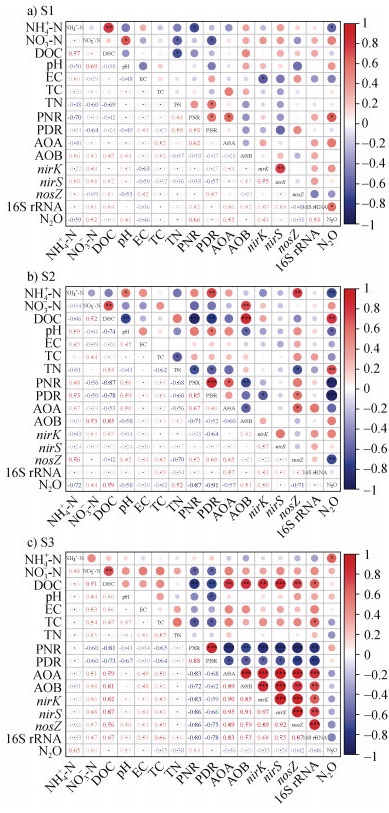
|
注:PNR:硝化潜势;PDR:反硝化潜势。*、**和***分别表示显著性水平在P < 0.05、P < 0.01和P < 0.001。 Note:PNR:Potential nitrification rate;PDR:Potential denitrification rate. * indicates a significant correlation at the 0.05 level,** at the 0.01 level,and *** at the 0.001 level. 图 4 土壤N2O排放与土壤理化性质、微生物基因丰度间的相关性 Fig. 4 Correlation between soil N2O emissions, soil physicochemical properties, and microbial gene abundance |
土壤微生物是调节氮素循环和维持土壤生态系统稳定性的主要驱动力。微生物功能基因丰度是预测潜在N2O产生速率的重要参数指标,已知微生物功能基因会影响N2O的产生和排放[20]。在S1土壤中,N2O排放量与PNR、16S rRNA呈显著正相关,与土壤NH4+-N含量呈显著负相关;在S2土壤中,N2O排放量与土壤DOC、TN含量呈极显著正相关,与pH呈显著负相关,与NH4+-N、PNR、PDR、nosZ基因丰度呈极显著负相关;在S3土壤中,N2O排放量与NH4+-N含量呈显著正相关(图 4)。表明土壤碳氮底物含量和微生物功能基因丰度是控制N2O排放的关键因子。有研究表明,在农业土壤中,N2O排放与土壤铵态氮、有机碳以及全氮含量呈显著正相关[21],这与本研究结果(图 4)一致。
本研究中,百菌清添加表现出促进或不影响土壤硝化和反硝化相关微生物功能基因丰度变化,在酸性土壤S1中,无论百菌清浓度如何,AOA基因丰度始终低于AOB(图 3),与前人研究结果[22]一致,在酸性较低的土壤中,AOB在N2O排放中占主导地位。
在S2土壤中,百菌清对AOA和AOB的影响不同(图 3),这可能是因为AOA对百菌清的敏感性低于AOB[23]。本研究发现,百菌清添加显著提高了S2土壤AOB基因丰度,但对AOA基因丰度无影响,且AOB基因丰度与DOC、硝态氮呈极显著正相关,与土壤pH呈显著负相关(图 3,图 4)。因此,推测百菌清添加诱导的土壤理化性质的变化可提高AOB基因丰度。百菌清在进入土壤后可通过矿化作用提高土壤DOC含量和微生物活性,进一步增强AOB的功能。该结果得到前人研究结果的支持,例如,有机质输入能增加土壤AOB基因丰度[24]。
在S3土壤中,中浓度(T10)和高浓度(T25)百菌清添加显著增加了土壤AOA和AOB基因丰度(图 3)。本研究还发现S3土壤硝化及反硝化微生物基因丰度主要受土壤DOC含量的影响(图 4),其原因可能是施用氮肥导致土壤中氮源增加,土壤C/N发生改变,此时反硝化过程将受到土壤碳源的制约,而百菌清作为外源碳加入可能为反硝化过程提供了电子供体。以往研究表明,有机肥额外的碳输入为微生物反硝化提供了能量[25-26]。百菌清的施用可通过为异养反硝化微生物提供丰富的底物来增强它们的活性。在本研究中,T10和T25显著增加了S3土壤中nirK、nirS基因丰度(图 3),部分解释了百菌清添加显著增加了N2O排放(图 1)。此外,百菌清添加还显著增加了S3土壤中nosZ基因丰度(图 3)。有相关研究表明,5倍推荐剂量(25 mg·kg–1)的百菌清添加在培养后期能显著增加土壤nosZ基因丰度[15]。这可能由于百菌清被微生物分解,可强化厌氧环境条件,有利于N2O还原菌的生长,导致百菌清添加处理中nosZ基因的丰度升高。需要指出的是,百菌清作为一种杀真菌剂,其主要靶标是真菌,可能通过抑制真菌活性导致细菌结构变化。遗憾的是,由于条件有限,本研究未能测定百菌清添加对土壤真菌群落的影响,有待后续进一步研究。
3.2 百菌清添加对不同土壤N2O排放的影响机制本研究发现,不同浓度梯度百菌清添加均不同程度增加三种土壤N2O排放(图 1),这与以往研究的结果[14]相一致,可能主要由于百菌清添加改变了土壤理化特性,进而通过影响氮循环相关微生物过程来促进N2O排放。该结果在百菌清添加后土壤中DOC和NO3–-N含量的变化(表 2)中得到了证实。研究结果表明,在添加百菌清后三种土壤DOC和NO3–-N含量均有升高趋势,尤其在S2和S3土壤中表现更明显,达到显著水平(表 2)。DOC和NO3–-N含量增加可能主要与百菌清添加至土壤后的自身降解过程及其介导的微生物代谢活动有关。具体而言,百菌清进入土壤后,可能通过两种途径影响土壤碳含量,一方面,由于百菌清本身为碳氮化合物,进入土壤后可通过生物或非生物途径而分解转化为土壤不同碳氮组分,从而增加土壤DOC和NO3–-N含量。另一方面,百菌清作为一种广谱性杀真菌剂,能够杀死土壤敏感微生物,导致微生物残体来源的土壤碳氮含量发生变化[27]。土壤有机质含量对N2O的产生和排放有显著影响,因为大多数异养微生物以土壤有机质为碳源和电子供体[28]。因此,土壤中有机质含量的增加可促进微生物的异养硝化和反硝化,进而导致N2O排放增加。综上,在控制土壤含水量相同的条件下,百菌清作为外源有机质输入可为土壤微生物提供碳氮活性底物,进而促进N2O的排放。
本研究结果表明,中性土壤中N2O排放量随百菌清浓度增加而增加,而在酸碱性土壤中则呈现相反趋势(图 1)。这种差异可能与土壤pH对微生物活性、氮循环过程以及百菌清降解的调控作用有关。在酸性或碱性土壤中,pH的极端条件可能抑制了部分参与氮循环的微生物群落活性,导致氮转化效率降低。百菌清作为一种广谱杀菌剂,在土壤中的持久性会随其浓度的增加而增加[7],可能会进一步加剧这种抑制效应。具体而言,低浓度百菌清不会对土壤硝化微生物产生显著影响,而百菌清在高浓度下会抑制硝化作用关键微生物,从而减少了N2O的产生和排放[29]。此外,百菌清在土壤中的快速耗散主要归因于土壤微生物[9],酸碱性土壤中微生物受极端pH环境条件胁迫,百菌清的降解速率可能较慢,导致其在土壤中积累并持续抑制微生物活性。相比之下,中性土壤中的微生物群落可能对百菌清的耐受性较强,且百菌清在中性条件下降解较快[30],百菌清的快速降解一方面降低了对土壤微生物毒害作用,另一方面可为土壤氮循环微生物提供碳源,进而出现中性土壤中N2O排放量随百菌清浓度增加而增加的现象(图 1)。综上所述,酸碱性土壤中N2O排放量随百菌清浓度增加而减少的现象,可能与百菌清和土壤pH对土壤微生物的胁迫存在协同作用有关。
硝化反硝化潜势在百菌清处理下有所下降,而N2O累积排放量显著增加(图 2),这可能与微生物群落结构变化、化学反硝化增加有关。土壤硝化及反硝化潜势反映了土壤产N2O的潜能,是由土壤整体微生物决定的,百菌清是一种杀菌剂,会对土壤部分微生物产生毒害作用[10],这可能导致土壤硝化及反硝化潜势降低。反硝化潜势测定方法主要基于微生物活性,通常不包括化学反硝化过程。因此,尽管反硝化潜势有所下降,但化学反硝化可能是导致N2O排放增加的重要原因之一。
此外,在三种农田土壤中,百菌清对硝化反硝化相关微生物功能基因丰度及16S rRNA影响不同(图 3)。但对比发现,百菌清添加对S2土壤微生物影响最小,仅显著影响了土壤AOB基因丰度,对其他微生物均无显著影响。而对S3土壤硝化反硝化微生物及16S rRNA影响最大,这可能与S2土壤黏粒含量高,而S3土壤黏粒含量低有关(表 1),原因是黏粒含量高的土壤增加了土壤对百菌清的吸附,降低了微生物的降解。百菌清作为一种有机化合物,其进入土壤后可能与土壤黏粒紧密结合,形成有机-矿物态结合物,进而增加SOC分子化合物的自身稳定性。这种物理保护作用影响微生物对有机质的分解利用,进而降低对土壤微生物的影响[31-32]。此外,百菌清在土壤中的降解率与土壤pH有关[33]。土壤黏粒含量及pH强烈影响百菌清在土壤中的耗散和累积,进而影响土壤硝化和反硝化微生物,因此,三种土壤质地及pH的明显差异可能是百菌清添加对N2O排放影响不一致的另一种原因。
4 结论百菌清添加刺激了土壤N2O排放,土壤N2O累积排放量随百菌清浓度的增加而变化,且在不同浓度百菌清处理间存在显著差异。不同浓度百菌清添加对三种类型农田土壤N2O排放影响的剂量效应不同,在S1和S3土壤中N2O排放量呈现出:T5 > T10 > T25 > CK,而在S2土壤中N2O排放量呈现出:T25 > T10 > T5 > CK。此外,不同浓度百菌清添加抑制土壤硝态氮还原,降低土壤反硝化潜势,且增加土壤DOC含量;T5处理显著降低了S1土壤中nosZ基因丰度;三种不同浓度的百菌清均增加了S2土壤AOB基因丰度;T10和T25显著增加了S3土壤中AOA、AOB、nirK、nirS和nosZ基因丰度。对于土壤总细菌丰度而言,T5和T25处理显著增加了S1土壤中16S rRNA基因丰度;T10和T25处理显著增加了S3土壤中16S rRNA基因丰度。相关性分析结果表明,土壤N2O排放与土壤碳氮含量和微生物基因丰度有关。然而,本研究也存在一些局限性,仅限于室内培养实验,尚未结合田间试验进行验证,未来可开展大田试验进一步验证大田施用条件下对土壤N2O排放的影响,以进一步明确农药百菌清施用对土壤N2O排放的作用机制。
| [1] |
Yang Y Y, Liu L, Zhang F, et al. Soil nitrous oxide emissions by atmospheric nitrogen deposition over global agricultural systems[J]. Environmental Science & Technology, 2021, 55(8): 4420-4429.
(  0) 0) |
| [2] |
Ma R Y, Yu K, Xiao S Q, et al. Data-driven estimates of fertilizer-induced soil NH3, NO and N2O emissions from croplands in China and their climate change impacts[J]. Global Change Biology, 2022, 28(3): 1008-1022. DOI:10.1111/gcb.15975
(  0) 0) |
| [3] |
Li Y, Wang R, Chen Z J, et al. Increasing net ecosystem carbon budget and mitigating global warming potential with improved irrigation and nitrogen fertilization management of a spring wheat farmland system in arid Northwest China[J]. Plant and Soil, 2023, 489(1/2): 193-209.
(  0) 0) |
| [4] |
Wang C, Shen Y, Fang X T, et al. Reducing soil nitrogen losses from fertilizer use in global maize and wheat production[J]. Nature Geoscience, 2024, 17(10): 1008-1015. DOI:10.1038/s41561-024-01542-x
(  0) 0) |
| [5] |
Xiang H, Hong Y G, Wu J P, et al. Denitrification contributes to N2O emission in paddy soils[J]. Frontiers in Microbiology, 2023, 14: 1218207. DOI:10.3389/fmicb.2023.1218207
(  0) 0) |
| [6] |
Jiang J L, Yang Y W, Wang L, et al. Effects of chlorothalonil application on the physio-biochemical properties and microbial community of a yellow–brown loam soil[J]. Agriculture, 2022, 12(5): 608. DOI:10.3390/agriculture12050608
(  0) 0) |
| [7] |
Wu X W, Cheng L Y, Cao Z Y, et al. Accumulation of chlorothalonil successively applied to soil and its effect on microbial activity in soil[J]. Ecotoxicology and Environmental Safety, 2012, 81: 65-69. DOI:10.1016/j.ecoenv.2012.04.017
(  0) 0) |
| [8] |
Su X X. The impacts mechanism of pesticide chlorothalonil on soil microbial denitrification and N2O Emission[D]. Chongqing: Chongqing University, 2019.[苏晓轩. 农药百菌清胁迫下土壤微生物反硝化及N2O释放机制研究[D]. 重庆: 重庆大学, 2019.]
(  0) 0) |
| [9] |
Regitano J B, Tornisielo V L, Lavorenti A, et al. Transformation pathways of 14C-chlorothalonil in tropical soils[J]. Archives of Environmental Contamination and Toxicology, 2001, 40(3): 295-302. DOI:10.1007/s002440010175
(  0) 0) |
| [10] |
Baćmaga M, Wyszkowska J, Kucharski J. The influence of chlorothalonil on the activity of soil microorganisms and enzymes[J]. Ecotoxicology, 2018, 27(9): 1188-1202. DOI:10.1007/s10646-018-1968-7
(  0) 0) |
| [11] |
Ding H, Zheng X Z, Zhang J, et al. Influence of chlorothalonil and carbendazim fungicides on the transformation processes of urea nitrogen and related microbial populations in soil[J]. Environmental Science and Pollution Research, 2019, 26(30): 31133-31141. DOI:10.1007/s11356-019-06213-8
(  0) 0) |
| [12] |
Yang Z, Deng Y, Zhong L, et al. Responses of soil bacterial and fungal denitrification and associated N2O emissions to organochloride pesticide[J]. Science of the Total Environment, 2023, 905: 167321. DOI:10.1016/j.scitotenv.2023.167321
(  0) 0) |
| [13] |
Lang M, Cai Z C. Effects of fungicide chlorothalonil on soil nitrous oxide and carbon dioxide emissions (In Chinese)[J]. Chinese Journal of Applied Ecology, 2008, 19(12): 2745-2750. [郎漫, 蔡祖聪. 百菌清对土壤氧化亚氮和二氧化碳排放的影响[J]. 应用生态学报, 2008, 19(12): 2745-2750.]
(  0) 0) |
| [14] |
Su X X, Chen Y, Wang Y Y, et al. Disturbances of electron production, transport and utilization caused by chlorothalonil are responsible for the deterioration of soil denitrification[J]. Soil Biology & Biochemistry, 2019, 134: 100-107.
(  0) 0) |
| [15] |
Teng Y, Zhang M Y, Yang G M, et al. Successive chlorothalonil applications inhibit soil nitrification and discrepantly affect abundances of functional genes in soil nitrogen cycling[J]. Environmental Science and Pollution Research, 2017, 24(4): 3562-3571. DOI:10.1007/s11356-016-8080-4
(  0) 0) |
| [16] |
Su X X, Wang Y Y, Peng G L, et al. Long-term effects of chlorothalonil on microbial denitrification and N2O emission in a tea field soil[J]. Environmental Science and Pollution Research, 2020, 27(14): 17370-17381. DOI:10.1007/s11356-020-07679-7
(  0) 0) |
| [17] |
Chen Y, Su X X, Wang Y Y, et al. Short-term responses of denitrification to chlorothalonil in riparian sediments: Process, mechanism and implication[J]. Chemical Engineering Journal, 2019, 358: 1390-1398. DOI:10.1016/j.cej.2018.10.148
(  0) 0) |
| [18] |
Hu H W, MacDonald C A, Trivedi P, et al. Effects of climate warming and elevated CO2 on autotrophic nitrification and nitrifiers in dryland ecosystems[J]. Soil Biology & Biochemistry, 2016, 92: 1-15.
(  0) 0) |
| [19] |
Petersen D G, Blazewicz S J, Firestone M, et al. Abundance of microbial genes associated with nitrogen cycling as indices of biogeochemical process rates across a vegetation gradient in Alaska[J]. Environmental Microbiology, 2012, 14(4): 993-1008. DOI:10.1111/j.1462-2920.2011.02679.x
(  0) 0) |
| [20] |
Lin J T, Xu Z Y, Xue Y H, et al. N2O emissions from soils under short-term straw return in a wheat-corn rotation system are associated with changes in the abundance of functional microbes[J]. Agriculture, Ecosystems & Environment, 2023, 341: 108217.
(  0) 0) |
| [21] |
Rubaiyat A, Hossain M L, Kabir M H, et al. Dynamics of greenhouse gas fluxes and soil physico-chemical properties in agricultural and forest soils[J]. Journal of Water and Climate Change, 2023, 14(10): 3791-3809. DOI:10.2166/wcc.2023.338
(  0) 0) |
| [22] |
Tzanakakis V A, Taylor A E, Bakken L R et al. Relative activity of ammonia oxidizing archaea and bacteria determine nitrification-dependent N2O emissions in Oregon forest soils[J]. Soil Biology & Biochemistry, 2019, 139: 107612.
(  0) 0) |
| [23] |
Zhang M Y, Xu Z H, Teng Y, et al. Non-target effects of repeated chlorothalonil application on soil nitrogen cycling: The key functional gene study[J]. Science of the Total Environment, 2016, 543: 636-643. DOI:10.1016/j.scitotenv.2015.11.053
(  0) 0) |
| [24] |
Fisk L M, Barton L, Maccarone L D, et al. Seasonal dynamics of ammonia-oxidizing bacteria but not Archaea influence soil nitrogen cycling in a semi-arid agricultural soil[J]. Scientific Reports, 2022, 12(1): 7299. DOI:10.1038/s41598-022-10711-0
(  0) 0) |
| [25] |
Wang J, Huang Q, Li Y, et al. A shift from nitrification to denitrification-dominated N2O emission in an acidic soil following organic amendment[J]. Biology and Fertility of Soils, 2023, 59(1): 117-122. DOI:10.1007/s00374-022-01680-7
(  0) 0) |
| [26] |
Shakoor A, Shakoor S, Rehman A, et al. Effect of animal manure, crop type, climate zone, and soil attributes on greenhouse gas emissions from agricultural soils—A global meta-analysis[J]. Journal of Cleaner Production, 2021, 278: 124019. DOI:10.1016/j.jclepro.2020.124019
(  0) 0) |
| [27] |
Cui J, Zhu Z K, Xu X L, et al. Carbon and nitrogen recycling from microbial necromass to cope with C: N stoichiometric imbalance by priming[J]. Soil Biology & Biochemistry, 2020, 142: 107720.
(  0) 0) |
| [28] |
Lazcano C, Zhu-Barker X, Decock C. Effects of organic fertilizers on the soil microorganisms responsible for N2O emissions: A review[J]. Microorganisms, 2021, 9(5): 983. DOI:10.3390/microorganisms9050983
(  0) 0) |
| [29] |
Lang M, Cai Z C. Effects of chlorothalonil and carbendazim on nitrification and denitrification in soils[J]. Journal of Environmental Sciences, 2009, 21(4): 458-467. DOI:10.1016/S1001-0742(08)62292-5
(  0) 0) |
| [30] |
Wang H L, Wang C J, Chen F, et al. Anaerobic degradation of chlorothalonil in four paddy soils[J]. Ecotoxicology and Environmental Safety, 2011, 74(4): 1000-1005. DOI:10.1016/j.ecoenv.2011.01.011
(  0) 0) |
| [31] |
Zhao J N, Feng X H, Hu J, et al. Mineral and microbial properties drive the formation of mineral-associated organic matter and its response to increased tempreture[J]. Global Change Biology, 2024, 30(12): e70004. DOI:10.1111/gcb.70004
(  0) 0) |
| [32] |
Wang W Y, Shen P F, Zhang H P, et al. Study on the relationship between total nitrogen and nitrogen functional microorganisms in soil aggregates under long-term conservation tillage (In Chinese)[J]. Acta Pedologica Sinica, 2024, 61(6): 1653-1667. DOI:10.11766/trxb202307120214 [王威雁, 沈鹏飞, 张侯平, 等. 长期保护性耕作下土壤团聚体全氮与氮功能微生物关系研究[J]. 土壤学报, 2024, 61(6): 1653-1667.]
(  0) 0) |
| [33] |
Zhang M Y, Teng Y, Zhu Y, et al. Residues of chlorothalonil successively applied to soil and effect on microbial communities in two soil types (In Chinese)[J]. Soils, 2014, 46(5): 839-844. [张满云, 滕应, 朱烨, 等. 百菌清重复施用后在土壤中的残留及其对微生物群落的影响[J]. 土壤, 2014, 46(5): 839-844.]
(  0) 0) |
 2025, Vol. 62
2025, Vol. 62


