2. 中国科学院大学, 北京 100049;
3. 中国科学院城市环境研究所, 福建厦门 361024;
4. 浙江大学环境与资源学院, 杭州 310058;
5. 中国科学技术大学环境科学与工程系, 合肥 230026;
6. 深圳大学高等研究院, 深圳 518060
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Urban Environment, Chinese Academy of Sciences, Xiamen, Fujian 361024, China;
4. College of Environmental and Resource Sciences, Zhejiang University, Hangzhou 310058, China;
5. Department of Environmental Science and Engineering, University of Science and Technology of China, Hefei 230026, China;
6. Institute for Advanced Study, Shenzhen University, Shenzhen 518060, China
土壤病毒是地球上最丰富的生物类群,每克土壤中含有约109个病毒颗粒[1]。以往人们将病毒视为一种病原体。然而随着研究的深入,科学界发现病毒在分解宿主释放养分、介导水平基因转移、重构微生物群落结构等方面对宿主细菌的碳氮循环、污染物降解、抗逆等具有重要作用,能直接或间接参与宿主细菌的代谢生理过程[2-5]。
随着生信技术的快速发展,越来越多的研究揭示了病毒在土壤生态系统服务中扮演着“隐形工程师”的角色。以“Soil”和“Virus”为关键词在Web of Science中进行检索,在2020—2024年时间段内共有4 845篇相关文献,近5年该领域发文量呈现整体上升趋势(图 1a);涉及领域主要包括环境科学、植物科学、免疫学和生态学等(图 1b);对关键词进行聚类分析,发现“抗生素抗性(Antibiotic resistance)”、“噬菌体疗法(Page therapy)”、“进化(Evolution)”是出现频率较高的关键词(图 1c),表明土壤病毒的研究主要聚焦于其功能和互作机制。值得注意的是,“水平基因转移(Horizontal gene transfer,HGT)”、“辅助代谢基因(Auxiliary Metabolic Genes,AMGs)”是近几年研究的高频关键词,说明土壤病毒领域的研究愈发受到关注。
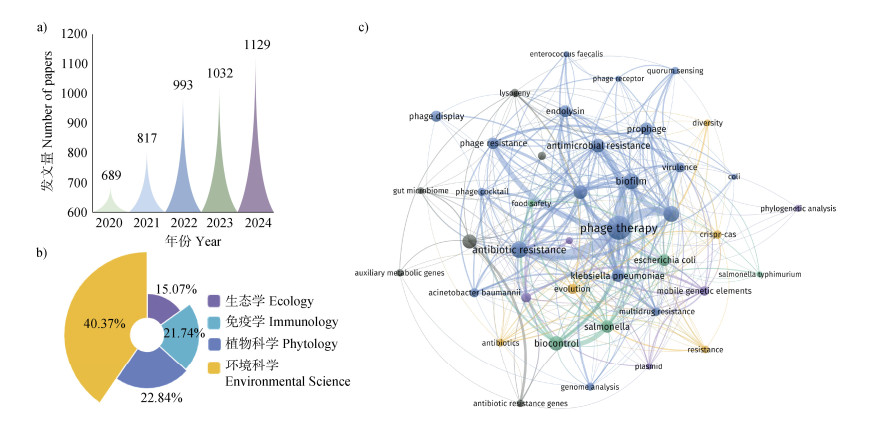
|
图 1 2020—2024年土壤病毒学研究热点 Fig. 1 Research hotspots in soil virology from 2020 to 2024 |
此外,基于合成生物学和多组学技术的发展,病毒在长寿基因调控、盐碱地治理及植物健康等方面的功能潜力也被逐渐发掘[6-8]。但是病毒调控机制的复杂性、宿主特异性及潜在的生态风险仍亟待解决。因此,本文从多个方面介绍土壤病毒在生态系统服务中的功能作用,揭示土壤病毒与宿主细菌互作的分子机制及生态价值,并对今后的研究方向进行展望,希望为开展有关土壤环境病毒组多维生态功能补偿及代谢调控方面研究提供科学指导。
1 土壤病毒组驱动养分元素循环 1.1 碳循环:病毒AMGs增强宿主碳循环潜力病毒通过携带编码碳水化合物活性酶(CAZymes),包括GT2、GT4和V-Csn等AMGs,直接参与宿主的碳代谢过程[9-10]。Zhu等[10]发现,在稻田土壤中噬菌体编码的固碳类AMGs(如丙酮酸代谢相关基因)可以通过水平转移整合到宿主基因组中,在重金属胁迫下显著提升宿主的碳固定效率。相关基因的表达不仅缓解了宿主的代谢压力,还促进了土壤有机碳的长期封存。在含有微塑料污染的土壤中,病毒会直接或通过AMGs促进宿主裂解效应释放可溶性碳源,或通过AMGs提高宿主对可降解微塑料碳源的利用能力,进而间接调节土壤碳储存与碳排放[11]。除上述外,珠峰高海拔土壤中病毒携带的GH23、GT4家族基因,通过降解植物残体中的纤维素加快复杂碳源矿化,并且其丰度与土壤C/N比呈显著正相关,可见病毒在碳循环中具有“分解者”和“调控者”的双重角色[12]。
1.2 氮循环:病毒裂解与基因转移的双重驱动病毒调控氮循环的特点是其具有“自上而下”的裂解效应和“自下而上”的代谢补偿作用。在稻田生态系统中,溶原噬菌体通过表达AMGs调节宿主的氮代谢通路,例如增强氨氧化细菌对羟胺的解毒能力,间接维持氮转化效率[13]。利用宏基因组-稳定性同位素探针技术研究发现,土壤中活性病毒通过侵染固氮细菌(如编码nifH的基因组),携带羟胺脱氢酶等AMGs,将15N同位素标记的氮素转移至病毒基因组,直接参与亚硝酸盐还原过程[14]。病毒裂解宿主之后释放出的氮被其他微生物所吸收,其内在连接关系形成了“病毒-宿主-环境”动态偶联的氮素周转网络。
1.3 磷与硫循环:病毒协同代谢的潜在贡献目前对病毒介导的P、S循环研究较少,但已有证据表明,土壤病毒通过HGT间接参与上述两种代谢活动。研究发现,土壤病毒携带的磷酸酶基因,能够促进土壤微生物在低磷条件下提高磷的吸收量[15]。Liang等[14]发现巨型病毒(Nucleocytoplasmic Large DNA Viruses,NCLDV)中携带碱性磷酸酶基因(alkaline phosphatase D,phoD),揭示了阔口罐病毒Pithovirus具有参与土壤磷循环的潜力。此外,已有研究发现硫氧化相关基因(如soxB、sat)的转移则可以促进硫化物的氧化过程[15]。Gu等[16]在土壤潘多拉病毒Pandoraviridae、巨型病毒Marseilleviridae和微小噬菌体Mimiviridae基因组中发现了硫氧化基因sat基因,系统进化树分析结果显示,这些AMGs是由真核生物或原核生物水平转移到病毒的。尽管它们在P、S循环上的贡献不如在C、N循环上大,但对于维持土壤元素循环和平衡同样发挥重要作用。
1.4 多元素代谢通路的多维调控病毒驱动的元素循环不是独立存在的,而是通过代谢通路的互作共同形成了一个协同网络(图 2)。碳固定过程(如Calvin-Benson-Bassham循环)需要氮代谢提供能量,而病毒携带的AMGs既可以增强宿主对C同化的能力也可以提升N同化能力。稻田病毒通过上调宿主固碳基因与氮还原基因的共表达,显著提升土壤C/N比,体现了病毒在多元素耦合中的枢纽作用[10,16]。此外,病毒裂解释放的胞内物质(如核酸、磷脂)为其他微生物提供了以C、N和P为主要元素组成的复合资源,进一步加强了养分循环的级联效应[13,16]。土壤病毒组通过水平基因转移与代谢补偿,推动养分元素自下而上的循环。病毒主要以C、N循环为主导,协同调控P、S代谢,组成一套复杂而高效的生态功能网络。因此,持续挖掘和丰富病毒组多样性的研究内容有助于拓展其生态学作用的广度和深度,并为后期的土壤健康管理和对抗全球气候变化提供新方法。
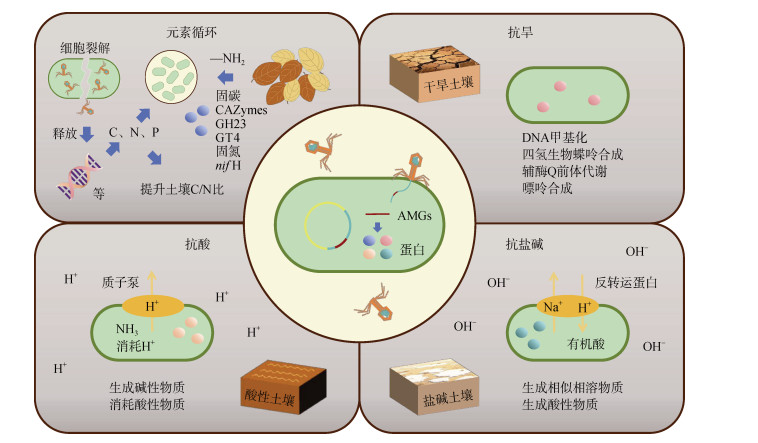
|
图 2 病毒编码的AMGs调控元素循环、抵御逆境胁迫 Fig. 2 Virus-encoded AMGs regulate biogeochemical cycling and confer stress resistance |
土壤病毒是土壤微生物逆境适应与群落重塑的工程师。病毒携带的辅助代谢基因在宿主适应酸化、盐碱、干旱等极端环境中发挥积极作用。
2.1 病毒编码产碱和耗酸基因,对冲土壤酸度酸性土壤(pH < 5.5)广泛分布于热带雨林、农业田及工业污染区域。低pH土壤会严重抑制植物生长、微生物活性及养分循环效率。土壤细菌能够通过质子泵、酸代谢、产生碱性物质来抵抗酸胁迫,而病毒携带的抗酸辅助代谢基因在酸性土壤改良中发挥缓冲作用(图 2)。研究发现,在酸性土壤中同时添加有机肥和碱渣后,病毒和细菌的α多样性显著增加,携带抗酸辅助代谢基因的病毒丰度显著上升,而病毒携带的coxC、asnB、sdhB等抗酸辅助代谢基因数量也显著增加。进一步向酸化红壤移植10倍病毒后,土壤pH较对照组高0.12,表明土壤病毒群落的整体移植不仅有利于细菌的生存,还有助于提升土壤的pH缓冲容量[17]。
2.2 病毒编码产酸和耗碱基因,对冲土壤碱度盐碱地(pH > 8.5,含盐量 > 0.1%)广泛分布在干旱、半干旱及沿海地区,其高盐碱度会使土壤出现板结现象、养分失衡、微生物活性受到抑制,对农业生产及生态恢复产生不利影响[18-19]。研究表明,土壤病毒组通过编码耐盐碱AMGs参与宿主抗逆性调控,形成了一套特殊的生态功能补偿机制[20]。通过对中国的松嫩平原盐碱土宏病毒组学比较发现,高盐碱组病毒携带的trkH(钾转运)、glgC(糖原合成)及PK(丙酮酸激酶)等关键AMGs丰度显著增加,以上基因可通过控制宿主的离子平衡(Na+/H+反转运)、渗透保护物质(相容性溶质)合成及酸性代谢物(丙酮酸、乳酸)生成等分子机制,有效中和土壤碱度(图 2)。值得注意的是,病毒AMGs的功能协同性在碳氮磷生物地球化学循环中尤为显著,通过将代谢模块协同表达可以提高宿主微生物对养分的竞争能力,实现对盐碱土微域环境的重构。进化选择压力分析进一步揭示,高盐碱组病毒的非同义突变率(pN/pS=0.82)显著高于对照组(pN/pS= 0.41,P < 0.001),说明病毒受到较强正向选择,快速进化是为宿主微生物群落提供应对胁迫的储备。此发现系统揭示了病毒组以“代谢调控-元素循环-进化驱动”的多维功能补偿机制,全面阐释了极端环境土壤病毒组适应性进化的生物学规律,为盐碱地生态功能定向调控提供理论依据。
2.3 病毒编码耐干旱基因干旱减少土壤中水分以及微生物生长速率,改变土壤微生物群落结构。Wu等[1]发现三个水分条件差异较大的土壤样品中,干旱及少雨地区土壤的病毒丰度远高于中雨区和多雨区土壤病毒丰度。Liu等[21]发现,在长期干旱条件下,病毒编码的AMGs数量显著增加,其中249个与碳利用有关、44个参与有机氮代谢。它们通过优化宿主的资源分配,帮助细菌在资源受限的环境中维持代谢活性。干旱条件下病毒富集的DNMT1、GCH1、queD和purD等基因,会分别参与DNA甲基化、四氢生物蝶呤合成、辅酶Q前体代谢及嘌呤合成,直接促进宿主在缺水状况下核苷酸的合成和能量供给(图 2)。长期干旱使得原核生物—病毒网络负连接的比例由20.43%增长为40.60%,病毒对低适应性宿主的捕食压力增强[22]。长期干旱下病毒AMGs使宿主获得的优势更多地反映在宿主提高生存机会上,并促进群落更强的抵御外部恶劣环境的能力,同时加快低适应性的裂解,优化整个群落对抗逆境的能力,甚至可能更为剧烈。
2.4 极端环境驱使病毒进入溶原状态病毒溶原化(lysogeny)作为极为有效的环境适应机制,在极端生态系统中展现出巨大的进化优势。当宿主细菌处于pH异常(< 5或 > 9)、高盐胁迫(Na+ > 0.5 mol·L–1)或营养限制(C/N > 30)等胁迫环境时,病毒倾向于以前噬菌体(prophage)的形式整合到宿主基因组中,和宿主建立共生存关系。研究表明中国安徽郎溪酸化土样沿纵向不同深度向下由pH从4.19(强酸性)逐渐升高为5.42(弱酸性),病毒溶原率从38.03%急剧下降至4.46%(P < 0.001),该动态变化与可培养宿主丰度呈显著负相关(r = –0.83,P=0.004)[1]。同样,松嫩平原盐碱土研究发现,高盐碱组溶原病毒占比显著高于低盐碱组(P < 0.05)[20]。
该生存策略的进化驱动力主要体现在两个方面:其一,物理化学保护机制,游离病毒颗粒在极端pH或高盐条件下DNA损伤率可达72%~89%·d–1,而整合态前噬菌体通过宿主DNA修复系统(如SOS响应)将遗传物质失活率降低至5%~8%·d–1;其二,能量代谢优化,溶原状态下病毒利用宿主复制机器实现被动增殖,其单位基因组复制能耗仅为裂解周期的1/15~1/20(0.8 vs. 12~16 ATP/bp)[18,20]。Santos-Medellín等[23]通过干旱实验证实,土壤含水量每降低10%,溶原标记基因(整合酶int、阻遏蛋白cI)丰度提升1.7倍~2.3倍(P < 0.05),提示干旱胁迫通过选择压驱动病毒群落结构重塑。
从生态系统层面解析,溶原化策略构建了“病毒-宿主-环境”三方互作的稳定结构:胁迫期寄生模式下利用基因组潜伏维持种群延续;当环境条件变好后进入裂解期寄生裂解促进病毒扩散增殖。这种动态平衡机制不仅保障了病毒组的遗传多样性储备,还通过前噬菌体编码的胁迫响应基因(如clpP蛋白酶、groEL分子伴侣)增强宿主的环境适应性,最终提升微生物群落的整体抗逆能力。这一结果说明病毒组能够在极端环境下具有其存在意义以及生态定位,也从另一个方面为阐释病毒组极端环境下生态位维持提供了一种新的理论基础。
3 病毒组学与污染土壤修复 3.1 有机污染治理中的病毒修复功能土壤中广泛存在的有机污染物(如有机氯农药和多环芳烃等)显著干扰微生物群落结构与功能[24],但病毒的参与为有机污染物的降解与污染土壤修复提供了新方向(图 3)。在中国有机氯农药污染农田的病毒宏基因组研究中发现,污染土壤的病毒多样化水平要高于对照组土壤,病毒组中AMGs显著富集,尤其是与有机氯农药降解密切相关的L-2- haloacid dehalogenase(L-DEX)基因[7]。这类基因能帮助宿主在有毒性的有机氯农药的亚抑制浓度下维护正常代谢水平及持续生物生长活性,即能够在污染的压力下辅助宿主实现“功能外包”的目的。Xia等[8]研究发现,在BaP(benzo[a]pyrene)污染下蚯蚓肠道病毒组中,低浓度BaP刺激AMGs表达、促进溶原性病毒积累,而高浓度污染则激活裂解周期,加剧噬菌体与宿主的代谢耦合,调动病毒编码的抗氧化和降解功能。因此病毒可以在不同污染条件下通过选择不同生存策略在裂解周期或者溶源状态之间灵活转换,从而能够有效地辅助降解有机污染物。
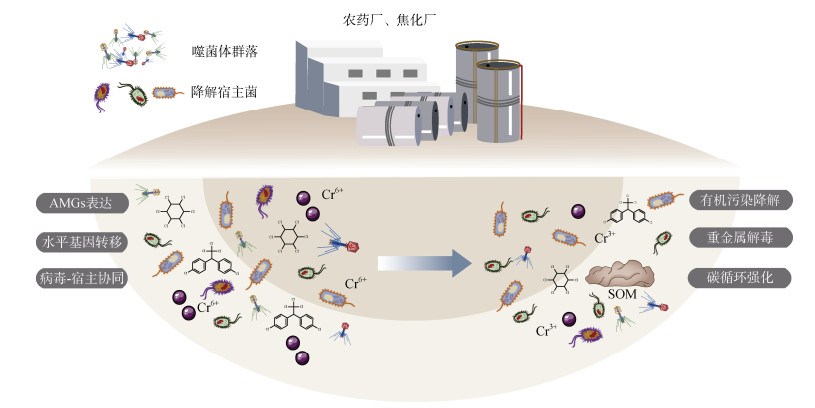
|
图 3 病毒协助宿主消纳土壤中污染物 Fig. 3 Viruses facilitate host degradation of contaminants in soil |
重金属污染下的土壤环境不仅构成代谢胁迫,也加剧了病毒-宿主相互作用的复杂性(图 3)。研究表明,病毒组在重金属胁迫下通过携带和转移重金属抗性基因,构建出一套“遗传净化器”系统。在高浓度Cr污染土壤中,原核生物的防御系统(如CRISPR系统)经历显著适应性重构,与微生物抗重金属基因共表达增强,病毒携带AMGs参与宿主重金属解毒代谢的频率上升。同时,病毒编码防御系统在移动遗传元件(如质粒和病毒)中的比例显著提升,增强了重金属抗性基因在群落间的“共享”效率[25]。另一项病毒群落结构与溶原性比例研究显示,污染梯度越高,病毒向溶原型演化的趋势越显著;尤其是广谱宿主噬菌体的增强,促进了重金属抗性遗传模块在微生物网络中的传播[26]。由此可见,病毒不仅能够保护宿主抵御重金属胁迫,还能作为载体通过水平基因转移传递重金属抗性基因,从而帮助宿主形成重金属抗性机制。
3.3 污染胁迫因子诱导环境中病毒驱动碳循环强化在污染物胁迫叠加气候骤变的背景下,固碳功能已成为考量土壤生态修复效果的重要指标。在众多土壤类型中,稻田土壤作为典型的间歇性厌氧环境,其碳循环受病毒调控的研究日益受到关注。最新研究表明,淹水生境病毒组中约23.7%的AMGs与碳固存有关[10]。碳固存AMGs在重金属污染胁迫下表达上调,并伴随13C标记的微生物生物量碳含量显著上升(提高约35.4%),而13CO2排放降低约17.9%,表明病毒在碳固定路径中的调控作用。病毒通过调节宿主的碳代谢潜力,成为污染土壤中兼具降解功能与生态修复潜力的“隐性净化器”。
4 病毒是促进植物生长发育的潜在驱动力近年来研究表明,病毒在调控植物健康中具有显著的益生功能(图 4)。通过感染病原微生物、调节土壤微生物群落结构、促进植物微生物的互作,病毒能够间接增强植物的抗病性、养分吸收能力和环境适应性。已有研究发现了噬菌体编码植物促生相关AMG的初步证据,例如,Braga等[27]从两种法国农业土壤中发现土壤噬菌体AMGs含有琥珀酸聚糖生物合成酶和3-氧酰基[酰基载体蛋白]还原酶,在根瘤形成过程中起重要作用。这些AMGs与有机质降解、植物根系发育、根际微生物生存策略有关。还有研究发现噬菌体编码钼储存蛋白(MosAB)和ProQ结构域,有助于植物提高氮素利用效率和抵御非生物胁迫[28]。Yang等[29]还发现了噬菌体编码与琥珀酸聚糖生物合成有关的AMGs以及3-酮脂酰ACP还原酶和乙酰乳酸合成酶,它们参与产生根瘤菌挥发性物质,刺激根系形成并增强抗病性。此外,还有大量研究发现噬菌体可用于治疗植物病害,例如:葡萄皮尔斯病、马蹄莲软腐病、水稻苍白褐变病、番茄细菌性斑点、柑橘腐烂病、葡萄柚柑橘细菌性斑点病等[30-33]。总体而言,土壤根际环境中,噬菌体和细菌相辅相成,互相影响,共同抵御外界环境胁迫,促进植物的生长发育。
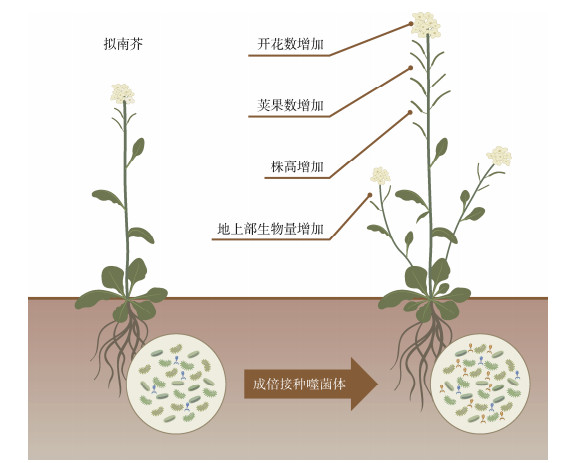
|
图 4 噬菌体成倍移植促进植物生长发育 Fig. 4 Phage multiplication promotes plant growth and development |
抗生素抗性基因(Antibiotic Resistance Genes,ARGs)在土壤环境中的迁移与扩散过程受到多种生物学机制的调控,HGT是介导微生物间遗传物质传递的一个重要过程[34]。噬菌体会直接影响HGT的发生频率。噬菌体通过直接基因转移事件或间接参与调节微生物群落动态,在ARGs的传播网络中展现出双重功能[35]:一方面噬菌体通过转导、裂解释放宿主DNA等方式直接促进ARGs的传播;另一方面噬菌体通过调控微生物群落结构或靶向裂解耐药菌间接抑制ARGs的扩散。这种复杂的相互作用使得噬菌体成为土壤ARGs传播的关键媒介,尤其在人类活动频繁的土壤生态系统中表现更为重要(图 5)。
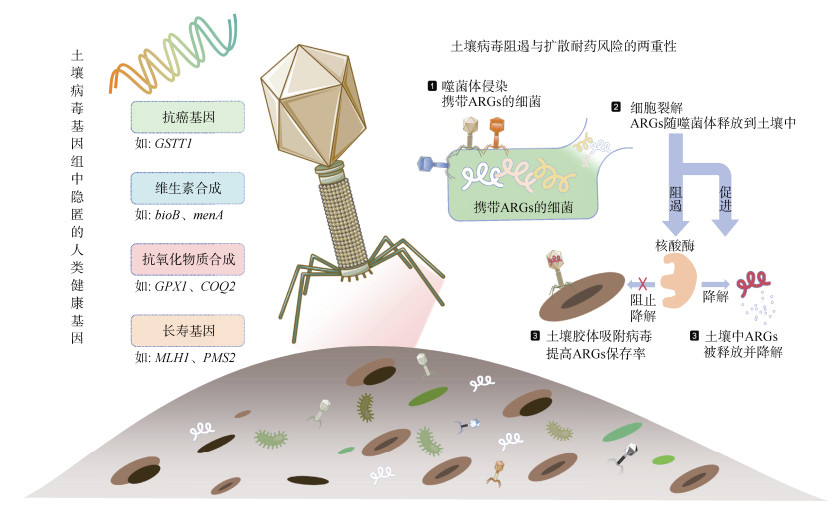
|
图 5 土壤病毒组中的抗生素抗性基因和人类健康基因 Fig. 5 Antibiotic resistance genes and human health-related genes in the soil virome |
研究表明,噬菌体与多重耐药菌共存时,ARGs丰度显著增加[36]。由噬菌体介导的基因转移可以跨宿主细胞进行,基因供体和受体细菌无须同时存在于土壤中,从而增加土壤中ARGs的丰度[37]。陈莫莲等[38]研究发现,噬菌体裂解宿主细胞释放出的子代噬菌体会随着土壤胶体组分(例如黏粒矿物和腐殖质)的吸附作用,减少携带ARGs的病毒被核酸酶的降解概率,不仅会延迟遗传信息的传递,还会扩大其影响的时间尺度。此外,农业活动(如粪肥施用)可能扩大噬菌体的宿主范围,提高ARGs转移的广度和AMGs的多样性。
5.2 噬菌体驱动的ARGs抑制策略噬菌体对ARGs的作用也值得重点关注。裂解性噬菌体通过靶向裂解携带ARGs的宿主细菌,从而降低耐药菌的丰度。噬菌体疗法已被用于靶向清除土壤病原菌(如青枯菌),从而减少ARGs的传播风险[39]。此外,噬菌体可通过调节土著微生物群落间接抑制耐药菌增殖。Yang等[29]研究发现根际土壤中的某些土著细菌可抑制青枯菌,但若其专性噬菌体过度侵染,会削弱这种抑制作用。因此,合理设
计噬菌体组合以维持生态平衡的稳定,成为一种潜在的干预措施。近年来,工程噬菌体的发展为精准干预ARGs传播提供了新思路。Ye等[39]研究发现携带CRISPR-Cas系统的工程噬菌体可特异性切割ARGs,Cas9噬菌体已成功清除病原菌内的耐药基因,有效抑制其水平转移。土壤病毒组阻遏与扩散耐药风险的机制受环境因素的动态调控,揭示噬菌体在ARGs生态网络中兼具传播与阻控的双重性,能够为土壤抗性基因风险管理提供理论及技术突破口。
6 土壤病毒基因组中隐匿的人类健康基因土壤病毒组在生态循环和环境污染修复中的作用已有众多研究,然而鲜为人知的是土壤病毒组中不仅携带对生态有益的基因,还携带一部分对人类健康有益的基因。在IMG/VR v4数据库中进行比对与检索,发现土壤病毒基因组中广泛存在多种对人类及宿主具有潜在益处的益生基因,涵盖抗癌、维生素合成、抗氧化及延长寿命四大功能类别。这些基因通过转导整合至病毒基因组,形成AMGs,并在宿主代谢过程中发挥关键作用(图 5)。
6.1 病毒编码人类健康基因土壤病毒编码的抗癌基因GSTT1通过催化谷胱甘肽与致癌物(如苯并芘)的结合,促进其排出体外,可有效地减少肺癌、结肠癌的发生率。病毒编码的维生素合成基因bioB和menA分别参与生物素(biotin)与维生素K2的合成,帮助进行宿主能量代谢与凝血[40-41]。同样可由病毒编码的抗氧化基因GPX1(谷胱甘肽过氧化物酶1),能够通过清除活性氧自由基,保护细胞膜与线粒体功能,延缓动脉粥样硬化以及神经退行性疾病进程[42]。此外,土壤病毒基因中广泛存在的长寿基因SIRT1可延缓糖尿病或阿尔茨海默病的发病进程[43]。这些基因的分布与功能显示病毒通过整合基因资源库从而达到保护人类身体健康的潜在作用。
6.2 病毒携带不同人类健康基因的协同效益不同类别的益生基因在病毒基因组中或能通过功能协同增强对人类健康的综合效益。例如,抗氧化基因GPX1与抗癌基因GSTT1在病毒基因组共现时,前者清除自由基以降低DNA氧化损伤,后者直接抑制肿瘤转移通路,形成双重抗癌屏障[44]。在土壤病毒中,携带arsC与menA的噬菌体可能通过双重策略影响人类健康:一方面通过砷解毒减少农作物中砷的富集,阻断砷通过食物链进入人体[45];另一方面通过宿主微生物(如根瘤菌)的维生素合成改善肠道菌群功能,间接促进人体对钙和维生素K的吸收[41]。土壤病毒基因组中多次检测到错配修复(MMR)基因MLH1与PMS2,能够修复宿主基因组错误,减少突变积累,延长宿主寿命[46-47]。这种基因协同性不仅体现了病毒基因组的进化智慧,也为靶向调控人类代谢网络提供了分子基础。
7 研究不足与展望尽管目前有关土壤环境病毒组的生态功能研究已取得了很大进步,但依然存在局限性与挑战。首先,功能解析的深度与广度不足。现有研究主要依靠基因预测性分析,病毒基因的实际表达活性、宿主特异性及环境适应权衡机制尚未明确,因此功能认知与实践应用间有一定脱节。其次,研究内容较为局限。研究病毒调节碳氮循环较多,对磷、硫等其他元素的代谢调控较少,对于地域生境以及宿主类型的覆盖也各有不同,在障碍土病毒组功能方面的研究几乎处于空白。最后,技术方法亟待突破。传统宏基因组技术对未培养病毒的功能注释能力有限,病毒-宿主互作过程的时间和空间动态特征不易被准确捕获,实验室模拟和实际土壤环境之间的复杂性差异会造成结果的生态相关性不足。
未来的相关研究需要围绕多学科交叉、多技术融合为核心,在理论创新和技术进步方面有所突破。在机制层面,应结合单病毒基因组学、原位代谢组学,揭示病毒组在元素循环中的全息调控网络,为污染土壤修复与碳中和提供理论支撑。在技术层面,需开发跨尺度环境响应模型,结合宏转录组与蛋白质组数据,突破病毒功能注释瓶颈;同时设计工程化噬菌体,通过靶向递送抗逆基因定向增强宿主适应性,并系统评估其生态安全性。在应用层面,应加强定位实验及长期监测的相关研究,建立病毒组数据库提升数据通量,挖掘有益微生物组提升作物品质。
致谢 感谢中国科学院南京土壤研究所吴云玲、刘舒月、李梦柯、钱旻和南京农业大学高卓凡、沈周舟对文章撰写提供的帮助。
| [1] |
Wu R N, Davison M R, Nelson W C, et al. DNA viral diversity, abundance, and functional potential vary across grassland soils with a range of historical moisture regimes[J]. mBio, 2021, 12(6): e02595-21.
(  0) 0) |
| [2] |
González-Villalobos E, Balcázar J L. Does phage- mediated horizontal gene transfer represent an environmental risk?[J]. Trends in Microbiology, 2022, 30(11): 1022-1024. DOI:10.1016/j.tim.2022.07.011
(  0) 0) |
| [3] |
Lee S, Sieradzki E T, Nicol G W, et al. Propagation of viral genomes by replicating ammonia-oxidising archaea during soil nitrification[J]. The ISME Journal, 2023, 17(2): 309-314. DOI:10.1038/s41396-022-01341-5
(  0) 0) |
| [4] |
Kuhlisch C, Schleyer G, Shahaf N, et al. Viral infection of algal blooms leaves a unique metabolic footprint on the dissolved organic matter in the ocean[J]. Science Advances, 2021, 7(25): eabf4680. DOI:10.1126/sciadv.abf4680
(  0) 0) |
| [5] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880. DOI:10.1038/s41564-018-0190-y
(  0) 0) |
| [6] |
Mao J, Zhang Q, Zhuang Y, et al. Reactivation of senescence-associated endogenous retroviruses by ATF3 drives interferon signaling in aging[J]. Nature Aging, 2024, 4(12): 1794-1812. DOI:10.1038/s43587-024-00745-6
(  0) 0) |
| [7] |
Zheng X X, Jahn M T, Sun M M, et al. Organochlorine contamination enriches virus-encoded metabolism and pesticide degradation associated auxiliary genes in soil microbiomes[J]. The ISME Journal, 2022, 16(5): 1397-1408. DOI:10.1038/s41396-022-01188-w
(  0) 0) |
| [8] |
Xia R, Sun M M, Balcázar J L, et al. Benzo [a] pyrene stress impacts adaptive strategies and ecological functions of earthworm intestinal viromes[J]. The ISME Journal, 2023, 17(7): 1004-1014. DOI:10.1038/s41396-023-01408-x
(  0) 0) |
| [9] |
Wu R N, Chai B L, Cole J R, et al. Targeted assemblies of cas1 suggest CRISPR-Cas's response to soil warming[J]. The ISME Journal, 2020, 14(7): 1651-1662. DOI:10.1038/s41396-020-0635-1
(  0) 0) |
| [10] |
Zhu D, Liu S Y, Sun M M, et al. Adaptive expression of phage auxiliary metabolic genes in paddy soils and their contribution toward global carbon sequestration[J]. Proceedings of the National Academy of Sciences of the United States of America, 2024, 121(49): e2419798121.
(  0) 0) |
| [11] |
Wang L, Lin D, Xiao K Q, et al. Soil viral-host interactions regulate microplastic-dependent carbon storage[J]. Proceedings of the National Academy of Sciences of the United States of America, 2024, 121(45): e2413245121.
(  0) 0) |
| [12] |
Zhu Y, Zhang Y, Yan S, et al. Viral community structure and functional potential vary with lifestyle and altitude in soils of Mt. Everest[J]. Environment International, 2023, 178: 108055. DOI:10.1016/j.envint.2023.108055
(  0) 0) |
| [13] |
Jia P, Liang J L, Lu J L, et al. Soil keystone viruses are regulators of ecosystem multifunctionality[J]. Environment International, 2024, 191: 108964. DOI:10.1016/j.envint.2024.108964
(  0) 0) |
| [14] |
Liang J L, Feng S W, Jia P, et al. Unraveling the habitat preferences, ecological drivers, potential hosts, and auxiliary metabolism of soil giant viruses across China[J]. Microbiome, 2024, 12(1): 136. DOI:10.1186/s40168-024-01851-8
(  0) 0) |
| [15] |
Hu X J, Gu H D, Semenov M, et al. Biogeographic patterns and adaptive strategies of microbial carbon metabolic profiles in paddy soils in the Chinese Mollisol region[J]. Geoderma, 2025, 456: 117265. DOI:10.1016/j.geoderma.2025.117265
(  0) 0) |
| [16] |
Gu H D, Hu X J, Zhang J Y, et al. Biogeographic patterns of viral communities, ARG profiles and virus-ARG associations in adjacent paddy and upland soils across black soil region[J]. Journal of Hazardous Materials, 2025, 485: 136909. DOI:10.1016/j.jhazmat.2024.136909
(  0) 0) |
| [17] |
Bi L, Du S, Fitzpatrick R, et al. Potential viral influence on sulfur metabolism in acid sulfate soils[J]. Soil Biology and Biochemistry, 2025, 205: 109773. DOI:10.1016/j.soilbio.2025.109773
(  0) 0) |
| [18] |
Meier D V, Pjevac P, Bach W, et al. Niche partitioning of diverse sulfur-oxidizing bacteria at hydrothermal vents[J]. The ISME Journal, 2017, 11(7): 1545-1558. DOI:10.1038/ismej.2017.37
(  0) 0) |
| [19] |
Zeng F F, Zhu Y H, Zhang D L, et al. Metagenomic analysis of the soil microbial composition and salt tolerance mechanism in Yuncheng Salt Lake, Shanxi Province[J]. Frontiers in Microbiology, 2022, 13: 1004556. DOI:10.3389/fmicb.2022.1004556
(  0) 0) |
| [20] |
Xu Y C, Magwanga R O, Yang X, et al. Genetic regulatory networks for salt-alkali stress in Gossypium hirsutum with differing morphological characteristics[J]. BMC Genomics, 2020, 21(1): 15. DOI:10.1186/s12864-019-6375-9
(  0) 0) |
| [21] |
Liu C, Chen Z J, Wang X L, et al. Response of soil phage communities and prokaryote-phage interactions to long-term drought[J]. Environmental Science & Technology, 2025, 59(6): 3054-3066.
(  0) 0) |
| [22] |
Aframian N, Omer Bendori S, Kabel S, et al. Dormant phages communicate via arbitrium to control exit from lysogeny[J]. Nature Microbiology, 2022, 7(1): 145-153.
(  0) 0) |
| [23] |
Santos-Medellín C, Blazewicz S J, Pett-Ridge J, et al. Viral but not bacterial community successional patterns reflect extreme turnover shortly after rewetting dry soils[J]. Nature Ecology & Evolution, 2023, 7(11): 1809-1822.
(  0) 0) |
| [24] |
Wang G H, Berdy B M, Velasquez O, et al. Changes in microbiome confer multigenerational host resistance after sub-toxic pesticide exposure[J]. Cell Host & Microbe, 2020, 27(2): 213-224.
(  0) 0) |
| [25] |
Huang D, Liao J Q, Balcazar J L, et al. Adaptive modification of antiviral defense systems in microbial community under Cr-induced stress[J]. Microbiome, 2025, 13(1): 34. DOI:10.1186/s40168-025-02030-z
(  0) 0) |
| [26] |
Huang D, Yu P F, Ye M, et al. Enhanced mutualistic symbiosis between soil phages and bacteria with elevated chromium-induced environmental stress[J]. Microbiome, 2021, 9(1): 150. DOI:10.1186/s40168-021-01074-1
(  0) 0) |
| [27] |
Braga L P P, Coutinho F H, Amgarten D E, et al. Novel virocell metabolic potential revealed in agricultural soils by virus-enriched soil metagenome analysis[J]. Environmental Microbiology Reports, 2021, 13(3): 348-354. DOI:10.1111/1758-2229.12939
(  0) 0) |
| [28] |
Braga L P P, Schumacher R I. Awaking the dormant virome in the rhizosphere[J]. Molecular Ecology, 2023, 32(11): 2985-2999. DOI:10.1111/mec.16893
(  0) 0) |
| [29] |
Yang K M, Wang X F, Hou R J, et al. Rhizosphere phage communities drive soil suppressiveness to bacterial wilt disease[J]. Microbiome, 2023, 11(1): 16. DOI:10.1186/s40168-023-01463-8
(  0) 0) |
| [30] |
Soares J M, Tanwir S E, Grosser J W, et al. Development of genetically modified citrus plants for the control of citrus canker and huanglongbing[J]. Tropical Plant Pathology, 2020, 45(3): 237-250. DOI:10.1007/s40858-020-00362-9
(  0) 0) |
| [31] |
Balogh B, Jones J B, Momol M T, et al. Improved efficacy of newly formulated bacteriophages for management of bacterial spot on tomato[J]. Plant Disease, 2003, 87(8): 949-954. DOI:10.1094/PDIS.2003.87.8.949
(  0) 0) |
| [32] |
Choudhary M, Pereira J, Davidson E B, et al. Improved persistence of bacteriophage formulation with nano N-acetylcysteine-zinc sulfide and tomato bacterial spot disease control[J]. Plant Disease, 2023, 107(12): 3933-3942. DOI:10.1094/PDIS-02-23-0255-RE
(  0) 0) |
| [33] |
Jiang H B, Li C X, Huang X F, et al. Phage combination alleviates bacterial leaf blight of rice(Oryza sativa L.)[J]. Frontiers in Plant Science, 2023, 14: 1147351. DOI:10.3389/fpls.2023.1147351
(  0) 0) |
| [34] |
Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440.
(  0) 0) |
| [35] |
Zhang Y, Guo Y J, Qiu T L, et al. Bacteriophages: Underestimated vehicles of antibiotic resistance genes in the soil[J]. Frontiers in Microbiology, 2022, 13: 936267. DOI:10.3389/fmicb.2022.936267
(  0) 0) |
| [36] |
Wang Q, Wang M, Yang Q X, et al. The role of bacteriophages in facilitating the horizontal transfer of antibiotic resistance genes in municipal wastewater treatment plants[J]. Water Research, 2025, 268: 122776. DOI:10.1016/j.watres.2024.122776
(  0) 0) |
| [37] |
Goyal S M, Gerba C P. Comparative adsorption of human enteroviruses, Simian rotavirus, and selected bacteriophages to soils[J]. Applied and Environmental Microbiology, 1979, 38(2): 241-247. DOI:10.1128/aem.38.2.241-247.1979
(  0) 0) |
| [38] |
Chen M L, An X L, Yang K, et al. Soil phage and their mediation on the horizontal transfer of antibiotic resistance genes: A review (In Chinese)[J]. Chinese Journal of Applied Ecology, 2021, 32(6): 2267-2274. [陈莫莲, 安新丽, 杨凯, 等. 土壤噬菌体及其介导的抗生素抗性基因水平转移研究进展[J]. 应用生态学报, 2021, 32(6): 2267-2274.]
(  0) 0) |
| [39] |
Ye M, Su J Q, An X L, et al. Silencing the silent pandemic: Eliminating antimicrobial resistance by using bacteriophages[J]. Science China Life Sciences, 2022, 65(9): 1890-1893. DOI:10.1007/s11427-022-2182-6
(  0) 0) |
| [40] |
Cleary P P, Campbell A, Chang R. Location of promoter and operator sites in the biotin gene cluster of Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 1972, 69(8): 2219-2223.
(  0) 0) |
| [41] |
Xv F, Chen J P, Duan L L, et al. Research progress on the anticancer effects of vitamin K2[J]. Oncology Letters, 2018, 15(6): 8926-8934.
(  0) 0) |
| [42] |
Schwarz M, Löser A, Cheng Q, et al. Side-by-side comparison of recombinant human glutathione peroxidases identifies overlapping substrate specificities for soluble hydroperoxides[J]. Redox Biology, 2023, 59: 102593. DOI:10.1016/j.redox.2022.102593
(  0) 0) |
| [43] |
Xu C Y, Wang L, Fozouni P, et al. SIRT1 is downregulated by autophagy in senescence and ageing[J]. Nature Cell Biology, 2020, 22(10): 1170-1179. DOI:10.1038/s41556-020-00579-5
(  0) 0) |
| [44] |
Maukonen M, Havulinna A, Partonen T, et al. Associations of chronotype with clock genes polymorphisms[J]. Proceedings of the Nutrition Society, 2020, 79(OCE2): E181. DOI:10.1017/S0029665120001299
(  0) 0) |
| [45] |
Tang X, Yu P F, Tang L, et al. Bacteriophages from arsenic-resistant bacteria transduced resistance genes, which changed arsenic speciation and increased soil toxicity[J]. Environmental Science & Technology Letters, 2019, 6(11): 675-680.
(  0) 0) |
| [46] |
Pretta A, Donisi C, Persano M, et al. Mismatch repair proteins(MMR)expression as predictive factor in locally advanced rectal cancer[J]. Journal of Clinical Oncology, 2022, 40(4_suppl): 182-182. DOI:10.1200/JCO.2022.40.4_suppl.182
(  0) 0) |
| [47] |
Pandey S, Rangarajan T, Zakalik D. Clinical characteristics of patients with PMS2 mutations[J]. Journal of Clinical Oncology, 2022, 40(16_suppl): 10610. DOI:10.1200/JCO.2022.40.16_suppl.10610
(  0) 0) |
 2026, Vol. 63
2026, Vol. 63


