2. 浙江省农业资源与环境重点实验室, 浙江大学环境与资源学院土水资源与环境研究所, 杭州 310058
2. Zhejiang Provincial Key Laboratory of Agricultural Resources and Environment, Institute of Soil and Water Resources and Environmental Science, College of Environmental and Resource Sciences, Zhejiang University, Hangzhou 310058, China
土壤作为地球关键带核心组分,其微生态系统(即土壤微生物组)是由土壤中所有的微生物(包括细菌、真菌、古菌、原生动物和病毒等)及其遗传信息和所处环境共同构成的复杂生态系统,是支撑粮食生产和生态系统服务的基础资源[1]。耕地土壤微生态健康对保障作物高产稳产、农产品质量及农业绿色转型至关重要[2-3]。然而,高强度人为活动的持续影响致使全球耕地土壤遭受传统农药和新污染物(如酞酸酯类塑化剂、抗生素和微塑料)等多种有机污染物的复合胁迫[4-6],严重威胁了土壤微生态的稳态、功能和恢复力[6-7],成为当前限制耕地产能提升和农业可持续发展的关键瓶颈。
有机复合污染通过协同、拮抗或改变环境行为等方式相互作用,其环境效应和生态毒性远超单一污染物简单叠加[6],从而对土壤微生态产生复杂的系统性影响[8-9],引发土壤肥力退化、土传病害加剧、作物减产及固碳等特定生态系统服务功能弱化,直接威胁农业生产的可持续性和区域生态安全[1,10]。阐明有机复合污染下土壤微生态的响应机制,并发展有效的微生态健康重塑策略,一直是土壤科学研究的前沿。
传统理化修复成本高昂且易致二次污染,基于单一菌株的生物修复对复合污染效果有限且不稳定[11-12]。面对有机复合污染挑战,融合微生物生态学、合成生物学、系统生物学及多组学技术的合成微生物组学(Synthetic Microbiome,SynMicro)为精准干预和重塑受损土壤微生态系统提供了新思路[11-12]。SynMicro通过“自上而下”(从复杂群落简化)或“自下而上”(从功能单菌或模块组装)的策略,可基于对微生物互作机制的理解,理性设计并构建结构明确、功能可预测、环境适应性强的定制化微生物群落[8-11]。这些合成菌群通过代谢协作、功能互补或生态位分化,更高效稳定地执行特定任务,如协同降解复合污染物、精准抑制病原菌、高效转化利用养分等,展现出重塑土壤微生态健康的巨大潜力[13-14]。随着宏基因组学、高通量筛选、单细胞生态学与AI驱动的生态设计方法的交叉融合,如何将这些创新工具与SynMicro相结合以重塑有机复合污染耕地土壤健康,成为亟待突破的核心问题[13-15]。
有鉴于此,本文旨在系统梳理耕地土壤有机复合污染的普遍性、复杂性及对土壤微生态健康的潜在风险;重点阐述如何利用SynMicro这一前沿策略,重塑有机复合污染胁迫下的耕地土壤微生态健康;深入探讨SynMicro在污染削减、病害防控、养分循环提升及多功能协同等方面的应用潜力;分析当前面临的关键挑战,并展望未来研究方向与技术路径;以期为耕地土壤微生态健康重塑提供理论与技术支撑,保障国家粮食安全和推动农业可持续发展。
1 耕地土壤有机复合污染:普遍性、复杂性与风险性 1.1 污染现状:全球视角与中国国情全球耕地土壤有机复合污染日益加剧,成为不容忽视的全球性挑战。全球约64%的耕地土壤受到多种农药的复合污染威胁,高风险区域占比达31%[6]。即使在有机管理农业区域,土壤中也普遍存在多种农药残留,凸显了这些污染物的持久性和广泛迁移特性[2]。欧洲监测数据显示,75%的农田土壤残留草甘膦及其代谢物氨甲基膦酸,43%的样点同时检出多种新烟碱类杀虫剂[16]。美国主要粮食带土壤中,除传统农药外,抗生素、酞酸酯等新污染物的检出率也在上升[16]。发展中国家,由于农业生产模式相对粗放,农药和化肥使用强度更高,面临更严峻的耕地土壤复合污染形势[6,17]。
中国作为农业大国和快速工业化国家,耕地土壤有机复合污染问题尤为突出复杂。Tang等[6]的研究表明,中国是全球农药污染高风险区域面积最大的国家之一,高达约290万平方千米,且遭受农药复合污染的种类最多,有8.4%的农业用地遭受着多于20种农药成分的污染风险,且污染谱系正从传统农药向多元化转变。除乙草胺、莠去津等传统农药的广泛残留外,农膜、畜禽养殖、工业活动等带来的酞酸酯、抗生素、微塑料等新污染物检出率不断攀升[9,18-19]。更值得警惕的是,被称为“永久化学品”的全氟和多氟烷基物质(Per- and Polyfluoroalkyl Substances,PFASs)也开始在耕地土壤中被检测到,并呈现累积趋势[20-21]。这些污染物共同构成了复杂的复合污染格局,对土壤生态系统造成潜在威胁。
1.2 污染复杂性:交互作用与累积效应耕地土壤中复合有机污染物之间复杂的交互作用及其在土壤环境中的长期累积效应,共同塑造了难以预测的生态环境风险(图 1)。首先,污染物间的交互作用导致非加和效应。多种有机污染物共存于土壤微环境时,其整体环境行为和生物毒性往往并非各组分独立效应的简单叠加,而是可能表现出协同增效、拮抗减效或加和等复杂关系[4-6]。这种交互作用可通过多种机制发生,包括通过改变环境行为(如微塑料吸附运输农药[18])、干扰降解(竞争酶系或共代谢[22-23],或抑制降解菌[24])及放大生物效应(协同毒性[25],加剧抗生素抗性基因(Antibiotic Resistance Genes,ARGs)传播[9])等。这些复杂的交互作用极大地增加了准确评估复合污染生态风险的难度,亟需深入研究其分子机制和环境行为。此外,由于许多有机污染物(如有机氯农药)具有持久性,长期低剂量农业投入(如农药施用)或持续的外部输入(如大气沉降),也可导致其在土壤中持续富集[26]。
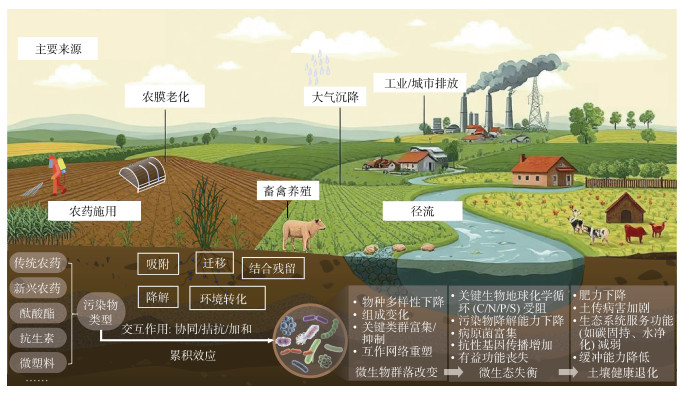
|
图 1 耕地土壤有机复合污染的普遍性、复杂性与风险性概念图 Fig. 1 A summary of the universality, complexity and risk of organic compound pollution in cultivated soil |
耕地土壤有机复合污染的深层风险,在于其长期化学压力所导致的微生态系统持久性失衡。这种失衡体现在多个层面:1)群落结构改变:如耐受菌富集,敏感菌抑制,功能冗余度下降[27];2)适应性演化:如筛选耐污耐药菌,增加ARGs丰度及扩散风险[28];3)理化性质改变:如影响土壤团聚体稳定性、有机质组成等[29];4)关键功能受损:如固碳和养分循环能力弱化[14]。因此,揭示复合污染长期累积的动态过程及其对土壤微生态的深层影响,是制定精准治理策略的关键前提。这些微观层面的紊乱将引发连锁效应,其影响从局部土壤扩展至区域生态乃至人类健康。例如,土壤固碳功能弱化会影响全球碳循环[7],而ARGs的扩散则可能通过食物链带来公共健康风险[28]。最终,这些效应共同导致土壤健康状态恶化,威胁农业生产的可持续性和粮食安全,尤其是在发展中国家[30]。因此,评估复合污染风险和制定修复策略,必须将重心置于其对微生态功能层面的冲击上(图 1)。
为全面揭示这一风险并实现精准评估,传统基于化学浓度和单一污染物毒性风险商的方法已显不足,亟需将污染物暴露水平与土壤微生态响应及健康状态深度耦合,构建综合指标体系[6,30]。为此,可以完善“微生态健康指数(Microbial Ecology Health Index,MEHI)”框架,涵盖结构多样性、功能基因丰度及实际代谢活性等多维度指标,通过加权整合(如主成分分析确定权重)生成量化分数,便于动态监测与早期预警[31]。同时,借助机器学习等数据科学工具,整合化学污染、土壤理化性质及MEHI等多源数据,利用随机森林、神经网络等模型,预测污染对微生态功能的非线性影响,精准识别高风险污染因子组合。进一步构建多参数风险评估矩阵,融合污染负荷指数、功能损伤指数及多样性衰减指数,结合主成分和聚类分析方法划分风险等级,实现风险空间可视化与优先管控因子的科学筛选。这一框架有望为精准识别污染驱动因子及制定针对性治理策略提供创新工具,助力土壤微生态健康风险的系统化评估与管理。
2 合成微生物组学:耕地土壤微生态健康重塑的前沿策略相较单一菌株,重组功能明确、结构简化的SynMicro可以更好地模拟自然群落互作,定向调控土壤多功能协同发展,实现污染削减、病害防控和土壤肥力提升等多重目标,为有机复合污染土壤微生态健康重塑提供可操作路径。
2.1 合成微生物组构建策略:“自上而下”与“自下而上”“自上而下”策略从复杂的自然微生物群落(如污染土壤中的土著菌群或高效功能富集培养物)出发,通过逐步简化和筛选,识别并保留维持核心功能的最小化有效微生物组合。常用方法包括连续稀释培养、功能驱动的富集筛选和基于互作网络的关键节点移除等[15]。例如,可以从长期受特定农药污染的土壤中,通过富集培养和高通量测序分析,识别出协同降解该农药及其代谢产物的关键微生物成员,进而构建一个结构精简但降解功能得以保留的SynMicro[32]。这种策略有助于挖掘自然界中已协同演化的功能模块。
“自下而上”策略则基于对单个微生物功能和互作机制的理解,从功能明确的分离菌株或基因工程菌出发,如同搭积木般理性地组装形成具有特定复合功能的微生物群落。这需要精确选择具有互补功能(如降解污染物不同环节、拮抗病原菌、促进植物生长等)的成员,并考虑它们之间的相互作用(协同或拮抗)[8]。例如,通过共培养实验和基因组尺度代谢模型预测,可以选择能够进行代谢“接力”的菌株组合,高效矿化单一菌株难以完全降解的复杂有机污染物[33]。这种策略提供了更高的设计自由度和功能可控性。
2.2 合成微生物组在耕地土壤健康重塑中的应用SynMicro在有机复合污染土壤中的应用已从单一功能(如污染降解)扩展到多目标协同(如病害防控与养分提升),通过微生物互作增强土壤恢复力。其优势在于功能冗余和协同效应,可在复合胁迫下维持稳定性。应用领域包括污染削减、病害防控、养分利用和多功能整合。
在污染削减方面,SynMicro通过成员间的代谢协作与功能互补,能够克服单一微生物在降解复杂污染物中面临的代谢瓶颈,拓展可降解污染物谱并提高降解效率。如,Ruan等[13]通过整合“自上而下”和“自下而上”方法,利用SuperCC代谢模型成功构建了降解溴苯腈辛酸酯的高效SynMicro,其降解效率显著高于单一菌株或自然微生物群落。此外,研究还发现某些辅助菌株(如Bacillus sp. P56,Lysinibacillus sp. LM5,Acinetobacter sp. AC6和Bradyrhizobium sp. BR1)和氮源的添加可以进一步提高SynMicro的降解性能,突出了通过代谢协作和功能互补优化SynMicro以增强污染物降解的可行性[13]。Xu等[34]通过微生物基因组尺度代谢模型指导构建的包含降解菌(Arthrobacter)及其互作菌(Halobacillus,Halomonas)的SynMicro,对阿特拉津的降解效率高于单一降解菌,且生物刺激策略(如添加碳源)能进一步提升效果。这些研究表明,SynMicro通过理性设计和环境优化,可高效应对复杂污染挑战,为土壤污染治理提供创新路径。
SynMicro在土传病害防控中也展现出显著应用潜力,能够通过多种机制有效抑制病原菌并提升作物抗病能力。研究表明,SynMicro能通过竞争排斥(如优势定殖和生物膜形成)占据根际生态位,限制病原菌生存空间;发挥抗生作用,产生抑制性化合物或分泌降解病原菌细胞壁的酶;激活植物自身防御系统,调控茉莉酸和水杨酸等信号通路,提升植物抗病水平[35-37]。这些机制还能通过SynMicro内部成员(包括高低丰度、不同物种乃至跨界组合)的功能互补和协同互作得以实现和放大,最终达到优于单一有益微生物的病害防控效果。未来,优化SynMicro构建策略、结合多组学技术解析交互机制,并开展田间试验验证其稳定性,将为现代农业提供绿色可持续的生物防控解决方案。
在养分高效利用方面,SynMicro能够通过促进土壤养分循环和增强植物养分吸收,显著提高养分利用效率。Compant等[38]指出,SynMicro可通过共生/非共生固氮、磷钾溶解及铁载体合成,直接增强植物营养获取。Jiang等[39]基于本土土壤核心微生物构建的SynMicro,在低肥力土壤中使玉米根冠比提高78%~121%,通过增强根际IAA分泌和氮磷代谢潜力优化养分利用。Zhou等[36]设计的烟草根际SynMicro在非灭菌土壤中使地上生物量增加76%~91%,并通过复合载体进一步提升净生物量至129%,凸显了从菌株筛选、群落构建到制剂优化的系统性策略对发挥SynMicro营养效率潜力的重要性。综合而言,通过精心设计和应用(特别是基于本土核心微生物的)SynMicro能够有效整合多种促营养机制,并确保其在复杂土壤环境中的定殖与功能发挥,从而显著提升作物对土壤养分的利用效率。
耕地土壤健康问题往往涉及污染、病害和养分失衡等多重因素,单一功能SynMicro难以全面应对。因此,构建多功能协同的SynMicro成为未来方向,通过整合污染降解、病害拮抗及养分循环等功能,有望实现污染土壤健康重建过程的多功能协同提升。
3 基于合成微生物组重塑土壤健康:前沿技术赋能将SynMicro从概念验证推向有效解决耕地有机复合污染及微生态失衡的实用技术,亟需摆脱传统试错法,转向基于机制理解的理性设计与精准优化。这高度依赖于一系列前沿技术的整合应用,这些技术从微生物资源挖掘、群落功能解析到理性设计与优化,贯穿SynMicro构建与应用的全链条,为应对有机复合污染胁迫下的土壤健康挑战提供了强有力的工具(图 2)。
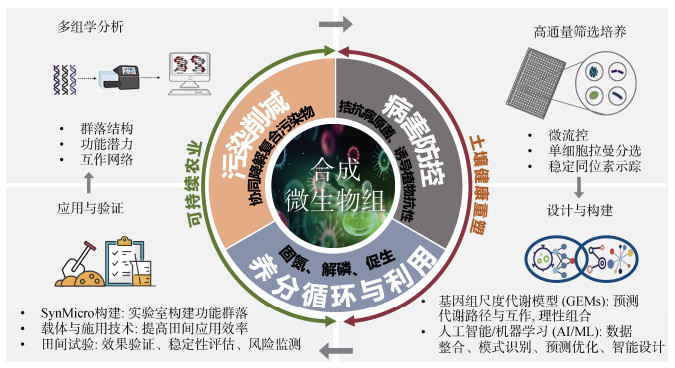
|
图 2 前沿技术赋能的合成微生物组设计与土壤健康重塑工作流程图 Fig. 2 Workflow diagram of synthetic microbiome design and soil health remodeling driven by cutting-edge technologies |
高通量组学技术是揭示有机复合污染对土壤微生态影响及其响应机制的核心工具。宏基因组学全面解析污染环境下微生物群落的组成结构、遗传多样性以及污染物降解、抗性传递、养分转化等潜在功能基因丰度,为识别受损功能和潜在修复菌群提供全局视角[27,40]。然而,基因潜力不等于实际功能,宏转录组学和宏蛋白质组学则可进一步揭示微生物在污染胁迫下的活性代谢通路和功能执行者[41]。代谢组学聚焦于土壤微环境中的代谢产物,能够捕捉微生物之间的代谢互馈及其与污染转化的直接关联[42]。将多组学数据整合分析,构建微生物互作网络模型,能够揭示有机污染胁迫如何重塑群落连接模式,并识别维持关键功能的微生物,为理性设计SynMicro提供数据支撑[11]。
3.2 高通量筛选与培养技术:加速功能微生物资源挖掘高通量筛选与培养技术的进步显著加速了功能微生物资源的挖掘。“下一代生理学方法”的兴起,特别是结合微流控、单细胞拉曼分选、稳定同位素示踪等技术,为快速获取功能微生物资源提供了新途径[43]。例如,Juang等[44]设计的微流控芯片可同时容纳多种微生物,并通过微井阵列测试不同微生物或化学物质间的交互作用。单细胞组学技术(单细胞基因组、转录组、拉曼光谱等)能够捕获复杂环境下未培养微生物的基因组与功能特征,揭示群落中“稀有但关键”的功能成员[45]。如,Liu等[46-47]联用传统分离和前沿的单细胞拉曼分选方法,开发了基于重水标记及钴酰胺特征峰的卤代有机物降解菌分选新方法,在此基础上成功分选获得具有降解效果的脱卤功能菌株。高通量培养组学结合自动化和机器学习,也为快速分离和鉴定植物根际等高度异质性土壤中的微生物提供了新策略[48]。
3.3 基因组尺度代谢模型:理性设计与精准调控基因组尺度代谢模型(Genome-scale Metabolic Models,GEMs)通过整合基因组信息和代谢网络数据,模拟微生物代谢过程和种间互作,为SynMicro的理性设计提供了理论框架。GEMs能够预测微生物群落中代谢“接力”路径和资源分配模式,优化成员组合以实现功能最大化。例如,可设计构建能够耐受高浓度污染物、具有多种脱卤途径、以及能够与其他环境微生物协同作用的合成菌群[8,33,49-50]。基于GEMs的设计思路,已成功构建出展现高稳定性、低竞争性和显著植物促生功能的SynMicro[51]。GEMs还可用于评估SynMicro在不同环境条件下的功能稳定性,如Schäfer等[14]通过模拟发现,添加特定辅助菌株可弥补代谢瓶颈,提升群落抗逆性。当前,GEMs的挑战在于模型精度受限于数据库完备性和环境变量复杂性,未来需整合代谢过程数据、多组学数据和代谢动态及通量分析,进一步提升预测能力[52]。
3.4 人工智能与机器学习:智能化设计与优化面对微生物组数据的爆炸式增长和系统复杂性,人工智能(Artificial Intelligence,AI)与机器学习(Machine Learning,ML)提供了更强大的数据整合、模式识别和预测优化能力,正成为SynMicro设计与优化的前沿驱动力。微生物组数据固有的成分性、稀疏性和高维度等挑战,致使传统方法难以捕捉其复杂关系,而AI/ML方法能够有效处理这些多维度数据,揭示隐藏的模式和关联[53-54]。例如,在土壤健康领域,ML已被成功应用于预测PFAS在土壤中的吸附行为,通过整合污染物和土壤的多种特性(如分子量、疏水性、pH、有机碳含量等),识别出影响吸附的关键因素并实现高精度预测[53]。更直接地,针对SynMicro在特定宿主性状上的应用(如植物对病原体的防护,与土壤微生物组改善植物健康的机制类似),研究者通过随机组装SynMicro并利用ML算法(如随机森林、弹性网络)分析其组成或丰度与宿主性状的关系,不仅能有效预测SynMicro群落功能,还能识别出驱动该性状的关键菌株甚至菌株组合的协同效应,为理性设计和构建高效SynMicro提供了数据驱动的方法[55]。此外,ML方法还能助力挖掘菌株组合的潜力,预测不同组合下的微生物群落功能。例如,借鉴代谢工程中利用高斯过程回归优化多基因拷贝数以提升产物产量和性能的策略,ML能显著加速高表现力菌株组合的构建过程[56]。这些研究共同表明,AI/ML能够整合复杂的多模态微生物组及环境数据,识别关键生物或非生物特征对特定功能的贡献,预测微生物组合表现,并指导优化设计,为重塑土壤健康的SynMicro工程设计提供强大智能工具。
4 挑战与展望尽管合成微生物组学可为有机复合污染下耕地土壤微生态健康重塑提供前所未有的机遇,但将其从概念推向普适有效的田间应用仍面临多重核心挑战(图 3)。首先,有机复合污染所致的高度时空异质性土壤微环境为精准解析菌群互作机制及环境影响带来巨大挑战[11,57]。其次,SynMicro构建应用鲁棒性与可预测性待提升,需高效筛选组装具稳定定殖的持久功能菌株,有效克服外源菌与本土菌的竞争[15]。再者,生态安全性与风险评估体系待完善,需评估外源菌潜在环境风险(基因转移、非靶标影响、抗性诱导)[25,28],建立健全风险评估框架和监管政策。最后,实验室到田间转化有距离,涉及菌剂生产、施用技术、长期监测等难题[12]。
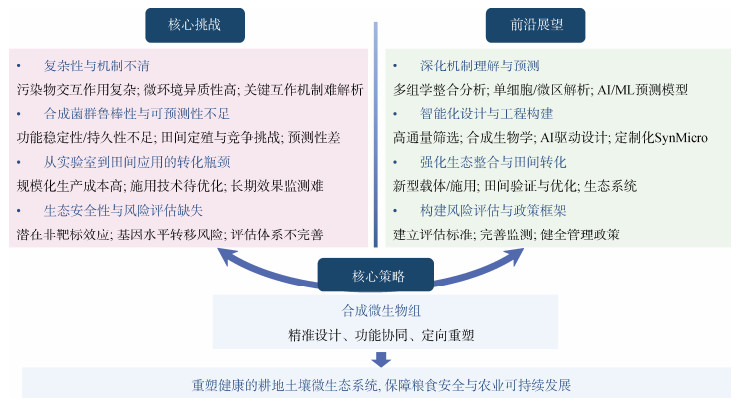
|
图 3 有机复合污染下耕地土壤微生态健康重塑:挑战驱动的前沿展望 Fig. 3 Reshaping arable soil microecological health under organic complex pollution: challenge-driven frontiers |
面向未来,耕地土壤微生态健康的重塑将高度依赖于多学科的深度交叉融合与前沿技术赋能下的集成创新(图 3),包括:
1)深化机制理解与预测建模:整合多组学与稳定同位素示踪技术,深入微观解析有机复合污染胁迫下菌群的代谢活动、种间互作及对环境变化的动态响应机制。在此基础上,进一步发展基于基因组尺度代谢模型(GEMs)和人工智能/机器学习(AI/ML)的预测模型,实现对污染环境下土壤微生态功能动态及其SynMicro干预效果的理性预测。
2)智能化设计与工程构建:利用高通量筛选技术、合成生物学工具以及AI驱动的设计平台,精准挖掘、组装功能性强、环境适应性好、结构精简的定制化SynMicro。重点攻克如何高效构建能够在复杂有机复合污染土壤环境中稳定定殖和发挥持久功能的合成菌群。
3)强化生态集成与田间转化:开发新型微生物载体与施用技术,提高合成菌群在复杂土壤环境中的定殖效率与功能稳定性;加强受控环境下的小规模和多点田间试验,验证合成菌群在多种污染类型、土壤类型和农业实践下的实际效能、持久性及对作物生产、土壤健康指标的综合影响;同时,关注SynMicro与土壤理化性质、植物根系互作的微观调控,提升其生态集成度。
4)构建风险评估与政策框架:建立一套科学、完善的合成微生物组环境风险评估标准体系,包括监测外源菌扩散、潜在基因转移、对非靶标生物的影响以及抗性诱导等风险。基于评估结果,推动建立健全相关的监管政策和法规框架,确保合成微生物组技术的研发、应用和推广安全、可持续。
综上,耕地土壤有机复合污染下微生态健康的重塑,需要跨越基础机制、技术瓶颈、生态整合和风险管理等多重障碍,未来发展路径在于依靠前沿技术驱动的理性设计、系统性的田间验证以及负责任的创新监管,从而充分释放合成微生物组在保障国家粮食安全和推动农业绿色转型中的巨大潜力。
5 结论耕地土壤有机复合污染存在普遍性、复杂性,其引发的微生物群落失衡、功能紊乱及土壤健康退化等深层影响对土壤微生态健康构成潜在风险,已成为全球性挑战,严重威胁土壤微生态健康和农业可持续发展。以合成微生物组学(SynMicro)为代表的现代分子生物学技术,为精准干预和重塑受损土壤微生态系统提供了前沿策略。面向上述挑战,通过理性设计SynMicro,构建功能明确、结构简化的微生物群落,可以实现污染削减、病害防控、养分利用及多功能的协同。后续应进一步整合多组学技术、高通量筛选、基因组尺度代谢模型、人工智能等前沿工具,推动SynMicro从机制理解走向智能化设计与工程构建,实现其在复杂土壤环境中的高效应用和田间转化;同时,还应进一步完善生态安全性评估和监管政策,确保该技术能可持续发展。未来,通过多学科交叉与技术集成创新,有望有效重塑耕地土壤微生态健康,为保障国家粮食安全和实现农业绿色可持续发展提供坚实支撑。
| [1] |
Banerjee S, van der Heijden M G A. Soil microbiomes and one health[J]. Nature Reviews Microbiology, 2023, 21(1): 6-20. DOI:10.1038/s41579-022-00779-w
(  0) 0) |
| [2] |
Riedo J, Wettstein F E, Rösch A, et al. Widespread occurrence of pesticides in organically managed agricultural soils-the ghost of a conventional agricultural past[J]. Environmental Science & Technology, 2021, 55(5): 2919-2928.
(  0) 0) |
| [3] |
Wu K N, Yang Q J, Zhao R. A discussion on soil health assessment of arable land in China (In Chinese)[J]. Acta Pedologica Sinica, 2021, 58(3): 537-544. DOI:10.11766/trxb202009280546 [吴克宁, 杨淇钧, 赵瑞. 耕地土壤健康及其评价探讨[J]. 土壤学报, 2021, 58(3): 537-544.]
(  0) 0) |
| [4] |
Navarro J, Hadjikakou M, Ridoutt B, et al. Pesticide toxicity hazard of agriculture: Regional and commodity hotspots in Australia[J]. Environmental Science & Technology, 2021, 55(2): 1290-1300.
(  0) 0) |
| [5] |
Stehle S, Schulz R. Agricultural insecticides threaten surface waters at the global scale[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(18): 5750-5755.
(  0) 0) |
| [6] |
Tang F H M, Lenzen M, McBratney A, et al. Risk of pesticide pollution at the global scale[J]. Nature Geoscience, 2021, 14(4): 206-210. DOI:10.1038/s41561-021-00712-5
(  0) 0) |
| [7] |
Wang F, Xiang L L, Sze-Yin Leung K, et al. Emerging contaminants: A one health perspective[J]. Innovation, 2024, 5(4): 100612.
(  0) 0) |
| [8] |
He Y, Li S Y, Yuan J, et al. A systematic understanding of microbial reductive dechlorination towards an improved "one health" soil bioremediation: A review and perspective[J]. Science China Technological Sciences, 2024, 67(10): 3009-3031. DOI:10.1007/s11431-024-2664-5
(  0) 0) |
| [9] |
Zheng X X, Jahn M T, Sun M M, et al. Organochlorine contamination enriches virus-encoded metabolism and pesticide degradation associated auxiliary genes in soil microbiomes[J]. The ISME Journal, 2022, 16(5): 1397-1408. DOI:10.1038/s41396-022-01188-w
(  0) 0) |
| [10] |
Yang X L, Yuan J, Li N N, et al. Loss of microbial diversity does not decrease γ-HCH degradation but increases methanogenesis in flooded paddy soil[J]. Soil Biology & Biochemistry, 2021, 156: 108210.
(  0) 0) |
| [11] |
Jing J Y, Garbeva P, Raaijmakers J M, et al. Strategies for tailoring functional microbial synthetic communities[J]. The ISME Journal, 2024, 18(1): wrae049. DOI:10.1093/ismejo/wrae049
(  0) 0) |
| [12] |
Lawson C E, Harcombe W R, Hatzenpichler R, et al. Common principles and best practices for engineering microbiomes[J]. Nature Reviews Microbiology, 2019, 17(12): 725-741. DOI:10.1038/s41579-019-0255-9
(  0) 0) |
| [13] |
Ruan Z P, Chen K, Cao W M, et al. Engineering natural microbiomes toward enhanced bioremediation by microbiome modeling[J]. Nature Communications, 2024, 15: 4694. DOI:10.1038/s41467-024-49098-z
(  0) 0) |
| [14] |
Schäfer M, Pacheco A R, Künzler R, et al. Metabolic interaction models recapitulate leaf microbiota ecology[J]. Science, 2023, 381(6653): 42-51.
(  0) 0) |
| [15] |
Jansson J K, McClure R, Egbert R G. Soil microbiome engineering for sustainability in a changing environment[J]. Nature Biotechnology, 2023, 41(12): 1716-1728. DOI:10.1038/s41587-023-01932-3
(  0) 0) |
| [16] |
Silva V, Mol H G J, Zomer P, et al. Pesticide residues in European agricultural soils–A hidden reality unfolded[J]. Science of the Total Environment, 2019, 653: 1532-1545. DOI:10.1016/j.scitotenv.2018.10.441
(  0) 0) |
| [17] |
Fernandes C L F, Volcão L M, Ramires P F, et al. Distribution of pesticides in agricultural and urban soils of Brazil: A critical review[J]. Environmental Science: Processes & Impacts, 2020, 22(2): 256-270.
(  0) 0) |
| [18] |
Su X, Liu M, Yuan J, et al. Potential risk of co-occurrence of microplastics and chlorinated persistent organic pollutants to coastal wetlands: Evidence from a case study[J]. Environmental Pollution, 2023, 320: 121087. DOI:10.1016/j.envpol.2023.121087
(  0) 0) |
| [19] |
He Y, Su X, Li S Y, et al. Pollution risk and regulation of organochlorine in soil: From the perspective of multi-process coupling (In Chinese)[J]. Acta Pedologica Sinica, 2024, 61(5): 1179-1187. DOI:10.11766/trxb202312010504 [何艳, 苏心, 李淑瑶, 等. 土壤有机氯污染风险与调控: 基于多过程耦合的视角[J]. 土壤学报, 2024, 61(5): 1179-1187.]
(  0) 0) |
| [20] |
Cheng Y, An Q, Qi H S, et al. Temporal trends of legacy and emerging PFASs from 2011 to 2021 in agricultural soils of Eastern China: Impacts of the Stockholm convention[J]. Environmental Science & Technology, 2023, 57(25): 9277-9286.
(  0) 0) |
| [21] |
Peter L G, Lee L S. Sources and pathways of PFAS occurrence in water sources: Relative contribution of land-applied biosolids in an agricultural dominated watershed[J]. Environmental Science & Technology, 2025, 59(2): 1344-1353.
(  0) 0) |
| [22] |
Weisner O, Frische T, Liebmann L, et al. Risk from pesticide mixtures–The gap between risk assessment and reality[J]. Science of the Total Environment, 2021, 796: 149017. DOI:10.1016/j.scitotenv.2021.149017
(  0) 0) |
| [23] |
Yang K W, Zhang L, Ruiz-Valencia A, et al. Heterogeneity in the composition and catabolism of indigenous microbiomes in subsurface soils cocontaminated with BTEX and chlorinated aliphatic hydrocarbons[J]. Environmental Science & Technology, 2025, 59(9): 4540-4550.
(  0) 0) |
| [24] |
Rumschlag S L, Mahon M B, Hoverman J T, et al. Consistent effects of pesticides on community structure and ecosystem function in freshwater systems[J]. Nature Communications, 2020, 11: 6333. DOI:10.1038/s41467-020-20192-2
(  0) 0) |
| [25] |
Wan N F, Fu L W, Dainese M, et al. Pesticides have negative effects on non-target organisms[J]. Nature Communications, 2025, 16: 1360. DOI:10.1038/s41467-025-56732-x
(  0) 0) |
| [26] |
Maggi F, Tang F H M, Tubiello F N. Agricultural pesticide land budget and river discharge to oceans[J]. Nature, 2023, 620(7976): 1013-1017. DOI:10.1038/s41586-023-06296-x
(  0) 0) |
| [27] |
Ni B, Xiao L, Lin D, et al. Increasing pesticide diversity impairs soil microbial functions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2025, 122(2): 24-35.
(  0) 0) |
| [28] |
Liu Z L, Wang Y F, Zhu D, et al. Increased transmission of antibiotic resistance occurs in a soil food chain under pesticide stress[J]. Environmental Science & Technology, 2024, 58(50): 21989-22001.
(  0) 0) |
| [29] |
Xu Y W, Zeng L Z, Tao Y F, et al. Release of additives from agricultural plastic films in water: Experiment and modeling[J]. Environmental Science & Technology, 2023, 57(27): 10053-10061.
(  0) 0) |
| [30] |
Xu J M, Ren C C, Zhang X M, et al. Soil health contributes to variations in crop production and nitrogen use efficiency[J]. Nature Food, 2025, 6(6): 597-609. DOI:10.1038/s43016-025-01155-6
(  0) 0) |
| [31] |
Xu N H, Chen B F, Wang Y, et al. Integrating anthropogenic-pesticide interactions into a soil health-microbial index for sustainable agriculture at global scale[J]. Global Change Biology, 2024, 30(11): 66-78.
(  0) 0) |
| [32] |
Wang S Q, Chen C, Zhao S Y, et al. Microbial synergistic interactions for reductive dechlorination of polychlorinated biphenyls[J]. Science of the Total Environment, 2019, 666: 368-376. DOI:10.1016/j.scitotenv.2019.02.283
(  0) 0) |
| [33] |
Puentes Jácome L A, Lomheim L, Gaspard S, et al. Biodegradation of lindane(γ-hexachlorocyclohexane)to nontoxic end products by sequential treatment with three mixed anaerobic microbial cultures[J]. Environmental Science & Technology, 2021, 55(5): 2968-2979.
(  0) 0) |
| [34] |
Xu X H, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5
(  0) 0) |
| [35] |
Qiao Y Z, Wang Z D, Sun H, et al. Synthetic community derived from grafted watermelon rhizosphere provides protection for ungrafted watermelon against Fusarium oxysporum via microbial synergistic effects[J]. Microbiome, 2024, 12(1): 101-113. DOI:10.1186/s40168-024-01814-z
(  0) 0) |
| [36] |
Zhou X, Wang J T, Liu F, et al. Cross-kingdom synthetic microbiota supports tomato suppression of Fusarium wilt disease[J]. Nature Communications, 2022, 13: 7890. DOI:10.1038/s41467-022-35452-6
(  0) 0) |
| [37] |
Li Z F, Bai X L, Jiao S, et al. A simplified synthetic community rescues Astragalus mongholicus from root rot disease by activating plant-induced systemic resistance[J]. Microbiome, 2021, 9(1): 217-229. DOI:10.1186/s40168-021-01169-9
(  0) 0) |
| [38] |
Compant S, Cassan F, Kostić T, et al. Harnessing the plant microbiome for sustainable crop production[J]. Nature Reviews Microbiology, 2025, 23(1): 9-23. DOI:10.1038/s41579-024-01079-1
(  0) 0) |
| [39] |
Jiang M T, Delgado-Baquerizo M, Yuan M M, et al. Home-based microbial solution to boost crop growth in low-fertility soil[J]. New Phytologist, 2023, 239(2): 752-765. DOI:10.1111/nph.18943
(  0) 0) |
| [40] |
Huang Y Q, Wen L Y, Zhang L G, et al. Community-integrated multi-omics facilitates screening and isolation of the organohalide dehalogenation microorganism[J]. Innovation, 2023, 4(1): 100355.
(  0) 0) |
| [41] |
Sharma B, Shukla P. Designing synthetic microbial communities for effectual bioremediation: A review[J]. Biocatalysis and Biotransformation, 2020, 38(6): 405-414. DOI:10.1080/10242422.2020.1813727
(  0) 0) |
| [42] |
Wang L J, Wang X Y, Wu H, et al. Integration of metagenomic analysis and metabolic modeling reveals microbial interactions in activated sludge systems in response to nanoplastics and plasticizers[J]. Water Research, 2025, 271: 122863. DOI:10.1016/j.watres.2024.122863
(  0) 0) |
| [43] |
Hatzenpichler R, Krukenberg V, Spietz R L, et al. Next-generation physiology approaches to study microbiome function at single cell level[J]. Nature Reviews Microbiology, 2020, 18(4): 241-256. DOI:10.1038/s41579-020-0323-1
(  0) 0) |
| [44] |
Juang D S, Wightman W E, Lozano G L, et al. Microbial community interactions on a chip[J]. Proceedings of the National Academy of Sciences of the United States of America, 2024, 121(39): e2403510121.
(  0) 0) |
| [45] |
Xu W J, Zhu W, Xia Y K, et al. Raman spectroscopy for cell analysis: Retrospect and prospect[J]. Talanta, 2025, 285: 127283. DOI:10.1016/j.talanta.2024.127283
(  0) 0) |
| [46] |
Liu M, Su X, Yuan J, et al. Unravelling the processes involved in biodegradation of chlorinated organic pollutant: From microbial community to isolated organohalide degraders[J]. Water Research, 2025, 268: 122730. DOI:10.1016/j.watres.2024.122730
(  0) 0) |
| [47] |
Liu M. Isolation of chlorinated organic matter-degrading bacteria from wetland sediments and enhancement of their degradation function[D]. Hangzhou: Zhejiang University, 2025.[刘孟. 湿地沉积物中氯代有机物降解菌挖掘及其降解功能强化机制[D]. 杭州: 浙江大学, 2025.]
(  0) 0) |
| [48] |
Huang Y M, Sheth R U, Zhao S J, et al. High-throughput microbial culturomics using automation and machine learning[J]. Nature Biotechnology, 2023, 41(10): 1424-1433. DOI:10.1038/s41587-023-01674-2
(  0) 0) |
| [49] |
Arora P K, Bae H H. Integration of bioinformatics to biodegradation[J]. Biological Procedures Online, 2014, 16: 88.
(  0) 0) |
| [50] |
Jaiswal S, Singh D K, Shukla P. Gene editing and systems biology tools for pesticide bioremediation: A review[J]. Frontiers in Microbiology, 2019, 10: 87-98. DOI:10.3389/fmicb.2019.00087
(  0) 0) |
| [51] |
Gonçalves O S, Creevey C J, Santana M F. Designing a synthetic microbial community through genome metabolic modeling to enhance plant-microbe interaction[J]. Environmental Microbiome, 2023, 18(1): 81-94. DOI:10.1186/s40793-023-00536-3
(  0) 0) |
| [52] |
Wang L J, Wang X Y, Wu H, et al. Metabolic modeling of synthetic microbial communities for bioremediation[J]. Critical Reviews in Environmental Science and Technology, 2023, 53(24): 2092-2111. DOI:10.1080/10643389.2023.2212569
(  0) 0) |
| [53] |
Fabregat-Palau J, Ershadi A, Finkel M, et al. Modeling PFAS sorption in soils using machine learning[J]. Environmental Science & Technology, 2025, 59(15): 7678-7687.
(  0) 0) |
| [54] |
Hernández Medina R, Kutuzova S, Nielsen K N, et al. Machine learning and deep learning applications in microbiome research[J]. ISME Communications, 2022, 2: 98-107. DOI:10.1038/s43705-022-00182-9
(  0) 0) |
| [55] |
Emmenegger B, Massoni J, Pestalozzi C M, et al. Identifying microbiota community patterns important for plant protection using synthetic communities and machine learning[J]. Nature Communications, 2023, 14: 7983. DOI:10.1038/s41467-023-43793-z
(  0) 0) |
| [56] |
Du F, Li Z J, Li X, et al. Optimizing multicopy chromosomal integration for stable high-performing strains[J]. Nature Chemical Biology, 2024, 20(12): 1670-1679. DOI:10.1038/s41589-024-01650-0
(  0) 0) |
| [57] |
Hartmann M, Six J. Soil structure and microbiome functions in agroecosystems[J]. Nature Reviews Earth & Environment, 2023, 4(1): 4-18.
(  0) 0) |
 2026, Vol. 63
2026, Vol. 63


