2. 南京农业大学生命科学学院, 南京 210095
2. College of Life Sciences, Nanjing Agricultural University, Nanjing 210095, China
石油烃、塑化剂、多环芳烃等有机污染物可通过多种途径进入土壤[1]。一些有机污染物自然条件下难降解,在土壤中长期存留,危害生态安全和人群健康[2]。我国污染农田土壤中低负荷有机污染场景最为多见,物理化学修复法往往成本较高、难以适用[3],而微生物由于其丰富的生物多样性和巨大的分解代谢潜力,在中低负荷有机污染土壤消减中更具优势[4]。1988年Whipps等提出了微生物组的定义[5],随着21世纪分子生物学和系统生物学的发展,研究中开始利用工程学方法对细胞生物进行改造控制,衍生出了合成生物学领域。整合不同微生物菌株,依据互作关系、生物竞争、协调稳定等原则,人工构建出可满足特定需求的微生物群落,这些群落被称为合成微生物组。美国在2016年公布了国家微生物组计划,中国科学院也在2017年启动了微生物组计划,合成微生物组的出现为生物技术的升级优化带来了新的可能性。
基于单一菌株的有机污染物消减效果经常受到各种因素的限制[6-9]。例如,与土著菌群成员的竞争或营养缺乏导致接种的外源降解菌株存活率低、单一菌株无法完全分解代谢复杂有机污染物、非目标污染物或其中间代谢产物对微生物代谢活动的抑制等[10]。与传统的单菌株方法相比,微生物组在有机污染物消减方面具有显著优势[11]。例如,微生物组表现出遗传和代谢多样性,能够通过协同代谢完全降解复杂有机污染物。
土壤环境中天然存在的微生物组会随环境条件变化而变化[12],其群落结构往往不是高效消减有机污染物的最佳结构。合成微生物组将合成生物学的原理从单细胞扩展到多细胞系统,使合成微生物组的从头设计成为可能。因此,设计优化微生物群落组成和结构,构建高效合成微生物组,有助于实现最佳的消减效果[13-14]。当前,合成微生物组相关技术已在农业、工业、发酵等领域开展应用。然而,目前合成微生物组在有机污染土壤生物修复方面的研究仍较为有限,得到的部分研究结果尚未充分总结和分析。
本文综述了现阶段微生物组在土壤有机污染物消减中的优势以及面临的问题,探讨了基于合成微生物组学的土壤中有机污染物消减作用及机制,并指出了该领域未来研究方向和所面临的挑战。
1 合成微生物组在土壤有机污染物消减中的优势目前,为解决微生物组应用于土壤有机污染物消减时遇到的问题,科学界在几类热点领域进行了大量研究(图 1)。本文借助Web of Science以“Soil”、“Bacterial degrading consortium”、“Synthetic microbiomes”、“Synthetic communities”为关键词进行检索,在2020—2024年时间段内共有1 167篇相关文献。发现近年该领域发文量呈现整体上升趋势(图 1a)。涉及领域中,主要包括环境科学、微生物学、农学和生态学等(图 1b)。对关键词和文章内容进行分析,得出基于合成微生物组消减土壤有机污染研究热点(图 1c)。微生物群落表征技术可帮助鉴定新的降解微生物;消减预测模型可以模拟物种相互作用并预测群落动态,有助于设计优化合成微生物组以实现有机污染物高效降解;跨学科方法利用植物-微生物相互作用与新型纳米材料,提高微生物活性、稳定性和污染物生物利用度;利用生物传感器、人工智能和物联网等技术可评估合成微生物组在长时间内生物降解能力变化,通过实时监测和管理策略,保持最高的消减效率和成本效益。研究热点表明土壤合成微生物组的研究主要聚焦于其功能和互作机制,值得注意的是,“Engineering”、“交叉喂养(cross-feeding)”是近几年研究的高频词,说明土壤合成微生物消减领域的研究愈发到关注。
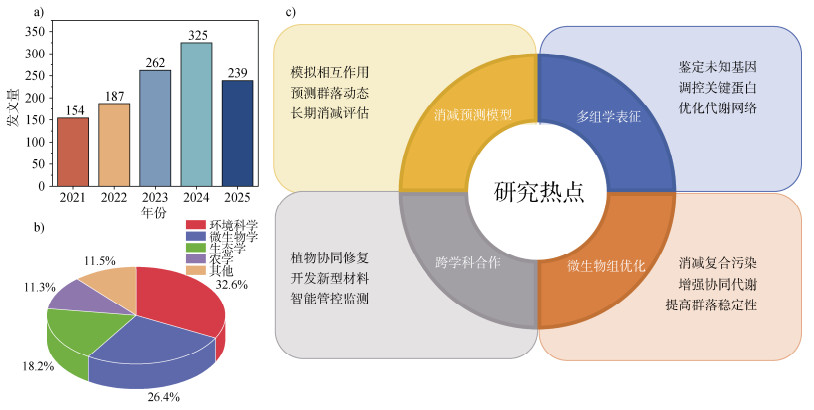
|
图 1 基于合成微生物组消减土壤中有机污染物的研究热点 Fig. 1 Research hotspots on elimination of organic pollutants in soil basing on synthetic microbiomes |
合成微生物组可通过组合多种功能菌株的代谢通路,实现复杂代谢功能重建,克服单一菌株在有机污染物代谢能力上的局限。在过去的几十年里,研究人员已经分离出许多能够降解有机污染物的纯培养微生物菌株,为污染环境修复提供了宝贵的微生物资源。常见的微生物修复策略包括生物强化(将降解菌株接种到污染环境)和生物刺激(向污染环境添加营养物质以促进菌株生长)[15]。基于单一菌株的生物修复大都存在效率受限的问题。与之相比,合成微生物组的协同代谢作用减少了中间代谢产物的积累,有效减少了反馈抑制作用,降低了反馈抑制作用对降解活性的阻碍[16]。此外,合成微生物组能够执行较单一菌株更多样化的功能。例如,通过组合针对不同有机污染物的菌株,可以设计合成微生物组以有效地矿化各种有机污染物的混合污染物[17];设计具有有机污染物消减能力菌株和重金属耐受菌株的微生物组可以有效用于有机物-重金属复合污染环境的修复[18]。
基于合成微生物组的有机消减过程具有更加稳定的消减效果。合成微生物组除了与其外部环境相互作用外,微生物组内的相互作用(直接或间接)也有利于促进稳定群落结构的建立。例如,合成微生物组内成员之间的交叉喂养可以创建紧密联系的菌群[19],相对于不相关的菌株组成的群落更具优势,从而增强接种微生物组的生态竞争力。此外,这种菌株间的交叉喂养关系还可以促进降解菌的生长,从而提高合成微生物组的整体降解效率[20]。
当应用于植物根际时,合成微生物组可促进微生物-植物共同消减有机污染物 [21]。根际作为围绕植物根系并受植物根系影响的狭窄土壤区域,应用合成微生物组可以执行多种功能,如有机污染物降解、重金属固定、植物病原体抑制、植物生长促进和抗逆性增强等,从而提高对土壤中有机污染物降解效率[22],同时减少植物对化学农药和肥料的依赖,促进植物在受有机污染土壤中的生长[23]。
2 合成微生物组消减土壤有机污染物中面临的问题原生微生物组不能直接应用于有机污染物消减是目前面临的首要问题。在有机污染物消减过程中,微生物组的群落结构不仅影响有机污染物消减的效率,还决定了最终的降解产物[24]。天然存在的微生物组往往不会针对有机污染物消减进行定向优化,因此原生微生物组通常不具备高效消减能力,其生物多样性也会随污染程度增加而降低。因此,需设计和优化合成微生物组的群落组成和结构,以制定有效的有机污染物消减策略,丰富土壤微生物种群丰度,在短期内获得较好消减效果的目的。实现这一目标的有效手段是利用合成微生物组技术将微生物组群落结构和微生物组内的代谢相互作用与生理功能关联。
剖析合成微生物组复杂的互作关系也是研究难点之一。即使在简单的合成微生物组(如双菌株成员组合)中,了解代谢相互作用并将其与生理表现相关联仍然具有较大挑战性[25]。合成生物学涉及对现有生物体进行工程改造或创造新的生命形式,被认为是能够应对人类重大挑战的革命性技术[26]。合成微生物组将合成生物学的原理从单细胞策略扩展到多细胞系统,使微生物组的从头设计成为可能[27]。这种方法旨在在多个菌株之间分配不同的代谢途径,为特定目的构建最佳组合。因此,合成微生物组是理性设计微生物组以增强消减效果的有力途径。
在实际应用中大多数研究仍需采用试错法来确定合成微生物组的最佳设计[28],然而,长期试错的时间和经济成本较高,因此,需要开发新的技术方法,减少对试错实验的过度依赖。此外,在设计和构建强化污染物消减的合成微生物组时,产生大量实验数据,其数据分析处理同样面临挑战。例如,相关数据库匮乏、实验室条件限制也是现阶段合成微生物组学研究中存在的难点[29]。
当前合成微生物组在土壤中存在生存压力大、稳定性差和功能发挥异常等问题。受有机污染的土壤微生物种群多样性较低,大部分对有机污染敏感的细菌被抑制或杀死,剩余的耐受菌成为土壤优势菌种,这些有机污染耐受菌可能会分泌有害物质对合成微生物组中的功能菌定植生长产生抑制作用。此外受有机污染的土壤往往营养物质匮乏,加入合成微生物组往往面临较大的竞争抑制,影响降解功能的稳定表达[30]。最后在污染土壤的实际应用中,往往难以达到实验室测试水平,在长期试验中,也要注意菌种的退化和变异风险。
3 设计合成微生物组学的方法策略由于微生物组高度复杂且对微生物组分子水平反应过程的了解有限,目前合成微生物组设计通常遵循自上而下的方法[31]。自上而下的方法使用特定的环境变量(例如某些底物含量、平均细胞培养时间和氧化还原条件等),驱动现有的微生物朝向适应给定环境条件并定向表达功能的方向演变。自上而下的方法可以用于预测环境中可能发生的生化反应过程[32]。通过施加有机污染压力对初始微生物菌群进行驯化,从而促进菌群向特定功能方向重组组装,可以获得具有特定消减功能的微生物菌群,进而从天然微生物菌群中识别关键消减菌株。在此基础上,进一步研究代谢通路,预测最佳菌株组合并进行实验验证,最终获取高效合成微生物组。
组学技术的快速发展使通过预测代谢通路及其相互作用来设计具有所需功能的微生物组成为可能,同时为自下而上设计微生物组提供了有效方法[33]。自下而上的方法重点在于设计微生物组代谢网络和相互作用,探索可行的生化转化过程和代谢反应。该设计过程通常包括获取微生物组中单个菌株的基因组、重构代谢网络、使用建模或网络分析工具模拟分析群落内部和种群之间代谢通量并进行优化。然而,目前实施自下而上的设计依然存在挑战。例如,重建的代谢网络往往不准确和/或不完整;许多基因、蛋白质和代谢物的功能未知;对驱动个体和群落水平表型的进化压力来源不明确;以及对基因、代谢和生态系统调控方案(例如,群体感应信号反应系统)的了解有限等[33]。自下而上的设计大多基于具有模式生物(例如大肠杆菌和酿酒酵母)设计简单群落[34]。然而,为了将简单群落扩展到具有数十到数百个不同物种的非模式生物的系统[35],需要深入了解微生物代谢网络和调控相互作用与功能表达的原理。自下而上的设计往往需要大量关于单个菌株和群落代谢相互作用的实验数据,可通过高通量分析,表型筛选和多组学分析等方法,以获取设计所需要的数据[36]。
在实际应用中,合成微生物组设计往往采用自上而下和自下而上相结合的方法[37],通过集成设计方法设计合成微生物组(图 2)。合成微生物组设计需遵循设计-构建-测试-学习(DBTL)循环原则[38]。首先通过富集或高通量分析筛选出消减作用强的微生物,采用人工选择或定向进化等方法通过一个或多个自上而下的DBTL循环获取具有有机污染物降解能力的初步微生物组;再利用组学方法(宏基因组学、宏转录组学、蛋白质组学和代谢组学)研究降解机制,并将初步结果纳入预测模型以预测菌株之间的相互作用;最后基于已获得结果,进行自下而上的设计-构建-测试-学习(DBTL)循环,添加辅助菌株,设计出一种新的自下而上合成微生物组,其往往具有更高的降解效率,可评估其在实际应用中的消减有效性[39]。在实际应用于土壤污染修复过程中,要保证应用成本较低,对土壤肥力和代谢活性负面影响较小。合成微生物组包含的功能微生物需具有良好的遗传稳定性,不易发生退化变异,在面对各种内外部干扰胁迫时,仍能保持其丰度和功能稳定。
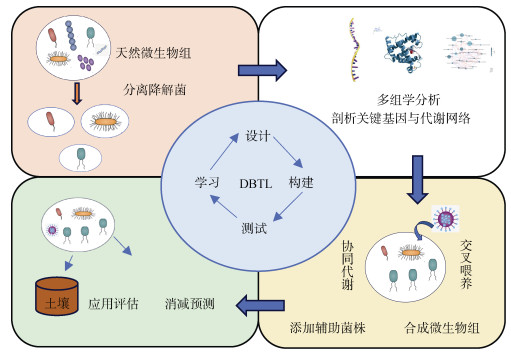
|
图 2 集成设计合成微生物组的方法 Fig. 2 Methods for synthesizing microbiomes by integrated design |
目前,合成微生物组设计的一个新兴目标是开发新的工具,使研究人员能够在一系列理想的条件下直接原位添加、删除或修改特定功能和表型[40]。原位宏基因组工程是一种有望实现这种灵活性的新兴技术[41]。不仅如此,将此技术与现有的CRISPR-Cas基因编辑技术相结合并进一步开发将可在原位精确操控微生物组的代谢网络,可以实现精准有效地设计合成微生物组[42]。此外,高质量基因组代谢模型(GSMM)技术被证实可以有效用于设计合成微生物组。该技术通过模拟不同菌株组合的代谢和生长性能,基于计算机模拟预测出最佳群落结构组合。例如,最新开发的代谢模型分析工具SuperCC,作为一种预测代谢相互作用的建模工具,其可为实验验证提供一系列可测试的预测。它可以预测共培养基中检测到的交换代谢物;还可以通过单培养中交换的代谢物改善菌株生长;可以采用SIP实验来验证交换代谢物的同化;最后利用转录组谱测试代谢反应所需基因的表达。建模工具与验证策略相结合可以极大地促进代谢建模的应用,模拟降解菌和辅助菌之间的相互作用,精准预测降解有机污染物的最佳菌株组合[43]。基因组规模代谢模型技术应用可预测合成微生物组在污染环境中的代谢通路和群落内部的互作机制。例如利用该技术,阐明了Pseudoxanthomonas sp. X-1和 Comamonas sp. 7D-2组成的合成微生物组高效降解农药污染物的机制是基于营养物质交换的交叉喂养。这些模拟工具还可以预测出可加速有机污染物降解的外源营养添加剂,为基于合成微生物组的生物刺激修复提供新策略。
4 基于合成微生物组的土壤中有机污染物消减作用及机制 4.1 基于合成微生物组的土壤中有机污染物消减作用设计构建合成微生物组可以对土壤中的有机污染物产生稳定高效的消减作用。研究表明,将降解菌株与特定的辅助菌株相结合可以显著增强其对有机污染物的降解能力。然而,并非所有降解菌株的组合均会提高对有机污染物的降解效率。比如,由两种Bacillus sp.细菌构成的微生物组对烷烃和芳烃的降解率分别较单菌降解率降低了55.3%和39.0%[51]。但目前“降解菌和辅助菌”策略仍是用来构建高效的有机污染物降解微生物组最优策略。例如,合成微生物组YJ-1对土壤中邻苯二甲酸酯(PAEs)降解率达85.78%,较未接种组提高34.11% [52];合成微生物组对土壤中除草剂二氟苯尼康降解率达到82.2%,而单菌最高70.1%[56]。目前已报道的基于合成微生物组学的土壤有机污染物消减作用见表 1。而在贫瘠的污染土壤中,可添加由非降解菌组成的合成菌群,并为其提供丰富的碳和氮源,合成微生物组将得到更好的生长并参与土壤有机氮和除草剂溴氧基等污染物的代谢,分解并释放有利于植物吸收的营养元素,间接促进对农药的降解[62]。因此为保障合成微生物组具有稳定高效的消减作用,在构建时需充分了解微生物组内菌株间的相互作用和消减机制。
|
|
表 1 基于合成微生物组学的土壤中有机污染物消减作用 Table 1 The elemination effect of organic pollutants in soil based on synthetic microbiome |
通过优化合成微生物组菌种组成和培养条件,可显著提高其对复杂有机污染物的消减能力。例如,在全氟辛酸(PFOA)消减实验中,难点主要在于破坏稳定的碳-氟(C-F)键。暴露于PFOA作为一种选择压力,促进了拟杆菌门、甲烷菌门和Petrimonas等微生物的生长,且部分低丰度物种,如Flexilinea flocculi、Bacteriovorax stolpii和Sphingomonas,与氟离子的产生呈正相关关系,这意味着这些菌株的潜在协作功能有助于生物脱氟反应。在此基础上加入玄武岩,可进一步提高约3%的PFOA去除率,且功能性细菌Dechloromonas、Thauera、Zoogloea、Gemmobacter和 Pseudomonas,可在玄武岩载体中大量生长。引入各种铁矿物(磁铁矿、菱铁矿和赤铁矿)后,PFOA去除率提高16.42%,这表明矿物添加也可以促进有机污染物消减。此外,铁矿物还促进了微生物多样性的增加,特别是Geobacter、Shewanella和Pseudomonas等降解相关属的丰富上升。进一步研究揭示,这与细胞外蛋白分泌的增加以及细胞色素合成途径、细胞外聚合物物质(EPS)合成和脱氟相关的基因的相应上调有关[63]。
4.2 基于合成微生物组学的土壤中有机污染物消减机制多组学方法(代谢组学、蛋白质组学、转录组学和宏基因组学)可用于研究合成微生物组对土壤中有机污染物消减机制。宏基因组学可以揭示合成微生物组的全部遗传物质,识别参与特定环境下有机污染物消减的不同微生物组及其功能基因。此外,宏基因组学分析有助于了解不可培养细菌的特征及其潜在的代谢途径,鉴定新的分解代谢基因和代谢通路。转录组学或宏转录组学可以检测单个微生物或微生物组响应环境变化的基因表达,识别参与降解途径的特定差异表达基因,进而揭示微生物组消减有机污染物可能的降解反应和途径。蛋白质组学的重点是研究蛋白质在合成微生物组中的作用,包括单加氧酶、双加氧酶、过氧化氢酶-过氧化物酶和醛脱氢酶等。比较不同条件下的蛋白质表达水平有助于深入了解驱动有机污染物降解的关键蛋白质。代谢组学研究细胞或生物体中的整套小分子量代谢物,分析微生物组的代谢特征,进而识别有机污染物降解的中间代谢产物和终产物,全面了解降解途径。多组学方法可结合基于代谢模型的计算机模拟技术,追踪土壤微生物信号分子、解析代谢物的交换过程,为深入研究复杂污染土壤中微生物组对复合有机污染物的共代谢过程、代谢互作机制提供了可能。
微生物间的协同代谢和交叉喂养是合成微生物组高效消减土壤中有机污染物的重要机制。合成微生物组菌株之间可通过多种机制强化降解有机污染物。1)获得更广谱的降解能力。使用P. putida F1和斯氏假单胞菌OX1(P. stutzeri OX1)两种菌株组建合成微生物组,两种菌株均能以苯和甲苯为底物生长,此外,还分别表现出对于乙苯和邻二甲苯的降解能力,使合成的微生物组具备更广谱的降解能力。2)提供营养物质,促进降解菌株生长。阿特拉津降解菌Arthrobacter sp. AT5在降解阿特拉津过程中会分泌氨基乙醇、乙胺和次黄嘌呤;而这些物质可被非降解菌Halobacillus sp. NY15利用。作为回报,菌株NY15分泌铵和亮氨酸;这些分泌物可被菌株AT5利用,促进其生长和对阿特拉津降解[64]。3)降低中间代谢产物对降解菌株的毒害作用。菌株D. anilini可在缺氧的条件下降解芳香族化合物苯胺,但D. anilini降解苯胺会积累中间产物硫化物,而硫化物浓度的增加,会抑制细胞的生长,引入硫化物代谢细菌T. roseopersicina组建合成微生物组,可显著提高对苯胺的降解效率。4)降低代谢产物积累对降解的反馈抑制。在四氢呋喃(THF)降解菌Rhodococcus ruber YYL与非降解菌Bacillus cereus MLY1组成的微生物组中,菌株YYL产生THF降解中间体,这些中间体可作为菌株MLY1的营养物质,缓解中间体对YYL的产物限制。5)提高菌群对环境胁迫耐受性。YYL对低pH胁迫非常敏感,这会降低其生长速率和THF降解效率。而菌株MLY1消耗YYL产生的酸代谢物,从而提高pH,帮助YYL抵抗酸胁迫[64]。6)协同实现有机污染物矿化。脱卤素杆菌FTH1(Dehalobacter sp. FTH1)具有还原性脱溴能力,可将2,4,6-TBP脱溴转化为苯酚,合成微生物组中另一菌株Clostridium sp. Ma13通过发酵葡萄糖为Dehalobacter sp. FTH1提供氢,最后在脱硫酸盐橡菌DS菌株(Desulfatiglans parachlorophenolica DS)的参与下将苯酚矿化为CO2[65]。然而,合成微生物组内部多样的协同代谢机制难以通过传统的实验室研究方法快速解析,需要借用计算机代谢通路模拟技术,降低合成微生物组设计构建的时间和数据成本,促进合成微生物组在土壤有机消减领域的应用。
群体感应(QS)机制可调控合成微生物组降解有机污染物。微生物之间通过释放信号分子进行种间交流,调控基因表达。QS系统激活时,微生物表达相关酶的基因,催化降解有机污染物。敲除Pseudomonas aeruginosa的信号分子AHLs合成酶基因rhlI,会使双加氧酶的nahH基因表达大幅下调,致使菌株降解率降低。利用基因敲除技术破坏双酚A降解菌Sphingonomas sp. YK5的QS系统时,使得细胞色素酶等与降解相关酶的基因表达水平显著下调,导致YK5无法降解双酚A[66]。QS可使微生物产生更多的胞外聚合物(EPS)以形成生物膜,生物膜有利于促进疏水性有机污染物的溶解,提高有机污染物的生物可利用性,同时还能帮助维持微生物组稳定活性[67]。制备菌剂时,生物炭可吸附酰化高丝氨酸内酯类(AHLs)信号分子,有利于维持细胞活性和细胞聚集,增强细胞间的QS交流,强化生物膜的形成。QS可调节微生物细胞膜表面疏水性,促进污染物附着,在利用Novosphingobium pentaromativorans US6-1及其QS系统缺失突变体分别组建合成微生物组对菲进行降解时,发现US6-1突变体可提高其细胞表面疏水性,使对菲具有更高的降解率[68]。随着研究的不断深入,群体感应相关基因元件及调控原理逐渐清晰。近年来通过合成生物学手段构建包含细菌QS系统组成部分的基因线路,实现了种内和种间的人工控制通讯,这些基于QS的基因线路在土壤有机污染消减领域有着巨大的应用潜力。
越来越多的研究表明,合成微生物组的构建并非简单的菌株组合。需要深入探究不同菌株间复杂的代谢关系和相互作用。合成微生物组内菌种种类越多,组内关系就越复杂,竞争抑制作用也越明显,所以在获得合成微生物组前,往往会开展菌株共培养实验,以确定合适的合成微生物组成员[69]。由于田间实验具有很大的不确定性,在实验室实验后往往需要将合成微生物组负载于合适的载体上(如生物炭,水凝胶等)制备成固定化菌剂,载体材料不仅提供了充足的微生物附着位点,还可以改善土壤物理性质,发挥一定的吸附作用,固定化菌剂克服了功能微生物易受外界环境、温度等条件影响以及生物有效性低的问题,提高了微生物对有机物的降解效能。应用固定化菌剂后可提高有机污染物结合态在土壤中的占比,从而减少了其对植物和微生物的毒性,还能提高土壤中细菌群落的丰富度和多样性,提高过氧化氢酶,过氧化物酶等酶活活性,从而促进土壤中有机污染物的消减。
5 展望近些年来,随着微生物组学技术发展,土壤有机污染消减研究取得显著进展。通过实验室规模和田间规模两种类型的污染生态系统测试合成微生物组的有机污染物消减能力。在田间实验中添加合成微生物组后土壤细菌群落的α多样性增加,但β多样性没有显著变化。此外,合成微生物组添加后部分官能团和氮循环相关基因在植物幼苗期受到显著抑制,可能不利于土壤养分循环过程。合成微生物组往往可以在实验室中有效地降解污染物,但在田间场景中可能效果较差。因此,土壤中有机污染物消减既涉及微生物降解有机污染物,也涉及在污染环境中合理部署合成微生物组,通常需要引入负载载体、分配营养物质和强化消减作用,保证在实际应用时具有良好稳定的消减效果。未来研究需深入探讨合成微生物组的设计、构建与优化的理论和方法,进一步提升消减效率和效果的可持续性。未来以下研究方向仍需关注:1)合成微生物组的优化:合成微生物组方法显著提高了对复杂有机污染土壤的污染物消减能力。未来研究应进一步剖析代谢网络,识别并精准调控消减有机污染物的关键基因,发现更多潜在功能基因,进一步提高合成微生物组对有机污染物消减能力。2)植物与合成微生物组联合作用:合成微生物组与植物的联合作用在消减土壤有机污染方面潜力突出。未来研究应探索植物与微生物的物质交换与养分传递机制,筛选出更适合特定有机污染消减的植物和微生物组合。3)多学科交叉研究与技术创新:通过多学科交叉研究和技术创新,提出有机污染消减强化策略,开发消减新方法,有望探索出更环保和高效的有机污染消减体系。在土壤大规模应用中需要借助大数据分析平台和云端远程监管技术,因地制宜,建立完整的土壤修复评价体系指标。随着更多研究创新与应用案例的出现,合成微生物组技术在土壤有机污染消减领域会迎来更大的突破和发展。
| [1] |
Gao Y Z, Wu Y M, Tang L, et al. Progress in bioremediation of organic contaminated soil based on microbiome (In Chinese)[J]. Chinese Journal of Applied Ecology, 2024, 35(12): 3225-3234. [高彦征, 吴一铭, 汤磊, 等. 基于微生物组学的有机污染土壤生物修复研究进展[J]. 应用生态学报, 2024, 35(12): 3225-3234.]
(  0) 0) |
| [2] |
Tyagi M, da Fonseca M M, de Carvalho C C. Bioaugmentation and biostimulation strategies to improve the effectiveness of bioremediation processes[J]. Biodegradation, 2011, 22(2): 231-241.
(  0) 0) |
| [3] |
Huang X, He J, Yan X, et al. Microbial catabolism of chemical herbicides: Microbial resources, metabolic pathways and catabolic genes[J]. Pesticide Biochemistry and Physiology, 2017, 143: 272-297.
(  0) 0) |
| [4] |
Xu X, Zarecki R, Medina S, et al. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions[J]. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5
(  0) 0) |
| [5] |
Berg G, Rybakova D, Fischer D, et al. Microbiome definition re-visited: Old concepts and new challenges[J]. Microbiome, 2020, 8(1): 103.
(  0) 0) |
| [6] |
Liu C, Jiang M, Yuan M M, et al. Root microbiota confers rice resistance to aluminum toxicity and phosphorus deficiency in acidic soils[J]. Nature Food, 2023, 4(10): 912-924.
(  0) 0) |
| [7] |
Kato S, Haruta S, Cui Z J, et al. Stable coexistence of five bacterial strains as a cellulose-degrading community[J]. Applied and Environmental Microbiology, 2005, 71(11): 7099-7106.
(  0) 0) |
| [8] |
Khalil A S, Collins J J. Synthetic biology: Applications come of age[J]. Nature Reviews Genetics, 2010, 11(5): 367-379.
(  0) 0) |
| [9] |
Lawson C E, Harcombe W R, Hatzenpichler R, et al. Common principles and best practices for engineering microbiomes[J]. Nature Reviews Microbiology, 2019, 17(12): 725-741.
(  0) 0) |
| [10] |
Liu X, Chen K, Chuang S, et al. Shift in bacterial community structure drives different Atrazine-degrading efficiencies[J]. Frontiers in Microbiology, 2019, 10: 88.
(  0) 0) |
| [11] |
Lin X Y, Li C R, Xu X H, et al
. Synthetic microbiome design: Applications and challenges from theoretical construction to environmental bioremediation (In Chinese)[J]. Chinese Journal of Biotechnology, 2025. DOI:10.13345/j.cjb.250436 [林心雨, 李超然, 徐希辉, 等
. 合成微生物组设计: 从理论构建到环境生物修复的应用与挑战[J]. 生物工程学报, 2025.]
(  0) 0) |
| [12] |
Henry C S, DeJongh M, Best A A, et al. High-throughput generation, optimization and analysis of genome-scale metabolic models[J]. Nature Biotechnology, 2010, 28(9): 977-982.
(  0) 0) |
| [13] |
Zhao X Z, Jiang C Y, Liu S J. Synthetic microbiomes: Rational design, engineering strategies, and application prospects (In Chinese)[J]. Chinese Journal of Biotechnology, 2025, 41(6): 2221-2235. [赵晞泽, 姜成英, 刘双江. 人工合成微生物组: 理性设计、精准构建与应用前景[J]. 生物工程学报, 2025, 41(6): 2221-2235.]
(  0) 0) |
| [14] |
Yao Y, Lv Z, Min H, et al. Isolation, identification and characterization of a novel Rhodococcus sp. strain in biodegradation of tetrahydrofuran and its medium optimization using sequential statistics-based experimental designs[J]. Bioresource Technology, 2009, 100(11): 2762-2769.
(  0) 0) |
| [15] |
Ma Z, Jiang M, Liu C, et al. Quinolone-mediated metabolic cross-feeding develops aluminium tolerance in soil microbial consortia[J]. Nature Communications, 2024, 15(1): 10148.
(  0) 0) |
| [16] |
Feng J R, Ni H G. Effects of heavy metals and metalloids on the biodegradation of organic contaminants[J]. Environmental Research, 2024, 246: 118069.
(  0) 0) |
| [17] |
Ojuederie O B, Babalola O O. Microbial and plant-assisted bioremediation of heavy metal polluted environments: A review[J]. International Journal of Environmental Research and Public Health, 2017, 14(12): 1504.
(  0) 0) |
| [18] |
Wu C, Li F, Yi S, et al. Genetically engineered microbial remediation of soils co-contaminated by heavy metals and polycyclic aromatic hydrocarbons: Advances and ecological risk assessment[J]. Journal of Environmental Management, 2021, 296: 113185.
(  0) 0) |
| [19] |
Sharma P, Bano A, Singh S P, et al. Engineered microbes as effective tools for the remediation of polyaromatic aromatic hydrocarbons and heavy metals[J]. Chemosphere, 2022, 306: 135538.
(  0) 0) |
| [20] |
Sarma H, Sonowal S, Prasad M N V. Plant-microbiome assisted and biochar-amended remediation of heavy metals and polyaromatic compounds ─ a microcosmic study[J]. Ecotoxicology and Environmental Safety, 2019, 176: 288-299.
(  0) 0) |
| [21] |
Fan X, Ge A H, Qi S, et al. Root exudates and microbial metabolites: signals and nutrients in plant-microbe interactions[J]. Science China Life Sciences, Published online March 11, 2025.
(  0) 0) |
| [22] |
Muhammad A, Khan M H U, Kong X, et al. Rhizospheric crosstalk: A mechanistic overview of how plant secondary metabolites alleviate abiotic stresses[J]. Plant Science, 2025, 354: 112431.
(  0) 0) |
| [23] |
Fang L C, Hu Z Y, Cui Q L, et al. Construction and application of synthetic communities: A new strategy to improve soil health (In Chinese)[J]. Acta Pedologica Sinica, 2025, 62(5): 1233-1245. DOI:10.11766/trxb202410120392 [方临川, 胡紫莹, 崔庆亮, 等. 合成菌群构建与应用: 提升土壤健康新策略[J]. 土壤学报, 2025, 62(5): 1233-1245.]
(  0) 0) |
| [24] |
Hu H, Wang M, Huang Y, et al. Guided by the principles of microbiome engineering: Accomplishments and perspectives for environmental use[J]. mLife, 2022, 1(4): 382-398.
(  0) 0) |
| [25] |
Xu X H, Liu X W, Jiang J D. Enhanced bioremediation of organic pollutant contaminated environment by microbial consortia: Current situations and challenges (In Chinese)[J]. Journal of Nanjing Agricultural University, 2020, 43(1): 10-17. [徐希辉, 刘晓伟, 蒋建东. 微生物菌群强化修复有机污染物污染环境: 现状与挑战[J]. 南京农业大学学报, 2020, 43(1): 10-17.]
(  0) 0) |
| [26] |
Haque S, Srivastava N, Pal D B, et al. Functional microbiome strategies for the bioremediation of petroleum-hydrocarbon and heavy metal contaminated soils: A review[J]. Science of the Total Environment, 2022, 833: 155222.
(  0) 0) |
| [27] |
Heckmann D, Lloyd C J, Mih N, et al. Machine learning applied to enzyme turnover numbers reveals protein structural correlates and improves metabolic models[J]. Nature Communications, 2018, 9(1): 5252.
(  0) 0) |
| [28] |
Cuperlovic-Culf M, Nguyen-Tran T, Bennett S A L. Machine learning and hybrid methods for metabolic pathway modeling[J]. Methods in Molecular Biology, 2023, 2553: 417-439.
(  0) 0) |
| [29] |
Grosskopf T, Soyer O S. Synthetic microbial communities[J]. Current Opinion in Microbiology, 2014, 18(100): 72-77.
(  0) 0) |
| [30] |
Araujo A S F, Pereira A P A, de Medeiros E V, et al. Root architecture and the rhizosphere microbiome: Shaping sustainable agriculture[J]. Plant Science, 2025, 359: 112599.
(  0) 0) |
| [31] |
Schmitz L, Yan Z, Schneijderberg M, et al. Synthetic bacterial community derived from a desert rhizosphere confers salt stress resilience to tomato in the presence of a soil microbiome[J]. The ISME Journal, 2022, 16(8): 1907-1920.
(  0) 0) |
| [32] |
Pradhan S, Bhattacharjee A, Dubey S, Sharma S. Ameliorating salt stress in tomato by a top-down approach of acclimatizing the rhizosphere microbiome[J]. Plant Physiology, 2025, 177(1): e70071.
(  0) 0) |
| [33] |
Xu Z Y, Hu H Y, Xu P, et al. Development and application of synthetic microbiome (In Chinese)[J]. Synthetic Biology Journal, 2021, 2(2): 181-193. [徐昭勇, 胡海洋, 许平, 等. 人工合成微生物组的构建与应用[J]. 合成生物学, 2021, 2(2): 181-193.]
(  0) 0) |
| [34] |
Berruto C A, Demirer G S. Engineering agricultural soil microbiomes and predicting plant phenotypes[J]. Trends in Microbiology, 2024, 32(9): 858-873.
(  0) 0) |
| [35] |
Behera P R, Behera K K, Sethi G, et al. Enhancing agricultural sustainability through Rhizomicrobiome: A review[J]. Journal of Basic Microbiology, 2024, 64(11): e2400100.
(  0) 0) |
| [36] |
Dahlstrom K M, McRose D L, Newman D K. Keystone metabolites of crop rhizosphere microbiomes[J]. Current Biology, 2020, 30(19): R1131-R1137.
(  0) 0) |
| [37] |
Johns N I, Blazejewski T, Gomes A L, et al. Principles for designing synthetic microbial communities[J]. Current Opinion in Microbiology, 2016, 31: 146-153.
(  0) 0) |
| [38] |
Chen M X, Wei Z, Tian L, et al. Development and application of synthetic microbial community (In Chinese)[J]. Science Bulletin, 2021, 66(3): 273-283. [陈沫先, 韦中, 田亮, 等. 合成微生物群落的构建与应用[J]. 科学通报, 2021, 66(3): 273-283.]
(  0) 0) |
| [39] |
Che S, Men Y. Synthetic microbial consortia for biosynthesis and biodegradation: promises and challenges[J]. Journal of Industrial Microbiology and Biotechnology, 2019, 46(9/10): 1343-1358.
(  0) 0) |
| [40] |
Lear G, Kingsbury J M, Franchini S, et al. Plastics and the microbiome: Impacts and solutions[J]. Environmental Microbiome, 2021, 16(1): 2.
(  0) 0) |
| [41] |
Chen X, He C, Zhang Q, et al. Modularized design and construction of tunable microbial consortia with flexible topologies[J]. ACS Synthetic Biology, 2024, 13(1): 183-194.
(  0) 0) |
| [42] |
Hu Y C, Cao Z H, Zheng L G, et al. Applications of CRISPR-Cas technologies in microbiome engineering (In Chinese)[J]. Chemical Journal of Chinese Universities, 2023, 44(3): 65-77. [胡玉灿, 曹朝辉, 郑灵刚, 等. CRISPR-Cas基因编辑技术在微生物组工程中的应用[J]. 高等学校化学学报, 2023, 44(3): 65-77.]
(  0) 0) |
| [43] |
Ruan Z, Chen K, Cao W, et al. Engineering natural microbiomes toward enhanced bioremediation by microbiome modeling[J]. Nature Communications, 2024, 15(1): 4694.
(  0) 0) |
| [44] |
Li X, Li P, Lin X, et al. Biodegradation of aged polycyclic aromatic hydrocarbons(PAHs)by microbial consortia in soil and slurry phases[J]. Journal of Hazardous Materials, 2008, 150(1): 21-26.
(  0) 0) |
| [45] |
Mukherjee A K, Bordoloi N K. Biodegradation of benzene, toluene, and xylene(BTX)in liquid culture and in soil by Bacillus subtilis and Pseudomonas aeruginosa strains and a formulated bacterial consortium[J]. Environmental Science and Pollution Research, 2012, 19(8): 3380-3388.
(  0) 0) |
| [46] |
Bai N, Li S, Zhang J, et al
. Efficient biodegradation of DEHP by CM9 consortium and shifts in the bacterial community structure during bioremediation of contaminated soil[J]. Environmental Pollution, 2020, 266(Pt 2): 115112.
(  0) 0) |
| [47] |
Huang Y H, Liu Y, Geng J, et al. Maize root-associated niches determine the response variation in bacterial community assembly and function to phthalate pollution[J]. Journal of Hazardous Materials, 2022, 429: 128280.
(  0) 0) |
| [48] |
Qin R, Xu T, Jia X. Engineering Pseudomonas putida to produce rhamnolipid biosurfactants for promoting phenanthrene biodegradation by a two-species microbial consortium[J]. Microbiology Spectrum, 2022, 10(4): e0091022.
(  0) 0) |
| [49] |
Liu T, Ning L, Mei C, et al. Synthetic bacterial consortia enhanced the degradation of mixed priority phthalate ester pollutants[J]. Environmental Research, 2023, 235: 116666.
(  0) 0) |
| [50] |
Khan G, Tiwari A, Patel D K, et al. Bioremediation of polyaromatic hydrocarbon polluted sewage sludge soil employing a bacterial consortium and phytotoxicity evaluation[J]. Letters in Applied Microbiology, 2024, 77(12): ovae130.
(  0) 0) |
| [51] |
Li J, Luo C, Cai X, et al. Microbial consortium assembly and functional analysis via isotope labelling and single-cell manipulation of polycyclic aromatic hydrocarbon degraders[J]. The ISME Journal, 2024, 18(1): wrae115.
(  0) 0) |
| [52] |
Wang L, Li Z, Li M, et al. Mechanisms of synthetic bacterial flora YJ-1 to enhance cucumber resistance under combined phthalate-disease stresses[J]. Journal of Environmental Management, 2024, 365: 121564.
(  0) 0) |
| [53] |
Wu T, Guo S Z, Zhang Y, et al. The engineering of TBBPA-degrading synthetic microbiomes with integrated strategies[J]. NPJ Biofilms and Microbiomes, 2025, 11(1): 139.
(  0) 0) |
| [54] |
Yadav R, Yadav S K, Mishra D, et al. An exploration of bacterial consortia in chlorpyrifos degradation, soil remediation, and promotion of plant growth[J]. Biodegradation, 2025, 36(3): 37.
(  0) 0) |
| [55] |
Wang T, Sui J, Zhou Y, et al. Difenoconazole degradation by novel microbial consortium TA01: Metabolic pathway and microbial community analysis[J]. International Journal of Molecular Sciences, 2025, 26(7): 3142.
(  0) 0) |
| [56] |
Książek-Trela P, Potocki L, Szpyrka E. The impact of novel bacterial strains and their consortium on diflufenican degradation in the mineral medium and soil[J]. Scientific Reports, 2025, 15(1): 18051.
(  0) 0) |
| [57] |
Mokale Kognou A L, Ngono Ngane R A, Jiang Z H, et al. Harnessing the power of microbial consortia for the biodegradation of per- and polyfluoroalkyl substances: Challenges and opportunities[J]. Chemosphere, 2025, 374: 144221.
(  0) 0) |
| [58] |
Ge Y, Yang A, Dong Y, et al. Efficient degradation mechanism of fomesafen by earthworms and gut degrading bacteria synthetic community[J]. Pesticide Biochemistry and Physiology, 2025, 214: 106607.
(  0) 0) |
| [59] |
Huang C, Zhao X, Zhang H, et al. SynCom-mediated herbicide degradation activates microbial carbon metabolism in soils[J]. IMeta, 2025, 426: 103390.
(  0) 0) |
| [60] |
Ma Q, Zhou Y, Parales R E, et al. Effects of herbicide mixtures on the diversity and composition of microbial community and nitrogen cycling function on agricultural soil: A field experiment in Northeast China[J]. Environment Pollution, 2025, 372: 125965.
(  0) 0) |
| [61] |
Ruan Z, Xu M, Xing Y, et al. Enhanced growth of wheat in contaminated fields via synthetic microbiome as revealed by genome-scale metabolic modeling[J]. Science of the Total Environment, 2024, 953: 176047.
(  0) 0) |
| [62] |
Tang Z, Vogel T M, Wang Q, et al. Microbial defluorination of TFA, PFOA, and HFPO-DA by a native microbial consortium under anoxic conditions[J]. Journal of Hazardous Materials, 2024, 465: 133217.
(  0) 0) |
| [63] |
Liu Z, Huang H, Qi M, et al. Metabolite cross-feeding between Rhodococcus ruber YYL and Bacillus cereus MLY1 in the biodegradation of tetrahydrofuran under pH Stress[J]. Applied and Environmental Microbiology, 2019, 85(19): e01196-19.
(  0) 0) |
| [64] |
Yu Y, Huang W, Tang S, et al. Metagenomic and enzymatic mechanisms underpinning efficient water treatment of 2-ethylhexyl diphenyl phosphate(EHDPP)by the microbial consortium 8-ZY[J]. Water Research, 2025, 275: 123178.
(  0) 0) |
| [65] |
Tahir K, Ali A S, Kim J, et al. Enhanced biodegradation of perfluorooctanoic acid in a dual biocatalyzed microbial electrosynthesis system[J]. Chemosphere, 2023, 328: 138584.
(  0) 0) |
| [66] |
Qian B, Rayner J L, Davis G B, et al. Per- and poly-fluoroalkyl substances (PFAS) sensing: A focus on representatively sampling soil vadose zones linked to nano-sensors[J]. Ecotoxicology and Environmental Safety, 2024, 284: 116932.
(  0) 0) |
| [67] |
Lorah M M, He K, Blaney L, et al. Anaerobic biodegradation of perfluorooctane sulfonate (PFOS) and microbial community composition in soil amended with a dechlorinating culture and chlorinated solvents[J]. Science of the Total Environment, 2024, 932: 172996.
(  0) 0) |
| [68] |
Mohammad N E, Mumtahina R, Md Nahid P, et al. PFAS contamination in soil and sediment: Contribution of sources and environmental impacts on soil biota[J]. Case Studies in Chemical and Environmental Engineering, 2024, 9: 100643.
(  0) 0) |
| [69] |
Gonçalves O S, Creevey C J, Santana M F. Designing a synthetic microbial community through genome metabolic modeling to enhance plant-microbe interaction[J]. Environmental Microbiome, 2023, 18(1): 81.
(  0) 0) |
 2026, Vol. 63
2026, Vol. 63


