2. 合肥工业大学土木与水利工程学院,合肥 230009;
3. Department of Plant,Soil and Microbial Sciences,Michigan State University,East Lansing,Michigan 48824,United States of America
土壤中的细菌常常主动利用自身特性进行取食并趋向有利(或躲避有害)环境。细菌的这种趋向有利化学物质(引诱剂,正趋化作用)、规避有害化学物质(驱避剂,负趋化作用)的本能简称为趋化性[1]。细菌趋化现象普遍存在于土壤生态系统中,受土壤物理、化学和生物因素的综合影响。它调节土壤微生物多样性及土壤养分的循环、转化和分布等[2],并在土壤污染生态防治及微生物过程修复等应用研究中意义重大[3]。
自Pfeffer[4]发现细菌趋化性以来,国内外众多学者就针对其展开了广泛的研究,其中,土壤中细菌的趋化现象备受人们关注。但由于土壤本身具有难观测性、空间异质性和时空变化不稳定等特性,土壤中细菌趋化性研究极具困难。近几十年以来,荧光原位杂交技术、微流控技术和光学显微技术的快速发展,为土壤细菌趋化性研究提供了技术支撑和光明的研究前景,极大地推动了其研究进程[5-7]。本文就国内外有关土壤细菌趋化性的研究进展进行总结和探讨,并就将来进一步的研究进行了展望,旨在为今后的相关研究提供一定的参考。
1 细菌趋化行为模式及信号传导通路由于自然环境异质复杂,细菌进化了多种趋化行为模式以适应其周围微环境的变动[8-9]。趋化性是细菌在复杂异质环境中寻找最佳化学环境的能力。当细菌处于存在引诱剂梯度的饱和液体环境中时,细菌会通过调节自身鞭毛旋转方向进行具有偏向性的趋化游动(swimming);当细菌位于含水粗糙表面时,细菌仍依赖鞭毛旋转进行种群涌动(swarming);而当其处于固体界面时,细菌则依靠四型菌毛(type Ⅳ pili, TfP)的收缩或拉伸进行蹭行(twitching)[9]。下面简要介绍上述三种细菌常见的趋化行为模式(游动、涌动和蹭行)及其相应的信号传导通路。
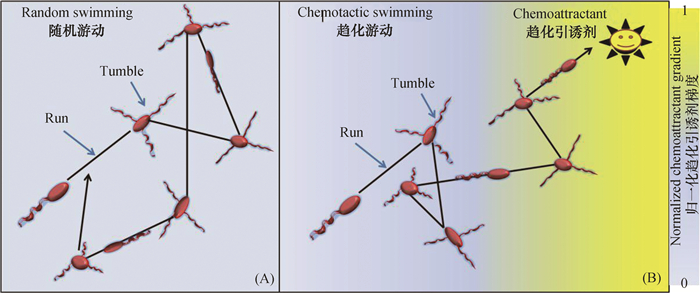
1.1 个体水平上的游动细菌借助鞭毛旋转在小雷诺数(约10−5)液体环境中游动[10]。前人以革兰氏阴性周生鞭毛菌大肠杆菌(Escherichia coli)为模式菌株就细菌鞭毛结构及其介导细菌游动的机制展开了深入研究[1, 8, 11-15]。Berg和Brown[11]利用3D跟踪细胞轨迹显微镜分别追踪E. coli在化学物质均匀和存在梯度的饱和液体环境中的运动行为,发现E. coli在均匀液体环境中的运动类似于随机游动(图 1A),包括近似直线的快速泳动(run)和突然转向的翻滚(tumble)。E. coli直线运行的速度约20 μm·s-1、时间0.8 s左右,此时周生鞭毛(5~10根)合成一束、鞭毛束逆时针方向旋转(从细胞体后面观察);E. coli突然转向翻滚的时间约0.2 s,此时鞭毛束散开、顺时针方向旋转(从细胞体后面观察)。当细菌置身于引诱剂(或驱避剂)浓度梯度中时,其沿着引诱剂(或反向驱避剂)浓度梯度上直线运动的总时间增加,进而实现具有偏向性的趋化游动(图 1B)[11]。周生鞭毛菌一般执行“泳动-翻滚”(run−tumble)运动形式,而极性鞭毛菌则进行“前进−后退”(forward-reverse)(如Shewanell aputrefaciens,Pseudoalteromonas haloplanktis,Vibrio coralliilyticus和Pseudomonas aeruginosa)或“前进-后退-跳转”(forward-reverse-flick)运动形式(如Vibrio alginolyticus)[12]。由于极性鞭毛菌不能像E. coli一样通过周生鞭毛束的“分−合”实现重新定位,便进化了另一种机制,即通过单根鞭毛钩的离轴变形(off-axis deformation)、短暂逆转(brief reversal)来改变游动方向,不过极性鞭毛菌亦是通过增加沿着引诱剂梯度上的总运动时间而实现趋化游动的[12]。
|
图 1 饱和液体微环境中,趋化性细菌(E. coli)在无引诱剂梯度条件下的随机游动(A)或引诱剂梯度条件下的趋化游动(B) Fig. 1 Random swimming of chemotactic bacteria (E. coli) in the absence ofchemoattractant gradient(A) or chemotactic swimming (B) in the presenceofchemoattractant gradientin saturated liquid microenvironment[11, 13] |
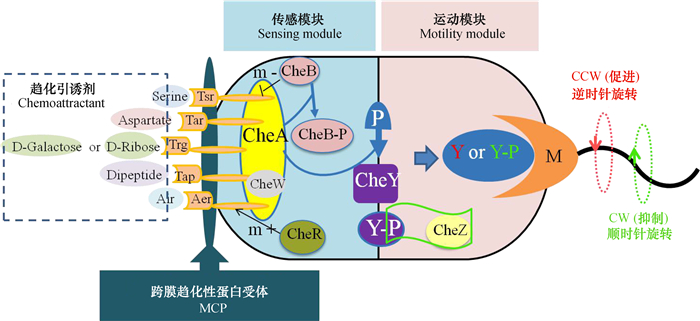
E.coli整个趋化信号传导通路由感受环境中化学物质浓度的跨膜趋化性受体蛋白MCP(Tar, Tsr, Trg, Tap, Aer)和细胞质内的六种调节蛋白(CheA, CheW, CheY, CheB, CheR和CheZ)参与,任何一个MCP和调节蛋白控制基因的突变或缺失均能致使细菌失去对特定引诱剂趋化信号的感知或传递能力,最终使细菌趋化功能受损或彻底丧失[14]。整体上,E. coli整个传导通路由传感模块和运动模块两部分组成(图 2),两模块经关键节点CheY蛋白连接[15]。一般情况下,有活性的CheA蛋白能够进行自磷酸化,一旦CheA蛋白通过自磷酸化得到磷酸基团,则将其转移给响应调节器CheY蛋白,或者去甲基化激酶CheB蛋白。然后磷酸化的CheY蛋白在细胞质中扩散并结合细胞膜上的鞭毛马达,促进鞭毛顺时针旋转[16]。而引诱剂小分子与受体蛋白的结合,会抑制CheA蛋白自磷酸化,降低鞭毛马达顺时针转动比率,从而增加细菌向高浓度引诱剂直线运动的时间,最终实现具有偏向性的随机游动[17]。
虽然极性鞭毛菌趋化信号传导通路与E. coli大体相似,但更为复杂,比如铜绿假单胞菌(Pseudomonas aeruginosa)控制MCPs的基因簇数量较E. coli多,且其细胞质内的调节蛋白也更多样[9]。简单来说,细菌趋化游动主要包括三步:1)MCP精确感受化学刺激并实现信号放大[18];2)细胞质内调节蛋白接收化学信号并通过CheY传递给鞭毛马达;3)鞭毛马达感受趋化信号并调整鞭毛旋转方向,最终产生具有偏向性的趋化游动[19]。需要说明的是,细菌鞭毛马达旋转由离子(周生鞭毛菌为H+,极性鞭毛菌为Na+)穿过细胞质膜沿着电化学梯度推动。H+或Na+沿着电化学梯度通过固定在细胞壁上的特定motAB蛋白复合物,motAB蛋白复合物沿着鞭毛马达前进并使其旋转[20]。
1.2 种群水平上的涌动当细菌位于含水粗糙表面时,细胞经历分裂增殖并依靠鞭毛旋转进行种群涌动,其速度(2~10 μm·s-1)远低于游动。科学工作者在研究细菌涌动机制时发现:1)涌动依赖于鞭毛旋转,并且鞭毛表达更充分的菌株往往表现出更强的涌动能力;2)涌动需要表面活性剂参与,而合成表面活性剂通常受细菌种群感知(quorum sensing, QS)机制的调节[9]。目前,关于细菌涌动的具体机制及其生物学意义尚未明确,比如细菌TfP在细菌涌动过程中的作用机制就较为模糊。Shrout等[23]研究P. aeruginosa的野生型菌株及其缺失pilA基因(编码TfP蛋白)菌株的涌动行为时发现,pilA基因的缺失显著促进了细菌涌动;但Murray和Kazmierczak[24]却发现pilA基因缺失对细菌涌动影响不大。随后,Anyan等[25]利用数学模拟与实验相结合的方法,从物理学角度进一步检验TfP在细菌涌动过程中的作用时发现,细菌种群涌动时TfP会通过调控细胞间排列(cell-cell arrangement)而限制其单细胞运动和种群运动。
当前的研究结果表明,细菌涌动和游动的趋化通路控制基因可能存在显著差异[26-27],但由于细菌涌动不仅涉及细胞分裂增殖,还往往受尚未明确的QS机制的影响,因此,人们对细菌涌动的趋化信号传导通路仍然知之甚少。
1.3 种群水平上的蹭行细菌通过将TfP发射出去—与表面结合—再收缩的方式牵引细菌在固体界面上蹭行,该过程依赖TfP的伸缩而不受制于鞭毛的旋转[24-25]。由于执行运动的功能性附属物不同,细菌蹭行速度(不大于0.1 μm·s-1)远低于游动和涌动。但是,蹭行对细菌生命过程具有多重意义:1)无法蹭行的细菌不能形成成熟的三维结构生物膜;2)蹭行很可能启动了其从饱和液态到固体界面态转变的信号通路[28](当细菌处在较硬固体界面间时,鞭毛作用大大减弱,蹭行接管细菌迁移的任务)。
对于E.coli,其TfP介导的趋化蹭行与鞭毛介导的趋化游动通路大体相近,首先MCP感受细胞外趋化物梯度,然后细胞质内系列调节蛋白(ChpA, ChpB, PilK, ChpC, PilH, PilG, PilG-P和PilH-P)接收化学信号并通过PilG-P和PilH-P传递给TfP,TfP接收信号并通过拉伸或收缩进行趋化蹭行[9]。虽然人们了解了E. coli的趋化蹭行传导通路,但此通路是否能代表所有细菌,有待一一证实。
土壤环境复杂多变,可能存在多种趋化机制。Laganenka等[29]发现E. coli分泌的胞外信使分子(autoinducer)能协调细胞与细胞间的通讯交流,进而增强细菌种群的趋化性迁移[30],这可能是目前尚不清楚的另一种趋化信号传导机制。土壤中细菌数量多、组成复杂(1 g土中细菌数量约1010个[31]),深入探究细菌适应复杂环境的多样化生存策略,将有助于揭示土壤微生物多样性的内在机制,并对微生物学研究及其相关交叉科学研究工作的深入开展具有重要意义。
2 定量细菌趋化性的数学模型定量模型与实验结果相互检验共同促进了人们对细菌趋化性的理解。数学模型可以精确描述细菌特性,例如伊辛模型(Duke和Bray)、Monnod-Wyman-Changeux(MWC)模型[32-33]和受体变量动力学性质模型可分别用于模拟细菌受体簇信号放大和精确适应特性。利用此类模型,不仅可以通过数值模拟仿真单细胞趋化运动,还可以从种群水平上模拟细菌的趋化性。Bray等[18]开启了用数值模拟的方法描述单个细菌趋化性的先河,发现了细菌(E. coli)对很大范围内的引诱剂(5 nmol·L-1~1 mmol·L-1天冬氨酸)具有趋化运动。Emonet等[34]提出了仿真单细胞趋化行为的模拟器AgentCell,Bary等[35]随后提出了类似的模拟器E.solo。细菌种群水平趋化性的模拟始于Keller-Segel(KS)模型。该模型是1971年Keller和Segel[36]提出的基于扩散和定向运动描述细菌种群趋化行为的一维数学模型:
| $ \frac{\partial b}{\partial t}=\nabla \cdot \left( \mu \nabla b-{{x}_{o}}b\nabla s \right) $ | (1) |
| $ \frac{\partial b}{\partial t}=D{{\nabla }^{2}}s-f\left( b, s \right) $ | (2) |
式中,b为细菌种群密度(bacterial population density),ind·mL-1,s为引诱剂浓度(attractant concentration),mol·L-1,μ为细菌随机运动系数(bacterial random motility coefficient),mm2·s-1,xo为细菌趋化感应系数(bacterial chemotactic sensitivity coefficient),mm/receptor,D为引诱剂扩散系数(attractant diffusion coefficient),mm2·s-1,f(b,s)描述引诱剂的消耗或降解。KS经典模型能很好地描述细菌在缓慢变化环境中的趋化行为,一直被沿用至今。但也有不足之处,如不能解释细菌在快速变化环境中的趋化行为。Si等[37]在研究时空变化环境下细菌趋化行为时发现,在高频引诱剂浓度变化下,细菌种群响应滞后于浓度变化,甚至出现细菌在低浓度区域聚集的反常现象。
1975年Lovely和Dahlquist[38]首次将细菌个体细胞观察结果与随机运动系数(μ)联系起来,提供了描述细菌种群运动的宏观运输参数。
| $ \mu =\frac{{{v}^{2}}t}{3\left( 1-\cos \theta \right)} $ | (3) |
式中,v为单个细菌平均游动速度(themeanindividual-cell swimming speed),µm·s-1,t表示单个细菌平均游动时间(the mean individual-cell run time),单位s,θ表示单个细菌平均旋转角度(the mean individual-cell turn angle),单位°。
在种群规模(population scale)上,细菌趋化运动可用类似于对流运输系数描述[36, 39]。
| $ {{v }_{c}}=\frac{2}{3}v\tanh \left( \frac{{{x}_{o}}}{2v}\frac{{{K}_{d}}}{{{\left( {{K}_{d}}+s \right)}^{2}}}\frac{\partial s}{\partial x} \right) $ | (4) |
式中,vc表示细菌趋化运动速度(bacterial chemotactic speed),µm·s-1。Kd是受体解离平衡常数(receptor dissociation equilibrium constant),mol·L-1。
在上述研究基础上,Zaval’skiĭ[40]建立了细菌趋化性动力学模型:
| $ \underbrace{\frac{\partial b}{\partial t}-}_{\left( \text{Ⅰ} \right)}\underbrace{\frac{5{{v}^{2}}}{3{{\gamma }_{0}}}\frac{{{\partial }^{2}}b}{\partial {{x}^{2}}}+\frac{v}{3}}_{\left( \text{Ⅱ} \right)}\underbrace{\left( \psi \frac{\partial b}{\partial t}+\frac{\partial \psi }{\partial t}b \right)=}_{\left( \text{Ⅲ} \right)}0 $ | (5) |
式中,γo(Hz)为细菌在化学均匀的介质中细胞运动方向改变的频率,ψ为趋化作用函数,用式(6)表示。
| $ \psi = \frac{{{\rm{ \mathsf{ δ} }}{K_d}}}{{{{\left( {{K_D} + C} \right)}^2}}}\frac{{\partial s}}{{\partial x}}V $ | (6) |
式中,δ为经验系数,假如趋化因子能够被细菌代谢,式(6)应该用描述趋化因子消耗或降解的式(7)代替。
| $ \frac{{\partial C\left( {x, t} \right)}}{{\partial t}} = - K\left( C \right)b\left( {x, t} \right) + D\frac{{{\partial ^2}C\left( {x, t} \right)}}{{\partial {x^2}}} $ | (7) |
式中,K(C)为细菌消耗引诱剂速率,mol·s-1。
式(5)描述细菌在线性化学梯度下的运动行为,其中(Ⅰ)项是对细菌浓度随时间的动态变化的描述;(Ⅱ)项表示细菌在均匀化学介质中的随机运动;(Ⅲ)项表示细菌向较高浓度引诱剂的迁移行为,取决于引诱剂分子的梯度和细菌受体与引诱剂分子的密切程度。
Lewus和Ford[41]于2001年将数学模型与实验数据相结合,分别基于细菌种群水平和单细胞水平实现了对细菌的随机运动系数和趋化运动系数的定量化,并且提出了无化学梯度下细菌的随机运动守恒方程(式(8))和化学梯度下细菌的偏向性趋化运动守恒方程(式(9)),
| $ \frac{{\partial b}}{{\partial t}} = {\mu _0}\frac{{{\partial ^2}b}}{{\partial {x^2}}} $ | (8) |
| $ \frac{{\partial b}}{{\partial t}} = \mu \frac{{{\partial ^2}b}}{{\partial {x^2}}} - \frac{\partial }{{\partial x}}\left( {{v_c}b} \right) $ | (9) |
式中,μ由式(3)计算得到,v由式(4)计算得到。
结合实验数据得到细菌个体水平参数,将其应用到细菌种群水平的宏观运动中可描述细菌种群趋化运动。Joanna等[42]将一维数学模型进行优化,实现了细菌趋化运动的二维、三维模拟,建议今后的模型使用者根据实际实验条件将模型进行修正优化,使数学模拟结果尽可能接近实验真实情况。
3 土壤中影响细菌趋化性的因素土壤中存在大量孔隙结构,孔隙大小和孔隙内物理、化学及生物环境共同作用影响微生物生命历程以及其群落结构[3, 43-44]。细菌常常利用自身趋化性寻找土壤中的营养源(如植物根系分泌或渗出物、其他微生物的胞外分泌物、土壤中的有机物质和化肥等),或远离其中的有害化学物质(如化学农药、抗生素和有害重金属离子等)。
3.1 物理因素土壤的时空异质性决定了细菌趋化现象普遍存在,但由于土壤孔隙大小不一、形状各异,并且其孔隙内微环境条件随时间和空间动态变化,因此,细菌在土壤中的运动并不像在自由水体环境中那么简单。当孔隙尺寸小于细菌鞭毛长度时,即使存在引诱剂或抑制剂,细菌也很难实现趋化运动(由于细菌鞭毛无法旋转不能改变运动方向)[45]。此外,当细菌个体尺寸大于孔隙大小时,往往容易阻塞(straining)孔隙、因而阻碍其进行趋化运动[46]。除阻塞外,细菌在土壤多孔介面的吸附是妨碍细菌进行趋化运动的另一个重要原因,其中,细菌和土壤多孔界面之间的Derjaguin-Landau-Verwey-Overbeek(DLVO)相互作用力[47-48]是关键影响因子之一。DLVO理论指出,细菌或胶体和土壤多孔界面之间的相互作用力主要包括三种力,即范德华吸引力、双电层力和短距离排斥力(包括水合力和空间排斥力等)[49-50]。当细菌和土壤多孔介面的电荷符号相同时,双电层力为排斥力,此时,存在排斥势垒会阻碍细菌吸附在土壤多孔介面。如果细菌和土壤多孔介面的电荷符号相反时,双电层力为吸引力,此时,总的相互作用力在任何相互作用距离均为吸引力,因此细菌极易被吸附在土壤多孔介面。
水分条件亦是影响土壤中细菌趋化运动的另一关键因素。水分条件影响并调节细菌趋化运动的模式:当土壤水分饱和时,土壤孔隙间水膜连续,细菌可在适宜大小的孔隙内趋化游动;当土壤水分不饱和时,部分孔隙间水膜不连续,细菌在粗糙表面上进行趋化涌动;而当土壤相对干旱时,蹭行很可能是细菌趋化运动的主要方式;同时,由于土壤孔隙结构及其内部环境在亚毫米尺度就差异很大,细菌很可能会根据周围微环境变化灵活更换趋化运动模式[9, 43-44],但细菌是如何感知周围水分变化并灵活调节运动附属物来执行适宜的趋化运动呢?详细机制还有待进一步探究。
此外,细菌的趋化性还受土壤温度的显著影响。Adler[51]使用毛细管法研究不同温度(0~40℃)条件下E. coli趋化性时发现,当温度低于15℃时,E. coli不再具有趋化性;而温度从20℃上升至30℃时,聚集在毛细管附近的细菌数量则增加了20倍。这是关于温度影响细菌趋化性的首次报道,那么温度影响细菌趋化运动的机制是什么呢?Larsen等[52]发现,随着环境温度的降低(从25℃降低至5℃),细菌(Vibrio anguillarum)运动速度明显减小,其对引诱剂的趋化响应也变得迟钝。当时,Larsen等[52]猜测,温度通过影响参与细菌趋化信号传导的跨膜受体和酶活性,以及其甲基化水平,而改变细菌趋化信号的传导速度。随后,Oleksiuk等[53]验证了上述猜想,并且量化了细菌运动速度与温度的关系,发现细菌的趋化运动速度在5~40℃范围内与环境温度线性正相关。
3.2 化学因素适宜的pH是细菌生长和执行趋化运动的必要条件之一,主要原因是引诱剂具有特定的pH适宜范围,大部分引诱剂适宜pH范围为6~9.5,pH小于6或大于9.5时细菌趋化运动能力下降,而当pH小于或等于4时其趋化能力基本丧失[54]。此外,离子浓度也显著影响细菌趋化能力,Larsen等[52]研发现细菌(V. anguillarum)在低盐溶液中的趋化性响应更为灵敏。此外,细菌趋化运动也受土壤中溶解氧浓度的调控[9]。例如,Shioi等[55]研究发现,当溶解氧浓度较高时,Salmonella typhimurimu、E. coli和Bacillus subtilis进行远离高浓度溶解氧的负趋化运动,而当溶解氧浓度低于0.25 mmol·L-1时,则进行趋向高浓度溶解氧的正趋化运动。同时,Tao[56]利用KS数学模型对细菌趋化运动中的耗氧量进行了计算。但是,人们对氧气浓度影响细菌趋化性的具体机制尚不明确。此外,土壤溶液重金属离子和污染物浓度也是影响细菌趋化运动的重要化学因素[54]。
细菌趋化性是土壤中物质循环的关键,受土壤理化性质的显著影响。因此,通过调节土壤理化条件(土壤孔隙度、水分、温度、pH、盐碱度、氧气浓度等)来调控细菌趋化运动,最终实现土壤养分的高效利用是一项长远而具有深刻意义的重要研究课题。
4 土壤中细菌趋化现象及其意义 4.1 趋化性影响土壤中微生物群落结构环境中的细菌一般会对多种化学物质具有趋化性,细菌种类不同,引诱剂也存在差别。土壤中微生物群落结构容易受细菌趋化作用的调节,其在土壤根际微环境中尤为显著[57]。20世纪80年代,Caetano-Anollés等[58]发现了细菌运动性和趋化性在根系定殖过程中的重要作用,表明细菌运动性和趋化性是细菌与植物根表面建立初始“化学-物理接触”的重要因素。De Weert等[59]也发现了趋化性在细菌竞争定殖(competitive colonization)中的必要作用。随后,Juárez-Hernández等[60]对比了野生型Pseudomonas putida KT 2440及其趋化受体缺失的突变体在番茄根系的定殖能力,发现趋化功能健全的野生型菌株更容易定殖到根系表面。综上可知,细菌趋化性是细菌在作物根系成功定殖的关键。细菌之所以能借助自身趋化性成功定殖于作物根系,主要是因为植物根系分泌的化学物质被微生物视作一种趋化信号(引诱剂-正趋化运动信号或驱避剂-负趋化运动信号)[61],它可以影响并调节细菌在根系的定殖水平和其在根际土壤中生存的能力,改变土壤微生物群落结构[62]。此外,Wu等[63]研究连作障碍产生的原因时发现,连作会导致根系有益微生物数量减少,而增加致病菌趋化蛋白CheA的表达,具体机制是,连作累计时间效应增加了土壤中致病真菌的营养引诱剂(有机酸),显著促进了致病真菌的生长,致使根系土壤微生物群落发生了改变。因此,进一步研究细菌趋化性在连作障碍中的作用机制将有助于明确单作系统土壤微生物群落结构的演变规律。
今后可从基因水平上研究趋化基因表达(通过改变土壤理化条件)与土壤微生物群落结构间的内在定量关系。此外,目前关于细菌趋化性对土壤微生物多样性影响的相关研究还不够深入,人们较多关注单一菌株的趋化作用。然而,自然环境中的细菌通常不是单独生活,而是与其他种群存在竞争、互利共生或交叉喂养等综合作用。研究多种菌株趋化作用过程中的互作或协同作用,有助于揭示土壤微生物多样性内在机制。
4.2 细菌趋化性对土壤养分循环的影响前人研究表明,细菌趋化性促进海洋生态系统中养分的转化与循环[2, 64]。单从物理作用的角度分析,细菌鞭毛运动能够使其周围液体产生流场(flow field)[65],该流场会影响化学物质的运输、细胞与其临近界面间的物理作用以及细胞间的耦合运动[66];而事实上这一过程往往是更为复杂的生物−物理−化学综合作用。Smriga等[2]发现,死亡浮游植物细胞裂解后释放大量溶解性有机物,而这些养分物质则会吸引具有趋化能力的细菌,该趋化过程仅仅持续约5~10 min,期间,细菌将裂解细胞捕食完后逐渐趋于随机运动,直至下一个捕食目标的出现。Pedler等[64]进一步说明了细菌趋化性具有促进海洋中碳循环的功能。
与海洋生态系统相比,土壤生态环境中养分的转化与循环更为复杂,其中,微生物的活动显得至关重要。在这一过程中,微生物通过释放胞外活性酶,分解大部分有机质或促进养分物质的矿化,以便从复杂的有机化合物中获取营养[31]。土壤中死亡的微生物、动物和植物枯枝落叶以及部分有机胶体颗粒均是细菌的营养源,细菌能将其矿化、固持,将植物不可直接利用的营养转化为微生物量碳、氮等。该过程加速了土壤中养分循环和能量流动,促进作物对养分的吸收利用。目前,我们关于细菌趋化性如何促进土壤养分循环的研究还处于定性化初级研究阶段。
4.3 细菌趋化性促进土壤有机污染物降解微生物在自然环境中进化出了完善的趋化作用机制来应对生存环境变化,例如,细胞通过MCP感知周围化学信号的变化,借助鞭毛和TfP执行偏向性运动,以便寻找适宜的生存环境[20]。近几十年来,科研工作者在细菌对环境污染物趋化性方面开展了大量卓有成效的研究[3, 67],并分离和鉴定了多种对环境异源污染物具有趋化性的微生物(表 1)。越来越多的证据显示,具有运动能力的微生物对其可降解的环境污染物几乎均具有趋化性,且大多细菌拥有明确探测和响应化学污染物的感官系统[68]。细菌对特定污染物的趋化基因与降解基因间有着密切联系,这点已从细菌对其大多降解底物具有趋化性的表观现象中得到初步证实[69]。Harwood等[70]也从分子生物学水平找到了直接证据,发现Pseudomonas putida PRS2000的4−基苯甲酸降解基因与趋化基因是被质粒共同调节的。随后,Hawkins和Harwood [71]发现Ralstonia eutropha JMP134的质粒上同时具有2, 4−二氯苯氧乙酸的趋化基因和降解基因。
|
|
表 1 对土壤异源污染物具有正趋化作用的微生物一览表 Table 1 A list of microbes showing positive chemotaxis to soil heterologous pollutants |
细菌通过对可代谢污染物的趋化作用可促进其对污染物的降解,该过程是生物修复受污染土壤的重要内容。Adadevoh等[3, 67]结合土柱实验和数学模型,在研究萘降解菌Pseudomonas putida G7和其趋化性突变体Pseudomonas putida G7 Y1对萘的趋化和降解作用时发现,细菌趋化能力延长了其在萘分布均匀的土柱中的穿透时间,进而提高了对萘的降解效率(萘的回收率降低了45%)。实际上,细菌对污染物的趋化运动实现了降解菌株与目标污染物之间的直接接触,而这样的接触是实现生物修复的关键。同时,趋化性增加降解菌寻找合适碳源、氮源和其他养分或能源的机会,使降解菌在与土著微生物的营养竞争过程中不至于一败涂地(甚至表现出优势),这对于低浓度污染物的生物修复尤为重要[68-69]。总之,土壤细菌趋化性研究将为土壤污染的生态防治和生物修复等应用研究提供理论基础和技术支撑。然而,利用土壤细菌趋化性实现对土壤污染的原位修复还有很长的一段路要走。
5 土壤细菌趋化性的研究方法作为天然的多孔介质,土壤由复杂的物理拓扑结构和孔隙网络内各种各样的化学物质、养分或电子受体等各类动态梯度组成,土壤中孔隙结构不仅数量众多而且时空差异性大。这些特征在亚毫米尺度上就差异很大,使得土壤细菌趋化性的宏观研究结果难以解释,同时,土壤的不透明性和复杂的内部结构使得土壤微生物过程的原位观测变得尤为困难,一定程度上限制了土壤细菌趋化性的研究[111]。为实现细菌趋化运动的定性或定量化研究,学者们研发了系列细菌趋化性研究方法,如毛细管法(图 3A)、琼脂平板法(图 3B)和砂柱实验系统(图 3C)及荧光原位杂交微流体显微术(图 3D)。下面简要概述前三种方法并重点介绍荧光原位杂交微流体显微术及其在土壤细菌趋化性研究中的应用。

|
图 3 细菌趋化性研究方法:改进的毛细管法(A);琼脂平板法(B);砂柱实验系统(C);荧光原位杂交微流体显微术(D) Fig. 3 Bacterialchemotaxis assays: Modified capillary assay (A); Agar-plate method (B); Sand-column experimental system (C); and Fluorescence in situ hybridization microfluidic microscopy (D) |
1884年,Pfeffer[4]将装有天冬氨酸溶液的毛细管水平放置到细菌悬浮液中,发现细菌会跟踪扩散出来的天冬氨酸分子,聚集在毛细管管口并进入毛细管内,由此发现了细菌的趋化现象。Sherris等[112]使用相似的扁平毛细管(flat capillary tube)观察了细菌对空气或精氨酸趋化游动的现象。毛细管法是细菌趋化性研究最原始的方法,该方法虽然能实现细菌趋化游动的半定量化[1],但不能用于研究细菌的趋化涌动和趋化蹭行。
5.2 琼脂平板法(agar-plate assay)琼脂平板法常用于研究细菌对可代谢引诱剂的趋化运动。如将菌液接种到琼脂培养基中心,趋化细菌消耗中心处营养物后会向外散开寻找新营养源;又或是将引诱剂置于琼脂培养基中心,一定时间形成浓度梯度后于特定位置接种菌液,记录细菌在引诱剂梯度上的趋化运动[1, 9]。该方法的优势在于调节琼脂培养基中琼脂浓度(w/v)可实现细菌运动模式的研究。普遍认为当琼脂浓度小于0.3%时,细菌在液体环境中游动;当琼脂浓度介于0.3%与1.0%之间时,细菌进行种群涌动;而琼脂浓度大于1.0%时,大量细菌在固体界面间蹭行[9, 25, 27-28]。人们常通过对比细菌在不同化学梯度琼脂培养基上的菌落形貌来分析细菌是否对该物质具有趋化作用,而事实上,菌落扩增不能单单归因于细菌的趋化运动,细菌分裂增殖引起的细胞排挤也很可能促使细胞向外沿移动,因此需结合细菌生长动力学模型、计算养分扩散与消耗速率等,综合分析后再做判定[44]。此外,该方法的优点在于简便、经济,但缺点是难以精确控制细菌运动环境条件(水、养分、氧气等)[9]。
5.3 砂柱实验系统(sand-column experimental system)砂柱实验系统常用于模拟细菌在多孔异质介质(土壤)中与水和溶质共同迁移的特征,借助化学物质浓度梯度再现土壤异质特性,调控石英砂粒径分布生成一定范围内的孔隙分布的砂土介质[42]。该系统操作便捷,价格便宜,赢得了众多研究者的青睐,不足之处在于1)无法实现细菌在含有养分梯度砂柱中趋化过程的可视化;2)不适于细质地土壤的模拟;3)恒定控制砂柱内非饱和水环境十分困难[67]。
5.4 荧光原位杂交微流体显微术(Fluorescence in situ hybridization microfluidic microscopy, FISHMM)荧光原位杂交技术、微流控技术和近代光学显微术相结合的集合技术简称为FISHMM,该集合不仅具有非接触、无损伤的特点,还快速、便捷,实现了细菌在可控微环境中趋化运动的可视化、定量化和数据化。DNA荧光原位杂交技术(Fluorescence in situ hybridization, FISH)是一种应用非放射性荧光物质依靠核酸探针杂交原理在核中或染色体上显示DNA序列位置的方法。该项技术安全、快速、准确、原位以及可动态观察等特点使其成为环境微生物学、微生物生态学和微生物诊断学研究的可靠工具[113]。但传统的荧光蛋白分子探针(绿色荧光蛋-GFP、红色荧光蛋白-RFP、黄色荧光蛋白-YFP等)标记细菌,存在长时间跟踪细胞轨迹激发光激发条件下荧光易淬灭的缺点。新型的纳米生物探针量子点具有光化学稳定性高、抗光漂白能力强、荧光不易淬灭等优良光学特性,能对完整的活细胞进行定性、定量和定位的测量,功能强大,可操作性强[114],预计新一代量子点技术将在土壤细菌趋化性及高清晰度活细胞成像、长时程观察活细胞迁移等方面发挥巨大潜力。
相比较传统的细菌趋化性研究方法,微流体技术能更精确地控制细菌趋化运动的微环境,开启了从单细胞尺度原位探究细菌趋化性的大门。微流控技术能够在微米尺度上定量分析细菌的趋化运动过程。Mao等[115]首次将微流技术应用到细菌趋化性研究中来,模拟构建了微生物栖息地的微观结构,实现了细菌趋化过程中细胞运动轨迹的可视化。为模拟土壤中微孔结构,越来越多的科学工作者专注于微流体设备平台的搭建,不仅能制出比线性结构外更复杂结构的微流体,还能控制微流体表面的湿度、粗糙度和亲疏水性等(分辨率可达到2 μm)[5-6]。总之,微流控技术具有能够精确控制微通道反应器中流体速度、水分含量、温度大小和化学梯度及表面特性等物理化学生物环境的能力。
简言之,首先利用微流体技术在透明且生物相容的有机材料(如:聚二甲基硅氧烷—PDMS或聚乙二醇—PEGDA等水凝胶)上拓印出几微米至数百微米的微沟道,用以模拟土壤中大小不一、形状各异的孔隙结构,再结合荧光原位杂交技术和光学显微技术3D追踪细胞的运动轨迹,二者结合实现了基于多细胞和微孔尺度上土壤细菌趋化作用机制的研究。荧光显微镜研究的快速发展,自动成像和细菌追踪技术的进步,为细胞内部形态、结构和生命运动现象观测提供技术支撑。值得一提的是,细菌运动轨迹的3D追踪一直是细菌趋化运动研究的核心技术。传统光学显微镜捕捉到的照片只是细菌空间运动轨迹的2D投影。不过,3D轨迹跟踪显微镜能够得到单个细菌的时空运动信息,其工作原理是基于通过3D显微镜的电动载物台的自动运动确保单个细菌的自动聚焦,该类显微镜为我们提供了周生鞭毛菌E. coli和单鞭毛菌Caulobacter crescentus单个细胞运动的基础信息[13],此技术的缺点在于一次只能追踪一个细菌。近阶段,数字全息显微术(digital holographic microscopy)和离焦显微术(defocused microscopy)[116]能够在不移动显微镜载物台的情况下,同时捕获上百个微生物的位置,二者功能强大,应用前景广阔。
总之,由现代分子生物学和生物物理学研究手段结合的荧光原位杂交微流体显微术,实现了从土壤孔隙水平和细胞尺度上探究土壤中微生物运动,能够可视化土壤内部生物过程,为定量生物学研究提供了强有力的工具,外加操作简便、节约空间,未来必将受到各个研究实验室的青睐。
6 研究展望虽然土壤细菌趋化性研究取得了一定的进展,但还有很多工作值得科学研究人员的重视并深入探究,例如:1)土壤孔隙和细胞水平上探究细菌对土壤动植物分泌物、土壤异源污染物的趋化机理,剖析细菌趋化基因与降解和代谢基因间的分子生物学关系;2)探究QS影响细菌种群的趋化性迁移的机制,进一步解析细菌趋化过程中细胞间的信号传递机制;3)探究细菌趋化性在细菌生存调控、种间关系调节和土壤微生物多样性维持及土壤养分再分配中的作用机制;4)探究细菌TfP介导细菌趋化运动的机制,并分析探讨其与鞭毛介导趋化运动的协作机制;5)研究细菌趋化性与土壤物理化学因素(土壤含水量、质地、孔隙结构、矿物组分等)的相互作用。这些问题均有待更多的科研工作去揭示。总之,对土壤细菌趋化性的深入研究有助于揭示细菌对环境的适应机制,在土壤微生物多样性维持、土壤养分分配和土壤污染生态防治及微生物过程修复等应用研究中意义重大,值得进行更多的深入研究。
致谢 感谢博士生王燕和韩天富对文献收集和讨论方面提供帮助。
| [1] |
Adler J. Chemotaxis in bacteria . Science, 1966, 153(3737): 708-716. DOI:10.1126/science.153.3737.708
(  0) 0) |
| [2] |
Smriga S, Fernandez V I, Mitchell J G, et al. Chemotaxis toward phytoplankton drives organic matter partitioning among marine bacteria . Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(6): 1576-1581. DOI:10.1073/pnas.1512307113
(  0) 0) |
| [3] |
Adadevoh J S T, Triolo S, Ramsburg C A, et al. Chemotaxis increases the residence time of bacteria in granular media containing distributed contaminant sources . Environmental Science and Technology, 2015, 50(1): 181-187.
(  0) 0) |
| [4] |
Pfeffer W. Locomotorische richtungsbewegungen durch chemische reize . Untersuchungen aus dem Botanischen Institut, in Tübingen, 1884, 3: 363-482.
(  0) 0) |
| [5] |
Deng J, Orner E P, Chau J F, et al. Synergistic effects of soil microstructure and bacterial EPS on drying rate in emulated soil micromodels . Soil Biology and Biochemistry, 2015, 83: 116-124. DOI:10.1016/j.soilbio.2014.12.006
(  0) 0) |
| [6] |
Cruz B C, Furrer J M, Guo Y, et al. Pore-scale water dynamics during drying and the impacts of structure and surface wettability . Water Resources Research, 2017, 53(7): 5585-5600. DOI:10.1002/2016WR019862
(  0) 0) |
| [7] |
Lambert B S, Raina J B, Fernandez V I, et al. A microfluidics-based in situ chemotaxis assay to study the behaviour of aquatic microbial communities . Nature Microbiology, 2017, 2(10): 1344-1349. DOI:10.1038/s41564-017-0010-9
(  0) 0) |
| [8] |
Adler J. Chemoreceptors in bacteria . Science, 1969, 166(3913): 1588-1597. DOI:10.1126/science.166.3913.1588
(  0) 0) |
| [9] |
Sampedro I, Parales R E, Krell T, et al. Pseudomonas chemotaxis . FEMS Microbiology Reviews, 2015, 39(1): 17-46.
(  0) 0) |
| [10] |
Purcell E M. Life at low Reynolds number . American Journal of Physics, 1977, 45(1): 3-11. DOI:10.1119/1.10903
(  0) 0) |
| [11] |
Berg H C, Brown D A. Chemotaxis in Escherichia coli analysed by three-dimensional tracking . Nature, 1972, 239(5374): 500-504. DOI:10.1038/239500a0
(  0) 0) |
| [12] |
Xie L, Berg H C. From the Cover:Bacterial flagellum as a propeller and as a rudder for efficient chemotaxis . Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(6): 2246-2251. DOI:10.1073/pnas.1011953108
(  0) 0) |
| [13] |
Berg H C. E. coli in motion . Physics Today, 2004, 58(2): 64-65.
(  0) 0) |
| [14] |
Bi S, Lai L. Bacterial chemoreceptors and chemoeffectors . Cellular and Molecular Life Sciences, 2015, 72(4): 691-708. DOI:10.1007/s00018-014-1770-5
(  0) 0) |
| [15] |
Sourjik V, Berg H C. Binding of the Escherichia coli response regulator CheY to its target measured in vivo by fluorescence resonance energy transfer . Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(20): 12669-12674. DOI:10.1073/pnas.192463199
(  0) 0) |
| [16] |
Bi S, Sourjik V. Stimulus sensing and signal processing in bacterial chemotaxis . Current Opinion in Microbiology, 2018, 45: 22-29. DOI:10.1016/j.mib.2018.02.002
(  0) 0) |
| [17] |
Hazelbauer G L. Bacterial chemotaxis:The early years of molecular studies . Annual Review of Microbiology, 2012, 66(1): 285-303. DOI:10.1146/annurev-micro-092611-150120
(  0) 0) |
| [18] |
Bray D, Levin M D, Mortonfirth C J. Receptor clustering as a cellular mechanism to control sensitivity . Nature, 1998, 393(6570): 85-88.
(  0) 0) |
| [19] |
Porter S L, Wadhams G H, Armitage J P. Signal processing in complex chemotaxis pathways . Nature Reviews Microbiology, 2011, 9(3): 153-165. DOI:10.1038/nrmicro2505
(  0) 0) |
| [20] |
Gabel C V, Berg H C. The speed of the flagellar rotary motor of Escherichia coli varies linearly with proton motive force . Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(15): 8748-8751. DOI:10.1073/pnas.1533395100
(  0) 0) |
| [21] |
Shimizu T S, Tu Y, Berg H C. A modular gradient-sensing network for chemotaxis in Escherichia coli revealed by responses to time-varying stimuli . Molecular Systems Biology, 2014, 6(1): 382-396.
(  0) 0) |
| [22] |
Alexandre G, Zhulin I B. More than one way to sense chemicals . Journal of Bacteriology, 2001, 183(16): 4681-4686. DOI:10.1128/JB.183.16.4681-4686.2001
(  0) 0) |
| [23] |
Shrout J D, Chopp D L, Just C L, et al. The impact of quorum sensing and swarming motility on Pseudomonas aeruginosa biofilm formation is nutritionally conditional . Molecular Microbiology, 2010, 62(5): 1264-1277.
(  0) 0) |
| [24] |
Murray T S, Kazmierczak B I. Pseudomonas aeruginosa exhibits sliding motility in the absence of type Ⅳ pili and flagella . Journal of Bacteriology, 2008, 190(8): 2700-2708. DOI:10.1128/JB.01620-07
(  0) 0) |
| [25] |
Anyan M E, Amiri A, Harvey C W, et al. Type Ⅳ pili interactions promote intercellular association and moderate swarming of Pseudomonas aeruginosa . Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(50): 18013-18018. DOI:10.1073/pnas.1414661111
(  0) 0) |
| [26] |
BurkartM, Toguchi A, Harshey R M. The chemotaxis system, but not chemotaxis, is essential for swarming motility in Escherichia coli . Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(5): 2568-2573. DOI:10.1073/pnas.95.5.2568
(  0) 0) |
| [27] |
Mariconda S, Wang Q, Harshey R M. A mechanical role for the chemotaxis system in swarming motility . Molecular Microbiology, 2010, 60(6): 1590-1602.
(  0) 0) |
| [28] |
Burrows L L. Pseudomonas aeruginosa twitching motility:Type Ⅳ pili in action . Annual Review of Microbiology, 2012, 66(4): 493-520.
(  0) 0) |
| [29] |
Laganenka L, Colin R, Sourjik V. Corrigendum:Chemotaxis towards autoinducer 2 mediates autoaggregation in Escherichia coli . Nature Communications, 2016, 7: 13979. DOI:10.1038/ncomms13979
(  0) 0) |
| [30] |
Long Z, Quaife B, Salman H, et al. Cell-cell communication enhances bacterial chemotaxis toward external attractants . Scientific Reports, 2017, 7(1): 12855. DOI:10.1038/s41598-017-13183-9
(  0) 0) |
| [31] |
Paul E A. Soil microbiology, ecology and biochemistry . Oxford:Academic Press, 2014.
(  0) 0) |
| [32] |
Monod J, Wyman J, Changeux J P. On the nature of allosteric transitions:A plausible model . Journal of Molecular Biolog, 1965, 12(1): 88-118. DOI:10.1016/S0022-2836(65)80285-6
(  0) 0) |
| [33] |
Changeux J P, Edelstein S J. Allosteric mechanisms of signal transduction . Science, 2005, 308(5727): 1424-1428. DOI:10.1126/science.1108595
(  0) 0) |
| [34] |
Emonet T, Macal C M, North M J, et al. AgentCell:A digital single-cell assay for bacterial chemotaxis . Bioinformatics, 2005, 21(11): 2714-2721. DOI:10.1093/bioinformatics/bti391
(  0) 0) |
| [35] |
Bray D, Levin M D, Lipkow K. The chemotactic behavior of computer-based surrogate bacteria . Current Biology, 2007, 17(1): 12-29. DOI:10.1016/j.cub.2006.11.027
(  0) 0) |
| [36] |
Keller E F, Segel L A. Model for chemotaxis . Journal of Theoretical Biology, 1971, 30(2): 225-234. DOI:10.1016/0022-5193(71)90050-6
(  0) 0) |
| [37] |
Si G, Tang M, Yang X. A pathway-based mean-field model for E. coli chemotaxis:Mathematical derivation and its hyperbolic and parabolic limits . Multiscale Modeling Simulation, 2014, 12(2): 907-926. DOI:10.1137/130944199
(  0) 0) |
| [38] |
Lovely P S, Dahlquist F W. Statistical measures of bacterial motility and chemotaxis . Journal of Theoretical Biology, 1975, 50(2): 477-496. DOI:10.1016/0022-5193(75)90094-6
(  0) 0) |
| [39] |
Rivero M A, Tranquillo R T, Buettner H M, et al. Transport models for chemotactic cell populations based on individual cell behavior . Chemical Engineering Science, 1989, 44(12): 2881-2897. DOI:10.1016/0009-2509(89)85098-5
(  0) 0) |
| [40] |
Zaval'skiĭ L I. Kinetic analysis of the chemotaxis of bacteria . Biofizika, 1988, 33(2): 328-332.
(  0) 0) |
| [41] |
Lewus P, Ford R M. Quantification of random motility and chemotaxis bacterial transport coefficients using individual-cell and population-scale assays . Biotechnology and Bioengineering, 2001, 75(3): 292-304. DOI:10.1002/(ISSN)1097-0290
(  0) 0) |
| [42] |
Joanna S T, Adadevoh S O, Brian D, et al. Modeling transport of chemotactic bacteria in granular media containing distributed contaminant sources . Environmental Science and Technology, 2017, 51(24): 14192-14198. DOI:10.1021/acs.est.7b04443
(  0) 0) |
| [43] |
Tecon R, Or D. Bacterial flagellar motility on hydrated rough surfaces controlled by aqueous film thickness and connectedness . Scientific Reports, 2015, 6: 19409.
(  0) 0) |
| [44] |
Wang G, Or D. Hydration dynamics promote bacterial coexistence on rough surfaces . The ISME Journal, 2013, 7(2): 395-404. DOI:10.1038/ismej.2012.115
(  0) 0) |
| [45] |
Berg H C, Turner L. Chemotaxis of bacteria in glass capillary arrays. Escherichia coli, motility, microchannel plate, and light scattering . Biophysical Journal, 1990, 58(4): 919-930. DOI:10.1016/S0006-3495(90)82436-X
(  0) 0) |
| [46] |
Diaz J, Rendueles M, Diaz M. Straining phenomena in bacteria transport through natural porous media . Environmental Science and Pollution Research, 2010, 17: 400-409. DOI:10.1007/s11356-009-0160-2
(  0) 0) |
| [47] |
Bhattacharjee S, Ko C H, Elimelech M. DLVO interaction between rough surfaces . Langmuir, 1998, 14(12): 3365-3375. DOI:10.1021/la971360b
(  0) 0) |
| [48] |
Choi N C, Choi J W, Kwon K S, et al. Quantifying bacterial attachment and detachment using leaching solutions of various ionic strengths after bacterial pulse . AMB Express, 2017, 7(1): 38-47. DOI:10.1186/s13568-017-0340-2
(  0) 0) |
| [49] |
Hahn M W, O'Melia C R. Deposition and reentrainment of Brownian particles in porous media under unfavorable chemical conditions:Some concepts and applications . Environmental Science and Technology, 2004, 38(1): 210-220. DOI:10.1021/es030416n
(  0) 0) |
| [50] |
Hahn M W, Abadzic D, O'Melia C R. Aquasols:on the role of secondary minima . Environmental Science and Technology, 2004, 38: 5915-5924. DOI:10.1021/es049746d
(  0) 0) |
| [51] |
Adler J. A method for measuring chemotaxis and use of the method to determine optimum conditions for chemotaxis by Escherichia coli . Journal of General Microbiology, 1973, 74(1): 77-91. DOI:10.1099/00221287-74-1-77
(  0) 0) |
| [52] |
Larsen M H, Blackburn N, Larsen J L, et al. Influences of temperature, salinity and starvation on the motility and chemotactic response of Vibrio anguillarum . Microbiology, 2004, 150(5): 1283-1290. DOI:10.1099/mic.0.26379-0
(  0) 0) |
| [53] |
Oleksiuk O, Jakovljevic V, Vladimirov N, et al. Thermal robustness of signaling in bacterial chemotaxis . Cell, 2011, 145(2): 312-321. DOI:10.1016/j.cell.2011.03.013
(  0) 0) |
| [54] |
Adler J, Templeton B. The effect of environmental conditions on the motility of Escherichia coli . Journal of General Microbiology, 1967, 46(2): 175-184. DOI:10.1099/00221287-46-2-175
(  0) 0) |
| [55] |
Shioi J, Dang C V, Taylor B L. Oxygen as attractant and repellent in bacterial chemotaxis . Journal of Bacteriology, 1987, 169(7): 3118-3123. DOI:10.1128/jb.169.7.3118-3123.1987
(  0) 0) |
| [56] |
Tao Y. Boundedness in a chemotaxis model with oxygen consumption by bacteria . Journal of Mathematical Analysis and Applications, 2011, 381(2): 521-529. DOI:10.1016/j.jmaa.2011.02.041
(  0) 0) |
| [57] |
Ofek-Lalzar M, Sela N, Goldman-Voronov M, et al. Niche and host-associated functional signatures of the root surface microbiome . Nature Communications, 2013, 5: 4950.
(  0) 0) |
| [58] |
Caetano-Anollés G, Bauer W D, Favelukes G. Role of motility and chemotaxis in efficiency of nodulation by rhizohiummeliloti . Plant Physiology, 1988, 86(4): 1228-1235. DOI:10.1104/pp.86.4.1228
(  0) 0) |
| [59] |
De Weert S, Vermeiren H, Mulders I H, et al. Flagella-driven chemotaxis towards exudate components is an important trait for tomato root colonization by Pseudomonas fluorescens . Molecular Plant-Microbe Interactions, 2002, 15(11): 1173-1180. DOI:10.1094/MPMI.2002.15.11.1173
(  0) 0) |
| [60] |
Juárez-Hernández D, Krell T, Corral-Lugo A, et al. Chemotaxis studies of Pseudomonas putida KT 2440 and mutants in chemoreceptors for efficient colonization of tomato plants . Biotechnology World Symposium, 2014.
(  0) 0) |
| [61] |
Mark G L, Dow J M, Kiely P D, et al. Transcriptome profiling of bacterial responses to root exudates identifies genes involved in microbe-plant interactions . Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(48): 17454-17459. DOI:10.1073/pnas.0506407102
(  0) 0) |
| [62] |
Zhang N, Wang D, Liu Y, et al. Effects of different plant root exudates and their organic acid components on chemotaxis, biofilm formation and colonization by beneficial rhizosphere-associated bacterial strains . Plant and Soil, 2014, 374(1/2): 689-700.
(  0) 0) |
| [63] |
Wu L, Chen J, Xiao Z, et al. Barcoded pyrosequencing reveals a shift in the bacterial community in the rhizosphere and rhizoplane of rehmannia glutinosa under consecutive monoculture . International Journal of Molecular Sciences, 2018, 19(3): 850-867. DOI:10.3390/ijms19030850
(  0) 0) |
| [64] |
Pedler B E, Aluwihare L I, Farooq A. Single bacterial strain capable of significant contribution to carbon cycling in the surface ocean . Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(20): 7202-7207. DOI:10.1073/pnas.1401887111
(  0) 0) |
| [65] |
Elgeti J, Winkler R G, Gompper G. Physics of microswimmers-single particle motion and collective behavior:A review . Reports on Progress in Physics, 2015, 78(5): 1-50.
(  0) 0) |
| [66] |
Drescher K, Dunkel J, Cisneros L H, et al. Fluid dynamics and noise in bacterial cell-cell and cell-surface scattering . Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(27): 10940-10945. DOI:10.1073/pnas.1019079108
(  0) 0) |
| [67] |
Adadevoh J S T, Ramsburg C A, Ford R M. Chemotaxis increases the retention of bacteria in porous media with residual NAPL entrapment . Environmental Science and Technology, 2018, 52(13): 7289-7295. DOI:10.1021/acs.est.8b01172
(  0) 0) |
| [68] |
Gupta S, Pathak B, Fulekar M H. Molecular approaches for biodegradation of polycyclic aromatic hydrocarbon compounds:A review . Reviews in Environmental Science and Bio/Technology, 2015, 14(2): 241-269. DOI:10.1007/s11157-014-9353-3
(  0) 0) |
| [69] |
Krell T, Lacal J, Reyesdarias J A, et al. Bioavailability of pollutants and chemotaxis . Current Opinion Biotechnology, 2013, 24(3): 451-456. DOI:10.1016/j.copbio.2012.08.011
(  0) 0) |
| [70] |
Harwood C S, Nichols N N, Kim M K, et al. Identification of the pca RKF gene cluster from Pseudomonas putida:Involvement in chemotaxis, biodegradation, and transport of 4-hydroxybenzoate . Journal of Bacteriology, 1994, 176(21): 6479-6488. DOI:10.1128/jb.176.21.6479-6488.1994
(  0) 0) |
| [71] |
Hawkins A C, Harwood C S. Chemotaxis of Ralstonia eutropha JMP134(pJP4)to the herbicide 2, 4-dichlorophenoxyacetate . Applied and Environmental Microbiology, 2002, 68(2): 968-972. DOI:10.1128/AEM.68.2.968-972.2002
(  0) 0) |
| [72] |
Coates J D, Chakraborty R, Lack J G, et al. Anaerobic benzene oxidation coupled to nitrate reduction in pure culture by two strains of Dechloromonas . Nature, 2001, 411(6841): 1039-1043. DOI:10.1038/35082545
(  0) 0) |
| [73] |
Estévez E, Veiga M C, Kennes C. Biodegradation of toluene by the new fungal isolates Paecilomyces variotii and Exophiala oligosperma . Journal of Industrial Microbiology and Biotechnology, 2005, 32(1): 33-37. DOI:10.1007/s10295-004-0203-0
(  0) 0) |
| [74] |
Chakraborty R, Coates J D. Anaerobic degradation of monoaromatic hydrocarbons . Applied Microbiology and Biotechnology, 2004, 64(4): 437-446.
(  0) 0) |
| [75] |
Lovley D R, Baedecker M J, Lonergan D J, et al. Oxidation of aromatic contaminants coupled to microbial iron reduction . Nature, 1989, 339(6222): 297-300. DOI:10.1038/339297a0
(  0) 0) |
| [76] |
Harms G, Zengler K, Rabus R, et al. Anaerobic oxidation of o-xylene, m-xylene, and homologous alkylbenzenes by new types of sulfate-reducing bacteria . Applied and Environmental Microbiology, 1999, 65(3): 999-1004.
(  0) 0) |
| [77] |
Hess A, Zarda B, Hahn D, et al. In situ analysis of denitrifying toluene-and m-xylene-degrading bacteria in a diesel fuel-contaminated laboratory aquifer column . Applied and Environmental Microbiology, 1997, 63(6): 2136-2141.
(  0) 0) |
| [78] |
Rabus R, Widdel F. Anaerobic degradation of ethylbenzene and other aromatic hydrocarbons by new denitrifying bacteria . Archives of Microbiology, 1995, 163(2): 96-103. DOI:10.1007/BF00381782
(  0) 0) |
| [79] |
Ball H A, Johnson H A, Reinhard M, et al. Initial reactions in anaerobic ethylbenzene oxidation by a denitrifying bacterium, strain EB1 . Journal of Bacteriology, 1996, 178(19): 5755-5761. DOI:10.1128/jb.178.19.5755-5761.1996
(  0) 0) |
| [80] |
Demir G. Degradation of toluene and benzene by Trametes versicolor . Journal of Environmental Biology, 2004, 25(1): 19-25.
(  0) 0) |
| [81] |
Alagappan G, Cowan R M. Effect of temperature and dissolved oxygen on the growth kinetics of Pseudomonas putida F1 growing on benzene and toluene . Chemosphere, 2004, 54(8): 1255-1265. DOI:10.1016/j.chemosphere.2003.09.013
(  0) 0) |
| [82] |
Rui L, Kwon Y M, Fishman A, et al. Saturation mutagenesis of toluene ortho-monooxygenase of Burkholderiacepacia G4 for enhanced 1-naphthol synthesis and chloroform degradation . Applied and Environmental Microbiology, 2004, 70(6): 3246-3252. DOI:10.1128/AEM.70.6.3246-3252.2004
(  0) 0) |
| [83] |
Parales R E, Ditty J L, Harwood C S. Toluene-degrading bacteria are chemotactic towards the environmental pollutants benzene, toluene, and trichloroethylene . Applied and Environmental Microbiology, 2000, 66(9): 4098-4104. DOI:10.1128/AEM.66.9.4098-4104.2000
(  0) 0) |
| [84] |
Kim J M, Jeon C O. Isolation and characterization of a new benzene, toluene, and ethylbenzene degrading bacterium, Acinetobacter sp. B113 . Current Microbiology, 2009, 58(1): 70-75. DOI:10.1007/s00284-008-9268-8
(  0) 0) |
| [85] |
Jung I G, Park C H. Characteristics of Rhodococcus pyridinovorans PYJ-1 for the biodegradation of benzene, toluene, m-xylene(BTX), and their mixtures . Journal of Bioscience and Bioengineering, 2004, 97(6): 429-431. DOI:10.1016/S1389-1723(04)70232-7
(  0) 0) |
| [86] |
Alvarez P J, Vogel T M. Substrate interactions of benzene, toluene, and para-xylene during microbial degradation by pure cultures and mixed culture aquifer slurries . Applied and Environmental Microbiology, 1991, 57(10): 2981-2985.
(  0) 0) |
| [87] |
Oh Y S, Bartha R. Construction of a bacterial consortium for the biofiltration of benzene, toluene and xylene emissions . World Journal of Microbiology and Biotechnology, 1997, 13(6): 627-632. DOI:10.1023/A:1018506602004
(  0) 0) |
| [88] |
Amor L, Kennes C, Veiga M C. Kinetics of inhibition in the biodegradation of monoaromatic hydrocarbons in presence of heavy metals . Bioresource Technology, 2001, 78(2): 181-185. DOI:10.1016/S0960-8524(00)00182-6
(  0) 0) |
| [89] |
Deeb R A, Alvarezcohen L. Temperature effects and substrate interactions during the aerobic biotransformation of BTEX mixtures by toluene-enriched consortia and Rhodococcus rhodochrous . Biotechnology and Bioengineering, 2015, 62(5): 526-536.
(  0) 0) |
| [90] |
Prenafetaboldú F X, Vervoort J, Grotenhuis J T, et al. Substrate interactions during the biodegradation of benzene, toluene, ethylbenzene, and xylene(BTEX)hydrocarbons by the fungus Cladophialophora sp. strain T1 . Applied and Environmental Microbiology, 2002, 68(6): 2660-2665. DOI:10.1128/AEM.68.6.2660-2665.2002
(  0) 0) |
| [91] |
Kim J M, Jeon C O. Isolation and characterization of a new benzene, toluene, and ethylbenzene degrading bacterium, Acinetobacter sp. B113 . Current Microbiology, 2009, 58(1): 70-75. DOI:10.1007/s00284-008-9268-8
(  0) 0) |
| [92] |
Stoecker M A, Herwig R P, Staley J T. Rhodococcus zopfii sp. nov., a toxicant-degrading bacterium . International Journal of Systematic Bacteriology, 1994, 44(1): 106-110. DOI:10.1099/00207713-44-1-106
(  0) 0) |
| [93] |
Chang B V, Wu W B, Yuan S Y. Biodegradation of benzene, toluene; and other aromatic compounds by Pseudmonas sp. D8 . Chemosphere, 1997, 65(12): 2807-2815.
(  0) 0) |
| [94] |
Eriksson M, Sodersten E, Yu Z, et al. Degradation of polycyclic aromatic hydrocarbons at low temperature under aerobic and nitrate-reducing conditions in enrichment cultures from northern soils . Applied and Environmental Microbiology, 2003, 69(1): 275-284. DOI:10.1128/AEM.69.1.275-284.2003
(  0) 0) |
| [95] |
Rockne K J, Cheesanford J C, Sanford R A, et al. Anaerobic naphthalene degradation by microbial pure cultures under nitrate-reducing conditions . Applied and Environmental Microbiology, 2000, 66(4): 1595-1601. DOI:10.1128/AEM.66.4.1595-1601.2000
(  0) 0) |
| [96] |
Lacal J, Muñoz-Martínez F, Reyes-Darías J A, et al. Bacterial chemotaxis towards aromatic hydrocarbons in Pseudomonas . Environmental Microbiology, 2011, 13(7): 1733-1744. DOI:10.1111/j.1462-2920.2011.02493.x
(  0) 0) |
| [97] |
Tartakovsky B, Levesque M, Dumortier R, et al. Biodegradation of pentachlorophenol in a continuous anaerobic reactor augmented with Desulfitobacterium frappieri PCP-1 . Applied and Environmental Microbiology, 1999, 65(10): 4357-4362.
(  0) 0) |
| [98] |
Beaudet R, Lévesque M J, Villemur R, et al. Anaerobic biodegradation of pentachlorophenol in a contaminated soil inoculated with a methanogenic consortium or with Desulfitobacterium frappieri strain PCP-1 . Applied Microbiology and Biotechnology, 1998, 50(1): 135-141. DOI:10.1007/s002530051268
(  0) 0) |
| [99] |
Chiu T C, Yen J H, Liu T L, et al. Anaerobic degradation of the organochlorine pesticides DDT and heptachlor in river sediment of Taiwan . Bulletin of Environmental Contamination and Toxicology, 2004, 72(4): 821-828.
(  0) 0) |
| [100] |
Bouchard B, Beaudet R, Villemur R, et al. Isolation and characterization of Desulfitobacterium frappieri sp. nov., an anaerobic bacterium which reductively dechlorinates pentachlorophenol to 3-chlorophenol . International Journal of Systematic Bacteriology, 1996, 46(4): 1010-1015. DOI:10.1099/00207713-46-4-1010
(  0) 0) |
| [101] |
Palmira D B, Mariana T, Miriam H, et al. Biodegradation of ochratoxin a by bacterial strains isolated from vineyard soils . Toxins, 2015, 7(12): 5079-5093. DOI:10.3390/toxins7124864
(  0) 0) |
| [102] |
Zhang H H, Wang Y, Zhao C, et al. Biodegradation of ochratoxin by Alcaligenes faecalis isolated from soil . Journal of Applied Microbiology, 2017, 123(3): 661-668.
(  0) 0) |
| [103] |
Gordillo F, Chávez F P, Jerez C A. Motility and chemotaxis of Pseudomonas sp. B4 towards polychlorobiphenyls and chlorobenzoates . FEMS Microbiology Ecology, 2010, 60(2): 322-328.
(  0) 0) |
| [104] |
Tremaroli V, Suzzi C V, Fedi S, et al. Tolerance of Pseudomonas pseudoalcaligenes KF707 to metals, polychlorobiphenyls and chlorobenzoates:effects on chemotaxis-, biofilm-and planktonic-grown cells . FEMS Microbiology Ecology, 2010, 74(2): 291-301.
(  0) 0) |
| [105] |
Parales R E, Harwood C S. Toluene-degrading bacteria are chemotactic towards the environmental pollutants benzene, toluene, and trichloroethylene . Applied and Environmental Microbiology, 2000, 66(9): 4098-4104. DOI:10.1128/AEM.66.9.4098-4104.2000
(  0) 0) |
| [106] |
Lanfranconi M P, Alvarez H M, Studdert C A. A strain isolated from gas oil-contaminated soil displays chemotaxis towards gas oil and hexadecane . Environmental Microbiology, 2010, 5(10): 1002-1008.
(  0) 0) |
| [107] |
Liu X, Parales R E. Bacterial chemotaxis to atrazine and related s-triazines . Applied Environmental Microbiology, 2009, 75(17): 5481-5488. DOI:10.1128/AEM.01030-09
(  0) 0) |
| [108] |
Pandey J, Sharma N K, Khan F, et al. Chemotaxis of Burkholderia sp. strain SJ98 towards chloronitroaromatic compounds that it can metabolise . BMC Microbiology, 2012, 12(1): 19-28. DOI:10.1186/1471-2180-12-19
(  0) 0) |
| [109] |
Leungsakul T, Keenan B G, Smets B F, et al. TNT and nitroaromatic compounds are chemoattractants for Burkholderiacepacia R34 and Burkholderia sp. strain DNT . Applied Microbiology and Biotechnology, 2005, 69(3): 321-325. DOI:10.1007/s00253-005-1983-5
(  0) 0) |
| [110] |
Vardar G, Barbieri P, Wood T K. Chemotaxis of Pseudomonas stutzeri OX1 and Burkholderia cepacia G4 toward chlorinated ethenes . Applied Microbiology and Biotechnology, 2005, 66(6): 696-701. DOI:10.1007/s00253-004-1685-4
(  0) 0) |
| [111] |
Kemner K M, O'Brien S, Whiteside M D, et al. Synchrotron-based micro and nanotomographic investigations of soil aggregate microbial and pore structure . American Geophysical Union:AGU Fall Meeting Abstracts, 2017.
(  0) 0) |
| [112] |
Sherris J C, Preston N W, Shoesmith J G. The influence of oxygen and arginine on the motility of a strain of Pseudomonas sp . Journal of General Microbiology, 1957, 16(1): 86-96. DOI:10.1099/00221287-16-1-86
(  0) 0) |
| [113] |
Liu B, Gulino M, Morse M, et al. Helical motion of the cell body enhances Caulobactercrescentus motility . Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(31): 11252-11256. DOI:10.1073/pnas.1407636111
(  0) 0) |
| [114] |
Roy P, Periasamy A P, Lin C Y, et al. Photoluminescent graphene quantum dots for in vivo imaging of apoptotic cells . Nanoscale, 2015, 7(6): 2504-2510. DOI:10.1039/C4NR07005D
(  0) 0) |
| [115] |
Mao H, Cremer P S, Manson M D. A sensitive, versatile microfluidic assay for bacterial chemotaxis . Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(9): 5449-5454. DOI:10.1073/pnas.0931258100
(  0) 0) |
| [116] |
Arai R, Otsuka T, Mori K, et al. Live from under the lens:Exploring microbial motility with dynamic imaging and microfluidics . Nature Reviews Microbiology, 2015, 13(12): 761-775. DOI:10.1038/nrmicro3567
(  0) 0) |
2. College of Civil Engineering, Hefei University of Technology, Hefei 230009, China;
3. Department of Plan, Soil and Microbial Sciences, Michigan State University, East Lansing, Michigan 48824, United States of America
 2019, Vol. 56
2019, Vol. 56