由土壤病原细菌、真菌或线虫等病原物引起的作物土传病害, 是全球作物生产的主要限制因子之一。近年来, 我国土传病害频繁爆发, 每年对我国近5亿亩次(1公顷合15亩)经济作物生产造成的损失达数百亿元。以茄科劳尔氏菌(Ralstonia solanacearum)引起的土传细菌性青枯病为例, 目前我国仅有4个省市未报道过该病害, 该病原菌能够侵染番茄、花生、生姜和烟草等几十种重要经济作物, 造成作物减产和重大经济损失[1]。在自然条件下, 植物与土壤微生物之间联系紧密, 在根土交界面——根际蕴含着大量的微生物, 它们对植物的生长和健康有重要影响[2], 促进植物营养元素周转和激素代谢[3], 辅助有毒有害物质的消减和吸收转运[4]。土传病原菌一般从根系入侵植物, 然而在进入根内之前, 必须要先突破根际土壤微生物组成的生物屏障, 因此认为土壤微生物群落在抵御病原菌入侵植物根系中发挥重要作用[5]。
土壤微生物包括细菌、真菌、原生动物和病毒等微小生物, 它们在根际形成复杂的生物互作网络, 对作物的生长和健康有重要的影响。原生动物以细菌及真菌为食, 在土壤食物网中处于初级消费者的地位, 其在防治土传病害方面具有一定潜力。目前, 关于土传病害防控的研究主要集中于有益细菌和真菌的防控效果及作用机制[6-9]等方面, 而原生动物防治土传病害的研究鲜有报道[10], 我国学者的关注则更少。本文梳理了土壤原生动物的功能及其在土传病害防控中的作用机制研究, 总结了原生动物的研究方法, 提出了一些值得重视的科学问题和研究方向, 以期引起学者对原生动物及其生态功能的关注和研究。
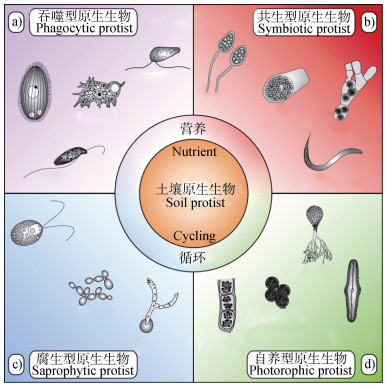
1 土壤原生动物及其生态功能 1.1 土壤原生动物在原生生物中的分类原生生物(Protist)是除植物、真菌和动物外的真核生物。土壤原生生物一般生活在土壤中或土壤表面覆盖的凋落物中, 是土壤食物网的重要成员。按照其在食物网中的生态系统功能, 原生生物被分为自养型(Autotrophic)、腐生型(Saprobiotic)、共生型(Symbiotic)和吞噬型(Phagocytic)四大类[11](图 1)。世界上大约四分之一的光合作用是由自养型原生生物进行的。在土壤生态系统中, 虽然自养型原生生物仅占土壤原生生物的一小部分, 但它们对土壤有机碳输入的贡献不可忽略[12]。此外, 一些腐生型原生生物也参与有机物质的降解, 在碳氮循环和养分转化过程中发挥着重要作用[13-15]。预测分析发现, 目前所有已知的原生生物中, 有约15%表现出共生(寄生或互惠)的生活方式, 大多数对其寄主无明显危害, 但其中一些会引起动植物病害[16], 比如锥虫属原生动物不仅可以引起人类的克氏锥虫病, 还能导致咖啡、椰子等植物韧皮部坏死病[17-18]。虽然这类有害原生生物所占比例较小, 但其风险仍值得关注。吞噬型原生生物, 即原生动物(Protozoa), 是土壤食物网中的消费者。在温室种植模式下表现为鞭毛虫占优势, 而室外农田中的肉足虫占比较高[19]。它们不仅可以捕食细菌, 也可捕食真菌[20-21], 甚至能捕食线虫[22]。Clarke和Maddera[23]利用荧光探针监测网柄菌变形虫(Dictyostelium amoebae)吞噬细菌时发现, 吞噬由变形虫体内的肌动蛋白驱动, 被吞噬的细菌内陷形成密封的吞噬泡, 并在新的肌动蛋白丝推动下进入变形虫体内完成消化过程。基于其捕食特性, 研究者开始探究土壤原生动物在土传病害防控中的作用效果和机制。
|
图 1 土壤原生动物在原生生物中的分类地位与生态功能(改自Geisen等[11]) Fig. 1 Taxonomic status and ecological functions of soil protozoa among protist (modified from Geisen et al[11]) |
土壤原生动物在土传病害防控中的作用研究始于20世纪60年代, 但相对于土壤细菌和真菌而言, 相关报道非常少。巨型肉足虫能够捕食多种病原真菌以及非病原真菌的菌丝和孢子等, 纤毛虫可降低棉籽受立枯丝核菌(Rhizoetonia solani)侵染的风险[24]。Foissner[25]发现, 当土壤原生动物存在时, 即使加入植物致病细菌(Bacillus aorideae)也不会影响马铃薯的正常生长, 而不接种原生动物的对照组则在几天之内发病枯萎。而后Chakraborty和Old[20]的研究表明, 一种Leptomyxiada目的变形虫通过裂解小麦全蚀病病原真菌的菌丝与孢子, 进而抑制小麦全蚀病;原生动物肾形虫的代谢产物可抑制黄萎病菌(Vertieillium dahiiae), 减少侵染率7%~11%, 增加棉花产量30%;齿脊肾形虫(Colpda steinii)、僧帽肾形虫(C. cueullus)或白色变形虫(Amoeba alibda)可裂解黄萎病菌菌丝或使它的孢子及菌核不发芽。应用快速生长的纤毛虫(Gross glockneriaaluta)可使12种病原真菌迅速死亡或形成胞囊[26]。作者研究发现在限菌条件下, 番茄根际接种变形虫Vahlkampfiidae paravahlkampfia和Vahlkampfiidae naegleria可以显著降低土传青枯病的发病率(未发表数据)。
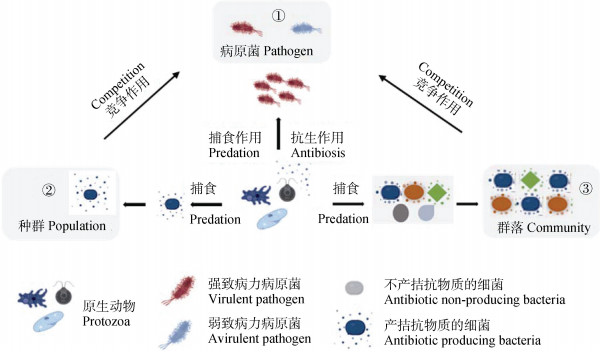
2 原生动物防控土传病害的作用机制 2.1 原生动物直接抑制土壤病原菌原生动物可直接捕食根际土壤中的病原细菌或真菌, 降低其数量, 进而抑制土传病害发生和发展(图 2)。Xiong等[27]通过宏基因组数据共发生网络分析发现, 番茄根际原生动物与病原青枯菌数量之间呈现负相关关系, 推测原生动物可能会直接捕食病原青枯菌。随后宋宇琦等(未发表数据)通过室内研究明确了原生动物对病原青枯菌具有很高的捕食效率, 与捕食模式大肠杆菌的效率相当。变形虫在土壤中捕食小麦全蚀病病原真菌(Gaeumannomyces graminis var. tritici)时, 首先进行接触, 然后捕食真菌的孢子或完整的真菌, 最后直接穿透真菌细胞壁或通过特殊的食物液泡将其消化[28]。
|
图 2 土壤原生动物参与土传病害防控的可能机制 Fig. 2 The possible mechanisms of soil protozoa to prevent and control soil-borne diseases |
除直接捕食作用外, 研究发现一些原生动物分泌的代谢产物也具有抑菌作用。Rosenthal等[29]发现, 卡式棘阿米巴(Acanthamoeba castellanii)的代谢产物可裂解9种革兰氏阳性菌的细胞壁, 其功能代谢产物由1-己糖胺酶、3-半乳糖苷酶、肽水解酶及淀粉酶组成。Riekenberg等[30]发现, 溶组织内阿米巴(Entamoeba histolytica)的β-N-乙酰基己糖胺酶能直接参与裂解细菌细胞的过程。Long等[31]发现, 变形虫也可能利用代谢产物抑制水稻黄单胞菌(Xanthomonas oryzae)的两个致病变种, 而不是通过直接的吞噬作用。
此外, 在原生动物的捕食胁迫下, 病原菌为了生存, 提高耐受性的同时还可能降低其致病性。黏质沙雷氏菌(Serratia marcescens ssp. marcescens)是一种机会致病菌, 因产生灵菌红素致病因子而在平板上形成红色菌落, 当施加原生动物嗜热四膜虫(Tetrahymena thermophila)时, 出现白色无毒菌落的频率显著提高, 而白色菌株对原生动物捕食的抵御能力更强, 但其致病力却显著降低[32]。
2.2 原生动物间接抑制土壤病原菌原生动物的捕食具有一定的选择性, 这可能由于不同细菌对原生动物的抵御能力不同。原生动物的捕食可刺激某些细菌产生更多的拮抗物质, 而其中一些拮抗物质对病原菌具有抑制作用, 进而减少病害的发生(图 2)。例如, 荧光假单胞菌(Pseudomonas fluorescens)通过产生2, 4-二乙酰基间苯三酚(DAPG)有效抑制病原青枯菌的生长, 从而减弱土传青枯病的发生[33]。Jousset和Bonkowski[34]发现, 当变形虫与荧光假单胞菌共培养时, 会促进间苯三酚衍生物的产生来应对变形虫的捕食, 进而提高DAPG的分泌。Song等[35]发现, 纳式虫Naegleria americana与荧光假单胞菌SS101和SBW25共培养时, 参与环脂肽合成的相关基因massA、massB与massC的表达均较细菌单独培养的对照组上调两倍以上。此外, 原生动物捕食能刺激细菌产生更多的次生代谢产物, 并对植物免疫产生不同程度的影响。例如, Jousset等[36]发现原生动物捕食荧光假单胞菌CHA0时, 细菌DAPG、吡咯硝酸盐、氰化氢及焦磷酸叶黄素合成相关基因的表达会上调, 同时通过影响茉莉酸途径来刺激植物的免疫系统[37]。
2.3 原生动物改变土著微生物群落结构和功能原生动物捕食可引起细菌群落结构的转变, 可能会影响群落抑病功能。研究发现异养鞭毛虫能够改变细菌的多样性和群落组成, 同时影响细菌对营养物质利用的能力[38]。原生动物Cercomonad cercozoa的捕食改变了细菌群落组成, 影响了细菌群落14种核心代谢功能, 包括膜转运, 尤其是VI型分泌系统的相关功能[39]。Rosenberg等[40]研究土壤变形虫Acanthamoeba castellanii捕食对拟南芥根际细菌群落组成的影响时发现, 变形虫对特定的细菌类群表现出不同的捕食偏好, 尽管变形虫使细菌的丰度大大降低, 但其中活性细菌的相对比例增加, 如细菌群落中的假单胞菌, 同时观察到功能基因(gacA)也发生了相应的变化。在稻田土壤系统研究中, 发现在丝足虫的捕食下, 变形杆菌被优先捕食, 而胞嘧啶和胸腺嘧啶含量低的革兰氏阳性细菌在原生动物捕食压力下更占优势[41]。由此可以推测, 原生动物对细菌群落的捕食具有高度的选择性, 而由原生动物捕食胁迫筛选出来的竞争能力强的细菌还可能有效抵御其他病原菌的入侵(图 2)。
尽管已有研究者提出土壤原生动物抑制土传病害的几种机制, 但目前并无直接证据证明土壤原生动物在抑制土传病害中具体如何发挥作用及其发生条件, 也无应用推广抑制土传病害的土壤原生动物制品, 因此, 未来尚需加强相关研究。
3 原生动物的研究方法由于方法学上的局限性, 相对于细菌和真菌, 土壤原生动物受到的关注较少。传统方法研究土壤原生动物, 主要依赖于分离培养与形态学观察。随着现代分子技术的发展, 土壤原生动物的研究方法发生了翻天覆地的变化。为了从多角度多功能更好地研究土壤食物链中的原生动物, 本文总结了当前研究土壤原生动物生物量、多样性、群落结构和生态功能的几种主要方法, 包括培养学/形态学方法鉴定、生物化学鉴定、现代分子技术手段以及利用流式细胞仪对原生动物进行筛分和计数。
3.1 形态学方法传统的土壤原生动物分离和培养方法如三级十倍稀释法[42]、活体培养法[43]等, 对于阐明原生动物的特征和生态功能是非常重要的。由于土壤原生动物种类繁多、数量庞大, 传统的形态学鉴定/培养学已不能满足当今研究的需要。近年来, 培养组学(Culturomics)得到快速发展。Bai[44]于2015年建立了拟南芥叶和根来源的细菌群落纯培养资源库, 它们大部分可被不依赖培养的16S扩增子测序稳定检测到。此外, 科研人员开发了一种新的高通量平台用于大量分离和培养环境中不可人工培养的微生物, 这种平台称为分离芯片[45]。借鉴以上的研究方法, 可建立土壤原生动物培养组学, 这对于探明土壤原生动物群落构成、个体行为与功能以及群落成员互作具有重要意义。需要指出的是, 无菌原生动物的构建也是当前原生动物与细菌互作研究中的难题, 此处不再讨论。
3.2 生物化学鉴定使用BIOLOG微平板[46]和磷脂脂肪酸(Phospholipid fatty acid analysis, PLFA)法[47]等生物化学手段来定量描述土壤微生物的多样性和群落结构组成也是解析土壤原生动物最常用的技术方法之一。Biolog分析是一种群落水平生理特性分析方法, 它基于微生物利用碳源能力的不同, 利用Biolog系统来研究微生物功能多样性。该方法对快速生长和适合在Biolog实验条件下生长的小部分群落成员有强烈的选择性, 而且被测试的底物不能准确地代表生态系统中的底物类型。因此, Biolog反应特征仅能粗略地代表实际土壤微生物群体的底物利用动力学特征[48]。有研究表明, PLFAs可定量检测土壤原生动物的生物量[49], 常用的原生动物特征性PLFAs种类有20:2ω6、20:3ω6、20:4、20:4ω6、20:4ω6, 9, 12, 15和22:6ω4c[50], 但目前在很多情况下, 仍无法完全确定每种特征脂肪酸与原生动物群落的对应关系[51]。Mauclaire等[52]研究表明, 利用13C富集的PLFA生物标记物与生物量计算法相结合来统计生物量, 是一种在原生动物食物链中追踪碳通量的合适方法。在土壤微生物生物量的应用, PLFA法偏重反映活性微生物的生物量。饱和与不饱和脂肪酸比(S/M)、反式顺式比(Trans/Cis)、含环丙基脂肪酸与含前体单不饱和脂肪酸比(Cy/Pre)和聚羟基丁酸与总脂肪酸比(Ph/T)均为土壤微生物生理状态和代谢活动方面常用的检测指标[53]。生化分析方法相对简便、效率高, 适用于多种生态系统, 缺点是不同生物之间成分的组成重合、样品的处理方式等均会影响准确评估摄食信息。
3.3 现代分子技术近年来, 土壤微生物学家紧随第二代高通量测序技术的迅猛发展, 也越来越多地使用第二代测序技术来解析土壤原生动物。通过进化保守基因18S rRNA, 可调查土壤食物网中原生动物的群落结构和多样性。此外, 通过原生动物的捕食模式, 还可将传统的原生动物系统分类地位划分至不同的功能群落, 这将大力推动土壤食物链的功能研究。稳定性同位素探针技术与高通量测序结合, 能够定向发掘土壤食物网中原生动物参与的特定功能, 比如, 可追踪土壤食物网中碳的周转和去向[54-55]。此外, 宏转录组测序技术也开始应用到土壤食物网中原生动物的检测, 但目前关于原生动物分类学信息仍比较缺乏, 需要基于培养的方法来弥补这一空白[56]。随着分类学信息的完善, 无偏见精确定量的PCR-free宏基因组测序将会是下一个全面解析土壤食物链中原生动物群落和功能的技术手段。总之, 高通量测序的应用为研究土壤原生动物提供了前所未有的机遇, 人们对土壤食物链中的微生物分类及其功能的了解也越来越深入。
3.4 流式细胞仪技术流式细胞仪(Flow cytometer)是对细胞进行自动分析和分选的装置。它可快速测量、存贮并显示悬浮于液体中的分散细胞的一系列重要生物物理、生物化学方面的特征参量, 还能预选、优化参量范围进而分选指定的细胞亚群。流式细胞仪主要以单细胞悬浮液(球形颗粒最佳)为研究对象, 因此起初主要应用于哺乳动物细胞的常规检测, 至20纪80年代, 流式细胞仪开始应用于水体浮游颗粒研究[57-58]。原生动物的流式应用相对其他微生物而言较为复杂, 因为其中不存在可被流式细胞仪识别的特征光合色素, 所以很难将其与其他颗粒区分开来, 此外, 由于细菌等引起的背景浓度过高, 很难区分原生动物细胞的信号。近年来, 研究者通过染色的方法利用流式细胞仪测定鞭毛虫或纤毛虫的数量。Zubkov等[59]通过SYBR Green I染色, 利用流式细胞仪测定了鞭毛虫的数量;Lindström等[60]将PicoGreen、TO-PRO-1及YO-YO-1三种核酸染料直接加入到分离自水体的纤毛虫样品中, 发现PicoGreen和TP-PRO-1两种染料可将纤毛虫与其他部分区分开, 且后者会使纤毛虫与细菌间差异更明显。Rifà等[61]通过采用提高荧光信号的阈值并降低检测电压的方法将原生动物与细菌的信号区分开, 但主要限制条件是细菌与鞭毛虫的比值不能高于1 000。由于变形虫本身的移动与生活习性的复杂性, 具体如何应用流式细胞仪检测变形虫数量有待进一步研究。
4 展望尽管原生动物在土传病害的防控中发挥着重要作用, 但相对于其他土壤微生物, 其相关研究仍处于起步阶段, 尚需大量深入的研究。对于土壤生物学研究工作者, 今后原生动物与土传病害的研究可从以下几个方面开展。
(1) 揭示土壤原生动物群落多样性、结构及其与土传病害发生之间的关系。探明土壤原生动物对作物健康的影响, 需结合当前高通量测序等非培养方法手段, 了解不同土壤环境中原生动物的多样性及群落结构特征, 掌握不同土壤生境下主导的原生动物种类和丰度;揭示水、热等土壤局域环境变化、施肥、耕作等人类活动以及全球变化等对土壤原生动物多样性和群落结构的影响。最后, 需要通过系统研究阐明原生动物多样性、群落结构以及关键原生动物在土传病害发生和防控中的作用机制, 尤其是原生动物在土壤微食物网中的重要贡献, 探究其在调控土壤微生物群落结构和功能中发挥的作用。
(2) 建立与作物健康相关的关键原生动物鉴定、分离和培养技术。近些年, 宏基因组学的快速发展, 使人们对土壤原生动物群落结构和空间分布规律等有了较好的认知, 但可培养原生动物的研究却逐渐被忽略。传统的土壤原生动物分离和培养方法对于阐明原生动物的特征和生态功能非常重要, 但由于方法的局限性, 难以全面解读原生动物群体的结构和功能特点。因此, 需要基于日益高效低廉的宏基因测序信息, 结合流式细胞仪等设备, 逐步建立高通量的土壤原生动物分离、鉴定、特性研究体系, 为构建土壤原生动物资源库, 全面解读关键土壤原生动物的抑病机制打好基础。
(3) 探明土壤原生动物与其他土壤生物互作对作物健康的影响。这部分主要有以下几个方面的重要问题:原生动物是直接影响作物健康还是通过与其他微生物作用从而间接影响作物健康?原生动物如何影响土壤微生物群落的组成和互作关系?土壤原生动物与其他土壤微生物有着怎样的互作关系?这些互作关系对抵御土传病原菌入侵有什么影响?这一互作关系是否受到土壤原生动物分类地位、捕食特性等影响?此外, 原生动物的捕食压力是环境微生物进化的基本驱动力之一, 因此, 探究原生动物捕食胁迫下, 被捕食者(如细菌)的演化规律和抵御捕食的机制也非常重要。由于获得无菌原生动物是一个很大的挑战, 在研究原生动物与细菌生态互作时, 通常受到杂菌的干扰。
(4) 加强原生动物资源收集和应用技术研发。开展土壤原生动物资源库的构建以及功能性原生动物的筛选、鉴定与保存等工作。同时加强应用技术研究, 比如原生动物的发酵技术、产品形式与技术标准、施用方式以及产品的风险评估, 形成相对完善的应用与开发流程。目前我国尚无土壤原生动物开发利用的公司, 但是荷兰Soil Tech公司已经开发出多款原生动物制品(https://soiltech.nl/)。希望相关部门可以制定相应政策并给予一定资金支持, 使更多科技工作者深入探究原生动物功能开发, 让土壤原生动物在土传病害防控和可持续农业发展中发挥其更多的作用。
| [1] |
Jiang G F, Wei Z, Xu J, et al. Bacterial wilt in China:History, current status, and future perspectives[J]. Frontiers in Plant Science, 2017, 8: 1549. DOI:10.3389/fpls.2017.01549
(  0) 0) |
| [2] |
Mendes R, Kruijt M, de Bruijn I, et al. Deciphering the rhizosphere microbiome for disease-suppressive bacteria[J]. Science, 2011, 332(6033): 1097-1100. DOI:10.1126/science.1203980
(  0) 0) |
| [3] |
Dodd I C, Zinovkina N Y, Safronova V I, et al. Rhizobacterial mediation of plant hormone status[J]. Annals of Applied Biology, 2010, 157(3): 361-379. DOI:10.1111/j.1744-7348.2010.00439.x
(  0) 0) |
| [4] |
Trivedi P, He Z L, van Nostrand J D, et al. Huanglongbing alters the structure and functional diversity of microbial communities associated with Citrus rhizosphere[J]. The ISME Journal, 2012, 6(2): 363-383. DOI:10.1038/ismej.2011.100
(  0) 0) |
| [5] |
Huang X Q, Cai Z C. Soil microbes and control of soil-borne diseases (In Chinese)[J]. Bulletin of Chinese Academy of Sciences, 2017, 32(6): 593-600. [黄新琦, 蔡祖聪. 土壤微生物与作物土传病害控制[J]. 中国科学院院刊, 2017, 32(6): 593-600.]
(  0) 0) |
| [6] |
Carrión V J, Perez-Jaramillo J, Cordovez V, et al. Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome[J]. Science, 2019, 366(6465): 606-612. DOI:10.1126/science.aaw9285
(  0) 0) |
| [7] |
Cha J Y, Han S, Hong H J, et al. Microbial and biochemical basis of a Fusarium wilt-suppressive soil[J]. The ISME Journal, 2016, 10(1): 119-129. DOI:10.1038/ismej.2015.95
(  0) 0) |
| [8] |
Klein E, Katan J, Gamliel A. Soil suppressiveness to Fusarium disease following organic amendments and solarization[J]. Plant Disease, 2011, 95(9): 1116-1123. DOI:10.1094/PDIS-01-11-0065
(  0) 0) |
| [9] |
Kwak M J, Kong H G, Choi K, et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature Biotechnology, 2018, 36(11): 1117. DOI:10.1038/nbt1118-1117
(  0) 0) |
| [10] |
Geisen S, Mitchell E A D, Wilkinson D M, et al. Soil protistology rebooted:30 fundamental questions to start with[J]. Soil Biology and Biochemistry, 2017, 111: 94-103. DOI:10.1016/j.soilbio.2017.04.001
(  0) 0) |
| [11] |
Geisen S, Mitchell E A D, Adl S, et al. Soil protists:A fertile frontier in soil biology research[J]. FEMS Microbiology Reviews, 2018, 42(3): 293-323. DOI:10.1093/femsre/fuy006
(  0) 0) |
| [12] |
Seppey C V W, Singer D, Dumack K, et al. Distribution patterns of soil microbial eukaryotes suggests widespread algivory by phagotrophic protists as an alternative pathway for nutrient cycling[J]. Soil Biology and Biochemistry, 2017, 112: 68-76. DOI:10.1016/j.soilbio.2017.05.002
(  0) 0) |
| [13] |
Clarholm M. Interactions of bacteria, protozoa and plants leading to mineralization of soil nitrogen[J]. Soil Biology and Biochemistry, 1985, 17(2): 181-187.
(  0) 0) |
| [14] |
Trap J, Bonkowski M, Plassard C, et al. Ecological importance of soil bacterivores for ecosystem functions[J]. Plant and Soil, 2016, 398(1/2): 1-24.
(  0) 0) |
| [15] |
Woods L E, Cole C V, Elliott E T, et al. Nitrogen transformations in soil as affected by bacterial- microfaunal interactions[J]. Soil Biology and Biochemistry, 1982, 14(2): 93-98.
(  0) 0) |
| [16] |
Sun Y H, Luo Y M. Environmental and health risks of pathogens in sewage sludge and their abating approaches (In Chinese)[J]. Soils, 2005, 37(5): 474-481. [孙玉焕, 骆永明. 污泥中病原物的环境与健康风险及其削减途径[J]. 土壤, 2005, 37(5): 474-481.]
(  0) 0) |
| [17] |
Bates S T, Clemente J C, Flores G E, et al. Global biogeography of highly diverse protistan communities in soil[J]. The ISME Journal, 2013, 7(3): 652-659. DOI:10.1038/ismej.2012.147
(  0) 0) |
| [18] |
Dupont A Ö C, Griffiths R I, Bell T, et al. Differences in soil micro-eukaryotic communities over soil pH gradients are strongly driven by parasites and saprotrophs[J]. Environmental Microbiology, 2016, 18(6): 2010-2024. DOI:10.1111/1462-2920.13220
(  0) 0) |
| [19] |
Li Y F, Li J, Li J J, et al. Effects of different management practices on microbial biomass carbon and protozoa abundance under greenhouse conditions (In Chinese)[J]. Soils, 2018, 50(4): 696-704. [李钰飞, 李季, 李吉进, 等. 温室不同管理模式对土壤微生物生物量碳和原生动物丰度的影响[J]. 土壤, 2018, 50(4): 696-704.]
(  0) 0) |
| [20] |
Chakraborty S, Old K M. Mycophagous soil amoeba:Interactions with three plant pathogenic fungi[J]. Soil Biology and Biochemistry, 1982, 14(3): 247-255. DOI:10.1016/0038-0717(82)90034-7
(  0) 0) |
| [21] |
Geisen S, Koller R, Hünninghaus M, et al. The soil food web revisited:Diverse and widespread mycophagous soil protists[J]. Soil Biology and Biochemistry, 2016, 94: 10-18. DOI:10.1016/j.soilbio.2015.11.010
(  0) 0) |
| [22] |
Geisen S, Rosengarten J, Koller R, et al. Pack hunting by a common soil amoeba on nematodes[J]. Environmental Microbiology, 2015, 17(11): 4538-4546. DOI:10.1111/1462-2920.12949
(  0) 0) |
| [23] |
Clarke M, Maddera L. Phagocyte meets prey:Uptake, internalization, and killing of bacteria by Dictyostelium amoebae[J]. European Journal of Cell Biology, 2006, 85(9/10): 1001-1010.
(  0) 0) |
| [24] |
Nikolyuk V. Some aspects of the study of soil protozoa[J]. Acta Protozoologica, 1969, 7: 99-108.
(  0) 0) |
| [25] |
Foissner W. Soil protozoa:Fundamental problems, ecological significance. Adaptations in ciliates and tetaceans, bioindicators. And guide to the literature[J]. Prog. Protistology, 1987, 2: 69-212.
(  0) 0) |
| [26] |
Petz W, Foissner W, Adam H. Culture, food selection and growth rate in the mycophagous ciliate Grossglockneria acuta Foissner, 1980:First evidence of autochthonous soil ciliates[J]. Soil Biology and Biochemistry, 1985, 17(6): 871-875. DOI:10.1016/0038-0717(85)90149-X
(  0) 0) |
| [27] |
Xiong W, Song Y Q, Yang K M, et al. Rhizosphere protists are key determinants of plant health[J]. Microbiome, 2020, 8: 27. DOI:10.1186/s40168-020-00799-9
(  0) 0) |
| [28] |
Chakraborty S, Old K M, Warcup J H. Amoebae from a take-all suppressive soil which feed on Gaeumannomyces graminis tritici and other soil fungi[J]. Soil Biology and Biochemistry, 1983, 15(1): 17-24.
(  0) 0) |
| [29] |
Rosenthal S, Reed E J, Weisman R A. Effect of lytic enzymes of Acanthamoeba castellanii on bacterial cell walls[J]. Journal of Bacteriology, 1969, 98(1): 182-189. DOI:10.1128/JB.98.1.182-189.1969
(  0) 0) |
| [30] |
Riekenberg S, Flockenhaus B, Vahrmann A, et al. The β-N-acetylhexosaminidase of Entamoeba histolytica is composed of two homologous chains and has been localized to cytoplasmic granules[J]. Molecular and Biochemical Parasitology, 2004, 138(2): 217-225.
(  0) 0) |
| [31] |
Long J J, Jahn C E, Sánchez-Hidalgo A, et al. Interactions of free-living amoebae with rice bacterial pathogens Xanthomonas oryzae pathovars oryzae and oryzicola[J]. PLoS One, 2018, 13(8): e0202941. DOI:10.1371/journal.pone.0202941
(  0) 0) |
| [32] |
Mikonranta L, Friman V P, Laakso J. Life history trade-offs and relaxed selection can decrease bacterial virulence in environmental reservoirs[J]. PLoS One, 2012, 7(8): e43801. DOI:10.1371/journal.pone.0043801
(  0) 0) |
| [33] |
Hu J, Wei Z, Friman V P, et al. Probiotic diversity enhances rhizosphere microbiome function and plant disease suppression[J]. mBio, 2016, 7(6): e01790-16. DOI:10.1128/mbio.01790-16
(  0) 0) |
| [34] |
Jousset A, Bonkowski M. The model predator Acanthamoeba castellanii induces the production of 2, 4, DAPG by the biocontrol strain Pseudomonas fluorescens Q2-87[J]. Soil Biology and Biochemistry, 2010, 42(9): 1647-1649. DOI:10.1016/j.soilbio.2010.05.018
(  0) 0) |
| [35] |
Song C X, Mazzola M, Cheng X, et al. Molecular and chemical dialogues in bacteria-protozoa interactions[J]. Scientific Reports, 2015, 5: 12837. DOI:10.1038/srep12837
(  0) 0) |
| [36] |
Jousset A, Rochat L, Scheu S, et al. Predator-prey chemical warfare determines the expression of biocontrol genes by rhizosphere-associated Pseudomonas fluorescens[J]. Applied and Environmental Microbiology, 2010, 76(15): 5263-5268. DOI:10.1128/AEM.02941-09
(  0) 0) |
| [37] |
Iavicoli A, Boutet E, Buchala A, et al. Induced systemic resistance in Arabidopsis thaliana in response to root inoculation with Pseudomonas fluorescens CHA0[J]. Molecular Plant-Microbe Interactions, 2003, 16(10): 851-858. DOI:10.1094/MPMI.2003.16.10.851
(  0) 0) |
| [38] |
Bonkowski M. Protozoa and plant growth:The microbial loop in soil revisited[J]. New Phytologist, 2004, 162(3): 617-631. DOI:10.1111/j.1469-8137.2004.01066.x
(  0) 0) |
| [39] |
Flues S, Bass D, Bonkowski M. Grazing of leaf-associated Cercomonads (Protists:Rhizaria structures bacterial community composition and function[J]. Environmental Microbiology, 2017, 19(8): 3297-3309. DOI:10.1111/1462-2920.13824
(  0) 0) |
| [40] |
Rosenberg K, Bertaux J, Krome K, et al. Soil amoebae rapidly change bacterial community composition in the rhizosphere of Arabidopsis thaliana[J]. The ISME Journal, 2009, 3(6): 675-684. DOI:10.1038/ismej.2009.11
(  0) 0) |
| [41] |
Murase J, Noll M, Frenzel P. Impact of protists on the activity and structure of the bacterial community in a rice field soil[J]. Applied and Environmental Microbiology, 2006, 72(8): 5436-5444. DOI:10.1128/AEM.00207-06
(  0) 0) |
| [42] |
Singh B N. A method of estimating the numbers of soil protozoa, especially amoebae, based on their differential feeding on bacteria[J]. Annals of Applied Biology, 1946, 33(1): 112-119. DOI:10.1111/j.1744-7348.1946.tb06282.x
(  0) 0) |
| [43] |
Foissner W. Basic light and scanning electron microscopic methods for taxonomic studies of ciliated protozoa[J]. European Journal of Protistology, 1991, 27(4): 313-330. DOI:10.1016/S0932-4739(11)80248-8
(  0) 0) |
| [44] |
Bai P. Biology of poly(ADP-ribose) polymerases:The factotums of cell maintenance[J]. Molecular Cell, 2015, 58(6): 947-958. DOI:10.1016/j.molcel.2015.01.034
(  0) 0) |
| [45] |
Nichols D, Cahoon N, Trakhtenberg E M, et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species[J]. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09
(  0) 0) |
| [46] |
Stefanowicz A. The Biolog plates technique as a tool in ecological studies of microbial communities[J]. Polish Journal of Environmental Studies, 2006, 15(5): 669-676.
(  0) 0) |
| [47] |
Zelles L. Fatty acid patterns of phospholipids and lipopolysaccharides in the characterisation of microbial communities in soil:A review[J]. Biology and Fertility of Soils, 1999, 29(2): 111-129. DOI:10.1007/s003740050533
(  0) 0) |
| [48] |
Zheng H, Ouyang Z Y, Fang Z G, et al. Application of biolog to study on soil microbial community functional diversity (In Chinese)[J]. Acta Pedologica Sinica, 2004, 41(3): 456-461. [郑华, 欧阳志云, 方治国, 等. BIOLOG在土壤微生物群落功能多样性研究中的应用[J]. 土壤学报, 2004, 41(3): 456-461.]
(  0) 0) |
| [49] |
Breulmann M, Schulz E, Weißhuhn K, et al. Impact of the plant community composition on labile soil organic carbon, soil microbial activity and community structure in semi-natural grassland ecosystems of different productivity[J]. Plant and Soil, 2012, 352(1/2): 253-265.
(  0) 0) |
| [50] |
Tischer A, Blagodatskaya E, Hamer U. Microbial community structure and resource availability drive the catalytic efficiency of soil enzymes under land-use change conditions[J]. Soil Biology and Biochemistry, 2015, 89: 226-237. DOI:10.1016/j.soilbio.2015.07.011
(  0) 0) |
| [51] |
Yan H, Cai Z C, Zhong W H. PLFA analysis and its applications in the study of soil microbial diversity (In Chinese)[J]. Acta Pedologica Sinica, 2006, 43(5): 851-859. [颜慧, 蔡祖聪, 钟文辉. 磷脂脂肪酸分析方法及其在土壤微生物多样性研究中的应用[J]. 土壤学报, 2006, 43(5): 851-859.]
(  0) 0) |
| [52] |
Mauclaire L, Pelz O, Thullner M, et al. Assimilation of toluene carbon along a bacteria-protist food chain determined by 13C-enrichment of biomarker fatty acids[J]. Journal of Microbiological Methods, 2003, 55(3): 635-649. DOI:10.1016/S0167-7012(03)00205-7
(  0) 0) |
| [53] |
Yao X D, Wang W, Zeng H. Application of phospholipid fatty acid method in analyzing soil microbial community composition (In Chinese)[J]. Microbiology China, 2016, 43(9): 2086-2095. [姚晓东, 王娓, 曾辉. 磷脂脂肪酸法在土壤微生物群落分析中的应用[J]. 微生物学通报, 2016, 43(9): 2086-2095.]
(  0) 0) |
| [54] |
Kramer S, Dibbern D, Moll J, et al. Resource partitioning between bacteria, fungi, and protists in the detritusphere of an agricultural soil[J]. Frontiers in Microbiology, 2016, 7: 1524. DOI:10.3389/fmicb.2016.01524
(  0) 0) |
| [55] |
Morriën E. Understanding soil food web dynamics, how close do we get?[J]. Soil Biology and Biochemistry, 2016, 102: 10-13. DOI:10.1016/j.soilbio.2016.06.022
(  0) 0) |
| [56] |
Geisen S, Tveit A T, Clark I M, et al. Metatranscriptomic census of active protists in soils[J]. The ISME Journal, 2015, 9(10): 2178-2190. DOI:10.1038/ismej.2015.30
(  0) 0) |
| [57] |
Legendre L, Yentsch C M. Overview of flow cytometry and image analysis in biological oceanography and limnology[J]. Cytometry, 1989, 10(5): 501-510. DOI:10.1002/cyto.990100505
(  0) 0) |
| [58] |
Trask B, van den Engh G, Elgershuizen J. Analysis of phytoplankton by flow cytometry[J]. Cytometry:The Journal of the International Society for Analytical Cytology, 1982, 2(4): 258-264.
(  0) 0) |
| [59] |
Zubkov M V, Zöllner E, Jürgens K. Digestion of bacterial macromolecules by a mixotrophic flagellate, Ochromonas sp., compared with that by two heterotrophic flagellates, Spumella pudica and Bodo saltans[J]. European Journal of Protistology, 2001, 37(2): 155-166. DOI:10.1078/0932-4739-00815
(  0) 0) |
| [60] |
Lindström E S, Weisse T, Stadler P. Enumeration of small ciliates in culture by flow cytometry and nucleic acid staining[J]. Journal of Microbiological Methods, 2002, 49(2): 173-182. DOI:10.1016/S0167-7012(01)00366-9
(  0) 0) |
| [61] |
Rifà T G, Latatu A, Ayo B, et al. Flow cytometric detection and quantification of heterotrophic nanoflagellates in enriched seawater and cultures[J]. Systematic and Applied Microbiology, 2002, 25(1): 100-108. DOI:10.1078/0723-2020-00085
(  0) 0) |
 2021, Vol. 58
2021, Vol. 58


